烟曲霉钙调蛋白的红色荧光蛋白标记及其在菌株极性生长的动态分布
曾荣 童建波 刘宇甄 陈青 段志敏 刘彩霞 吕桂霞 林彤 李岷
210042南京,中国医学科学院 北京协和医学院 皮肤病研究所激光科 江苏省皮肤性病学分子生物学重点实验室(曾荣、刘宇甄、林彤),真菌科(童建波、段志敏、刘彩霞、吕桂霞、李岷);江苏省血液中心(陈青)
烟曲霉是系统性曲霉病常见的致病菌[1],极性生长在其致病性中发挥重要作用。近年发现,钙信号系统不仅参与真菌生长、发育调控,同时在调控真菌菌丝的极性生长方面也有作用[2-3]。钙调蛋白(CaM)是该通路的核心成分[4],对其进行研究将有助于了解钙信号系统。CaM基因在烟曲霉中是一个必需基因,无法直接敲除该基因进行相关研究[5],但可通过基因重组、添加标记基因并表达标记蛋白来进行研究。我们拟对烟曲霉CaM进行红色荧光蛋白(red fluorescent protein,RFP)标记,构建CaMRFP烟曲霉菌株,观察该合成菌株菌丝极性生长过程中CaM动态分布,以期了解CaM在烟曲霉致病及耐药方面的作用。
材料与方法
一、材料
1.菌株和质粒:烟曲霉菌株A1160(△Ku80;pyrG)作为烟曲霉转化受体菌株来自美国真菌保藏中心,具有尿苷、尿嘧啶缺陷及nku基因缺失的特点。质粒pXDRFP4同样来自美国真菌保藏中心(ATCC),质粒图谱见图1。
2.主要试剂和仪器:自配YAG培养液,每升含酵母粉5 g、葡萄糖20 g、微量元素1m l,固体培养基需加入20 g/L琼脂粉;在YAG基础上加入1.1 g/L尿苷及1.2 g/L尿嘧啶制备YUU培养基;在YUU培养基基础上加入44.7 g/L氯化钾制备YUUK培养基;RPMI 1640液体培养基(美国Gibco公司)。rTaq聚合酶(日本TaKaRa公司),AccuPrime Taq High Fidelity DNA聚合酶(美国Invitrogen公司),PTC-100TMPCR仪(美国MJResearch公司)。
3.引物设计:构建含有红色荧光蛋白-烟曲霉pyrG营养标记(mRFP-AfpyrG)的转化子片段,对烟曲霉CaM基因序列的前后两端侧翼序列及含有mRFP-AfpyrG营养标记的质粒进行PCR扩增。融合PCR产物线性片段包括3部分,分别为上游组蛋白H1侧翼序列部分,mRFP-AfpyrG质粒部分,下游组蛋白染色体侧翼序列部分,见图2。先对上述3个片段分别进行PCR扩增。设计9条引物(包括P1~P6),见表1。上游侧翼序列使用P1、P3扩增,下游侧翼序列使用P4、P6扩增,设计引物时在P3、P4引物上添加Cassette序列作为接头。

图1 pXDRFP4质粒图谱 红色为红色荧光蛋白(RFP)序列,蓝色为AfpyrG营养筛选标记
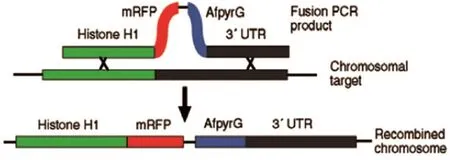
图2 钙调蛋白-红色荧光蛋白(CaM-RFP)烟曲霉菌株构建原理图 绿色和黑色均为所要替换基因组序列两边的侧翼序列,红色和蓝色序列为pXDRFP4质粒上Cassette片段,红色为RFP序列,蓝色为AfpyrG营养筛选标记

表1 引物序列
二、方法
1.转化子线性片段合成:扩增各组成片段[6],①上游侧翼序列(L-flank),PCR步骤:95℃ 2min,95 ℃ 15 s,58 ℃ 30 s,68 ℃ 1 min,共30个循环,10℃延伸1 h。PCR反应体系由100 ng模板DNA、300 nmol/L引物和1.25 U AccuPrime Pfx DNA聚合酶等组成;②下游侧翼序列(R-flank),扩增步骤:94 ℃ 3min,94 ℃ 45 s,49℃30 s,72 ℃ 1min(5个循环),94℃ 45 s,54℃ 1min,72℃ 1min(30个循环),72℃10min,10℃延伸。PCR反应体系由100 ng模板DNA、300 nmol/L引物、5 U rTaq DNA聚合酶等组成;③Cassette上RFP-AfpyrG片段PCR步骤:94℃2min,94 ℃ 20 s,70 ℃1 s,55 ℃ 30 s,68 ℃ 3min,共30个循环,10℃延伸。PCR反应体系由100 ng模板DNA、300 nmol/L引物、5 U rTaq DNA聚合酶等组成。PCR产物均用Takara切胶回收试剂盒纯化,冻于-80℃备用。
3个片段连接融合PCR:用于产生转化用的线性片段。反应体系:模板(共3部分,即2个侧翼序列和Cassette片段,每部分0.5μl),P2和P5引物(每种终浓度 400 nmol/L),2.5 U AccuPrime Taq High Fidelity DNA聚合酶,缓冲液20μl。PCR步骤:94 ℃2min,94 ℃20 s,70℃ 1 s,55℃ 30 s,68 ℃4min,9个循环;94 ℃ 20 s,70 ℃ 1 s,55 ℃ 30 s,68 ℃ 5min,4个循环;94 ℃20 s,70 ℃ 1 s,55 ℃ 30 s,68 ℃ 5min,9个循环,10℃延伸。切胶回收目的片段,以fusion产物片段为模板,用AccuPrime Pfx DNA聚合酶再次扩增,然后切胶回收目的片段,以获得足量转化用片段。
2.烟曲霉原生质体制备及转化:采用原生质体转化的方法将整合的融合PCR片段转入受体菌株A1160(△Ku80;pyrG)中。按照文献配制转化液,进行转化子的制备及筛选[6],操作过程如下:收集在YUU固体培养基上生长2.5 d的A1160菌株的孢子,将孢子以5×108个/m l加入YUUK液体培养基中,37℃培养。镜检,待50%以上孢子萌发时,收集孢子,将孢子重悬于原生质体溶液。镜检,待绝大多数孢子都已经形成原生质体,离心收集。在原生质体悬液中加入质粒DNA,先后经历冰浴和热激。再向原生质体悬液中加入YAGK(2%琼脂粉),倒入无菌平板下过夜。3 d后观察,转化子计数,并筛选转化子。
3.转化子初步筛选:将含1%琼脂糖的固体培养基液化,吸取50μl,在无菌载玻片上均匀涂一薄层,待冷却凝固后用牙签将所有的转化子分别接种于YAG培养基上,并同步点接在涂有一层YAG琼脂糖培养基的载玻片上,转化子号一一对应,放于湿盒中37℃培养14 h后,用荧光显微镜观察,采用DsRed通道,发现菌株有明显荧光后,从YAG平板上挑出对应号码菌株在YAG培养基上划线分离单菌落,培养2 d,再取单菌落于YAG培养基上划线分离,并同步点接在涂有一层YAG培养基的载玻片上,菌株号一一对应,以保证每次所挑菌落都有荧光,重复3次取均值以保证菌落的纯度以及荧光表型的稳定性,选取生长最为旺盛、荧光表型最稳定的1株菌,命名为CaM-RFP烟曲霉菌株。
4.转化子验证:使用引物CaM-RFP-P1和RFP-pyrG(R)进行PCR扩增,检测组蛋白H1侧翼序列部分和mRFP-AfpyrG质粒部分是否正确整合,PCR步骤:95 ℃ 2min,95 ℃ 10 s,56 ℃ 15 s,72 ℃ 1min,共30个循环,10℃延伸,反应体系由100 ng模板DNA、300 nmol/L引物、1.25 U AccuPrime Pfx DNA聚合酶等组成。用引物CaM-RFP-P6和F-RFP-pyrG进行PCR扩增,检测mRFP-AfpyrG质粒部分与下游组蛋白染色体侧翼序列部分是否正确整合,PCR步骤:95 ℃ 2min,95 ℃ 10 s,56 ℃ 15 s,72 ℃ 1min,共30个循环,10℃延伸,反应体系同上。野生菌株也使用上述引物进行PCR扩增。PCR扩增产物置于琼脂糖凝胶中电泳检测。
5.生长动力学及形态学分析:野生菌株烟曲霉A1160和标记菌株CaM-RFP烟曲霉在YAG固体培养基平板上培养72 h后,挑取单菌落,研磨,用RPMI 1640液体培养基将菌悬液调至1.0×105/ml,置于96孔细胞培养板中,37℃培养[7-8]。分别在4、8、10、16、24、48及72 h时使用0.5 g/L四甲基氮盐(XTT)试剂检测菌株生长活力,使用酶标仪在490 nm波长下测定吸光度(A值)。同时将上述菌悬液置于预先放置细胞爬片的24孔板中培养。分别在4、8、10、16、24 h使用荧光显微镜观察孢子出芽、菌丝形态及长度等。每个结果至少重复3次取均值。
6.荧光显微镜观测:使用蔡司显微镜(德国Zeiss公司)观察菌丝荧光表达,开启DsRed通道后先使用10×低倍镜找荧光位置,后使用40×高倍镜定位。
7.CaM在烟曲霉生长过程中的动态分布观察:将孢子点种在培养基上,置于37℃培养,在孢子接种后6 h开始每隔1 h采用显微镜动态观察。孢子经过培养可以在固体培养基上方形成小菌落,通过观察长在边缘的菌丝观察荧光表型。
三、统计方法
用SPSS18.0软件进行数据统计分析,计量资料比较采用t检验,P<0.05为差异有统计学意义。
结 果
一、转化子验证结果
使用引物CaM-RFP-P1和RFP-pyrG(R)可以自CaM-RFP烟曲霉菌株中扩增出1条与预期大小一致、长度为4 368 bp的DNA片段,而野生株无法扩增出DNA片段。使用引物CaM-RFP-P6和F-RFP-pyrG可以自CaM-RFP烟曲霉菌株中扩增出1条与预期大小一致、长度为2 278 bp的DNA片段,而野生株无法扩增出DNA片段。证明CaM-RFP烟曲霉菌株已经发生了同源整合,CaM的C端RFP荧光蛋白标记成功。见图3。
二、CaM-RFP烟曲霉菌株生长动力学及形态学特征
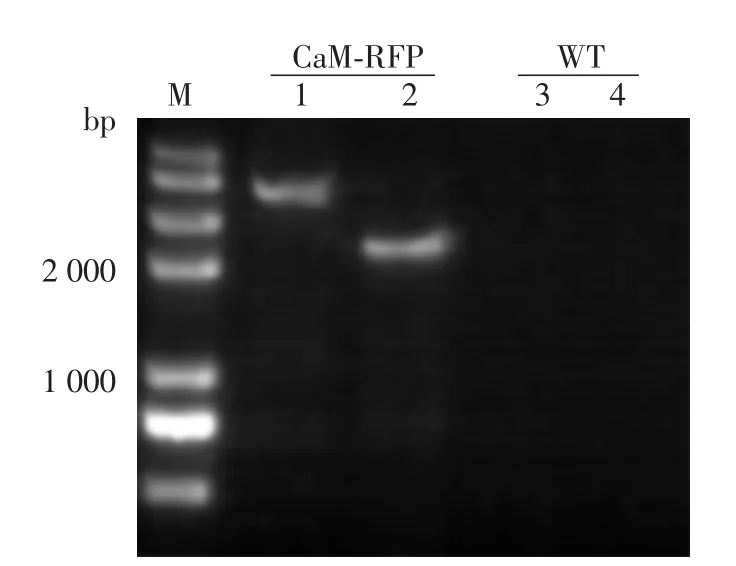
图3 重组株烟曲霉PCR验证结果 M:标准参照物;1、2:重组烟曲霉菌株;3、4:A1160野生烟曲霉菌株;1、3使用引物CaM-RFPP1/RFP-pyrG(R)扩增;2、4使用引物CaM-RFP-P6/F-RFP+pyrG扩增
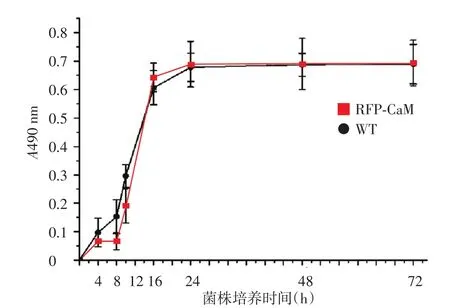
图4 野生株与重组株烟曲霉生长动力学比较 WT代表烟曲霉野生株;RFP-CaM代表红色荧光蛋白标记钙调蛋白的烟曲霉重组株

图5 钙调蛋白在烟曲霉孢子出芽(5A)、菌丝伸长(5B)、极性生长(5C)和分支(5D~5F)形成过程中的动态定位 5A~5F:分别代表培养6、7、8、9、10和11 h。箭头示钙调蛋白定位方向
在4、8、10、16、24、48、72 h时重组株和野生株烟曲霉间活力值(A490)差异均无统计学意义(t值分别为2.16、0.865、2.83、4.51、1.32、3.29、1.96,均P>0.05)。见图4。烟曲霉孢子的生长特性见图5。8 h时重组株和野生株出芽率分别为8.92%±1.51%和8.75%±1.42%,两组之间差异无统计学意义(t=0.553,P>0.05)。8 h时重组株和野生株菌丝长度分别为(64.92±7.61)μm和(63.54±8.17)μm,10 h时为(177.23±7.43)μm和(176.47±9.79)μm,两时间点两菌株间比较,差异均无统计学意义(t值分别为2.55、3.75,均P>0.05)。16 h后,菌丝相互缠绕,长度无法测量,但2株菌的生长形态无显著差异。
三、CaM在烟曲霉生长过程中的动态分布
见图5。在固体培养基上接种孢子后6 h,CaM定位于孢子的一侧,培养7~8 h后CaM所定位的位点分生出菌丝,9~10 h后分生出菌丝分支,形成新的生长极。继续培养,CaM一直位于生长菌丝的顶端。
讨 论
RFP标记技术是研究蛋白在细胞内定位的最新方法。要实现该技术需通过分子克隆技术将目的基因与RFP基因融合,在活细胞内表达后,融合蛋白上的RFP即可显示目的蛋白的分布,无需任何外加的酶或底物即可方便地动态观察活细胞。目前该技术广泛用于研究细菌在宿主中的动态定位等[8-9],但烟曲霉领域的研究刚起步。我们构建CaMRFP烟曲霉菌株来观察CaM在烟曲霉极性生长过程中的动态定位情况,为开展烟曲霉基础及应用研究提供良好的材料。
我们利用融合PCR方法成功进行CaM的C端RFP荧光蛋白标记,并对转化子进行筛选和鉴定。融合PCR可便捷地进行C端荧光蛋白标记,其中引物设计是关键环节。将3个片段连接的融合PCR效率不高,非特异性条带较多,可进一步纯化;纯化后得到的产物量不够时,可以此为模板进行再次扩增,以得到足够的转化片段。
菌丝的极性生长是烟曲霉致病能力的表现,具有极性生长表现的细胞能够承受强大的外力从而穿透其生长介质。我们前期[7]研究也发现,极性生长能促进烟曲霉生物膜的形成。Brand等[10]发现,白念珠菌钙信号系统能够使其从酵母状态转变为菌丝状态,即进入极性生长模式,该生长模式会增加其对宿主细胞的侵袭性。这一现象在其他类似研究中得到进一步证实[11-12]。我们在不加任何酶和底物的情况下动态观察CaM荧光,发现在孢子出芽阶段,红色荧光主要聚集在出芽的一端。随着菌丝的延长,红色荧光仍然处于菌丝的生长顶端。当部分荧光集中定位于除菌丝顶端外的点时,意味着新的分枝在该定位点将出现。表明CaM在一定程度上调控了孢子的萌发及菌丝的生长方向。
总之,我们成功构建CaM-RFP烟曲霉菌株。对该菌株生长发育阶段CaM分布的动态观察揭示了CaM参与调控烟曲霉孢子出芽和极性生长等生物学过程的功能,为进一步探索CaM在烟曲霉对宿主侵袭过程中的动态变化情况及可能的抗真菌药物靶点提供依据。
[1] EllisM,Richardson M,de Pauw B.Epidemiology[J].Hosp Med,2000,61(9):605-609.DOI:10.12968/hosp.2000.61.9.1415.
[2] Berridge MJ,Lipp P,Bootman MD.The versatility and universality of calcium signalling[J].Nat Rev Mol Cell Biol,2000,1(1):11-21.DOI:10.1038/35036035.
[3] Carafoli E.Calcium signaling:a tale for all seasons[J].Proc Natl Acad Sci U S A,2002,99(3):1115-1122.DOI:10.1073/pnas.032427999.
[4] Chin D,Means AR.Calmodulin:a prototypical calcium sensor[J].Trends Cell Biol,2000,10(8):322-328.DOI:10.1016/S0962-8924(00)01800-6.
[5] Itadani A,Nakamura T,Hirata A,et al.Schizosaccharomyces pombe calmodulin,Cam1,plays a crucial role in sporulation by recruiting and stabilizing the spindle pole body components responsible for assembly of the foresporemembrane[J].Eukaryot Cell,2010,9(12):1925-1935.DOI:10.1128/EC.00022-10.
[6] Chen S,Song Y,Cao J,et al.Localization and function of calmodulin in live-cells ofAspergillusnidulans[J].Fungal Genet Biol,2010,47(3):268-278.DOI:10.1016/j.fgb.2009.12.008.
[7] Zeng R,LiM,Chen Q,etal.In vitroanalyses ofmild heat stress in combination with antifungal agents againstAspergillus fumigatusbiofilm[J].Antimicrob Agents Chemother,2014,58(3):1443-1450.DOI:10.1128/AAC.01007-13.
[8] Kuhn DM,BalkisM,Chandra J,etal.Uses and limitations of the XTT assay in studies ofCandidagrowth and metabolism[J].J Clin Microbiol,2003,41(1):506-508.DOI:10.1128/JCM.41.1.506-508.2003.
[9] Sabuquillo P,Gea A,Matas IM,et al.The use of stable and unstable green fluorescent proteins for studies in two bacterial models:Agrobacterium tumefaciensandXanthomonas campestrispv.campestris[J].Arch Microbiol,2017,199(4):581-590.DOI:10.1007/s00203-016-1327-0.
[10] Brand A,Vacharaksa A,Bendel C,et al.An internal polarity landmark is important for externally induced hyphal behaviors inCandida albicans[J].Eukaryot Cell,2008,7(4):712-720.DOI:10.1128/EC.00453-07.
[11] Wilson D,Mayer FL,Miramón P,et al.Distinct roles ofCandida albicans-specific genes in host-pathogen interactions[J].EukaryotCell,2014,13(8):977-989.DOI:10.1128/EC.00051-14.
[12] Brand A.Hyphal growth in human fungal pathogens and its role in virulence[J].Int J Microbiol,2012,2012:517529.DOI:10.1155/2012/517529.

