星状病毒鸽群分离株的全基因分析
王楷宬,王素春,庄青叶,邱 源
(中国动物卫生与流行病学中心,山东青岛 266032)
星状病毒是一类无囊膜,呈球形,直径为28~30 nm的单股正链RNA病毒。该病毒最早于腹泻儿童的粪便中被发现。其名称源于病毒粒子在电镜下呈五角或六角星形[1]。本科病毒包含禽星状病毒属和哺乳动物星状病毒属。其基因组长度为6.11~7.72 kb[2]。所有星状病毒的基因组均含3个开放阅读框(Open reading frame,ORF),即ORFla、ORFlb和ORF2,基因组两端为5´和3´非翻译区(Untranslated region,UTR)。ORFla和ORFlb编码非结构蛋白(Nonstructural protein,NSP),ORF2编码衣壳蛋白(Capsid protein,CP)[3]。迄今为止,对星状病毒易感的动物多达22种。在养禽业中,星状病毒通常引起幼龄个体的胃肠道疾病,可导致鸡[4]、火鸡[5]、珍珠鸡[6]、鸭[2]、鸽子[7]和野生水禽[8]的肠炎、肾炎或病毒型肝炎等。
2011年,中国[9]和挪威[10]的研究人员均报道从鸽子中检测到星状病毒,并发现该病原属禽肾炎病毒(Avian nephritis virus,ANV)[7],并能引起鸽子腹泻。Tung等[11]于2013年通过高通量测序在野鸽中检测到星状病毒。此后,未再见有其他关于鸽子感染星状病毒的报道。
本实验室于2014年在青岛市的肉鸽中检测到星状病毒。为深入了解我国鸽群中的星状病毒特性,采用高通量测序方法,对该毒株进行了测序和基因组分析。
1 材料和方法
1.1 材料和仪器
Ion PGM Template OT2 400 Kit、Ion PGM Sequencing 400 Kit V2、E-Gel SizeSelect 2%Agarose、Ion 316 Chip Kit V2、Dynabeads MyOne Streptavidin C1 Beads、Qubit核酸浓度测定仪及配套试剂:购自Life technologies公司。其他设备包括:超净工作台(美国Forma Scientific)、移液器(Eppendorf)、孵化器(德州诚信孵化设备有限公司)、高速台式离心机(德国Heraeus Biofuge primoR)、PCR扩增仪(Perkin Elmeter Gen Amp PCR System 9600)。
高性能计算平台为Dell T630塔式服务器,具有2颗Intel(R)Xeon(R)CPU E5-2620 v3 @2.40 GHz;内存264 G,存储23 T;操作系统版本为 CentOS Linux release 7.1.1503(Core)。
1.2 核酸提取
2014年秋自山东省青岛市某活禽交易市场肉鸽的粪便样品中检测到鸽星状病毒(Pigeon/China/20/2014)[12]。将该份样品充分混匀,高速离心和过滤,去除大分子物质和细菌,使用QIAamp Viral RNA Mini Kit(Qiagen,德国)提取病毒核酸,-80 ℃保存。
1.3 高通量测序(NGS)文库构建
对提取的病毒核酸,采用文献[12]报道的方法进行NGS文库构建。具体方法为:在8 μL病毒RNA中,加入1 μL 100 μmol/L引物A15N6和2 μL无核酸酶的水进行混合,72 ℃ 5 min,然后将RNA引物混合物放在冰上孵育至少3 min;在 混 合 物 中, 加 入 4 μL 5×first-strand buffer、1 μL dNTP(100 μmol/L)、2 μL DTT(0.1 mol/L)、1 μL RNaseOUT ™ Recombinant Ribonuclease Inhibitor(40 U/μL) 和 1 μL SuperScript® III Reverse Transcriptase(200 U/μL)(Invitrogen,USA),25 ℃ 反应15 min,42 ℃反应 30 min,70 ℃ 15 min,终止反应;在反应产物中,加入1 μL RNase H(TaKaRa,Japan),37 ℃ 反应 20 min; 采 用 DynaMag ™ -2 Magnet 和 Agencourt®AMPure® XP Reagent(Beckman Coulter,USA)纯化合成第一链cDNA;在纯化的第一链cDNA中,加入引物B15N6,70 ℃ 5 min,再加入1 μL Klenow fragment(5 U)(NEB,USA)、5 μL 10×NEBuffer 2、2 μL dNTP(100 μmol/L) 和 1 μL DTT(0.1 mol/L),37 ℃反应30 min;最后采用 1×Phusion High-Fidelity Buffer、10 μmol/L 引物A30 和 B30(表1)、0.5 U Phusion High-Fidelity DNA Polymerase(NEB,USA)进行PCR扩增。将PCR产物采用E-Gel® SizeSelect™ Agarose Gel进行电泳,筛选和回收约450 bp的扩增产物,将其作为NGS文库。

表1 引物与引物序列
1.4 NGS测序
将NGS文库稀释为26 pmol/L;应用Ion PGM Template OT2 400 Kit,对DNA文库进行测序前的样品处理。将处理后的样品加样至Ion 316 芯片,置于PGM测序仪进行测序。
1.5 测序数据分析
对NGS测序数据经质控后,采用standalone NCBI BLASTn tool[13],将测序得到的reads与GenBank 核酸数据库进行比对(E值为10~5)。由MetaGenome Analyzer(MEGAN)[14]进行 BLASTn分析结果展示;提取其中与星状病毒序列比对上的reads,再采用CLC genomics workbench 8.5.1(Qiagen,Germany),将reads进行De Novo拼接,并分析拼接完成的基因组中的ORF;将注释后的序列提交GenBank。
1.6 基因组特性分析
采用MEGA 6.0[15]将Pigeon/China/20/2014的全基因和3个ORF氨基酸序列,分别与GenBank中星状病毒科代表毒株(表2)全基因和3个ORF氨基酸序列进行比对和分析,并分析这些毒株的衣壳蛋白氨基酸序列之间的遗传距离(p-distance)。
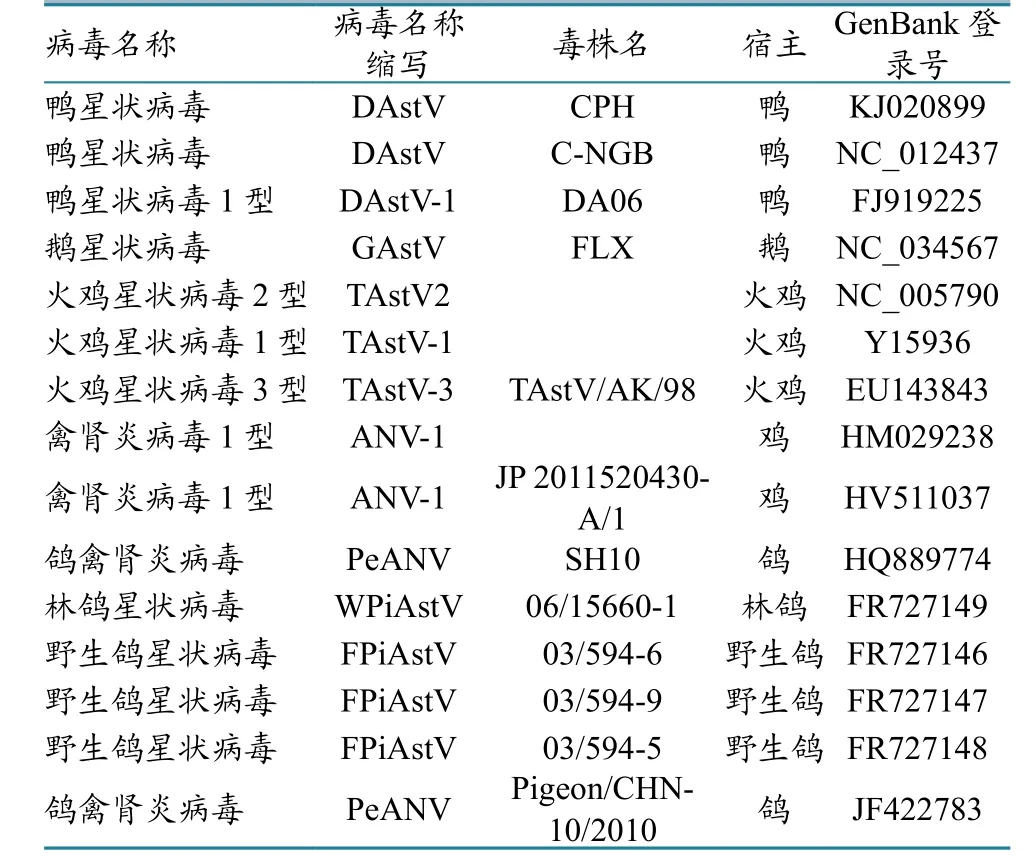
表2 基因比较分析使用的星状病毒毒株
2 结果与分析
2.1 基因组解析
芯片上样率为79%;测序文库得到质量较高的序列测序数据。CLC genomics workbench 8.5.1拼接得到的鸽星状病毒中国分离株全基因大小为6 872 bp,GenBank登录号为 MF768270。Pigeon/China/20/2014基因组具有与其他星状病毒相似的结构和基因顺序,其5´端有一段非编码区,3´端为聚A尾。基因组中主要包含3个ORF:ORF1a(14~3 028)编码1 005个氨基酸的多肽;ORF1b(3 028~4 554)编码509个氨基酸RNA依赖的RNA多聚酶;ORF2(4 573~6 594)编码674个氨基酸的衣壳蛋白。3´端(6 677~6 711)具有1个保守的茎环样模体(s2m)。有研究推测,该模体与病毒转录机制有关,在启动蛋白质合成过程中起重要作用。在ORF1a和ORF1b 重合区域存在1个核糖体移码序列(RFS)。该序列具有AAAAAAC特征(3 025~3 011)。
2.2 基因组比较分析
Pigeon/China/20/2014与15株禽星状病毒代表性毒株(表2)衣壳蛋白氨基酸序列的进化分析结果见图1;各毒株衣壳蛋白氨基酸序列之间的p-distance分析结果见图2。衣壳蛋白氨基酸序列进化分析结果显示,Pigeon/China/20/2014与2003年分离自挪威的野生鸽03/594-9亲缘关系最近(2011年报道)。按照国际病毒分类委员会(ICTV)第9次病毒分类报告中的星状病毒科分类标准,可将这些禽星状病毒分为两个基因群。所有鸽源星状病毒均属于Group Ⅰ。该基因群还包含从鸡群分离的禽肾炎病毒(图3),且不再分基因型。而GroupⅡ包含两个基因型(Group ⅡA 和 Group ⅡB),鸭、鹅和火鸡的星状病毒分离株均属此基因群。

图1 星状病毒衣壳蛋白(ORF2)氨基酸序列进化分析

图2 Pigeon/China/20/2014与星状病毒代表毒株衣壳蛋白氨基酸序列p-distance分析
Pigeon/China/20/2014与2003年分离自挪威的野生鸽03/594-9亲缘关系最近。全基因组进化分析结果见图3。
其与野生鸽分离的3株星状病毒(03/594-5、03/594-6和03/594-9)的亲缘关系较近,而与野生鸽分离的这3株星状病毒同源性更高。ORF1b进化分析结果见图4。
因从野生鸽中分离的3株星状病毒和从林鸽中分离的病毒缺少此基因序列,所以Pigeon/China/20/2014与ANV和PeANV亲缘关系较近。ORF1a进化分析结果见图5。

图3 星状病毒全基因进化分析

图4 星状病毒RNA 依赖的RNA多聚酶(ORF1b)氨基酸序列进化分析

图5 星状病毒ORF1a氨基酸序列进化分析
3 讨论
星状病毒能引起禽的肠炎,并增大幼龄火鸡、鸡、鹅的死亡率,或者引起鸡的肾炎和鸭的致死性肝炎[16]。本文对从我国华东地区活禽交易市场售卖的肉鸽中分离的1株星状病毒基因组进行了分析,发现该毒株属于Group Ⅰ基因群的禽星状病毒,且与2003年挪威野生鸽分离株(03/594-9)亲缘关系最近,与2011年我国分离的鸽源星状病毒(SH10)亲缘关系较远。挪威分离株(03/594-9)分离自2003年的野生鸽样品,且发表的文章仅报道了该毒株的部分基因组[10]。历经10余年,在我国活禽市场又检测到与此相似的病毒。这可能是由于国际贸易引起的。世界动物卫生组织(OIE)规定的动物贸易检疫名录中,未涉及星状病毒,因此携带该病原的鸽子通过国际贸易引入我国的可能性极大,也不排除通过跨国境赛鸽比赛逐步扩散到我国的可能。我国2011年分离的鸽源星状病毒(SH10)与鸡源的禽肾炎病毒亲缘关系较近。而本文报道的鸽源星状病毒与2011年分离株不同,说明我国鸽群中感染的星状病毒较为多样。我国虽然对鸭源星状病毒研究较多,但本文分离的Pigeon/China/20/2014与3株鸭源星状病毒衣壳蛋白的平均氨基酸遗传距离(p-distance)在0.735~0.756之间,差异较大。
星状病毒分为禽星状病毒属和哺乳动物星状病毒属。ICTV第9次病毒分类报告中星状病毒科的分类标准为:在氨基酸水平上对完整的衣壳蛋白进行遗传分析;两个基因组之间衣壳蛋白的平均氨基酸遗传距离(p-distance)在0.204~0.284之间,同一个基因型氨基酸遗传距离范围在0.576~0.741之间。本文对各种禽类感染的禽星状病毒属病毒进行了基因分析。按照以上标准,本文分析的禽星状病毒主要可分为两个基因群(Group Ⅰ 和GroupⅡ),Group Ⅱ又分为两个基因型(Group ⅡA 和Group ⅡB)。Group Ⅰ基因群中的星状病毒宿主为鸡和鸽;Group Ⅱ基因群中的星状病毒宿主为鸭、鹅和火鸡。衣壳蛋白是星状病毒的表面蛋白。分析结果显示该蛋白可能与宿主嗜型有关,但其机制还需进一步研究证实。
综上所述,本文报道了我国第2株鸽群星状病毒全基因,为禽源星状病毒的进一步分类提供了基础数据,有利于了解我国流行的鸽星状病毒特性。
[1] MADELEY C R,COSGROVE B P. Letter:28 nm particles in faeces in infantile gastroenteritis [J]. Lancet,1975,2(7932):451-452.
[2] FU Y,PAN M,WANG X,et al. Complete sequence of a duck astrovirus associated with fatal hepatitis in ducklings [J]. Journal of general virology,2009,90(5):1104-1108.
[3] MONROE S S,JIANG B,STINE S E,et al.Subgenomic RNA sequence of human astrovirus supports classification of Astroviridae as a new family of RNA viruses [J]. Journal of virology,1993,67(6):3611-3614.
[4] KANG K I,ICARD A H,LINNEMANN E,et al.Determination of the full length sequence of a chicken astrovirus suggests a different replication mechanism [J].Virus genes,2012,44(1):45-50.
[5] KOCI M D,SEAL B S,SCHULTZ-CHERRY S.Molecular characterization of an avian astrovirus [J].Journal of virology,2000,74(13):6173-6177.
[6] CANELLI E,CORDIOLI P,BARBIERI I,et al.Astroviruses as causative agents of poultry enteritis:genetic characterization and longitudinal studies on field conditions [J]. Avian diseases,2012,56(1):173-182.
[7] ZHAO W,ZHU A L,YU Y,et al. Complete sequence and genetic characterization of pigeon avian nephritis virus,a member of the family Astroviridae [J]. Archives of virology,2011,156(9):1559-1565.
[8] CHU D K,LEUNG C Y,PERERA H K,et al. A novel group of avian astroviruses in wild aquatic birds [J].Journal of virology,2012,86(24):13772-13778.
[9] ZHAO W,ZHU A L,YUAN C L,et al. Detection of astrovirus infection in pigeons(Columbia livia)during an outbreak of diarrhoea [J]. Avian pathol,2011,40(4):361-365.
[10] KOFSTAD T,JONASSEN C M. Screening of feral and wood pigeons for viruses harbouring a conserved mobile viral element:characterization of novel Astroviruses and Picornaviruses [J]. PLos one,2011,6(10):e25964.
[11] PHAN T G,VO N P,BOROS A,et al. The viruses of wild pigeon droppings [J]. PLos one,2013,8(9):e72787.
[12] QIU Y,CHEN J M,WANG T,et al. Detection of viromes of RNA viruses using the next generation sequencing libraries prepared by three methods [J]. Virus research,2017,237:22-26.
[13] MCGINNIS S,MADDEN T L. BLAST:at the core of a powerful and diverse set of sequence analysis tools [J].Nucleic acids research,2004,32:20-25.
[14] HUSON D H,AUCH A F,QI J,et al. MEGAN analysis of metagenomic data [J]. Genome research,2007,17(3):377-386.
[15] TAMURA K,STECHER G,PETERSON D,et al.MEGA6:Molecular evolutionary genetics analysis version 6.0 [J]. Molecular biology and evolution,2013,30(12):2725-2729.
[16] CATTOLI G,DE BATTISTI C,TOFFAN A,et al.Co-circulation of distinct genetic lineages of astroviruses in turkeys and guinea fowl [J]. Archives of virology,2007,152(3):595-602.

