红鳍东方鲀源哈维弧菌毒力基因检测及分型研究
王力+张吉鹏+刘美如+叶仕根+黎睿君+李华+李强


摘要 为探明红鳍东方鲀源哈维弧菌所携带毒力基因的种类对致病性的影响,以从辽宁地区患病红鳍东方鲀中分离得到的24株哈维弧菌为材料,根据GenBank公布的基因序列,设计合成引物,采用聚合酶链式反应(Polymerase Chain Reaction,PCR)对7种毒力基因flaB、flaC、toxS、zot、tcpA、tdh和tlh进行检测。结果表明,24株哈维弧菌中均检测出鞭毛蛋白flaB基因和flaC基因,其余5种基因均未检测到。同时对24株哈维弧菌进行了BOX-PCR扩增,结果将其分为4种基因型,记为A1-A4型。
关键词 红鳍东方鲀;哈维弧菌;毒力基因;BOX-PCR
中图分类号 S965.225 文献标识码 A 文章编号 1007-5739(2018)02-0237-03
Virulence Genes Detection and BOX-PCR Identification of Vibrio harveyi Strains Isolated from Takifugu rubripes
WANG Li 1 ZHANG Ji-peng 1 LIU Mei-ru 1 YE Shi-gen 1 LI Rui-jun 1 LI Hua 1 LI Qiang 2
(1 Key Laboratory of Mari-culture&Stock Enhancement in North China Sea,Ministry of Agriculture,Dalian Ocean University,Dalian Liaoning 116023; 2 College of Marine and Biology Engineering,Yancheng Institute of Technology)
Abstract In this paper,we detected virulence genetypes of Vibrio harvey isolated from Takifugu rubripes for further studying its pathogenicity.24 strains of V.harveyi isolated from infected Takifugu rubripes in Liaoning region were used for research.The PCR-primers were designed and synthesized according to the published sequence in the GenBank data.Polymerase Chain Reaction(PCR)was used to detect 7 virulence genes,including flaB,flaC,toxS,zot,tcpA,tdh and tlh.The results showed that both flaB and flaC gene were detected in all 24 strains of V.harveyi,and other 5 virulence genes were not detected.Meanwhile,the results of BOX-PCR indentation showed that 24 strains of V.harveyi were divided into 4 genotypes,respectively named A1-A4.
Key words Takifugu rubripes;Vibrio harvey;virulence gene;BOX-PCR
哈維弧菌(Vibrio harveyi)属于革兰氏阴性菌,已成为水产养殖上的主要致病菌,被哈维弧菌感染的对象有对虾[1]、大黄鱼[2]、鲈鱼[3]、石斑鱼[4]、半滑舌鳎[5]、大菱鲆[6]、鮸鱼[7]、鲍[8]、红鳍东方鲀[9]等水产动物,严重危害着养殖业的发展。随着分子生物学和免疫学技术的发展,哈维弧菌致病因子受到广泛的研究。研究表明,哈维弧菌内存在flaB、flaC、toxS、zot、tcpA、tdh和tlh基因,并且毒力基因的分布与分离菌株的来源有关[10-11]。同时,BOX-PCR指纹分析技术可以反映菌株间基因组的差异,广泛应用于生物多样性的研究[12]。因此,为了进一步探究哈维弧菌的致病机制,本文在原有工作的基础上,对辽宁地区患病红鳍东方鲀内分离鉴定的24株哈维弧菌进行flaB、flaC、toxS、zot、tcpA、tdh和tlh 7种毒力基因的研究,同时对其进行BOX-PCR探究,为有效防控红鳍东方鲀细菌性疾病及相关基因疫苗的研发奠定基础,同时对哈维弧菌的致病机制和防治提供参考。
1 材料与方法
1.1 试验材料
2014年对辽宁地区红鳍东方鲀细菌流行病进行为期1年的调查,发现红鳍东方鲀全年死亡率较低,主要表现为烂鳍烂尾、体表发黑,少数肝脏充血、脾脏肿大、肠道无食物并充满积液等。在104条病鱼的肝、脾、肾和体表溃烂处共分离到70株细菌,经16S rRNA基因序列分析及哈维弧菌特异性引物PCR扩增,共鉴定出24株哈维弧菌,经人工感染试验确定其为主要的致病菌,编号为2HWH001—2HWH024。
1.2 试验方法
1.2.1 哈维弧菌毒力基因检测。选取7种毒力基因,包括flaB、flaC、toxS、zot、tcpA、tdh和tlh,对哈维弧菌分离株进行毒力分析。PCR扩增采用25 μL的反应体系:10×PCR缓冲液5 μL,25 mmol/L MgCl2 3 μL,2.5 mmol/L 4×dNTP 1 μL,Taq聚合酶(5 U/μL)0.25 μL,DNA模版1 μL,引物各0.5 μL,双蒸水补足至25 μL。各毒力基因引物由上海生工生物工程有限公司合成,其序列如表1所示。endprint
PCR反应体系25 μL:10×PCR缓冲液5 μL,25 mmol/L MgCl2 3 μL,2.5 mmol/L 4×dNTP 1 μL,Taq聚合酶(5 U/μL)0.25 μL,DNA模版1 μL,引物各0.5 μL,双蒸水补足25 μL。PCR反应条件:94℃预变性5 min,30个循环,94℃变性1 min,toxS基因52 ℃退火1 min,zot、tcpA、tlh、flaC基因均58℃退火1 min,tdh基因55 ℃退火1 min,flaB基因50 ℃退火1 min,72 ℃延伸1 min,72 ℃再延伸7 min。PCR产物用1.0%琼脂糖凝胶电泳进行鉴定(120 V电压下电泳15 min),在凝胶成相系统下拍照分析。
1.2.2 哈维弧菌 Box分型。选择通用引物BoxA1R[14]对24株哈维弧菌进行BOX-PCR扩增。PCR反应体系25 μL:10×PCR缓冲液5 μL,25 mmol/L MgCl2 3 μL,2.5 mmol/L 4×dNTP 1 μL,Taq聚合酶(5 U/μL)0.25 μL,DNA模版1 μL,引物各0.5 μL,双蒸水补足25 μL。PCR扩增循环参数:95 ℃预变性 4 min,94 ℃变性30 s,52 ℃退火 1 min,65 ℃延伸8 min,35个循环,最后65 ℃再延伸10 min。PCR产物用1.5%琼脂糖凝胶电泳鉴定,在凝胶成相系统下拍照分析,用Gel-Pro analyzer分析电泳图,用Ntsys软件进行聚类分析。
2 结果与分析
2.1 毒力基因检测
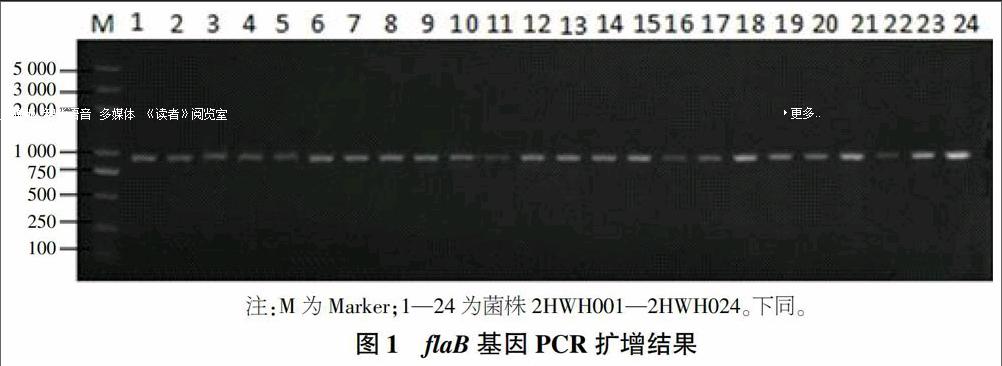
采用7种毒力基因的特异性引物对24株哈维弧菌进行PCR扩增,结果如图1、2所示,可以看出,24株分离株均存在flaB、flaC基因,而toxS、zot、tcpA、tdh和tlh基因均未被检出。
2.2 BOX-PCR分型
哈维弧菌分离株经BOX-PCR扩增,PCR产物为2~10条,大小介于100~5 000 bp之间,如图3所示。经聚类分析,24株哈维弧菌可分为4个基因型,如图4所示,分别标记为A1—A4。其中A1型有10株,分别为菌株2HWH001、2H-WH002、2HWH003、2HWH004、2HWH005、2HWH006、2H-WH008、2HWH009、2HWH010、2HWH012,主要是高温季节分离的病菌。A2型有11株,分别为菌株2HWH011、2HWH0-15、2HWH016、2HWH017、2HWH018、2HWH019、2HWH020、2HWH021、2HWH022、2HWH023、2HWH024,主要以低温季节分离的病菌为主,且主要分离于鱼的内脏。A3型只有1株菌,为菌株2HWH-007。A4型有2株,分别为菌株2HWH013、2HWH014。
3 讨论
3.1 毒力基因研究
对哈维弧菌致病机制的研究证实不同种弧菌间可能存在同一种毒力基因。这种致病性的强弱差异可能是由弧菌某种或某些可遗传的毒力因子决定的[14-15]。同时有研究表明,哈维弧菌分泌的毒力因子胞外酶或溶血素可能对斑节对虾(Penaeus monodon)的致病性起重要作用[16]。由于对哈维氏弧菌病认识较晚,对其毒力因子和致病机制的研究尚不够充分。为了初步探索菌株毒力因子与致病性强弱的关系,本研究选取7种常见的毒力因子进行研究,结果发现,我国辽宁地区患病红鳍东方鲀源哈维弧菌无论强弱毒株都未携带toxS、zot、tcpA、tdh和tlh基因,表明遼宁地区患病红鳍东方鲀源哈维氏弧菌与这5种毒力因子的相关度很低;而flaB和flaC基因在24株哈维弧菌中检出率为100%,笔者分析flaB和flaC基因可能与哈维氏弧菌致病性相关,具体相关性还需进一步分析。庞玲玲[10]探索了山东青岛沿海养殖场发病的鲈鱼和大菱鲆等海水养殖动物中哈维弧菌的5种毒力基因分布情况,结果证实toxS、zot和tcpA基因在哈维弧菌中不是普遍存在的,而flaB和flaC基因分布广泛,这与本研究结果有一定的相似性。flaB和flaC基因是编码鞭毛蛋白的基因,调控着细菌附着表面的能力,能够使哈维弧菌具有很强的附着力[17-18]。结合红鳍东方鲀发病临床症状和毒力基因检测结果,笔者认为红鳍东方鲀烂鳍烂尾病的发病机制是鱼体体表受损后,哈维弧菌附着于创伤处,使伤口发炎,当鱼体抵抗力降低时细菌侵入到内脏,进而引起死亡。徐芝亮[19]研究了广东、海南、广西地区患病的海水鱼类或水样中哈维弧菌的毒力基因分布情况,发现toxS、zot、tcpA、tlh和tdh基因在哈维弧菌分离株中的分布情况与地区有一定的相关性,zot基因在广东分离菌株中分布比较广泛,toxS基因在广西和广东分离菌株出现较频繁,tdh基因则广泛出现在海南分离菌株中,而tcpA和tlh基因在3个地区的分离菌株中都没有检测出。刘迪等[20]研究表明南方虾源哈维氏弧菌无论毒性强弱都很少携带zot和toxS基因,并认为菌株的致病性差异可能与其生化特性相关。研究表明[21-24],toxS、zot、tcpA、tdh和tlh基因在霍乱弧菌、拟态弧菌、溶藻弧菌等分布较多。本研究在辽宁地区养殖红鳍东方鲀源哈维弧菌中均未检测出toxS、zot、tcpA、tdh和tlh基因,笔者推测毒力基因与菌株的分离地区和动物源性相关,但尚需更多研究予以证明。
3.2 BOX-PCR分型研究
BOX-PCR指纹图谱分析技术,是根据BOX插入因子设计引物,扩增微生物基因组DNA的重复性片段,使不同大小的DNA片段与位于这些片段之间的序列得到扩增,最后经琼脂糖凝胶电泳检测其多态性的一种微生物鉴定方法[25]。随着微生物检测技术的不断发展,BOX-PCR技术在微生物多样性研究中已得到应用[26-28]。本研究通过引物BoxA1R将24株哈维弧菌分为4型,大部分属于A1和A2型,其中A1型细菌主要分离自高温季节,A2型细菌主要分离于低温季节,且分离自内脏的哈维弧菌大部分属于A2型,猜测BOX分型可能与水温和分离部位相关,但还需进一步验证。徐芝亮等[29]研究发现哈维弧菌BOX分型与地域有关,本研究由于调查地点距离较近,无法证实这一点。林海云等[30]研究显示,不同地理来源的青枯雷尔氏菌在BOX-PCR中可扩增出各自特异性条带,指出青枯雷尔氏菌的遗传分化与地理来源存在相关性。Louws等[31]采用BOX-PCR对多种植物病原体和假单胞菌进行分子分型分析,结果表明,BOX-PCR能很好地反映它们的基因结构,能对大量菌株进行分类分析。有研究表明[32],BOX-PCR指纹分析技术更适合于种及种以下水平遗传多样性的研究。本文结果为今后哈维弧菌基因分型提供了一定的参考,同时也为哈维弧菌疫苗的防治奠定了基础。endprint
4 参考文献
[1] 刘问,钱冬,杨国梁,等.南美白对虾虾苗淡化期间发光病病原研究[J].集美大学学报(自然科学版),2004(4):300-304.
[2] 石亚素,童国忠,薛超波,等.舟山养殖大黄鱼烂尾病中哈氏弧菌的分离鉴定及药敏试验[J].中国卫生检验杂志, 2005(3):267-269.
[3] 张静,施慧,谢建军,等.网箱养殖鲈鱼内脏白点病病原的分离与鉴定[J].浙江海洋学院学报(自然科学版),2009(2):176-182.
[4] 梅冰,周永灿,徐先栋,等.斜带石斑鱼烂尾病病原菌的分离与鉴定[J].热带海洋学报,2010(6):118-124.
[5] 陈政强,姚志贤,林茂,等.半滑舌鳎皮肤溃疡病病原研究[J].水产学报,2012,36(5):764-771.
[6] 范文辉,黄倢,王秀华,等.养殖大菱鲆溃疡症病原菌的分离鉴定及系统发育分析[J].微生物学报,2005(5):665-670.
[7] 张凤萍,彭志兰,张健,等.鮸鱼弧菌病病原菌(哈维氏弧菌)的分离与鉴定[J].微生物学报,2010(3):304-309.
[8] 陈月忠,黄万红,蔡清海,等.闽南地区鲍溃烂病的研究[J].福建水产,2008(4):31-37.
[9] 王斌,于兰萍,胡亮,等.红鳍东方鲀皮肤溃烂病病原菌的分离与鉴定[J].中国水产科学,2008(2):352-358.
[10] 庞玲玲.哈维氏弧菌毒力相关基因的筛查及快速检测技术研究[D].青岛:中国海洋大学,2007.
[11] 徐芝亮.哈维氏弧菌遗传多样性分析及毒力相关基因的检测[D].湛江:广东海洋大学,2012.
[12] 杨凤环,李正楠,姬惜珠,等.BOX-PCR技术在微生物多样性研究中的应用[J].微生物学通报,2008(8):1282-1286.
[13] SECHI L A,DUPRE I,DERIU A,et al.Distribution of Vibrio cholerae virulence genes among different Vibrio species isolated in Sardinia,Italy[J].J ApplMicrobiol,2000,88(3):475-481.
[14] VERSALOVIC J,SCHNEIDER M,BRUIJN F,et al.Genomic fingerprint of bacteria using repetitive sequence-based polymerase chain reaction[J].Methods in Molecular & Cellular Biology,1994,5(1):25-40.
[15] SAEED MO.Association of Vibrio harveyi,with mortalities in cultured marine fish in Kuwait [J].Aquaculture,1995,136:21-29.
[16] LIU P C,LEE K C,KOU G H,et al.Isolation of Vibrio harveyi,from Dis-eased Kuruma Prawns Penaeusjaponicus[J].Current Microbiology,1996,33(2):129-132.
[17] LEE S E,KIM S Y,JEONG B C,et al.A Bacterial Flagellin,Vibrio vuln-
ificusFlaB,Has a Strong Mucosal Adjuvant Activity To Induce Prote-ctive Immunity[J].Infection & Immunity,2006,74(1):694.
[18] MILLIKAN D S,RUBY E G.Vibriofischeri Flagellin A Is Essential for Normal Motility and for Symbiotic Competence during Initial Squid Light Organ Colonization[J].Journal of Bacteriology,2004,186(13):4315-4325.
[19] 徐芝亮.哈維氏弧菌遗传多样性分析及毒力相关基因的检测[D].湛江:广东海洋大学,2012.
[20] 刘迪,房文红,周红霞,等.虾源哈维氏弧菌的致病性与生物学特性比较分析[J].海洋渔业,2017,39(2):197-205.
[21] MILLER V L,MEKALANOS J J.Synthesis of cholera toxin is positively regulated at the transcriptional level by toxR[J].Proceedings of the National Academy of Sciences of the United States of America,1984,81(11):3471-3475.
[22] SHI L,MIYOSHI S,HIVRA M,et al.Detection of Genes Encoding Cho-lera Toxin(CT),Zonula Occludens Toxin(ZOT),Accessory Cholera Enterotoxin(ACE)and Heat-Stable Enterotoxin(ST) in Vibrio mimicus Clinical Strains[J].Microbiology & Immunology,1998,42(12):823-828.endprint
[23] RAJA N,SHAMSVDIN M N,SOMARANY W,et al.Detection and mole-cular characterization of the zot gene in Vibrio cholerae and V.alginol-yticus isolates[J].Southeast Asian Journal of Tropical Medicine & Public Health,2001,32(1):100.
[24] SECHI L A,DUPR?魬 I,DERIV A,et al.Distribution of Vibrio cholerae,virulence genes among different Vibrio,species isolated in Sardinia,Italy[J].Journal of Applied Microbiology,2000,88(3):475-481.
[25] 刘佳妍,金莉莉,王秋雨.细菌基因组重复序列PCR技术及其应用[J].微生物学杂志,2006(3):90-93.
[26] 刘洋,隋新华,陈文新.华北及西北地区岩黄芪根瘤菌的表型及遗传多样性[J].生态学报,2005(5):1088-1094.
[27] TAC?魨O M,AIVES A,SAAVEDRA M J,et al.BOX-PCR is an adequate tool for typing Aeromonasspp[J]. Antonie Van Leeuwenhoek,2005,88:173-179.
[28] PROUDY I,BOUGI?魪 D,COTON E,et al.Genotypic characterization of Enterobacter sakazakii isolates by PFGE,BOX-PCR and sequencing of the fliC gene[J].Journal of Applied Microbiology,2008,104(1):26-34.
[29] 徐芝亮,吴灶和,简纪常,等.哈维氏弧菌的分子遗传多样性研究[J].水产学报,2012(9):1450-1456.
[30] 林海云,车建美,刘波,等.基于BOX-PCR和REP-PCR技术青枯雷尔氏菌遗传多样性分析[J].农业生物技术学报,2011(6):1099-1109.
[31] LOUWS F J,FUIBRIGHT D W,STEPHENS C T,et al.Specific genomic fingerprints of phytopathogenicXanthomonas and Pseudomonas pathov-ars and strains generated with repetitive sequences and PCR[J].Applied & Environmental Microbiology,1994,60(7):2286.
[32] 張红侠,冯瑞华,李俊,等.黄土高原地区大豆根瘤菌的遗传多样性和系统发育[J].微生物学报,2010(11):1466-1473.endprint

