宏基因组文库新型β-葡萄糖苷酶的筛选、克隆及酶学性质分析
唐乐丽,王晓萌,吴秀玲,黄 庆,李 荷*
(广东药科大学药学院,广东 广州 510006)
微生物资源在地球上的含量十分丰富,其中原核细胞数量和种类高达到6×1030~8×1030个[1],微生物具有的多样性特质,为挖掘新酶、新的代谢物质和生物活性物质提供了一种新来源[2]。然而Bakken等[3]研究发现,自然界中高达99%的微生物是未培养微生物,即在目前的实验环境下是不能被培养生长的,如果利用传统培养微生物学的方法,只能获得地球上极少数微生物的活性物质,从而兴起了宏基因组技术,1985年,Pace等通过提取环境微生物总DNA法,成功研究了大量未培养微生物所携带的遗传学信息,这之后,许多研究者都通过此方法对未培养微生物进行了研究[4-5]。
β-葡萄糖苷酶(β-D-glucosidase,EC3.2.1.21),属于纤维素酶类,是3 种纤维素分解酶中的重要组成成分之一,它能够水解结合于末端非还原性的β-D-葡萄糖键,同时释放出β-D-葡萄糖和相应的配基。β-葡萄糖苷酶有较广泛的工业应用,可用来降解纤维素[6-8]生产生物乙醇、改善食品风味、转化大豆异黄酮糖苷化合物、制备低聚龙胆糖、防治病虫害[9-11]等。但目前工业用β-葡萄糖苷酶多数来源于木霉等真菌及茶树类植物[12-13],反应条件(如温度、pH值)适应范围相对较窄,且酶活力偏低,造成经济成本较高,制约β-葡萄糖苷酶的广泛应用[14-15]。而且生物酶应用于工业生产的条件比较苛刻,要求生物酶具有良好的热稳定性、有机溶剂耐受性、金属离子耐受性以及在特定pH值、温度条件下的高酶活力,因此需要发掘酶学性质更加优良的β-葡萄糖苷酶来提高工业生产效率,降低生产成本。宏基因组技术研究对象是微生物总DNA并独立于宿主之外进行研究,所获得的新基因一般都较完整,相似度也较低,可以用来大量筛选新的有开发前景的工业酶[16-18]。黑龙江玉米秸秆还田土壤中纤维素资源丰富,可能含有能够产生酶活力较高的β-葡萄糖苷酶微生物。本研究以黑龙江玉米秸秆还田土壤微生物为对象,构建其宏基因组文库,利用功能筛选方法,拟从文库中筛选具有酶活力较高的新型β-葡萄糖苷酶基因并对其酶学性质进行分析。
1 材料与方法
1.1 材料与试剂
土样采自黑龙江省牡丹江市郊区玉米秸秆还田土壤,采集表层下10~20 cm的土样,-20 ℃保存。Escherichia coli DH5α、E. coli BL21由本实验室保存。建库所用载体质粒pUC118、pET-32a(+)购自大连TaKaRa公司。
XhoⅠ、EcoRⅠ等限制性内切酶、T4 DNA连接酶以及各种DNA MarkerPrime、STARTMMax Premix、蛋白质分子质量Marker 大连TaKaRa公司;DNA凝胶回收试剂盒(E.Z.N.A®Gel Extraction Kit)、质粒提取试剂盒(E.Z.N.A®Plasmid Mini Kit)、聚合酶链式反应(polymerase chain reaction,PCR)产物回收试剂盒(E.Z.N.A®Cycle-Pure Kit) 美国Omega公司;氨苄青霉素(ampicillin,Amp)、5-溴-4-氯-3-吲哚-α-D-半乳糖苷(5-bromo-4-chloro-3-indolyl-α-D-galactopyranoside,X-gal)、异丙基-β-D-硫代吡喃半乳糖苷(isopropylβ-D-1-thiogalactopyranoside,IPTG) 美国Sigma公司;七叶苷、对硝基苯酚(p-nitrophenol,pNP),对硝基苯基-β-D-吡喃葡萄糖苷(p-nitrophenyl-β-D-glucopyranoside,pNPG) 生工生物工程(上海)有限公司;其他化学试剂均为国产分析纯。
1.2 仪器与设备
凝胶成像系统 美国BioTek公司;电泳仪 北京六一仪器公司;PCR仪 美国AB公司;恒温水浴锅上海精密实验设备公司;电击仪 美国Bio-Rad公司;Nanodrop2000c超微量紫外-可见光分光光度计 赛默飞世尔科技公司;PB-10型pH计 德国Sartorius公司。
1.3 方法
1.3.1 玉米秸秆还田土壤基因组总DNA的提取[19]及纯化
取4 个高压灭菌后的50 mL离心管,称取玉米土壤样品,每管6 g,各加DNA 提取缓冲液13.5 mL,涡旋振荡混匀后,放摇床220 r/min、37 ℃活化30 min。各管加1.5 mL质量浓度20 g/100 mL 的十二烷基硫酸钠(sodium dodecyl sulfate,SDS),使其终质量浓度达到2 g/100 mL。在65 ℃水浴锅中水浴2 h,每隔30 min上下轻轻颠倒数次混匀。25 ℃、6 000 r/min离心10 min。将上清液转移至高压灭菌的50 mL高速离心管,加入等体积的氯仿-异戊醇(24∶1)混合液,上下轻轻颠倒混匀。25 ℃、11 000 r/min离心30 min。收集上清液,加入0.6 倍体积的异丙醇上下轻轻颠倒混匀,室温静置3 h以上。25 ℃、11 000 r/min离心30 min。弃上清液,用4 ℃预冷的75%乙醇溶液洗涤沉淀1 次。4 ℃、8 000 r/min离心10 min,弃上清液,放入60 ℃烘箱烘干。每管加0.2 mL 65 ℃预热的ddH2O,并65 ℃温浴5 min,溶解基因组DNA。1.0%琼脂糖凝胶电泳检测提取的基因组DNA,并立即纯化回收基因组DNA。提取的粗基因组DNA用1.0%的琼脂糖进行琼脂糖凝胶电泳,使用Omega的DNA Extraction Kit回收并纯化所提取的玉米土壤基因组DNA。
1.3.2 玉米秸秆还田土壤宏基因组文库的构建
选择EcoRⅠ酶对提取纯化的玉米土壤DNA进行不完全酶切80 min,回收2.3~8 kb的DNA片段。将回收的酶切DNA片段连接到线性化的经EcoRⅠ酶切的克隆载体上,用TaKaRa T4 DNA Ligase于16 ℃连接12~16 h。对连接产物进行纯化:取出DNA酶切片段与载体的连接产物,加入2.5 倍体积-20 ℃提前预冷的无水乙醇和0.05 倍体积的3 mol/L乙酸钠轻轻混匀,-20 ℃沉淀1 h以上;4 ℃、12 000 r/min离心20 min;弃上清液,用-20 ℃预冷的70%乙醇溶液轻轻洗涤沉淀2 次。4 ℃、12 000 r/min离心20 min,收集管中沉淀,60 ℃烘箱中烘干;加入10 μL 65 ℃预热的dd H2O重悬连接的载体DNA。将纯化的连接产物的电击转化法转入大肠杆菌E. coli DH5α感受态细胞。取适量培养物涂布于LAXI培养基(100 mg/L Amp、20% IPTG和20 mg/L X-gal),37 ℃培养过夜,即得玉米土壤宏基因组文库。
1.3.3 土壤宏基因组文库β-葡萄糖苷酶的筛选
挑选白色菌落,剔除蓝色菌落,点到β-葡萄糖苷酶筛选培养(100 μg/mL Amp、0.25%柠檬酸铁铵、0.1%七叶苷)平板上,37 ℃培养24 h,观察菌落周围是否有黑色水解圈,有黑色水解圈的菌落为β-葡萄糖苷酶阳性克隆子[20]。
1.3.4 测序及序列的生物信息学分析
1.3.4.1 ORF Finder
挑选转化后仍然具有黑色透明圈的克隆子,提取质粒,EcoRⅠ单酶切,若仍旧能够切出载体和目的DNA条带,则将重组质粒送到Invitrogen公司进行序列测定。将测序得到的目的DNA序列用NCBI的ORF Finder在线工具查找该序列的开放阅读框(open reading frame,ORF),再将ORF序列经pBlast逐一比对,得到序列与已报道的功能蛋白的相似度等信息。
1.3.4.2 系统发育树和氨基酸保守性分析[21-22]
将疑似功能蛋白的基因与已报道的该蛋白各家族成员序列先用Clustal W软件比对序列并导出序列对比结果,再用MEGA 4.0软件中Unrooted Neighbor-Joining Method,the Bootstrap Test选择(1000 replicates)构建系统发育树,得到基因的种属信息和与已报道基因的亲缘关系。
1.3.4.3 等电点、信号肽等性质分析
用在线分析软件ExPASy ProtParam tool(http://web.expasy.org/protparam/)分析氨基酸等电点、半衰期、分子质量、不稳定系数和亲疏水性等。用TMHMM 2.0和SignalP 4.1 servers,分析氨基酸的跨膜区和信号肽序列。用SOPMA预测蛋白的二级结构。
1.3.5 bgl2238基因的PCR扩增与重组菌株的表达
根据测序结果设计一对引物:bgl2238-F和bgl2238-R,引物两端引入EcoRⅠ和XhoⅠ酶切位点,引物序列如下:bgl2238-fw:5′-CCGGAATTCATGAAAC ACATCCTAAACCTATGCC-3′(下划线部分为EcoRⅠ酶切位点);bgl2238-rv:5′-CCGCTCGAGTTATCGTACG CTAAAAGTAAGGGCTTC-3′(下划线部分为XhoⅠ酶切位点)。引物由Invitrogen公司合成;以包含pUC118-bgl2238的质粒为模板,采用Prime STARTMMax Premix进行PCR扩增,PCR回收试剂盒对扩增产物进行纯化回收。
分别用XhoⅠ和EcoRⅠ将纯化后的PCR产物和载体pET-32a(+)进行双酶切,用PCR回收试剂盒纯化双酶切产物,采用T4 DNA Ligase对双酶切处理的pET-32a(+)及PCR产物16 ℃连接12~16 h,直接转化E. coli BL21(DE3)。随机挑取转化子经培养后,提取其质粒并双酶切验证。挑取酶切验证正确的转化子于37 ℃摇床中培养,当OD值达到0.8左右时,加入IPTG,使其终浓度为1.0 mmol/L,25 ℃诱导表达11 h,收集菌体,超声破碎菌体后得粗酶液。粗酶液采用His·Bind®Resin试剂盒进行纯化。
1.3.6 β-葡萄糖苷酶活力的测定
将重组菌株接种于装有20 mL LB液体培养基中,25 ℃、220 r/min培养11 h,取菌液3 mL,6 000 r/min离心20 min,弃上清液,用冰预冷的无菌dd H2O洗涤沉淀两次,再用4 mL dd H2O重悬细胞沉淀,超声波破碎菌液直至变清,即为粗酶液。取10 μL一定稀释倍数的粗酶液、80 µL最适pH值的B-R缓冲液、50 mmol/L的pNPG 10 µL混合,最适温度下水浴20 min,取200 µL 1 mol/L Na2CO3溶液终止酶促反应,取上述混合物200 µL,测定OD405nm。一个酶活力单位(U)定义为每分钟分解pNPG产生1 µmol pNP所需的酶量。
2 结果与分析
2.1 玉米秸秆还田土壤总DNA的提取及纯化

图1 土壤基因组DNA的琼脂糖电泳分析Fig. 1 Agarose gel electrophoresis of soil genomic DNA
通过直接提取法获得玉米秸秆还田土壤基因组DNA,结果如图1所示,基因大小片段集中在15 kb以上,大约是23~30 kb之间,拖尾现象不明显,只有极少量DNA被降解,表明直接提取法获得的基因组DNA质量非常好,满足建库的需要。
2.2 土壤宏基因组文库的评估
将扩大酶切后的土壤基因组DNA与载体连接,电击转化入大肠杆菌E. coli DH5α,构建宏基因组文库,随机挑取16 个白色克隆子,EcoRⅠ酶切验证插入片段的大小,有14 个克隆子含有不同插入片段,文库阳性克隆率较高,重组率为87.5%,文库插入片段大小0.8~8.0 kb,该文库含有大约5 500 个阳性克隆子,其插入片段平均长度在4.5 kb左右,片段特异性较好。
2.3 土壤宏基因组文库中β-葡萄糖苷酶的筛选
利用底物七叶苷功能筛选法,对宏基因组文库进行初步筛选,从构建的宏基因组文库5 500 个克隆子中,发现1 个克隆子在七叶苷平板上有黑色水解圈,初步确定其具有β-葡萄糖苷酶活性,编号为44号克隆子,如图2所示。后期通过提质粒、酶切验证后送Invitrogen公司测序。
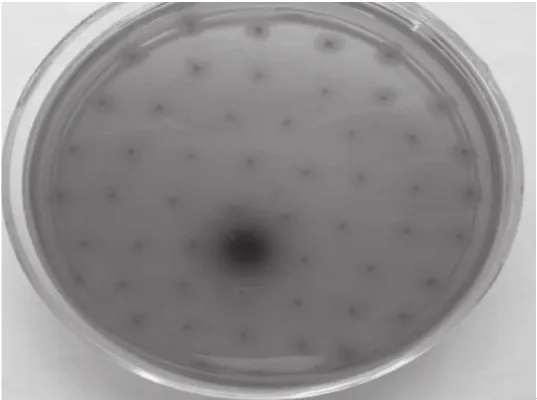
图 2β-葡萄糖苷酶阳性克隆子的筛选Fig. 2 Screening of positive clones with β-glucosidase activity
2.4 测序及生物信息学分析
2.4.1 NCBI的ORF Finder
将44号克隆子的质粒送交Invitrogen公司进行测序,测序结果显示44号克隆子基因序列全长为2 470 bp。利用NCBI网站中Sequence Analysis的在线查找工具ORF Finder(http://www.ncbi.nlm.nih.gov/gorf/gorf.html)对此序列进行ORF查找,从2 470 bp的插入片段中发现一个β-葡萄糖苷酶编码基因的ORF序列,该ORF基因序列全长2 238 bp,编码745 个氨基酸,基因命名为bgl2238,预测其蛋白分子质量为80.67 kDa,序列已提交至NCBI核酸数据库(GenBank),登录号为KU320675。经NCBI的BlastP在线序列比对分析,发现bgl2238基因序列所编码的蛋白bgl2238含有GH3超家族和GH3C超家族的结构域(图3),其与来源于Bacteroides thetaiotaomicron的β-葡萄糖苷酶(GenBank:WP_048691945.1)具有58%的相似性。

图3 推测的bgl2238的保守序列区域Fig. 3 Putative conserved domains of bgl2238
2.4.2 系统发育树
使用生物信息学软件Clustal W和MEGA 4.0对bgl2238与亲源性较近的蛋白质进行系统发育树的构建,结果如图4所示,可以看出,bgl2238与来源于Alistipes CAG: 435和未培养微生物(登录号:AAZ32298.1)的β-葡萄糖苷酶亲缘关系较近,与其他来源的β-葡萄糖苷酶亲缘关系都较远。
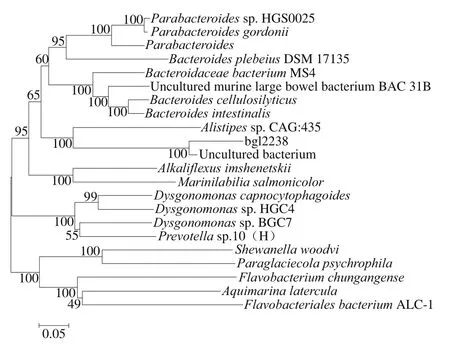
图4 bgl2238与亲缘性较近蛋白的系统发育分析Fig. 4 Phylogenetic analysis of bgl2238
2.4.3 bgl2238的氨基酸序列、二级结构分析
用生物信息学方法预测bgl2238的蛋白分子质量、等电点PI、半衰期、不稳定系数和亲疏水性等基本理化性质、氨基酸跨膜结构域和信号肽序列见表1,信号肽序列分析结果见图5,二级结构见图6。
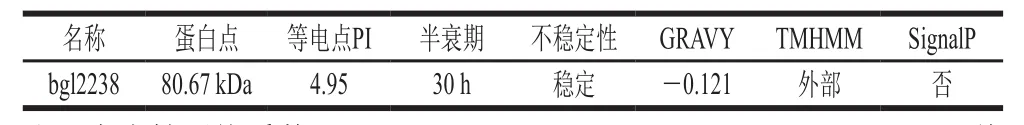
表1 bgl2238的氨基酸性质分析Table1 Amino acid properties of bgl2238
由表1可知,bgl2238的pI值为4.95,属酸性蛋白质,蛋白结构较稳定,疏水性高,位于细胞膜外且无信号肽序列。
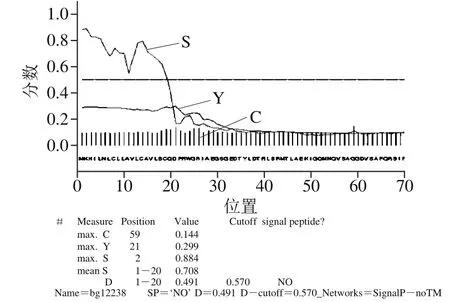
图5 预测的蛋白bgl2238信号肽分析Fig. 5 Signal peptide analysis prediction of protein bgl2238
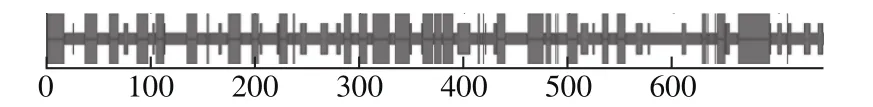
图6 预测蛋白bgl2238的二级结构Fig. 6 Secondary structure prediction of protein bgl2238
不同的mRNA结构可能编码不同的蛋白质二级结构,翻译慢的mRNA区域倾向于编码β折叠、无规则卷曲结构,翻译快的mRNA区域更倾向于编码α螺旋结构。由图6可知,bgl2238的二级结构中,α螺旋结构占37.05%,无规卷曲结构为49.93%,延伸片层结构占13.02%,说明蛋白bgl2238的mRNA翻译较快。
2.5 bgl2238基因克隆与表达
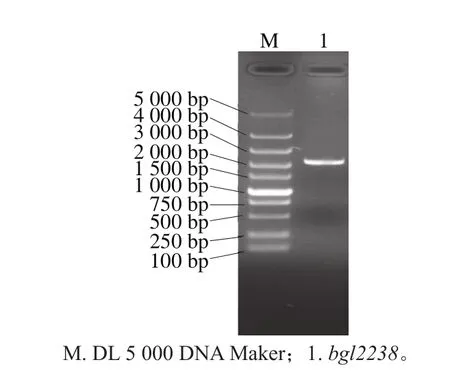
图7 基因bgl2238的PCR扩增产物Fig. 7 Amplified products of gene bgl2238
以pUC118-bgl2238重组质粒为模板,以bgl2238-F和bgl2238-R为引物进行PCR扩增,得到一条非常明显的DNA条带,大小位于2 000~3 000 bp之间,与预期基因大小2 238 bp一致(图7)。重组质粒pET-32a(+)-bgl2238直接化转E .coli BL21(DE3)感受态细胞,对重组菌株诱导表达得粗酶液。对粗酶液进行纯化,进行SDS-聚丙烯酰胺凝胶电泳检测,在大约100 kDa处出现了目标条带(图8),与理论计算得到β-葡萄糖苷酶分子质量大小(80.67 kDa)加上分子标签(18.15 kDa)相符合。
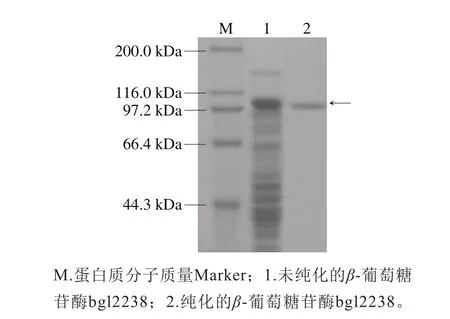
图8 bgl2238蛋白纯化SDS-聚丙烯酰胺凝胶电泳图Fig. 8 SDS-PAGE analysis of recombinant bgl2238
2.6 β-葡萄糖苷酶bgl2238酶学性质分析
2.6.1 pH值对酶活力及稳定性的影响
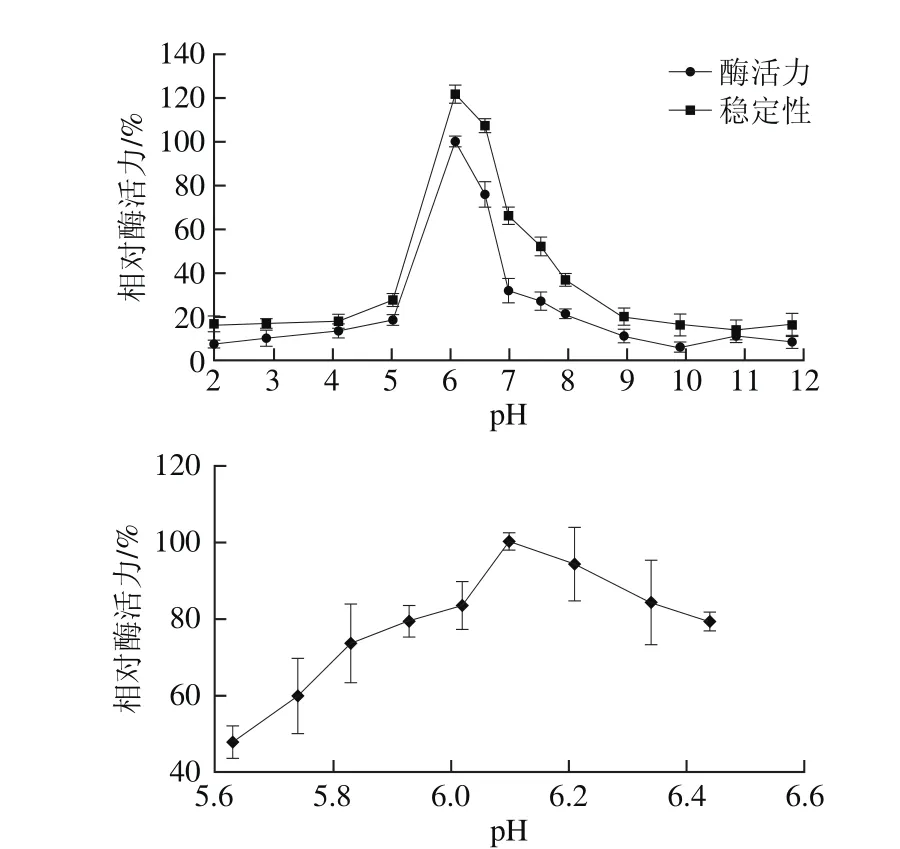
图9 pH值对β-葡萄糖苷酶bgl2238活力及稳定性的影响Fig. 9 Effect of pH value on the activity and stability of β-glucosidase bgl2238
保持反应温度为44℃,不同pH值缓冲液中(1.98、2.87、4.10、5.02、6.09、6.59、7.0、7.54、7.96、8.95、9.91、10.88、11.82),测定β-葡萄糖苷酶bgl2238活力,获得一级pH值梯度下的最适pH值范围后,将pH值梯度进一步缩小,以最终获得酶最适反应pH值。将经pH 2.0~12.0缓冲液稀释的粗酶液4 ℃放置4 h后,测定剩余酶活力。将最适pH值下未经4 h孵育的最高酶活力定义为100%。结果如图9所示,bgl2238酶的最适反应pH值为6.10,用pH 6.09~6.59缓冲液处理粗酶液4 h后仍能保持60%以上的酶活力,酶促反应的pH值范围较窄,在偏酸和偏碱性环境下bgl2238的酶活性和稳定性都很低,表明bgl2238属于一种偏中性酶。
2.6.2 温度对酶活力及稳定性的影响
于最适pH值的B-R缓冲液中,分别置于4、15、30、40、45、50、60、70 ℃和80 ℃水浴中进行酶活力测定,获得一级温度梯度下的最适温度范围,再将温度梯度进一步缩小,以获得酶最适反应温度。将适当稀释的粗酶液分别放于4~80 ℃不同水浴中保温4 h,测定剩余酶活力。以未经热处理(4 ℃)的酶液的酶活力定为100%。结果如图10所示,bgl2238酶促反应的最适温度为44 ℃,在32~44 ℃范围内,酶活力随温度的升高而增大,44 ℃后,酶活力随温度上升而急剧下降,至60 ℃时bgl2238完全失活;在4~50 ℃之间不同温度下热处理bgl2238 4 h,发现bgl2238仍保留70%以上的酶活力,表明β-葡萄糖苷酶活力在50 ℃以下稳定性良好。在44 ℃、pH 6.10时bgl2238酶活力最高,计算其酶活力为29.1 U/mg。
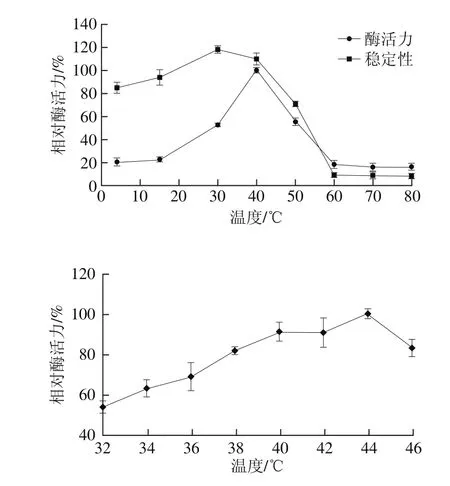
图10 温度对β-葡萄糖苷酶bgl2238活力及稳定性的影响Fig. 10 Effect of temperature on the activity and stability of β-glucosidase bgl2238
2.6.3 金属离子对酶活力的影响
在80 μL含金属离子终浓度分别为1 mmol/L或10 mmol/L的最适pH值缓冲液中加入10 μL经稀释的粗酶液,4 ℃放置4 h后,于最适温度条件下测定剩余酶活力。将未加金属离子的酶促反应中酶活力定义为100%。如图11所示,不同浓度的K+、Na+、Fe2+、Mg2+、Mn2+、Ca2+、Co2+和Ni2+对酶活力均有促进作用,其中Fe2+对bgl2238酶活力的促进作用最大,10 mmol/L Fe2+使相对酶活力高达260%,增加至酶的原始活性的2 倍多,而重金属离子Ag+、Hg2+及Cu2+对酶活力有强烈抑制作用。
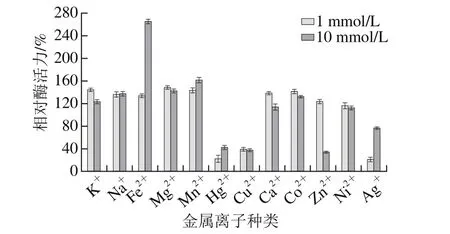
图11 金属离子对β-葡萄糖苷酶bgl2238活性的影响Fig. 11 Effect of metal ions on the activity of β-glucosidase bgl2238
2.6.4 β-葡萄糖苷酶bgl2238酶促动力学分析
以pNPG为底物,将粗酶液稀释50 倍,利用Lineweaver-Burk双倒数法测定bgl2238的米氏常数,以1/V为纵坐标、1/[S]为横坐标作图,曲线与x轴相交的点为-1/Km,与y轴相交的点为1/Vmax。结果如图12所示,β-葡萄糖苷酶bgl2238以pNPG为底物时,Vmax=576 μmoL/(L·min),Km= 0.296 mmol/L。
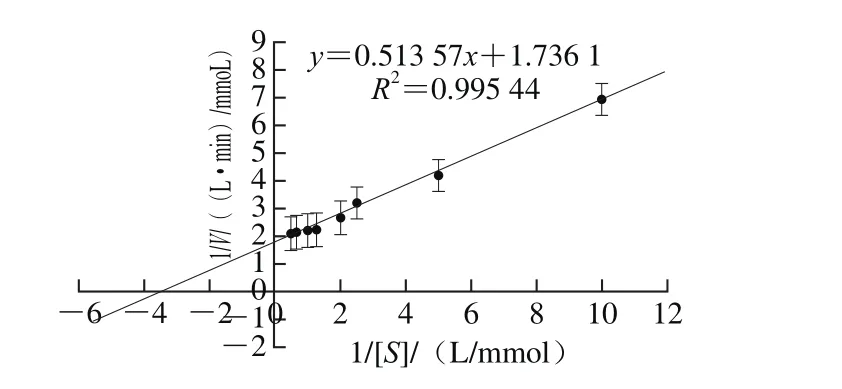
图12 β-葡萄糖苷酶bgl2238以pNPG为底物的动力学参数Fig. 12 Lineweaver-Burk plot for β-glucosidase bgl2238 with pNPG as substrate
3 结 论
宏基因组技术通过基因克隆、序列筛选或者功能筛选法、基因表达等手段,避开了绝大多数微生物难以分离培养的问题,为挖掘新型生物催化剂开辟了新途径[23-29]。本研究筛选到的β-葡萄糖苷酶bgl2238经测定,最适pH值为6.10,用pH 6.09~6.59缓冲液处理粗酶液4 h后仍能保持60%以上的酶活力,表明bgl2238属于一种偏中性酶,在生产过程中只需保持酶促反应的pH值在中性附近即可,对生产设备的酸碱度耐受性要求低,节约成本。β-葡萄糖苷酶bgl2238最适温度为44 ℃,当酶促反应温度升高到50 ℃时,相对酶活力不足40%,说明此酶不耐高温。不同的金属离子影响酶的活性,目前大约35%已知酶在金属离子存在下活性增加[30-31],金属离子通过与带负电的氨基酸残基结合而增加酶结构的稳定性。本研究中不同浓度的几种金属离子对β-葡萄糖苷酶bgl2238活力均有激活作用,其中Fe2+对酶活力的促进作用最大,10 mmol/L Fe2+使相对酶活力高达260%,增加至酶的原始活性的2 倍多,利用β-葡萄糖苷酶bgl2238这一特性,向酶促反应体系中加入适当浓度的K+、Na+、Fe2+可以明显提升bgl2238的酶活力,从而避免某些酶因严重依赖一些金属离子(如Mg2+、Mn2+、Cu2+、Ca2+、Co2+、Zn2+和Ni2+等)而造成的金属环境污染。为使得bgl2238能够更好、更广泛地应用于工业生产,后期可通过易错PCR对β-葡萄糖苷酶bgl2238进行改造并构建突变文库,以期获得对温度和pH值耐受性更好的酶。
[1] WHITMAN W B, COLEMAN D C, WIEBE W J. Prokaryotes:the unseen majority[J]. Proceedings of the National Academy of Sciences of the United States of America, 1998, 95(12): 6578-6583.DOI:10.1073/pans.95.12.6578.
[2] COWAN D A. Microbial genomes: the untapped resource[J].Trends in Biotechnology, 2000, 18(1): 14-16. DOI:10.1016/S0167-7799(99)01395-5.
[3] BAKKEN L R. Separation and purification of bacteria from soil[J].Applied & Environmental Microbiology, 1985, 49(6): 1482-1487.
[4] HANDELSMAN J, RONDON M R, BRADY S F, et al. Molecular biological access to the chemistry of unknown soil microbes: a new frontier for natural products[J]. Chemical & Biology, 1998, 5(10):R245-R249. DOI:10.1016/S1074-5521(98)90108-9.
[5] ANDERSON R E, SOGIN M L, BAROSS J A. Evolutionary strategies of viruses, bacteria and archaea in hydrothermal vent ecosystems revealed through metagenomics[J]. PLoS ONE, 2014, 9(10): e109696.DOI:10.1371/journal.pone.0109696.
[6] MCDONALD J E. The microbial ecology of anaerobic cellulose degradation in municipal waste landfill sites: evidence of a role for fibrobacters[J]. Environmental Microbiology, 2012, 14(4): 1077-1087.DOI:10.1111/j.1462-2920.2011.02688.x.
[7] 石元春. 生物质能源主导论: 为编制国家“十二五”规划建言献策[J]. 能源与节能, 2011(1): 1-7. DOI:10.3969/j.issn.2095-0802.2011.01.002.
[8] 段晓云. 代表性纤维素酶家族活性架构的功能位点分析[D]. 济南:山东大学, 2013: 1-70.
[9] 高蕾蕾, 李迎秋. 纤维素酶及其在食品行业中的应用[J]. 食品工业,2017(2): 271-274.
[10] HANDA C L, COUTO U R, VICENSOTI A H, et al. Optimisation of soy flour fermentation parameters to produce β-glucosidase for bioconversion into aglycones[J]. Food Chemistry, 2014, 152: 56-65.DOI:10.1016/j.foodchem.2013.11.101.
[11] TAMAKI F K, ARAUJO É M, ROZENBERG R, et al. A mutant β-glucosidase increases the rate of the cellulose enzymatic hydrolysis[J]. Biochemistry & Biophysics Reports, 2016, 7: 52-55.DOI:10.1016/j.bbrep.2016.05.014.
[12] ÜNAL M Ü, ŞENER A. Biochemical properties of β-glucosidase from Turkish Hacıhaliloğlu apricot (Prunus armenica L.) as affected by harvest year[J]. LWT-Food Science and Technology, 2017, 79: 190-196. DOI:10.1016/j.lwt.2017.01.033.
[13] 周汉琛, 雷攀登, 丁勇. 茶树β-葡萄糖苷酶研究进展[J]. 茶叶科学,2016, 36(2): 111-118. DOI:10.3969/j.issn.1000-369X.2016.02.001.
[14] RAHIKAINEN J L, MARTINSAMPEDRO R, HEIKKINEN H,et al. Inhibitory effect of lignin during cellulose bioconversion: the effect of lignin chemistry on non-productive enzyme adsorption[J].Bioresource Technology, 2013, 133(2): 270-278. DOI:10.1016/j.biortech.2013.01.075.
[15] RAHIKAINEN J L, MOILANEN U, NURMI-RANTALA S, et al.Effect of temperature on lignin-derived inhibition studied with three structurally different cellobiohydrolases[J]. Bioresource Technology,2013, 146(10): 118-125. DOI:10.1016/j.biortech.2013.07.069.
[16] POPOVIC A, TCHIGVINTSEV A, TRAN H, et al. Metagenomics as a tool for enzyme discovery: hydrolytic enzymes from marinerelated metagenomes[M]. Springer International Publishing, 2015: 1.DOI:10.1007/978-3-319-23603-2_1.
[17] MANIVASAGAN P, VENKATESAN J, SIVAKUMAR K, et al. Marine actinobacterial metabolites: current status and future perspectives[J]. Microbiological Research, 2013, 168(6): 311-332.DOI:10.1016/j.micres.2013.02.002.
[18] SUBRAMANI R, AALBERSBERG W. Culturable rare actinomycetes:diversity, isolation and marine natural product discovery[J]. Applied Microbiology and Biotechnology, 2013, 97(21): 9291-9321.DOI:10.1007/s00253-013-5229-7.
[19] ZHOU J Z, BRUNS M A, TIEDJE J M. DNA recovery from soils of diverse composition[J]. Applied & Environmental Microbiology, 1996,62(2): 316-322.
[20] 李松, 杨倩, 范李龙, 等. 碱性β-葡萄糖苷酶产生菌株的筛选、鉴定及部分酶学性质研究[J]. 食品工业科技, 2016, 37(2): 180-184.DOI:10.13386/j.issn1002-0306.2016.02.028.
[21] SIEVERS F, WILM A, DINEEN D, et al. Fast, scalable generation of high-quality protein multiple sequence alignments using Clustal Omega[J]. Molecular Systems Biology, 2011, 7(7): 1429-1432.DOI:10.1038/msb.2011.75.
[22] TAMURA K, STECHER G, PETERSON D, et al. MEGA6: Molecular evolutionary genetics analysis version 6.0[J]. Computer Applications in the Biosciences Cabios, 2013, 30(12): 2725-2729. DOI:10.1093/molbev/mst197.
[23] LIU J, JIA Z, LI S, et al. Identification and characterization of a chitin deacetylase from a metagenomic library of deep-sea sediments of the Arctic Ocean[J]. Gene, 2016, 590(1): 79-84. DOI:10.1016/j.gene.2016.06.007.
[24] MORI T, KAMEI I, HIRAI H, et al. Identification of novel glycosyl hydrolases with cellulolytic activity against crystalline cellulose from metagenomic libraries constructed from bacterial enrichment cultures[J].SpringerPlus, 2014, 3(1): 1-7. DOI:10.1186/2193-1801-3-365.
[25] LI Y, LIU N, YANG H, et al. Cloning and characterization of a new β-glucosidase from a metagenomic library of rumen of cattle feeding with Miscanthus sinensis[J]. BMC Biotechnology, 2014, 14(1): 1-9.DOI:10.1186/1472-6750-14-85.
[26] JIMÉNEZ D J, MONTAÑA J S, ÁLVAREZ D, et al. A novel cold active esterase derived from Colombian high Andean forest soil metagenome[J]. World Journal of Microbiology and Biotechnology,2012, 28(1): 361-370. DOI:10.1007/s11274-011-0828-x.
[27] ZHU Y B, LI J B, CAI H N, et al. Characterization of a new and thermostable esterase from a metagenomic library[J]. Microbiological Research, 2013, 168(9): 589-597. DOI:10.1016/j.micres.2013.04.004.
[28] SANTOS D F K D, ISTVAN P, NORONHA E F, et al. New dioxygenase from metagenomic library from Brazilian soil: insights into antibiotic resistance and bioremediation[J]. Biotechnology Letters,2015, 37(9): 1-9. DOI:10.1007/s10529-015-1861-x.
[29] 楚雍烈, 杨娥. 宏基因组学及其技术的研究进展[J]. 西安交通大学学报(医学版), 2008, 29(6): 601-608.
[30] 翁彩红, 冯宏昌, 赖春芬, 等. 松杉灵芝漆酶的分离纯化及酶学特性研究[J]. 药物生物技术, 2017(1): 21-26.
[31] 肖超, 张梁, 李颜颜, 等. 蜡样芽胞杆菌磷脂酶C在乳酸克鲁维酵母中重组表达、纯化及酶学性质分析[J]. 微生物学报, 2017, 57(1):87-96.

