干酪乳杆菌对北京黑猪保育阶段生长性能及肠道菌群的影响
王四新 季海峰* 石国华 刘 辉 王红卫 张董燕 王 晶 张 伟 王雅民
(1.北京市农林科学院畜牧兽医研究所,北京 100097;2.北京黑六牧业科技有限公司,北京 102211)
随着科技发展和社会进步,养殖业中饲用抗生素将逐渐被禁用,寻求抗生素替代品和替代技术备受关注。益生菌能够改善肠道菌群平衡,产生益生性物质,促进动物健康,提高生产性能,被人们寄予了厚望。干酪乳杆菌(Lactobacilluscasei)是益生菌的一种,农业部《饲料添加剂品种目录》(2013)允许其在养殖动物中添加使用。县怡涵等[1]、张魏伟等[2]和王齐桓等[3]给仔猪(大×长和杜×长×大)饲喂含干酪乳杆菌的饲粮后发现促进了仔猪肠道绒毛及黏膜上皮细胞发育,提高了仔猪采食量和日增重,降低了料重比。有关益生菌和抗生素对仔猪肠道菌群的影响,Zhang等[4]给仔猪(长×大)口服罗伊氏乳杆菌制剂或金霉素后发现,与金霉素组相比,罗伊氏乳杆菌组仔猪空肠内的菌群ACE指数和Chao1指数变大,菌群丰富度增加;Looft等[5]在仔猪饲粮中添加抗生素后发现肠道内容物中变形菌门的相对含量增加。然而,Poole等[6]在仔猪(杜×大)饲粮添加中低剂量(50 g/t)金霉素后没有发现仔猪粪便菌群结构和多样性发生显著变化;Li等[7]在仔猪(杜×长×大)饲粮中用粪肠球菌替代抗生素,发现猪肠道菌群变化规律与添加抗生素类似。对于不同猪种的肠道(粪便)菌群差异,杨柳等[8]、肖文萍[9]、杨丽娜[10]和杨伟平[11]分别研究了长白猪、大白猪和杜洛克猪以及藏猪、荣昌猪、巴马猪、二花脸和梅山猪的肠道(粪便)菌群情况,发现不同品种猪的粪样菌群种/属及数量存在较大差异,国内品种比国外瘦肉型品种的肠道(粪便)菌群多样性更丰富。北京黑猪是我国自主培育的特色品种,与引进品种杜洛克、长白猪、大白猪或其杂交猪相比,具有肉质好、耐粗饲和抗病力强等特点,越来越受到民众的欢迎。目前,有关干酪乳菌对北京黑猪的应用研究尚未见报道。开展干酪乳杆菌对北京黑猪保育阶段生长性能和肠道菌群的研究,将会为北京黑猪的科学养殖和绿色发展提供技术支撑。
1 材料与方法
1.1 试验材料
干酪乳杆菌活菌冻干制剂(活菌数2.0×1010CFU/g),由北京市农林科学院畜牧兽医研究所制备。15%金霉素预混剂购自金河集团。
1.2 试验动物分组与饲养管理
选用北京黑猪保育阶段猪120头,(35±2)日龄、体重为(7.53±0.21) kg,随机分成3组,分别为对照组、干酪乳杆菌组和金霉素组,每组4栏(重复),每栏10头。对照组试验猪饲喂基础饲粮(不添加干酪乳杆菌和金霉素);干酪乳杆菌组试验猪饲喂在基础饲粮中添加干酪乳杆菌活菌冻干制剂的饲料,每千克饲料中有效活菌数在4.0×109CFU;金霉素组试验猪饲喂在基础饲粮中添加金霉素预混剂的饲料,每千克饲料含金霉素75 mg。基础饲粮的组成及营养水平见表1。
试验在同一保育猪舍进行,实行高床分栏饲养,自由采食,自由饮水,舍内温度24~28 ℃,相对湿度60%~70%,猪群饲养管理按场内统一技术规程进行,试验期为30 d。
1.3 生长性能测定
分别在试验开始和试验结束时空腹称重,记录每天消耗饲粮情况,计算平均日增重、平均日采食量和料重比。
1.4 粪样采集与总DNA提取、16S rRNA测序
粪样采集:在试验的第28天清晨饲喂前采集粪样,每栏采集2头份,每组采集8头份,共采集粪样24头份。用无菌不锈钢勺采集猪自然排出的粪便,每头采集新鲜粪便20 g于无菌管中,置于干冰中,带回实验室,保存于-80 ℃冰箱,用于提取总DNA。
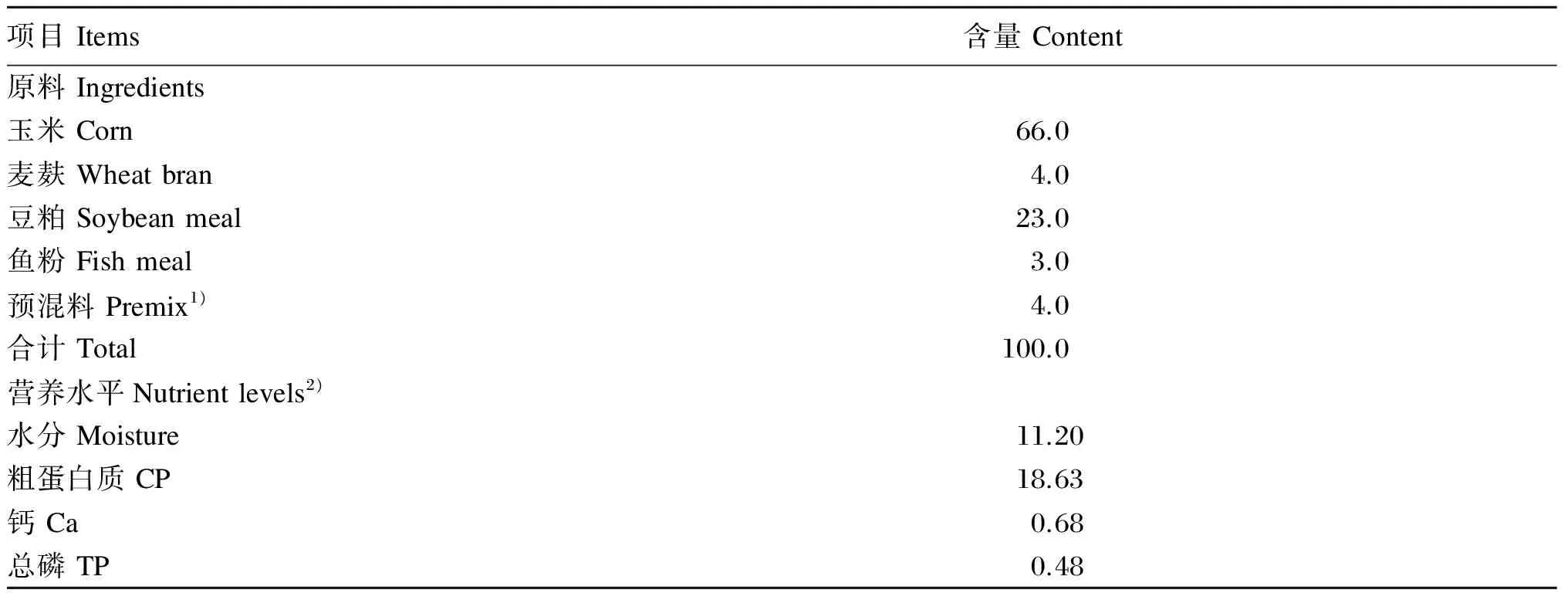
表1 基础饲粮组成及营养水平(风干基础)
1)预混料为每千克饲粮提供The premix provides the following per kilogram of the diet:Cu (as copper sulfate) 150 mg,Fe (as ferrous sulfate) 150 mg,Zn (as zinc sulfate) 140 mg,Mn(as manganese sulfate) 40 mg,Se (as sodium selenite) 0.3 mg,I (as potassium iodide) 0.6 mg,VA 8 000 IU,VD31 800 IU,VE 24 IU,VK 1.8 mg,VB11.6 mg,VB26 mg,VB63.2 mg,VB120.05 mg,生物素 biotin 0.3 mg,叶酸 folic acid 1.2 mg,烟酸 nicotinic acid 24 mg,泛酸 pantothenic acid 18 mg,氯化胆碱 choline chloride 0.4 g,L-赖氨酸盐L-lysine salt 2.2 g,苏氨酸盐 threonine salt 0.4 g,钙 calcium 5 g,总磷 total phosphorus 3 g。
2)水分、粗蛋白质、钙和总磷为实测值。Moisture, CP, Ca and TP are measured values.
总DNA提取:用TIANamp微生物DNA Kit[天根生化科技(北京)有限公司]提取猪粪便中细菌总DNA,具体提取步骤参照试剂盒说明书进行。用DNA纯化试剂盒对提取的总DNA进行纯化,用原子分光光度计和琼脂糖凝胶电泳检验总DNA浓度和纯度,然后置于-20 ℃冰箱保存备用。
16S rRNA基因扩增及测序:以北京黑猪保育阶段猪粪便细菌总DNA为模板,PCR扩增细菌16S rRNA的V3~V4区。扩增体系:4 μL的5×FastPfu Buffer,2 μL的2.5 mmol/L dNTPs,各0.8 μL的上、下游引物(5 μmol/L),0.4 μL的FastPfu DNA Polymerase,10 ng的DNA模板,最终体积20 μL。采用细菌通用引物:338F(5′-ACTCCTACGGGAGGCAGCA-3′)和806R(5′-GGACTACHVGGGTWTCTAAT-3′)。PCR反应条件为:95 ℃变性3 min;95 ℃ 30 s,55 ℃ 30 s,72 ℃ 45 s,进行27个循环;72 ℃,延伸10 min。每个样本3个重复,将同一样本的PCR产物混合后用2%琼脂糖凝胶电泳检测,使用AxyPrep DNA凝胶回收试剂盒(Axygen公司)切胶回收PCR产物,Tris-HCl洗脱;2%琼脂糖凝胶电泳检测。参照电泳初步定量结果,将PCR产物使用QuantiFluorTM-ST蓝色荧光定量系统(Promega公司)进行检测定量,之后按照每个样本的测序量要求,进行相应比例的混合;在Illumina Miseq平台进行PE250测序。
1.5 数据分析
过滤read尾部质量值20以下的碱基,设置50 bp的窗口,如果窗口内的平均质量值低于20,从窗口开始截去后端碱基,过滤质控后50 bp以下的reads;根据PE reads之间的overlap关系,将成对reads拼接(merge)成一条序列,最小overlap长度为10 bp;拼接序列的overlap区允许的最大错配比率为0.2,筛选不符合序列;根据序列首尾两端的barcode和引物区分样品,并调整序列方向,barcode允许的错配数为0,最大引物错配数为2。按照97%相似性对非重复序列(不含单序列)进行操作分类单元(OTU)聚类,在聚类过程中去除嵌合体,得到OTU的代表序列,将所有优化序列map至OTU代表序列,选出与OTU代表序列相似性在97%以上的序列,生成OTU表格。为了得到每个OTU对应的物种分类信息,采用RDP classifier贝叶斯算法对97%相似水平的OTU代表序列进行分类学分析,并在各个水平统计每个样本的菌群组成。
生长性能数据结果用平均值±标准误表示,数据处理与分析采用SPSS 19.0统计软件中的ANOVA程序进行单因素方差分析,采用Duncan氏法进行多重比较,以P<0.05作为差异显著性判断标准。
2 结果与分析
2.1 生长性能情况
由表2可知,在北京黑猪保育阶段饲粮中添加干酪乳杆菌和金霉素均能提高平均日增重、降低料重比。与对照组相比,干酪乳杆菌组和金霉素组的平均日增重分别提高了12.71%(P<0.05)和8.58%(P<0.05),料重比分别降低了7.34%(P<0.05)和4.52%(P>0.05)。与金霉素组相比,干酪乳杆菌组的平均日增重和料重比都有所改善,但差异不显著(P>0.05)。

表2 干酪乳杆菌对北京黑猪保育阶段生长性能的影响
同行数据肩标不同小写字母表示差异显著(P<0.05)。
In the same row, values with different small letter superscripts mean significant difference (P<0.05).
2.2 肠道菌群情况
2.2.1 序列及OTU统计
对照组、干酪乳杆菌组和金霉素组共采集鲜粪样24个,对其分析后共获得了1 439 917条高质量序列,有效长度为634 742 973 bp,平均长度为440.82 bp。对照组、干酪乳杆菌组和金霉素组的序列数分别为467 327、488 949和483 641,长度分别为206 235 445、215 290 320和213 217 208 bp。
按照97%相似性,对非重复序列(不含单序列)进行OTU聚类,聚类过程中去除嵌合体,得到所有24个粪样OTU的代表序列871 056、OTU数14 390,对照组、干酪乳杆菌组和金霉素组粪样的OTU数分别为4 782、4 920和4 688。由图1可知,对照组、干酪乳杆菌组和金霉素组共有的OTU数为823,对照组独有的OTU数为5,干酪乳杆菌组独有的OTU数为29,金霉素组独有的OTU数为3。
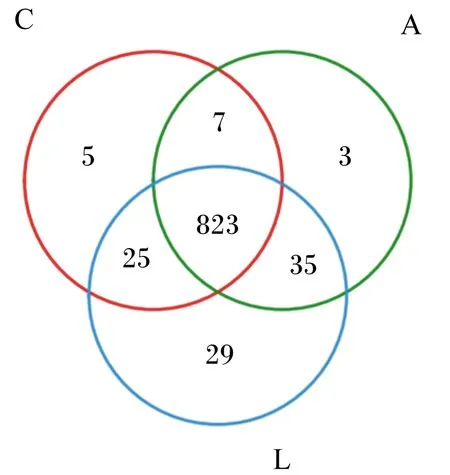
C:对照组;L:干酪乳杆菌组;A:金霉素组。下图同。
C: control group; L:Lactobacilluscaseigroup; A: chlortetracycline group. The same as below.
图13组粪样OTU维恩图
Fig.1 The OTU Venn diagram of fecal samples among three groups
2.2.2 肠道菌群多样性
由表3可知,对照组、干酪乳杆菌组和金霉素组粪样的测序覆盖率在99.68%~99.72%。3组粪
样中,对照组的ACE指数和Chao1指数最低;干酪乳杆菌组的ACE指数、Chao1指数和Shannon指数都是最高;金霉素组的Simpson指数最高,Shannon指数最低。通过对3组粪样中ACE指数、Chao1指数、Shannon指数和Simpson指数比较,发现干酪乳杆菌组的肠道菌群丰富度和多样性高于对照组和金霉素组。
2.2.3 肠道菌群组成
在门分类水平上,菌群组成情况见图2和图3。3组粪样中总共有17个菌门,其中拟杆菌门(Bacteroidetes)、厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)和螺旋体门(Spirochaetae)为优势菌门,它们的相对丰度在99.02%~99.07%。3组粪样中的优势菌门相对丰度有差异,其中金霉素组的拟杆菌门相对丰度为51.28%,厚壁菌门、变形菌门和螺旋体门的相对丰度分别为44.94%、2.38%和0.43%;干酪乳杆菌组和对照组的优势菌门相对丰度相近,它们的拟杆菌门、厚壁菌门、变形菌门和螺旋体门相对丰度分别为47.30%和47.30%、47.89%和46.86%、3.01%和3.89%、0.82%和1.02%。另外,干酪乳杆菌组独有疣微菌门(Verrucomicrobia),其相对丰度为0.000 69%。
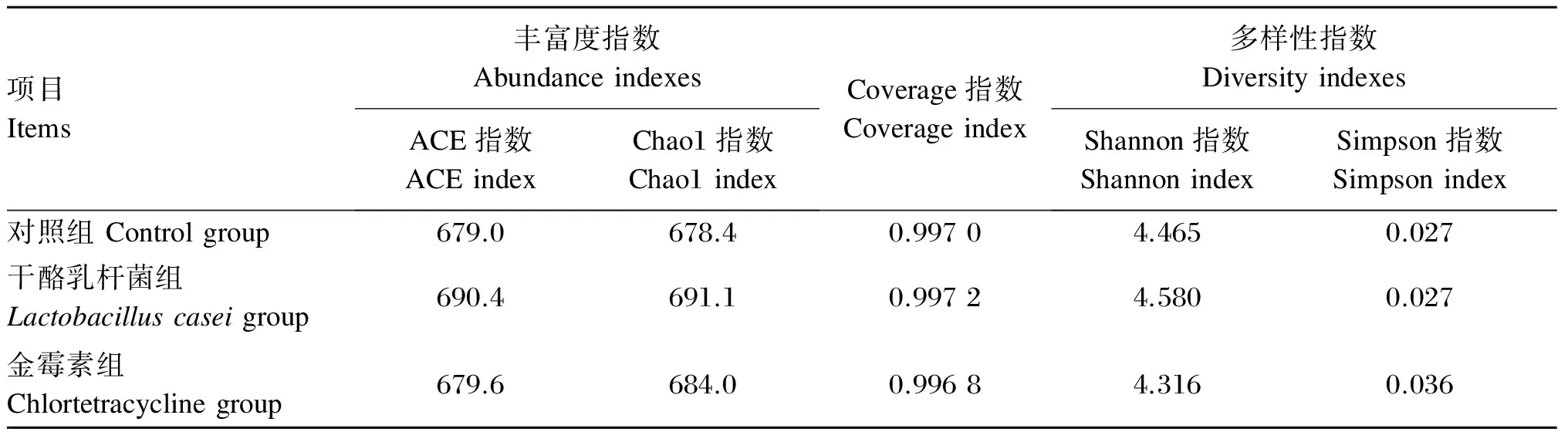
表3 3组粪样的菌群多样性指数
在属分类水平上,菌群组成情况见图4和图5。3组粪样中总共有206个菌属,对照组、干酪乳杆菌组和金霉素组粪样分别含有198、200和197个菌属,相对丰度大于1%的菌属有30个,总相对丰度在83.28%~86.02%。3组粪样中的菌属相对丰度不同,金霉素组中普氏菌属9(Prevotella_9)的相对丰度较高,为27.90%,干酪乳杆菌组和对照组的相近,分别为19.87%和20.27%;金霉素组中普雷沃氏菌科NK3B31群(Prevotellaceae_NK3B31_group)的相对丰度(5.12%)低于干酪乳杆菌组和对照组(分别为7.31%和7.22%)。另外,在对照组中,链球菌属(Streptococcus)和不可培养韦荣球菌科(Veillonellaceae_uncultured)的相对丰度较高,分别为5.31%和3.94%;在金霉素组中,巨型球菌属(Megasphaera)和乳酸杆菌属(Lactobacillus)的相对丰度较高,分别为6.47%和3.16%;在干酪乳杆菌组,未分类毛螺旋菌科(Lachnospiraceae_unclassified)和厌氧弧菌属(Anaerovibrio)的相对丰度较高,分别为5.76%和3.49%。

Bacteroidetes:拟杆菌门;Firmicutes:厚壁菌门;Proteobacteria:变形菌门;Spirochaetae:螺旋体门;Others:其他。
图2门水平上的优势菌群分布
Fig.2 Distribution of dominant flora at phylum level
2.2.4 相似性分析
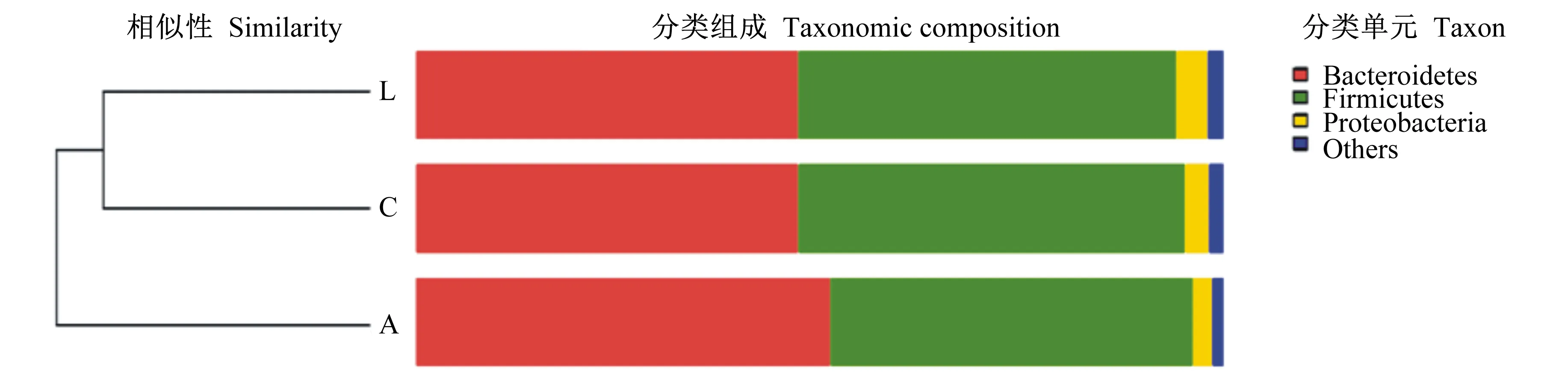
通过对3组粪样门水平上聚类树图分析,3组粪样中菌群组成和相对丰度有所不同,见图6。与对照组和干酪乳杆菌组相比,金霉素组粪样中拟杆菌门相对丰度较高,厚壁菌门和变形菌门相对丰度较低,对照组与干酪乳杆菌组的粪样中菌群组成和相对丰度较接近。
3 讨 论
3.1 干酪乳杆菌对北京黑猪保育阶段生长性能的影响
益生菌能够增强肠道屏障功能,增加小肠绒毛高度,增大小肠的吸收面积,促进机体对营养物质的消化吸收和健康生长。县怡涵等[1]、张魏伟等[2]和王齐桓等[3]的研究均表明干酪乳杆菌能够显著提高断奶仔猪小肠的绒毛高度,增加绒毛高度与肠隐窝深度的比值(V/C值),增加肌层厚度及增殖细胞核抗原(PCNA)含量,能有效维护断奶仔猪肠道健康,减轻大肠杆菌对肠黏膜结构的损害,促进肠道的发育,提高断奶仔猪的生长性能和饲料利用效率。本研究在北京黑猪保育阶段饲粮中分别添加干酪乳杆菌制剂和金霉素,发现干酪乳杆菌和金霉素均可提高仔猪的生长性能,干酪乳杆菌组的平均日增重比对照组提高了12.71%,料重比降低了7.34%;金霉素组的平均日增重比对照组提高了8.58%,料重比降低了4.52%;干酪乳杆菌组与金霉素组相比,平均日增重和料重比也有所改善,但差异不显著。本研究结果表明,北京黑猪保育阶段饲粮中添加干酪乳杆菌制剂可以显著提高仔猪的平均日增重,显著降低料重比,这与县怡涵等[1]给仔猪(大×长)饲喂干酪乳杆菌的效果基本一致,这也表明在猪保育阶段饲粮中适当添加干酪乳杆菌,不管是对国外引进猪种,还是国内培育猪种,均有助于维持肠道健康,提高仔猪生长性能。
3.2 干酪乳杆菌对北京黑猪保育阶段肠道菌群的影响
动物肠道中栖息着数量庞大且复杂多样的菌群,菌群与宿主之间存在着互利共生关系,宿主为菌群提供栖息地和营养,菌群协助宿主新陈代谢,肠道菌群对宿主营养消化和健康起着重要作用[12-14]。研究人员常常通过新鲜粪样中菌群的变化了解肠道内菌群的情况,在菌群多样性分析过程中,Coverage指数值越高,则样本中序列被测出的概率越高;ACE指数、Chao1指数和Shannon指数值越大,说明群落丰富度和多样性越高;Simpson指数值越大,说明群落多样性越低。本研究采用高通量测序技术对北京黑猪保育阶段24个鲜粪样进行了测序,测序覆盖率在99.68%~99.72%,并获得了1 439 917条高质量序列和14 390个OTU,利用维恩图对3组粪样进行比较发现,对照组、干酪乳杆菌组和金霉素组共有的OTU数为823个,干酪乳杆菌组独有的OTU数最多(29个),金霉素组独有的OTU数最少(3个)。对菌群进行多样性分析发现,干酪乳杆菌组粪样的ACE指数、Chao1指数和Shannon指数均高于对照组和金霉素组,表明干酪乳杆菌组的粪样菌群丰富度和多样性均高于对照组和金霉素组;而金霉素组的Simpson指数最高,表明金霉素组粪样中的菌群多样性是3组粪样中最低的。本研究结果说明,北京黑猪保育阶段饲粮中添加干酪乳杆菌可以增加肠道菌群的丰富度和多样性,而添加金霉素降低肠道菌群的多样性。Zhang等[4]研究了罗伊氏乳杆菌和金霉素对仔猪肠道菌群结构和多样性的影响,与金霉素组比较,罗伊氏乳杆菌组的空肠菌群ACE指数和Chao1指数增大,空肠菌群丰富度增加,这与本研究结果相一致。而Li等[7]在仔猪饲粮中用粪肠球菌代替抗生素,发现肠道菌群变化和添加抗生素时类似,其菌群丰富度和多样性均低于对照组。可见,不同微生物菌种对肠道菌群的影响效果是有差异的。
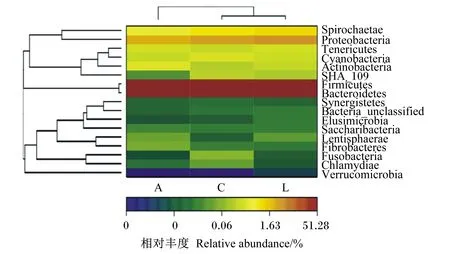
Spirochaetae:螺旋体门;Proteobacteria:变形菌门;Tenericutes:软壁菌门;Cyanobacteria:蓝细菌;Actinobacteria:放线菌门;Firmicutes:厚壁菌门;Bacteroidetes:拟杆菌门; Synergistetes:互养菌门;Bacteria_unclassified:未分类细菌;Lentisphaerae:黏胶球形菌门;Fibrobacteres:纤维杆菌门;Fusobacteria:梭杆菌门;Chlamydiae:衣原体门;Verrucomicrobia:疣微菌门。
图3菌群在门水平上的热图
Fig.3 Heatmap of microbiota at phylum level
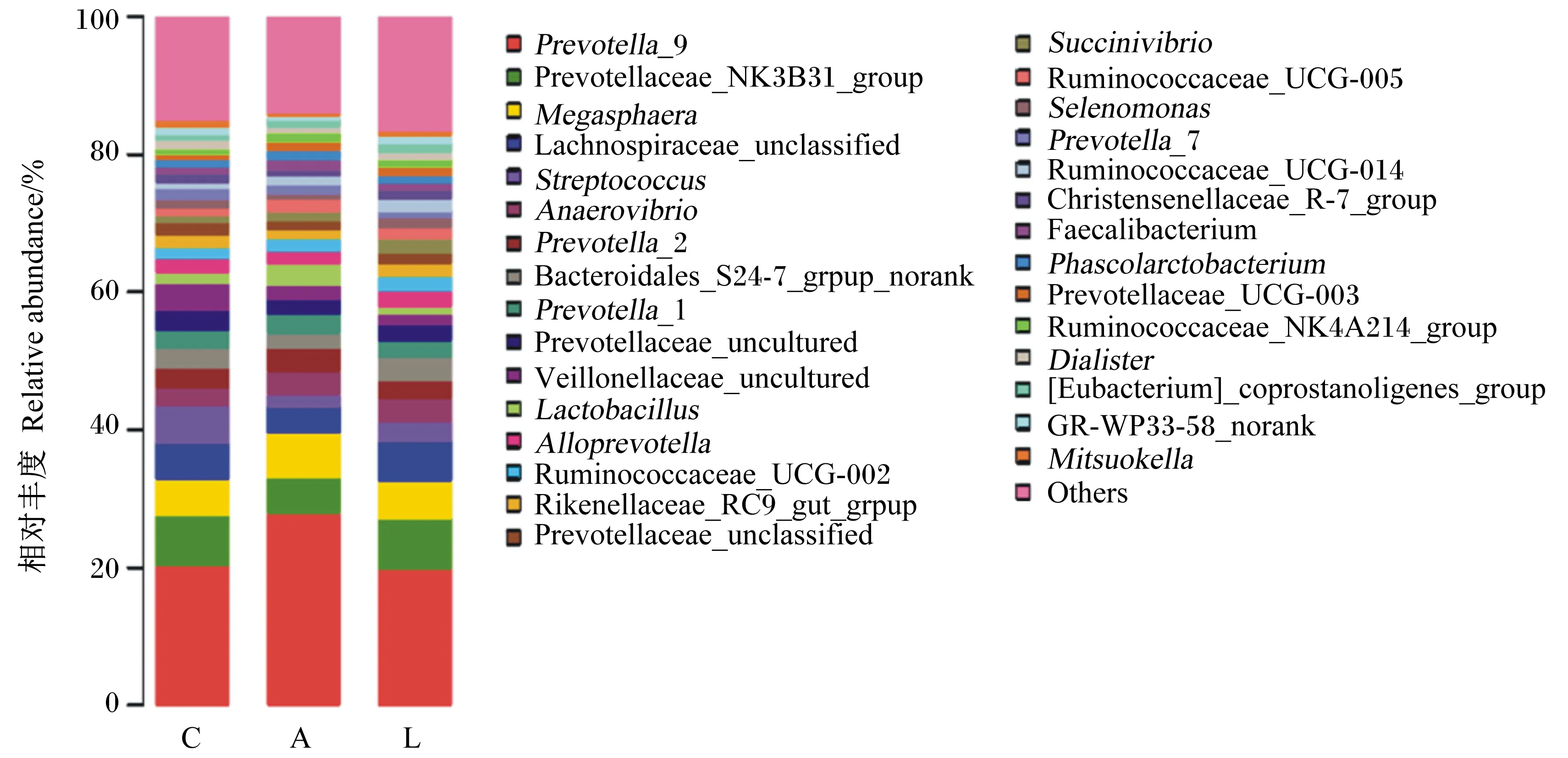
Prevotella_9:普氏菌属9;Prevotellaceae_NK3B31_group:普雷沃氏菌科NK3B31群;Megasphaera:巨型球菌属;Lachnospiraceae_unclassified:未分类毛螺旋菌科;Streptococcus:链球菌属;Anaerovibrio:厌氧弧菌属;Prevotella_2:普氏菌属2;Prevotella_1:普氏菌属1;Prevotellaceae_uncultured:不可培养普雷沃氏菌科;Veillonellaceae_uncultured:不可培养韦荣球菌科;Lactobacillus:乳杆菌属;Alloprevotella:拟普雷沃菌属;Ruminococcaceae_UCG-002:瘤胃菌科UCG-002;Prevotellaceae_unclassified:未分类普雷沃氏菌科;Succinivibrio:琥珀酸弧菌属;Ruminococcaceae_UCG-005:瘤胃菌科UCG-005;Selenomonas:月形单胞菌属;Prevotella_7:普氏菌属7;Ruminococcaceae_UCG-014:瘤胃菌科UCG-014;Phascolarctobacterium:考拉杆菌属;Prevotellaceae_UCG-003:普雷沃氏菌科UCG-003;Ruminococcaceae_NK4A214_group:瘤胃菌科NK4A214群;Dialister:小类杆菌属;Mitsuokella:光岗菌属;Others:其他。图5同 The same as Fig.5。
图4属水平上的优势菌群分布
Fig.4 Distribution of dominant flora at genus level

图5 菌群在属水平上的热图
Bacteroidetes:拟杆菌门;Firmicutes:厚壁菌门;Proteobacteria:变形菌门; Others:其他。
图6门水平上的菌群组成聚类树图
Fig.6 Microbiota community barplot with cluster tree at phylum level
国内外多位学者探讨了在饲粮中添加抗生素和益生菌对肠道菌群的影响,Kim等[15]在饲粮中添加泰乐菌素后发现可引起肠道内微生物种群的变化,特别是乳酸杆菌属、Sporacetigenium、Acetanaerobacterium和Eggerthella,比未添加组的检测频率高。Looft等[5]探讨了抗生素(金霉素、磺胺甲嘧啶和青霉素)对猪肠道菌群的影响,发现抗生素处理14 d后,与未处理组猪只相比,变形菌门相对含量增加1%~11%。而Poole等[6]在饲粮中添加金霉素,饲喂4周低剂量(50 g/t)的金霉素后发现其对仔猪(杜×大)肠道菌群结构无显著影响。Li等[7]以仔猪(杜×长×大)为对象,研究了饲粮中添加粪肠球菌代替抗生素的情况,发现猪肠道菌群变化规律与添加抗生素组类似。本研究发现,在北京黑猪保育阶段粪样中,厚壁菌门和拟杆菌门是优势菌门,普氏菌属和普雷沃氏菌科是优势菌属,这与Pajarillo等[16-17]、Kim等[18]和Zhao等[19]对杜洛克猪、长白猪和大白猪生长育肥阶段的粪便菌群的研究结果类似。3组粪样中菌群组成及相对丰度是有差异的,干酪乳杆菌组独有疣微菌门;金霉素组的拟杆菌门、放线菌门、纤维杆菌门、黏胶球形菌门和Saccharibacteria相对丰度比对照组和干酪乳杆菌组高,而厚壁菌门、变形菌门、螺旋体门和梭杆菌门的相对丰度低于其他2组;对照组粪样中链球菌属的相对丰度高于干酪乳杆菌组和金霉素组,说明饲粮中添加干酪乳杆菌或金霉素可以降低粪便中链球菌的数量;另外,研究发现饲粮中添加金霉素并没有降低粪样中乳酸杆菌属和埃希氏杆菌属-志贺氏菌属的相对丰度,添加干酪乳杆菌也没有提高粪样中乳酸杆菌属的相对丰度,这与Kim等[15]和Li等[7]的结论类似。
关于不同品种猪的肠道菌群差异情况,杨柳等[8]、肖文萍[9]、杨丽娜[10]和杨伟平[11]比较了藏猪、荣昌猪、巴马猪、二花脸、梅山猪以及长白猪、大白猪和杜洛克猪的肠道(粪便)菌群情况,发现不同品种猪的粪样菌群种/属及数量有较大差异,国内地方品种巴马猪、二花脸和梅山猪的粪样菌群聚为一簇,外来瘦肉型品种大白猪、约克夏和杜洛克的粪样菌群聚为一簇。国内猪种比国外瘦肉型猪种的肠道(粪便)菌群多样性更丰富,盲肠中的纤维素分解菌的数量更大。本研究也发现了类似规律,具有肉质好、耐粗饲和抗病力强等特点的北京黑猪,其保育阶段粪样中普氏菌属(Prevotella)、普雷沃氏菌科(Prevotellaceae)、瘤胃菌科(Ruminococcaceae)、毛螺旋菌科(Lachnospiraceae)和厌氧弧菌属等纤维素分解菌(或半纤维素分解菌)的相对丰度较高。
4 结 论
在北京黑猪保育阶段饲粮中添加干酪乳杆菌能增加仔猪肠道菌群的丰富度和多样性,有利于肠道健康,进而提高饲料利用率和仔猪的生长速度。
[1] 县怡涵,赵秀英,李晨博,等.植物乳杆菌和干酪乳杆菌对仔猪生长性能、器官指数及小肠形态的影响[J].动物营养学报,2015,27(12):3805-3811.
[2] 张魏伟,王安如,滕可导,等.干酪乳杆菌对断奶仔猪十二指肠发育的组织学影响[J].中国农业大学学报,2015,20(3):114-120.
[3] 王齐桓,王安如,周宁聪,等.干酪乳杆菌对断奶仔猪回肠黏膜结构及上皮内淋巴细胞数量的影响[J].中国兽医杂志,2016,52(2):34-37.
[4] ZHANG D Y,JI H F,LIU H,et al.Changes in the diversity and composition of gut microbiota of weaned piglets after oral administration ofLactobacillusor an antibiotic[J].Applied Microbiology and Biotechnology,2016,100(23):10081-10093.
[5] LOOFT T,JOHNSON T A,ALLEN H K,et al.In-feed antibiotic effects on the swine intestinal microbiome[J].Proceedings of the National Academy of Sciences of the United States of America,2012,109(5):1691-1696.
[6] POOLE T L,SUCHODOLSKI J S,CALLAWAY T R,et al.The effect of chlortetracycline on faecal microbial populations in growing swine[J].Journal of Global Antimicrobial Resistance,2013,1(3):171-174.
[7] LI P H,NIU Q,WEI Q T,et al.Microbial shifts in the porcine distal gut in response to diets supplemented withEnterococcusfaecalisas alternatives to antibiotics[J].Scientific Reports,20177:41395.
[8] 杨柳,张邑帆,郑华,等.荣昌、长白、杜洛克猪肠道微生物ERIC-PCR-DGGE指纹图谱比较分析[J].家畜生态学报,2011,32(5):21-25.
[9] 肖文萍.藏猪肠道微生物多样性的研究[D].硕士学位论文.杨凌:西北农林科技大学,2012:22-27.
[10] 杨丽娜.不同猪种间肠道微生物菌群以及生长相关指标的比较[D].硕士学位论文.南京:南京农业大学,2014:22-25.
[11] 杨伟平.藏猪肠道细菌群落组成与纤维素分解菌的研究[D].博士学位论文.杨凌:西北农林科技大学,2015:27-40.
[12] 朱伟云,余凯凡,慕春龙,等.猪的肠道微生物与宿主营养代谢[J].动物营养学报,2014,26(10):3046-3051.
[13] KIM H B,ISAACSON R E.The pig gut microbial diversity:Understanding the pig gut microbial ecology through the next generation high throughput sequencing[J].Veterinary Microbiology,2015,177(3/4):242-251.
[14] ISAACSON R,KIM H B.The intestinal microbiome of the pig[J].Animal Health Research Reviews,2012,13(1):100-109.
[15] KIM H B,BOREWICZ K,WHITE B A,et al.Microbial shifts in the swine distal gut in response to the treatment with antimicrobial growth promoter,tylosin[J].Proceedings of the National Academy of Sciences of the United States of America,2012,109(38):15485-15490.
[16] PAJARILLO E A B,CHAE J P,BALOLONG M P,et al.Assessment of fecal bacterial diversity among healthy piglets during the weaning transition[J].Journal of General and Applied Microbiology,2014,60(4):140-146.
[17] PAJARILLO E A B,CHAE J P,BALOLONG M P,et al.Pyrosequencing-based analysis of fecal microbial communities in three purebred pig lines[J].Journal of Microbiobogy,2014,52(8):646-651.
[18] KIM H B,BOREWICZ K,WHITE B A,et al.Longitudinal investigation of the age-related bacterial diversity in the feces of commercial pigs[J].Veterinary Microbiology,2011,153(1/2):124-133.
[19] ZHAO W J,WANG Y P,LIU S Y,et al.The dynamic distribution of porcine microbiota across different ages and gastrointestinal tract segments[J].PLoS One,2015,10(2):e0117441.
*Corresponding author, professor, E-mail: jhf207@126.com

