海洋放线菌酰基转移酶基因的克隆
□ 马艳玲 李海贤 翁 萍 刘泽璇 许小庆 曾 荣 佛山科学技术学院食品科学与工程学院

2000年后的海洋放线菌的研究受到极大的关注,强烈地吸引着各个领域的研究者[1]。其可以合成些具有结构新颖、功能多样且生物活性广泛的新型化合物[2-3],为一些新型的抗生素的发现提供了珍贵的资源指导。
在人体机体内,有着多种酰基转移酶,除了催化蛋白质的酰基化和去酰基化,还发挥着其他重要功能,例如脂酰辅酶A:胆固醇酰基转移酶,所催化的胆固醇的酯化反应无论在细胞还是个体水平的胆固醇代谢平衡中都发挥了极其重要的作用是细胞内已知的唯一一个催化游离胆固醇与长链脂肪酸连接形成胆固醇酯的酶,是参与吸收、转运和贮存胆固醇的一个及其重要的酶,可直接影响血液中胆固醇水平[4]。如何表达这些潜在的基因簇来解决食物污染及耐药性问题一直是现在的重要研究方向,异体表达这些基因簇可以成为解决这个问题的方法之一[5-6]。因此通过生物转基因技术研究海洋放线菌酰基转移酶基因的克隆,能够使海洋放线菌酰基转移酶更好地产生各种独特的具有生物活性的代谢物和酶。
1 试验材料与方法
1.1 试验材料
1.1.1 菌种来源
海洋放线菌Salinispora CNS-205,为本实验室保存。
1.1.2 主要试剂
大肠杆菌DH10B;限制性内切酶NdeI和EcoRI;DNA分子标准量marker;DNA聚合酶。DNA提取试剂盒,T4DNA 连接酶,凝胶回收试剂盒,质粒小提试剂盒,均购买自天根生化科技(北京)有限公司;其余均为国产分析纯试剂。
1.2 试验方法
1.2.1 重组质粒的构建
PCR反应体系:H2O 28.5 μL,5×FAST HiFidelity PCR Buffer 10 μL, 20×FAST PCR Enhancer 2.5 μL, DMSO 2 μL,Template 2 μL,PrimerL 2 μL, PrimerR 2 μL, FAST HiFidelity polymerase 1 μL。PCR程序:升温90 ℃,2 min;DNA变性95 ℃,30 s;退火53 ℃,30 s;引物延伸72 ℃,2 min;30周期后,72 ℃维持10 min,电泳。PCR产物的片段为双链平末端,需通过酶切的方式将粘性末端暴露出来。酶切反应体系如下(50 mL反应体系)。
水 11 mL
EcoR I Buffer 5 mL
PCR回收样品 30 mL
EcoR I 2 mL
Nde I 2 mL
然后放入37 ℃水浴锅中水浴2 h,经胶回收后的PCR产物连入pUC18载体。酶连体系(20 mL反应体系)。
灭菌水 6 mL
PCR样品回收片段 10 mL
PUC-18回收片段 1 mL
10×T4 DNA Ligase Buffer 2 mL
T4 DNA Ligase 1 mL
1.2.2 酶切验证
用EcoR I和NdeI酶切,酶切反应体系如下(10 mL反应体系)。
水 6.2 mL
EcoR I Buffer 1 mL
抽提的质粒样品 2 mL
EcoR I 0.4 mL
Nde I 0.4 mL
2 试验结果与分析
2.1 目的基因的PCR扩增
以海洋放线菌总DNA为模板, 用上述引物进行扩增,再EcoRI 和NdeI酶切回收。原PCR产物的目的条带应位于大约1100bp处,然而酶切回收后,扩增产物经凝胶电泳检测得出在约1100bp处有一条亮带(如图1所示),有待进一步鉴定。

图1 PCR电泳
2.2 重组质粒的验证
图2中第一个条带在约2 700 bp左右,为载体的酶切条带;第二个条带位于1 100 bp左右,为目的基因的酶切条带。得出该重组质粒后,测序委托南京金斯瑞生物科技有限公司完成。结果如图2所示。
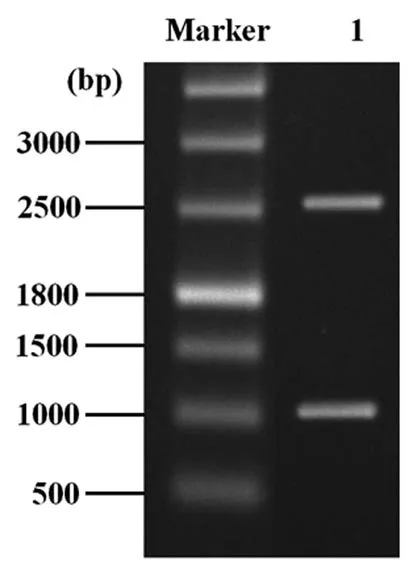
图2 酶切验证
3 讨论
凝胶回收中向吸附柱加入液体应垂直且正中加入,否则会对实验结果产生影响,特别是加入洗脱液时会决定实验的成败,同时避免桶破吸附柱。
[1]田新朋,张偲,李文均.海洋放线菌研究进展[J].微生物学报,2011(2):161-169.
[2]KinSLam.Discovery of Novelmetabolites from Marine Acctinomycete[J].Curr Opin Microbiol,2006(9):245-251.
[3]马艳玲.海洋放线菌基因组文库的构建和功能基因的克隆及序列分析[D].武汉:华中师范大学,2011.
[4]范东东,孙铭娟,王梁华,焦炳华.酰基转移酶研究[J].生命的化学 ,2008(6):701-703.
[5]马艳玲,邓海魏,箐箐,等.稀有海洋放线菌Salinispora arenicola非核糖体肽合成酶和卤代酶生物合成基因簇核心区的克隆及序列分析[J].生物技术通报 ,2011(2):157-162.
[6]JW.-H.Li, JohnC.Vederas.Drug Discovery and NaturalProd-ucts:End of an Era or an Endless Frontier[J].Science,2009(10):161-165.


