氯代烯烃胁迫下菌群SWA1的降解活性及群落结构
赵天涛 ,邢志林 ,张丽杰 ,项锦欣 ,何 芝 ,杨 旭 ,高艳辉 (.重庆理工大学化学化工学院,重庆400054;2.重庆大学城市建设与环境工程学院,重庆 400044)
氯代烯烃在生产生活中的不当存储和使用,使其在空气、水和土壤环境中广泛存在,由于它们具有扩散范围广、降解持续时间长和生物毒性强等特点,对自然环境造成了严重污染.生物降解具有反应条件温和、成本低和无二次污染等优势,长期以来开展了大量研究.目前研究者已经分离得到了可降解氯代烯烃的菌株,对纯菌株降解氯代烯烃的途径有了深入认识,如二氯乙烯(DCE)可以作为微生物唯一碳源实现去除[1];DCE和 TCE可通过共代谢途径发生降解[2-5];四氯乙烯(PCE)可在脱卤酶的作用下转化为低氯代化合物[6].
尽管分离的纯菌株在实验室条件下能够实现污染物降解,但在实际应用中往往面临环境适应性差和降解活性持续时间短的问题[7],如Dehalococcoides属微生物能够实现PCE的完全脱氯,但在高浓度污染物的复杂环境下极易失去活性[8].此外,环境中氯代烯烃污染物的完全降解往往是多种微生物共同的作用结果[9-10],已有研究证实多种微生物混合比单一培养微生物降解氯苯氧基苯酚,聚乙二醇和氯酚化合物等污染物效果更高[11].更重要的是,在现有技术条件下,可分离培养的微生物只占总数的 0.1%~1.0%,未分离培养的微生物在复杂环境中对污染物的作用可能更为重要[12].因此,筛选高活性混合菌在污染物降解中具有更大的应用潜力.
近年来,许多研究者基于水环境沉积物驯化富集的混合微生物分析了氯代烯烃耐受性及降解情况.大多以TCE作为氯代烯烃中的典型污染物,其耐受浓度变化范围为 9~146mg/L[4,13-15],结果表明高浓度TCE对微生物生长及活性有显著影响,当达到 146mg/L,已无甲烷氧化发生[13].实际污染场地中生物降解作用使污染物发生转化,降解不完全造成有毒副产物的显著积累,如 PCE的不完全脱氯会造成TCE和DCE的大量积累,所以系统考察微生物对多种不同类型氯代烯烃的耐受性是必要的.以混合菌为生物介质对氯代烯烃耐受性研究中,只发现Choi等[13]考察了混合菌群对TCE和PCE的耐受性,PCE浓度范围为0.199~0.796mg/L.当前氯代烯烃的生物降解研究中,浓度多维持在小于 10mg/L的范围[16-17],混合菌的低耐受性限制了其在高污染场地的应用,关于高耐受,可高效降解氯代烯烃混合菌的研究鲜有报道.
填埋场覆盖层长期受高浓度、成分复杂污染物驯化,衍生了多种环境适应能力强的功能微生物,在氯代烃污染物生物降解领域具有很大应用潜力[18-19].基于 TCE驯化后的填埋场覆盖层,课题组富集筛选了混合菌群 SWA1,前期研究表明菌群SWA1具有高的群落结构多样性,甲烷为底物可共代谢高效降解 TCE[20].在此基础上,本研究将利用菌群SWA1考察其对3种典型氯代烯烃的耐受性:通过监测高浓度氯代烯烃条件下甲烷氧化能力和菌体生长特性分析混合菌的生物活性;检测氯代烃浓度变化及氯离子产生速率评估氯代烃共代谢降解能力;利用高通量测序技术鉴定不同种类氯代烯烃不同浓度驯化后混合菌群的多样性变化,确定高耐受菌种丰度,有效降解氯代烃微生物种类,明确这些微生物的相互作用关系.研究结果以期为高耐氯代烯烃混合菌的筛选和应用提供基础.
1 材料与方法
1.1 混合菌的驯化富集
试验用混合菌分离于重庆长生桥(29°35'N,106°33'E)生活垃圾填埋场覆盖土.该混合菌命名为SWA1,最适生长温度为30℃,最佳pH值为7.0.可以甲烷为唯一碳源生长,生长特性参见前期研究[20].
1.2 氯代烯烃耐受性实验
量取25mL灭菌N-NMS培养基(配制方法参见文献[20])和5mL SWA1接种液于100mL血清瓶中,聚四氟乙烯塞子和铝盖密封,20mL无菌 CH4置换瓶中空气.分别添加不同浓度的二氯乙烯(t-1,2-DCE,阿法埃莎化学有限公司,98%)、三氯乙烯(TCE,天津市科密欧化学试剂厂,色谱纯)和四氯乙烯(PCE,成都市龙科化工试剂厂,分析纯).实验组中t-1,2-DCE浓度设置为150,250,350,450,580mg/L,分别编号为 tDCE150、tDCE250、tDCE350、tDCE450和tDCE580;TCE浓度为50,100,150,200,250mg/L,分别编号为 TCE50、TCE100、TCE150、TCE200和TCE250;PCE浓度为 100,200,300,400,500mg/L,分 别编号 为PCE100、PCE200、PCE300、PCE400和PCE500.以无氯代烃样品作为空白组,所有组分一式 2份.监测初始生物气和氯代烃浓度,各试样置于30℃,170r/min的培养箱中,监测微生物生长曲线,生物气浓度变化和氯代烃降解情况.
1.3 分析检测
甲烷、氧气和二氧化碳采用气相色谱(川仪SC-6000A)测定,色谱条件:不锈钢色谱柱 TDX 8-12-25 2m,进样口温度、柱温以及检测器(TCD)温度分别为 120,90,120℃,氮气为载气,载气流速为 25mL/min,进样量为 0.5mL.氯代烯烃由SC-3000B气相色谱仪 FID检测器检测,GDX-104 2m 色谱柱;氮气为载气,载气流速为35mL/min,尾吹气速为 10mL/min;进样口、柱箱和检测器温度分别为 200,80,200℃;进样量为0.1mL,基流补偿0.00nA.
1.4 DNA提取与高通量测序
分别选取空白组及不同浓度 tDCE(tDCE150、tDCE250、tDCE350、tDCE450 和tDCE580)、TCE(TCE50、TCE100、TCE150 和TCE200)和 PCE(PCE100、PCE200 和 PCE300)驯化后,生长到稳定期的共13个样品的菌液10mL,离心弃去上清液后于-80 ℃保藏用于多样性测序分析.用购自天根生化科技(北京)有限公司的细菌基因组DNA提取试剂盒(TANamp Bacteria DNA Kit)提取样品的基因组 DNA.样本定量、DNA 序列修饰及验证和混合文库后,将其定量稀释至4~5pmol/L后放于Illumina MiSeq测序仪中测序(Illumina MiSeq PE250测序仪,上海美吉生物医药科技有限公司),扩增16S rRNA基因V3~V4区,引物 338F:5´-ACTCCTACGGGA-GGCAGCA-3´,引物 806R:5´-GGACTACHVG-GGTWTCTAAT-3´[20]使用合成测序法,测定长度2×250bp.
过滤原始序列尾部质量值<20的碱基,设置50bp窗口过滤50bp以下的序列.将成对reads拼接成一条序列,最小重叠长度为 10bp,且重叠区允许的最大错配比率为 0.2.最后检测序列上的barcode并区分土样.利用R(vegan)、CANOCO、PAST等程序软件和NCBI、Mothur等数据平台进行数据解析,对基因数据进行操作分类单元(OTUs)聚类分析和分类学分析.
1.5 数据分析
相关性利用SPSS Statistics 21软件分析,多样性数据采用 i-sanger平台(http://www.isanger.com/)分析.
2 结果与讨论
2.1 氯代烯烃胁迫下混合菌群的生物特性
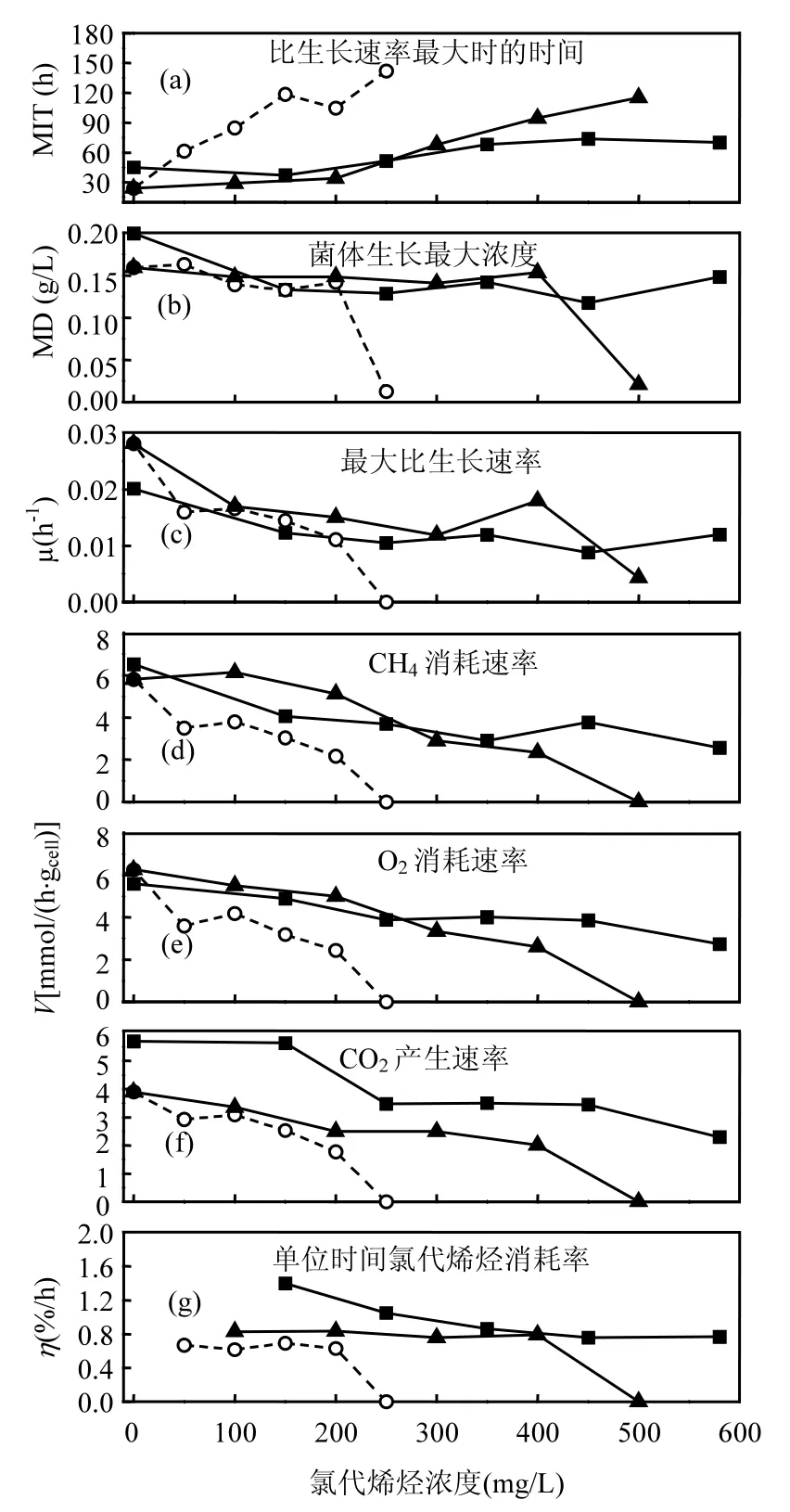
图1 氯代烯烃胁迫下菌体甲烷氧化活性,生长特性和氯代烃降解情况Fig.1 The methane oxidation activity, growth characteristic and chloroalkene biodegradation of SWA1under chloroalkenes acclimatization
不同浓度氯代烯烃胁迫下混合菌群 SWA1的生长特性,甲烷氧化能力及氯代烃降解情况如图1所示.主要通过监测比生长速率最大时的时间(MIT)、最高菌体浓度(MD)和最大比生长速率(μ)来考察SWA1的特性变化.随着t-1,2-DCE浓度的增大(0~580mg/L),MIT缓慢增大,变化范围为 37~73h;无 t-1,2-DCE 时,MD 和 μ 最大,为0.20g/L和 0.02h-1;当添加 t-1,2-DCE浓度为150mg/L时,MD和μ减小,但随着t-1,2-DCE增大,MD 和 μ均无显著变化,变化分别为 0.12~0.15g/L 和 0.009~0.012h-1.表明在该 t-1,2-DCE浓度范围对SWA1的调整期具有一定的影响,随着菌体耐受性的提高,能迅速生长.添加TCE(0~250mg/L)后,SWA1的 MIT 迅速增大,变化范围为 24~142h,增长近 6 倍;浓度为 0~200mg/L时,SWA1的 MD无显著变化,为 0.13~0.16g/L,μ显著减小,为0.028~0.01h-1;当TCE浓度增加到250mg/L时,SWA1几乎无生长,此时μ为0,表明最高耐受浓度约 250mg/L.添加不同浓度的 PCE(0~500mg/L)时,SWA1的 MIT显著增大,变化为 24~115h;当 PCE 浓度为 0~400mg/L时,MD和μ无显著性差异,当增加到500mg/L时几乎无生长,此时μ为0,表明对PCE最高耐受浓度为500mg/L(图1a,b和c).
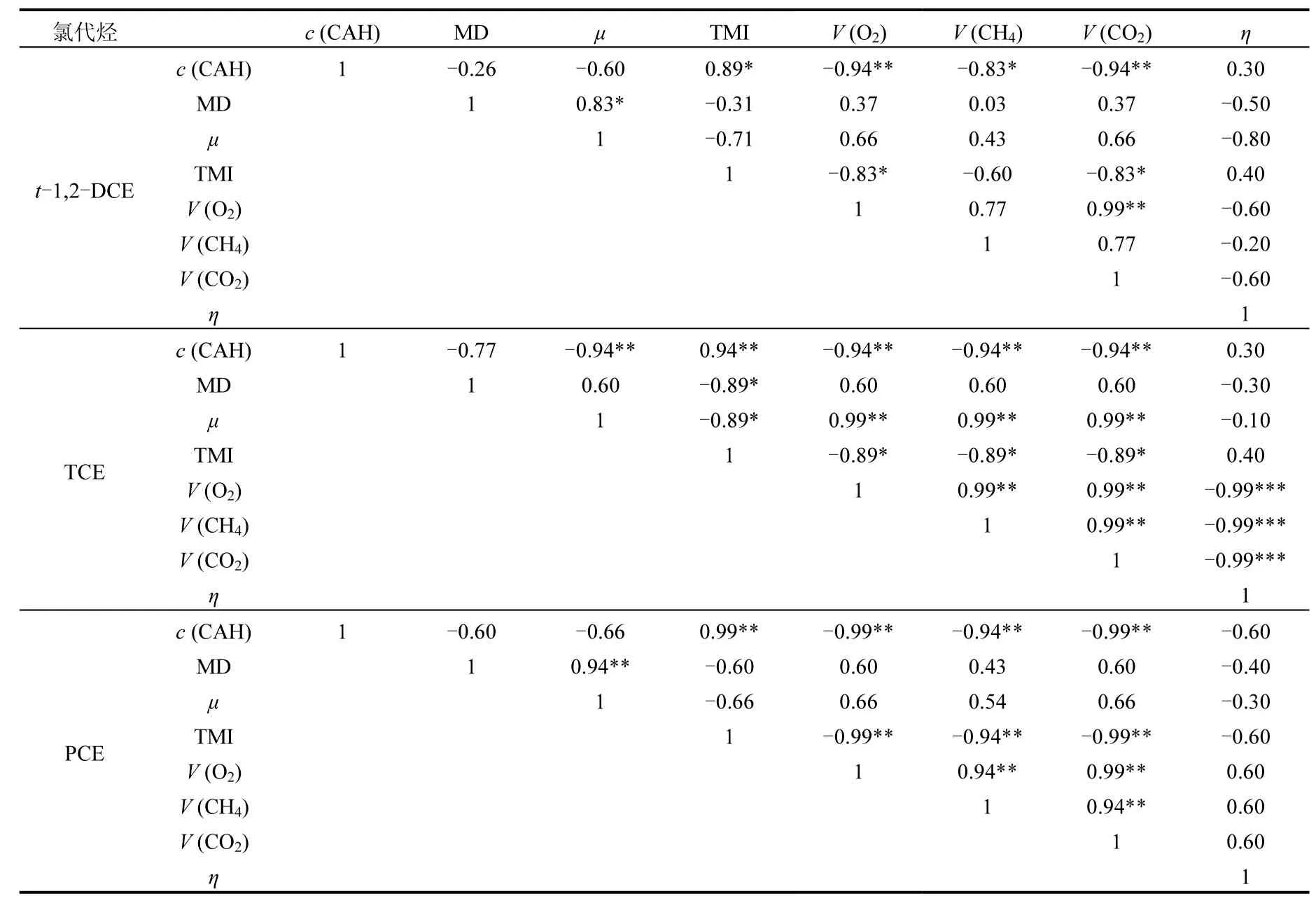
表1 氯代烯烃胁迫下甲烷氧化参数、菌群生长参数及氯代烃降解参数相关性Table 1 Correlation among the methane oxidation parameters, growth parameters and chloroalkene biodegradation parameters
SWA1生长过程中监测了甲烷消耗情况,甲烷、氧化消耗速率和二氧化碳的生成速率具有相同的变化趋势,均随氯代烃浓度的增大减小.t-1,2-DCE浓度为0~580mg/L时,甲烷氧化速率由 6.52mmol/(hgcell)减小为 2.56mmol/(hgcell);PCE浓度为0~400mg/L,甲烷氧化速率缓慢减小,当增加至 500mg/L时,无甲烷氧化;随着 TCE浓度的增加甲烷氧化速率迅速减小,当达到250mg/L时,无甲烷氧化.根据不同氯代烃时SWA1的生长特性和甲烷氧化特性变化可知对3种氯代烯烃的耐受性顺序为 t-1,2-DCE>PCE>TCE.现有研究最多的为微生物对 TCE耐受性,不同环境下所筛选微生物对TCE有不同耐受程度.Broholm等[15]研究了TCE对湖水沉积物分离的混合甲烷氧化菌的活性影响,发现当浓度为13mg/L时,已无甲烷氧化发生,而Choi等[13]研究发现湿地土壤中富集的甲烷氧化菌混合菌对TCE的最高耐受浓度为 146mg/L.对比结果表明SWA1具有更高的TCE耐受能力(图1d,e和 f).混合菌群SWA1生长到稳定期,对3种氯代烯烃的去除率均大于 90%,SWA1在污染物降解中具有高的应用潜力.单位时间(h-1)内对 t-1,2-DCE具有更高的降解效率,并随着浓度的增大而缓慢减小;当菌体活性未完全抑制时,TCE和 PCE的去除效率变化很小,菌体完全抑制后无氯代烃降解(图1g),表明混合菌群具有更强的环境适应能力,已有研究发现混合菌群中微生物间可形成如形态变化,修改细胞属性等多种对有机溶剂的自适应机制[21],同时最新研究还发现混合菌对底物具有交替利用的现象[22],这对于混合菌产生高耐受性具有重要意义.
氯代烯烃驯化过程中各参数间相互关系如表1所示.各氯代烯烃浓度c(CAH)与甲烷氧化速率 V(CH4)(r=-0.943~-0.829,P<0.05),氧气消耗速率 V(O2)(r=-0.999~-0.943,P<0.01)和二氧化碳产生速率 V(CO2)(r=-0.999~-0.943,P<0.01)呈显著负相关,与 MIT呈显著正相关(r=0.886~0.999,P<0.05),表明高浓度氯代烯烃均抑制菌体生长和生物氧化活性.V(CH4)和V(CO2)相关性结果显著,表明二氧化碳主要来源于甲烷的生物氧化.TCE和 t-1,2-DCE共代谢降解过程中,V(CH4)与氯代烃去除效率 η呈负相关,表明甲烷和氯代烃间存在竞争性抑制关系[4].而PCE胁迫条件下,V(CH4)与PCE去除效率为正相关关系,表明SWA1对氯代烯烃存在不同的降解机制.已有研究表明低氯取代烃更易发生共代谢生物降解,全氯代烃难以发生共代谢降解[18],关于PCE的去除机理还需进一步深入探究.
2.2 不同氯代烯烃胁迫下群落结构丰富度、多样性及聚类分析
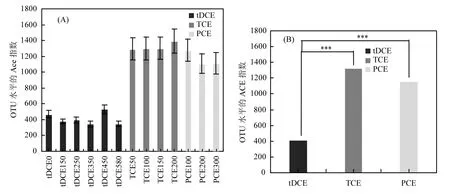
图2 Ace指数(A)及不同氯代烯烃胁迫下微生物群落结构种间差异(B)Fig.2 Ace index (A) and difference of community structure (B) among samples under chloroalkenes acclimatization***:差异性极显著(P<0.001)
选取不同氯代烯烃、不同浓度驯化后混合菌样品进行高通量测序,14个样品共获得 460598条16S rRNA基因片段序列.在97%的对比度下,共有1540个OTUs,覆盖度高于99.9%,表明抽样完全.不同氯代烯烃胁迫条件下样品多样性指数(Ace指数)及微生物群落结构种间差异如图2所示. t-1,2-DCE驯化后OUT水平的Ace指数无显著性变化,表明未显著改变微生物多样性.TCE和PCE胁迫下,Ace指数显著增大,且随TCE浓度的增大而增大,表明 TCE和 PCE显著增大 SWA1的微生物多样性.群落结构种间差异结果(图2B)显示TCE或PCE驯化后样品的微生物多样性与t-1,2-DCE驯化后微生物群落结构具有显著性差异(P<0.01),而TCE驯化和PCE驯化后的微生物群落结构无显著性差异,表明SWA1对不同结构和性质的污染物有不同响应.
不同氯代烃耐受条件下样品在各分类水平下的层级聚类和PCA分析如图3所示.不同分类水平下的层级聚类图和PCA图差别较大.门水平层级聚类和 PCA 分析结果(图3A和 a)显示SWA1在门水平的群落组成不受氯代烃种类和浓度的影响,表明SWA1门水平微生物结构稳定.纲水平层级聚类和PCA(图3B和b)结果表明该水平微生物群落结构在同一氯代烃驯化后具有相似性;属水平层级聚类和PCA(图3C和c)结果显示,同一氯代烃驯化后,微生物群落结构和多样性相似度最大,表明不同氯代烯烃对SWA1属组成和相对丰度有显著影响.TCE和PCE耐受性驯化后 SWA1的微生物群落结构相似度大,表明TCE和PCE对微生物的影响机理更相似.
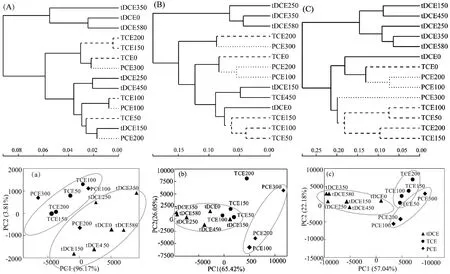
图3 氯代烯烃胁迫下不同分类水平下的层级聚类和PCA分析Fig. 3 Hierarchical clustering and PCA analysis of different level of classification under different chloroalkenes acclimatization
2.3 组间微生物群落结构组成及菌属差异
高通量测序技术分析不同氯代烯烃胁迫下SWA1群落结构组成.所有样品中变形菌门Proteobacteria和拟杆菌门bacteroidetes为主导菌门,相对丰度大于98%,而变形菌门Proteobacteria相对丰度大于85%.不同浓度不同氯代烯烃驯化样品中变形菌门 Proteobacteria和拟杆菌门 bacteroidetes相对丰度无显著性差异(P=0.45~0.52),表明 SWA1中大多数功能微生物均属于这两门.已有对覆盖土微生物群落结构的研究表明对覆盖土的多样性测序表明变形菌门 Proteobacteria、厚壁菌门 Firmicutes和拟杆菌门Bacteroidetes为主导菌门[23].
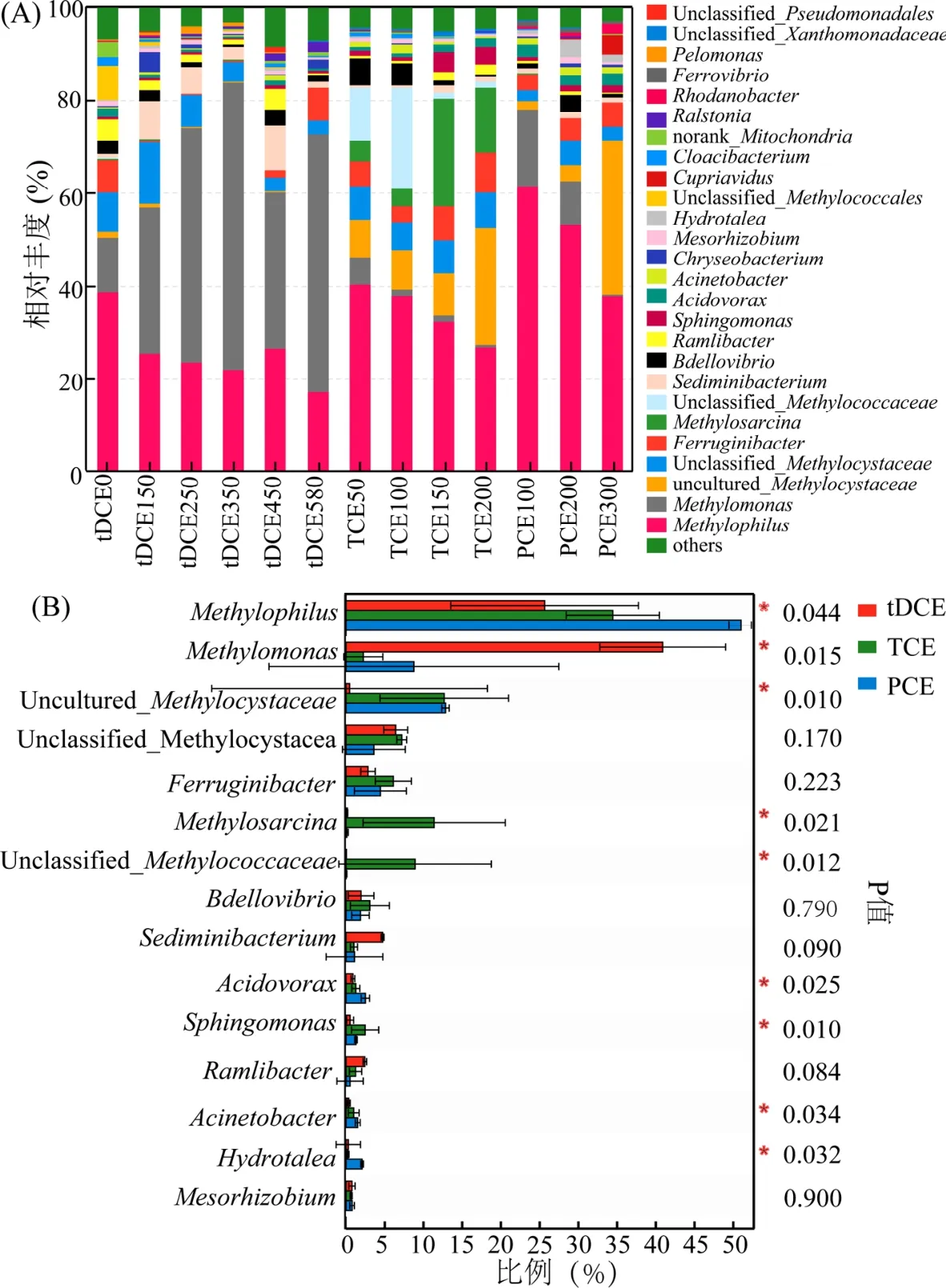
图4 不同氯代烃驯化后微生物群落结构(A)和多物种差异检验(B)Fig.4 Column charts of Microbial community structure (A) and diversity test of different species after chloroalkenes acclimatization (B)
不同氯代烯烃胁迫下 SWA1属水平群落组成及优势菌属差异性如图4所示.t-1,2-DCE胁迫下 SWA1中优势菌属为甲基单胞菌属Methylomonas(31.7%~62.2%)、 嗜 甲 基 菌 属Methylophilus(17.4%~26.6%)、甲基孢囊菌属(未分类)Methylocystaceae_Unclassified(2.9%~13.3%)和 Sediminibacterium(1.4%~9.5%).TCE胁迫下SWA1优势菌属为嗜甲基菌属 Methylophilus(26.9%~46.3%)、甲基孢囊菌属(未培养)Methylocystaceae_uncultured(8.1%~25.1%)、甲基八叠球菌属 Methylosarcina(3.8%~23.2%)和甲基孢囊菌属(未分类)Methylocystaceae_Unclassified(1.3%~21.8%).PCE胁迫下优势菌属为嗜甲基菌属 Methylophilus(37.9%~61.7%)、甲基孢囊菌属(未培养)Methylocystaceae_uncultured(1.7%~33.4%),甲 基 单 胞 菌 属Methylomonas(0.4%~16.6%)和 Ferruginibacter(3.4%~5.3%)(图4A).表明不同氯代烯烃胁迫下,SWA1中微生物群落结构多样性和相对丰度发生显著变化.Choi等[13]研究TCE和PCE对湿地土壤分离的混合甲烷氧化菌的影响,分析了其中甲烷氧化菌的群落结构组成及变化,发现甲基孢囊菌属 Methylocystis为优势菌属,对 TCE和PCE具有高耐受性.Shukla等[24]研究了由水稻田分离的混合甲烷氧化菌对TCE的降解,证明混合菌种Methylocystis对TCE降解起重要作用.而本研究中利用填埋场分离的混合菌在TCE和PCE胁迫下,Methylophilus在混合菌群相对丰度最大,对TCE和PCE具有高的耐受性,表明不同污染场地的微生物结构和功能具有很大差异.
不同氯代烯烃胁迫下,物种差异性分析结果如图4B所示.Methylophilus(P=0.044)、Methylomonas(P=0.015)、Methylocystaceae_Uncultured (P=0.010)、Methylosarcina(P=0.021)、Methylococcaceae_Unclassified (P=0.012)、嗜酸菌属 Acidovorax(P=0.025)、鞘氨醇单胞菌 Sphingomonas(P=0.010)、不动杆菌属 Acinetobacter(P=0.034)和 Hydrotalea(P=0.032)差异性显著,t-1,2-DCE胁迫下 Methylomonas的相对丰度最大,表明Methylomonas对 t-1,2-DCE有高的耐受性,在t-1,2-DCE的生物降解中起重要作用,已有关于Methylomonas methanica NCIB1113降解 t-1,2-DCE的报道[25].
2.4 混合菌群微生物在生物降解中的可能作用分析
氯代烃胁迫条件下生物降解参数与微生物群落结构相关性如表2所示.随t-1,2-DCE浓度的增大,金黄杆菌属 Chryseobacterium、Sediminibacterium和Methylomonas的相对丰度增加,该类菌株可耐受高浓度 t-1,2-DCE.甲烷氧化菌菌属Methylocystaceae_Unclassified、甲基球目菌属(未分类)Methylococcales_Unclassified、Methylophilus和非甲烷氧化菌菌属Ramlibacter、Acidovorax与 V(CH4)呈显著正相关(r=0.89~0.99,P<0.05),表明在t-1,2-DCE胁迫条件下这些菌属是SWA1中对甲烷氧化起重要作用.在混合菌属中,非甲烷氧化菌Ramlibacter和Acidovorax一方面可能通过与甲烷氧化菌的协同作用关系促进甲烷氧化,另一方面可能直接参与甲烷代谢,有研究显示 Acidovorax在甲烷产生环境中具有高的相对丰度[26-27],关于这些非甲烷氧化菌在甲烷消耗中的作用还需进一步验证.SWA1中只有Methylomonas(r=0.486)和Sediminibacterium (r=0.657)相对丰度与t-1,2-DCE降解效率η呈正相关,表明它们对t-1,2-DCE的降解起主要作用,已有研究证明含有Methylomonas的混合菌或分离的纯菌株 Methylomonas都能通过共代谢降解t-1,2-DCE等氯代烃[28-29].未见 Sediminibacterium降解氯代烃的报道,有研究表明Sediminibacterium在混合菌中可产生葡萄糖等化合物[30],因此Sediminibacterium可通过为其他微生物提供生长底物的协同作用方式促进氯代烃降解.纯种菌株共代谢降解氯代烃研究中发现氯代烃去除和甲烷氧化间存在不可避免的竞争性抑制[31-32],相关性结果显示SWA1中与甲烷氧化和 t-1,2-DCE去除相关菌属不尽相同(表4),这在一定程度上可缓解竞争性抑制的发生,更有利于氯代烃的去除.
TCE胁迫条件下,菌属 Methylocystaceae_Unclassified、Methylocystaceae_uncultured、Ramlibacter、Methylosarcina、Sphingomonas、Sediminibacterium和Acidovorax与TCE浓度呈显著正相关(r=0.50~0.90),表明这些菌属耐高浓度TCE.SWA1中甲烷氧化菌菌属Methylophilus、Methylomonas及非甲烷氧化菌菌属Cloacibacterium 对甲烷氧化起主要作用,有研究显示 Cloacibacterium为水环境沉积物和市政废水中的主要微生物[33-34],表明其在有机物降解中起重要作用.高耐受TCE微生物均与η呈正相关关 系,菌 属 Methylocystaceae_uncultured、Ramlibacter、Methylosarcina、Sediminibacterium和Acidovorax对TCE的去除起主要作用,甲烷氧化菌在TCE降解研究中已有许多报道[5],也有研究证明Acidovorax对地下水中TCE去除起重要作用[35],可知SWA1对于氯代烃的去除并非只有甲烷为底物的共代谢降解过程.同样,该条件下,甲烷氧化微生物和氯代烃降解微生物主要菌属不尽相同,生物降解过程中混合菌属SWA1中各菌株既有协同作用关系又相互独立.
PCE胁迫下,根据微生物群落结构变化,可知甲烷氧化相关微生物为 Methylophilus.Methylocystaceae_uncultured在高浓度PCE环境中具有高的相对丰度,表明其对PCE具有高的耐受性.在优势菌属中 Acidovorax有报道可进行PCE的生物降解.不同氯代烯烃胁迫条件下,SWA1群落结构具有显著性差异,甲烷氧化微生物和氯代烃降解微生物均不尽相同,说明群落结构改变是SWA1功能变化的重要原因.
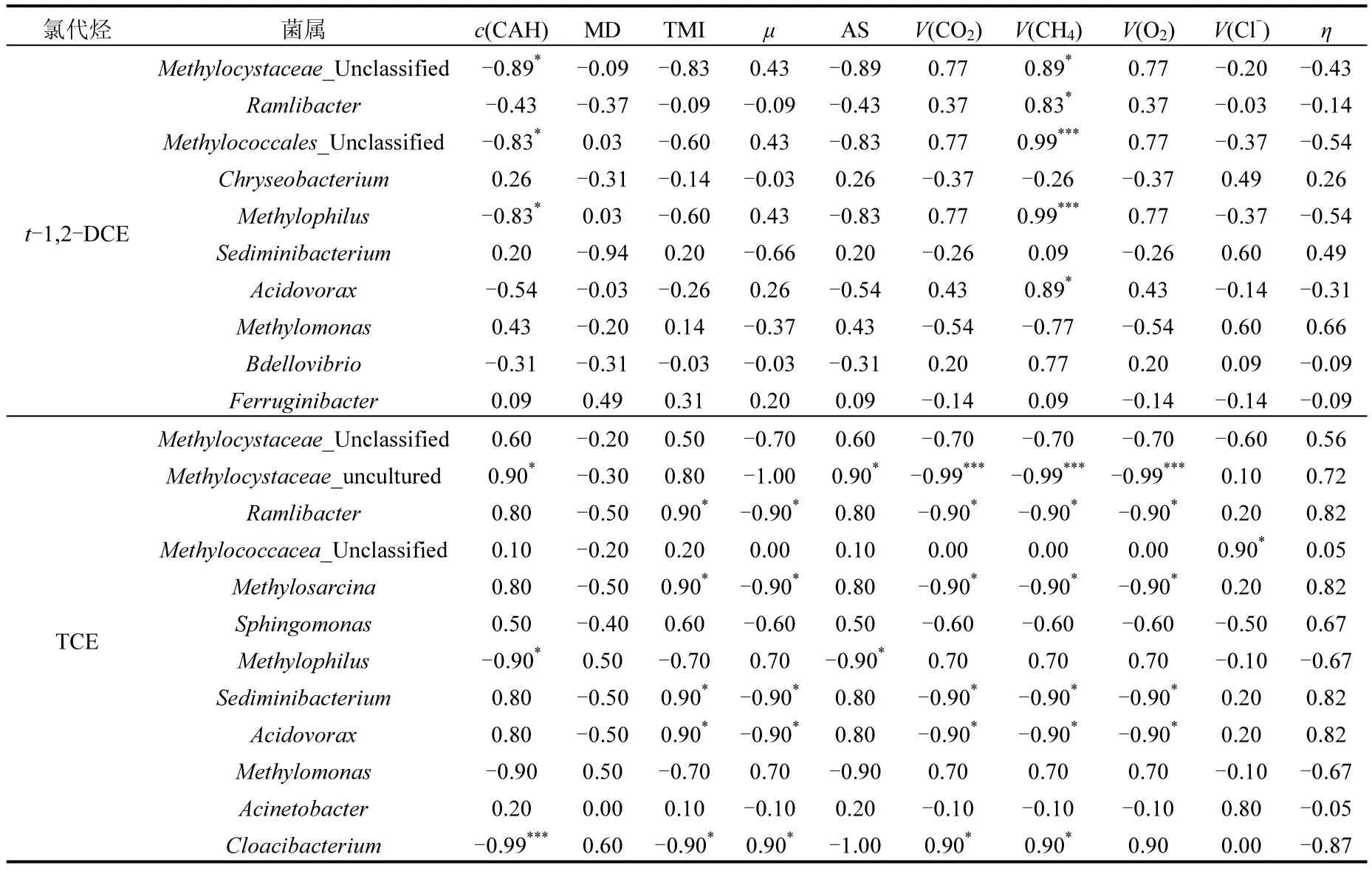
表2 氯代烃胁迫下降解参数与微生物相关性Table 2 Correlation between relative abundances of the microbial diversity at dominant bacterial taxa and SWA1characteristics
2.5 氯代烯烃降解过程微生物的可能相互作用分析
自然环境中,污染物的有效去除依赖于混合菌中多种微生物的协同作用关系[9-10].基于Spearman相关性系数,建立种群中不同菌属间的相互作用关系网,获得物种在环境样本中的共存关系,t-1,2-DCE和TCE胁迫下SWA1中优势菌属相互作用关系网如图5所示.SWA1中绝大多数优势菌属间为正相关,表明在生物降解过程中,协同作用是各菌属间的主要关系.不同甲烷氧化菌间,不同非甲烷氧化菌间及甲烷氧化菌和非甲烷氧化菌间均存在相互作用关系,各菌属间既有直接作用又有间接相互作用.t-1,2-DCE胁迫条件 下 Methylophilus、 Methylococcales_Unclassified、Methylocystaceae_Unclassified和Cloacibacterium各菌属间均存在正相关关系.TCE胁迫条件下,Ramlibacter黄单胞菌(未分类)Xanthomonadaceae_unclassified、Methylosarcina、Acidovorax和Sediminibacterium菌属间存在正相关关系.根据甲烷氧化菌代谢特性,二氧化碳产生速率为甲烷氧化速率的一半[36],该反应体系中,唯一生长底物为甲烷,共代谢底物为氯代烃,监测过程发现二氧化碳增加速率与甲烷消耗速率几乎相等,而所添加氯代烃中所含的当量二氧化碳远小于甲烷中的当量二氧化碳,这表明近一半的二氧化碳来源于非甲烷氧化菌的新陈代谢,生长底物来源于甲烷氧化菌代谢的有机物.已有相关研究表明甲烷氧化菌与非甲烷氧化菌相互作用关系可以为混合菌提供稳定的生长环境和合适的基质组成[37-38].
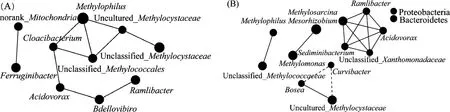
图5 不同氯代烯烃胁迫下微生物间的相互作用关系网Fig.5 The interaction networks of the microorganism in SWA1under different chloroalkenes acclimatization
3 结论
3.1 混合菌 SWA1对氯代烯烃有高的耐受性,对t-1,2-DCE的最高耐受浓度高于580mg/L;对TCE最高耐受浓度为250mg/L;对PCE最高耐受浓度为500mg/L.好氧共代谢条件下SWA1能有效降解氯代烃,对t-1,2-DCE去除率高于90%.3.2 不同氯代烯烃胁迫条件下,混合菌SWA1微生物群落结构种间差异显著,t-1,2-DCE驯化后优势菌属为 Methylomonas(31.7%~62.2%),TCE和PCE驯化后优势菌属为Methylophilus(26.9%~46.3%,37.9%~61.7%).
3.3 氯代烯烃共代谢降解过程中,直接参与甲烷氧化和氯代烃降解微生物差异较大,t-1,2-DCE 共代谢生物降解中 Methylophilus、Ramlibacter和 Acidovora促进甲烷氧化,Methylomonas参与t-1,2-DCE降解.TCE共代谢降解中参与甲烷氧化和氯代烃降解的优势菌属分别为 Methylophilus和 Cloacibacterium.Methylocystaceae_uncultured对TCE降解起重要作用.
3.4 微生物spearman相关性分析显示SWA1中t-1,2-DCE 胁 迫 条 件 下 Methylophilus、Methylococcales_Unclassified、Methylocystaceae_Unclassified和Cloacibacterium各菌属间均存在直接正相关关系.TCE胁迫条件下,Ramlibacter、Xanthomonadaceae_unclassified、Methylosarcina、Acidovorax和Sediminibacterium菌属间存在直接正相关关系.菌属间相互协同作用强化SWA1生物活性.
[1] Coleman N V, Mattes T E, Gossett J M, et al. Biodegradation of cis-dichloroethene as the sole carbon source by a betaproteobacterium [J]. Applied & Environmental Microbiology,2002,68(6):2726-2730.
[2] Pant P, Pant S. A review: Advances in microbial remediation of trichloroethylene (TCE) [J]. Journal of Environmental Sciences,2010,22(1):116-126.
[3] Le N B, Coleman N V. Biodegradation of vinyl chloride, cisdichloroethene and 1,2-dichloroethane in the alkene/alkaneoxidising mycobacterium strain NBB4 [J]. Biodegradation, 2011,22(6):1095-1108.
[4] Alvarez-Cohen L, Mccarty P L. Product toxicity and cometabolic competitive inhibition modeling of chloroform and trichloroethylene transformation by methanotrophic resting cells[J]. Applied and Environmental Microbiology, 1991,57(4):1031-1037.
[5] 邢志林,张丽杰,赵天涛.专一营养与兼性甲烷氧化菌降解氯代烃的研究现状、动力学分析及展望 [J]. 生物工程学报, 2014,30(4):531-544.
[6] Kao C M, Liao H Y, Chien C C, et al. The change of microbial community from chlorinated solvent-contaminated groundwater after biostimulation using the metagenome analysis. Journal of Hazardous Materials, 2016,302:144—150.
[7] El Fantroussi S, Agathos S. Is bioaugmentation a feasible strategy for pollutant removal and site remediation? [J]. Current Opinion in Microbiology, 2005,8(3):268-275.
[8] Frascari D, Fraraccio S, Nocentini M, et al. Aerobic/anaerobic/aerobic sequenced biodegradation of a mixture of chlorinated ethenes, ethanes and methanes in batch bioreactors [J].Bioresource Technology, 2012,128(1):479-486.
[9] 杨清香,贾振杰,李慧君,等.高效脱色菌群的脱色、产酶及群落分析 [J]. 中国环境科学, 2007,27(6):763-767.
[10] Shah V, Zakrzewski M, Wibberg D, et al. Taxonomic profiling and metagenome analysis of a microbial community from a habitat contaminated with industrial discharges [J]. Microbial Ecology, 2013,66(3):533-550.
[11] 张蔚文.混合菌培养在生物降解中的意义 [J]. 环境科学与技术, 1993,60(1):16-20.
[12] Witzig R, Junca H, Hecht H, et al. Assessment of toluene/biphenyl dioxygenase gene diversity in benzene-polluted soils:Links between benzene biodegradation and genes similar to those encoding isopropylbenzene dioxygenases [J]. Applied &Environmental Microbiology, 2006,72(5):3504-3514.
[13] Choi S A, Lee E H, Cho K S. Effect of trichloroethylene and tetrachloroethylene on methane oxidation and community structure of methanotrophic consortium [J]. Journal of Environmental Science & Health Part A Toxic/hazardous Substances & Environmental Engineering, 2013,48(13):1723-1731.
[14] Alvarez-Cohen L, Mccarty P L. Effects of toxicity, aeration, and reductant supply on trichloroethylene transformation by a mixed methanotrophic culture [J]. Applied & Environmental Microbiology, 1991,57(1):228-235.
[15] Broholm K, Jensen B K, Christensen T H, et al. Toxicity of 1,1,1-trichloroethane and trichloroethene on a mixed culture of methane-oxidizing bacteria [J]. Applied & Environmental Microbiology, 1990,56(8):2488-2493.
[16] Frascari D, Zanaroli G, Bucchi G, et al. Trichloroethylene aerobic cometabolism by suspended and immobilized butane-growing microbial consortia: A kinetic study [J]. Bioresource Technology,2013,144(6):529.
[17] Kocamemi B A, Ceçen F. Biological removal of the xenobiotic trichloroethylene (TCE) through cometabolism in nitrifying systems [J]. Bioresource Technology, 2010,101(1):430-433.
[18] 何 芝,赵天涛,邢志林,等.典型生活垃圾填埋场覆盖土微生物群落分析 [J]. 中国环境科学, 2015,35(12):3744-3753.
[19] 郭 敏,何品晶,吕 凡,等.垃圾填埋场覆土层Ⅱ型甲烷氧化菌的群落结构 [J]. 中国环境科学, 2008,28(6):536-541.
[20] 高艳辉,赵天涛,邢志林,等.铜离子对混合菌群降解三氯乙烯的影响与机制分析 [J]. 生物工程学报, 2016,32(5):621-634.
[21] Heipieper H J, Neumann G, Cornelissen S, et al. Solvent-tolerant bacteria for biotransformations in two-phase fermentation systems [J]. Applied Microbiology and Biotechnology, 2007,74(5):961-973.
[22] Liu J, Martinez-Corral R, Prindle A, et al. Coupling between distant biofilms and emergence of nutrient time-sharing [J].Science, 2017,356(6338):638-642.
[23] Xing Z, Zhao T, Gao Y, et al. Real-time monitoring of methane oxidation in a simulated landfill cover soil and MiSeq pyrosequencing analysis of the related bacterial community structure [J]. Waste Management, 2017,68:369-377.
[24] Shukla A K, Vishwakarma P, Upadhyay S N, et al. Biodegradation of trichloroethylene (TCE) by methanotrophic community [J].Bioresource Technology, 2009,100(9):2469-2474.
[25] Janssen D B, Grobben G, Hoekstra R, et al. Degradation of trans-1,2-dichloroethene by mixed and pure cultures of methanotrophic bacteria [J]. Applied Microbiology and Biotechnology, 1988,29(4):392-399.
[26] Van EertenJansen M C, Veldhoen A B, Plugge C M, et al.Microbial community analysis of a methane-producing biocathode in a bioelectrochemical system [J]. Archaea-an International Microbiological Journal, 2013,2013(9):1-12.
[27] Helm J, Wendlandt K D, Rogge G, et al. Characterizing a stable methane-utilizing mixed culture used in the synthesis of a high-quality biopolymer in an open system [J]. Journal of Applied Microbiology, 2006,101(2):387-395.
[28] Moore A T, Vira A, Fogel S. Biodegradation of trans-1,2-dichloroethylene by methane-utilizing bacteria in an aquifer simulator [J]. Environmental Science & Technology, 1989,23(4):403-406.
[29] Koh S C, Bowman J P, Sayler G S. Soluble methane monooxygenase production and trichloroethylene degradation by a type I methanotroph, Methylomonas methanica 68-1 [J].Applied & Environmental Microbiology, 1993,59(4):960-967.
[30] Kim Y J, Kim M K, Bui T P N, et al. Sediminibacterium ginsengisoli sp. Nov., a ß-glucosidase producing bacterium isolated from soil of a ginseng field [J]. International Journal of Systematic & Evolutionary Microbiology, 2009,63(5):173-174.
[31] Lewis S, Gary D H, Paul V R, et al. A field evaluation of in-situ biodegradation of chlorinated ethenes: Part 3, studies of competitive inhibition [J]. Ground Water, 1991,29(2):239-250.
[32] Charng M K, Voice T C, Criddle C S. Kinetics of competitive inhibition and cometabolism in the biodegradation of benzene,toluene, and p-xylene by two pseudomonas isolates [J].Biotechnology & Bioengineering, 1993,41(11):1057-1065.
[33] Allen T D, Lawson P A, Collins M D, et al. Cloacibacterium normanense gen. Nov., sp. Nov., a novel bacterium in the family flavobacteriaceae isolated from municipal wastewater [J].International Journal of Systematic & Evolutionary Microbiology,2006,56(6):1311-1316.
[34] Cao S J, Deng C P, Li B Z, et al. Cloacibacterium rupense sp.Nov., isolated from freshwater lake sediment [J]. International Journal of Systematic & Evolutionary Microbiology, 2010,60(9):2023-2026.
[35] Li K. Microbial bioremediation and monitoring of a TCE-contaminated site [J]. Journal of Applied Behavior Analysis,2011,36(2):187-204.
[36] 邢志林,赵天涛,陈新安,等.覆盖层氧气消耗通量模型及甲烷氧化能力预测 [J]. 化工学报, 2015,66(3):1117-1125.
[37] Petersen J M, Dubilier N. Methanotrophic symbioses in marine invertebrates [J]. Environmental Microbiology Reports, 2009,1(1):319-335.
[38] Stock M, Hoefman S, Kerckhof F M, et al. Exploration and prediction of interactions between methanotrophs and heterotrophs [J]. Research in Microbiology, 2013,164(10):1045-1054.

