双氯芬酸钠对苏云金芽孢杆菌毒性的分子机制
吴 双,秦华明,叶锦韶 (暨南大学环境学院,广东省环境污染控制与修复材料工程技术研究中心,广州市环境暴露与健康重点实验室,广州 广东 510632)
非甾体抗炎药(NSAIDs)在全世界得到广泛的应用,但也是一类日益受到人们关注的新兴污染物,由其引起的环境污染问题受到越来越多的关注[1].其中双氯芬酸(DCF)通常被公认为“世界上最受欢迎的止痛药”,是最常用的NSAIDs,主要以钠盐的形式存在,全球消费量已达上千吨[2-3].DCF是一种持久性有毒污染物[4],具有很强的环境污染性,欧盟早在2000年就将DCF列为环境中优先控制的污染物[5],它们可以通过多种途径进入环境,对水生生物造成严重的毒害作用[6],并且通过食物链的富集会对其他生物和人体健康造成重大威胁,有研究表明,DCF对生物体产生毒性[7-10],诱导小鼠肾细胞凋亡[11],上调促炎细胞因子,并诱导肾组织中 NF-κB 的活化[12].有研究结果表明由于线粒体功能障碍导致的ATP合成损伤可能是 DCF引起肝细胞毒性的关键原因[13-15].由于双氯芬酸钠的用量大而降解效率低[16-17],其残留物进入环境后对环境生态和人体健康存在风险,但目前对双氯芬酸钠毒理性质并未十分明确, 尚无将 iTRAQ技术应用于双氯芬酸的抑菌机理的研究报道,尚需进行更深入的研究.为此,本文选用模式微生物菌种革兰氏阳性菌苏云金芽胞杆菌为研究对象,基于iTRAQ的定量蛋白质组学技术[18],检测鉴定了苏云金芽胞杆菌在双氯芬酸钠胁迫下差异表达的蛋白质,并对其进行了生物信息学分析,以期揭示双氯芬酸钠对苏云金芽孢杆菌细胞毒性的分子机制.本研究对于深入评价双氯芬酸钠对生态安全及人类健康的影响具有参考意义.
1 材料和方法
1.1 菌种和培养基
B.thuringiensisGIMCC1.817为本实验室所有,储存在中国广东省微生物所.双氯芬酸钠购自Sigma Aldrich(St.Louis, MO, USA).牛肉膏蛋白胨培养基:牛肉膏3g/L、蛋白胨10g/L、NaCl 5g/L.无 机 盐 体 系 (MSM):K2HPO4·12H2O 4g/L 、KH2PO42g/L、(NH4)2SO420g/L、KCl 2g/L、MgSO41g/L.
1.2 菌体培养
将保存在斜面上的苏云金芽孢杆菌接种到100mL牛肉膏蛋白胨液体培养基中,置于 30 ℃、100r/min的摇床中震荡培养24h.随后,3500r/min离心10min,然后用磷酸盐缓冲溶液洗涤细胞3次.
1.3 生长抑制曲线的测定
将 1μmol/μL的双氯芬酸钠溶液 20μL和浓度为1g/L的苏云金芽孢杆菌加入装有20mL无机盐培养体系的 50mL锥形瓶中,置于 30℃、100r/min的摇床中震荡培养.以不使用双氯芬酸钠溶液的培养体系(其他条件不变)作为对照组.分别在培养 4,8,12,24,36,48,60,72,80h时用酶标仪于波长600nm处测定OD值并绘制生长曲线.
1.4 蛋白质提取
同生长抑制曲线测定培养条件,在双氯芬酸钠处理后,24h后3500r/min离心10min并用磷酸盐缓冲溶液洗涤菌体 3次进行全蛋白提取:将菌体悬浮在 1mL裂解液中,裂解液配方为15mmol/L Tris-HCl pH7.4、7mol/L尿素、2mol/L硫脲、4%CHAPS,使用前加 2mmol/L IPG缓冲液,1mmol/LDTT和1mmol/L PMSF混合物,冰上放置,每隔2min涡旋10s.样品在液氮中冻融3次,每次 15min,然后超声破碎 20min,超声破碎后的样品加1μL核酸酶冰上放置30min.4℃, 12000r/min离心 30min,取上清为总蛋白,使用 Bradford方法测量蛋白质的浓度[19].
1.5 蛋白质还原和酶解
用10mol/L DTT或2μL还原剂将每个样品的蛋白质在37℃下还原1h.用1μL半胱氨酸锁定剂将半胱氨酸在室温下封闭 10min.将蛋白质样品加入 10KD(Amicon Ultra-0.5)超滤离心管中,用 100μL溶解缓冲液洗涤,12000r/min离心20min,重复 3次.除去收集管中的液体后,37℃下用 50μL 胰蛋白酶(Promega,V5280,USA)将过滤管中的样品酶解14h过夜.随后,12000r/min离心20min,将 1μg胰蛋白酶加入到过滤管中二次酶解 2h.离心后,收集管中的液体,使用 Bradford方法测定肽段的浓度[20].
1.6 iTRAQ标记和脱盐
根据制造商的说明书,用 iTRAQ reagent多重试剂盒(Sigma,PN 4352135,USA)标记肽段.将150μL的乙醇加入到116和117iTRAQ试剂中,然后涡旋并用迷你离心机将溶液离心至管底.将肽段转移到新管后,用116的iTRAQ试剂标记对照组肽段,117的iTRAQ试剂标记处理组肽段,2h后向每个样品中加入100μL的水以终止反应.将1μL 溶液通过 ABI 4800MALDI TOF/TOF(Applied Biosystems,Foster City,CA)以检测标记效率.随后,将标记的样品与 Strata-X(Phenomenex, USA)混合,涡旋,脱盐,并通过强阳离子交换层析(SCX)分离,标记的肽段在真空浓缩仪中冷冻干燥.
1.7 蛋白质的LC-ESI-MS/MS分析
用溶液(2%乙腈,0.1%甲酸)复溶样品,12000r/min离心 20min,使用 NanosprayIII源(AB SCIEX)装有 AB SciexTripleTOF 5600质谱仪(AB SCIEX,Framingham,MA,USA)检测,质谱检测方式为MS/MS扫描.将鉴定的蛋白质在 NCBI(http://www.ncbi.nlm)进行内部 BLAST检索匹配.使用 ProteinPilot TM Software 4.5(AB SCIEX)进行Uniprot Database中的蛋白质鉴定和相对iTRAQ定量.对于iTRAQ定量,通过Pro GroupTM算法自动选择肽段,计算报告峰面积,误差因子和 P值.采用反向数据库搜索来估计肽段蛋白质阳性结果错误率 FDR.使用不低于 95%的肽段置信水平,变化倍数 1.2倍以上作为鉴定标准,认为二者之间差异具有统计学意义.
1.8 生物信息学分析
将所得到的蛋白质通过蛋白质资源数据库(UniProt)(http://www.uniprot.org/)得到基因列表,利 用 PANTHER 分 类 数 据 库(http://www.pantherdb)对变化倍数 1.2倍以上的差异表达蛋白质进行 GO分析(用于注释鉴定的蛋白质的生物过程,分子功能和细胞组分信息).通过STRING(http://www.string-db.org/)构 建 差 异 表达蛋白质的相互作用网络,使用 KEGG数据库(http://www.genome.jp/kegg/pathway)对鉴定到的差异蛋白质进行匹配,挖掘由差异蛋白质表达变化导致发生改变的重要代谢信号通路(P<0.05,即显著富集)[21].
2 结果与讨论
2.1 双氯芬酸钠对苏云金芽孢杆菌生长和繁殖的影响
如图1所示,由生长抑制曲线,处理组的 OD值从4h后持续比对照组OD值低,48h内对照组OD值较稳定,而处理组OD值一直处于下降趋势.可以看出双氯芬酸钠对苏云金芽孢杆菌的生长和繁殖具有明显的抑制作用.

图1 双氯芬酸钠对苏云金芽孢杆菌的生长抑制Fig.1 Inhibition of the growth of Bacillus thuringiensis by diclofenac sodium
2.2 蛋白质的鉴定
使用串联质谱(MS/MS)对苏云金芽孢杆菌总蛋白进行检测,并用Mascot软件共鉴定差异倍数大于 1.2、P值小于 0.05的差异蛋白质有 17种,其中表达上调的蛋白质5种,表达下调的蛋白质12种.这些蛋白质的详细信息列见表1.

表1 双氯芬酸钠胁迫下苏云金芽孢杆菌差异表达蛋白Table 1 Differential expressed proteins in Bacillus thuringiensis with diclofenac sodium treatment

续表1
2.3 蛋白质GO功能注释
通过蛋白质 GO 分析(图2)可以看出,得到的17种蛋白质中对应到生物过程,其中10种蛋白参与代谢过程,占58.82%,其次有5种蛋白参与细胞过程,占29.41%,主要涉及到酰基辅酶A脱氢酶、尿嘧啶磷酸核糖基转移酶、核糖体蛋白、转运ATP结合蛋白.细胞组分类以细胞部分和细胞器为主,分别占62.5%和25%,其中细胞部分主要包括翻译延伸因子、核苷酸激酶、磷酸核糖基转移酶.有16种差异蛋白对应分子功能,涉及结合(如酰基辅酶A脱氢酶、5'-甲基硫代腺苷/S-腺苷高半胱氨酸核苷酶、50S核糖体蛋白)、催化活性(如寡肽转运ATP结合蛋白、核苷酸激酶、丙酮酸脱氢酶)、结构分子活性、翻译调节器活动和转运蛋白活性.
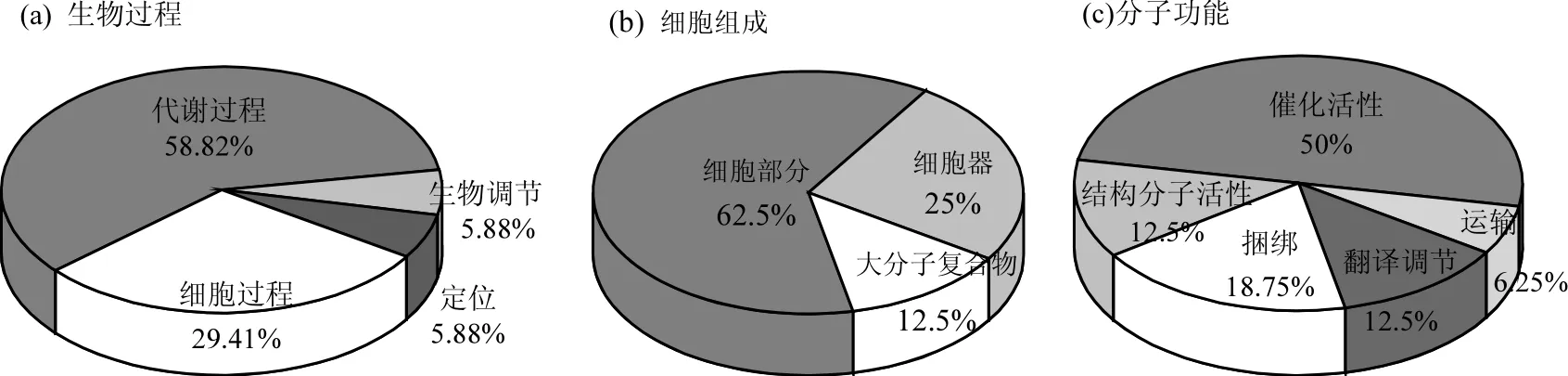
图2 差异表达蛋白的GO分析Fig.2 Protein GO analysis of Bacillus thuringiensis under diclofenac sodium stress
2.4 KEGG代谢通路分析
使用KEGG pathway对差异表达蛋白质可能参与的信号通路进行分析发现,差异表达的蛋白质参与的代谢通路中,其中上调蛋白,Upp涉及DNA和RNA的合成,核糖体蛋白RplM和RplL参与蛋白质的合成;下调蛋白,Prs、MtnN和PdhA分别参与磷酸戊糖途径、甲硫氨酸代谢和丙酮酸代谢,RpoA参与DNA和RNA的合成(图3),AtpH和OppF分别参与氧化磷酸化和群体感应.总体来看,差异蛋白质主要参与的代谢通路包含细胞核酸代谢、胞内蛋白质合成和能量代谢,这与 GO 功能注释结果类似,研究证实,双氯芬酸钠胁迫主要在细胞核心代谢网络能量代谢中抑制 ATP合成,蛋白质合成中在转录水平对功能蛋白质的合成造成了障碍,造成了有氧代谢受阻,直接影响到乙酰辅酶 A的形成,继而影响三羧酸循环和随之的能量代谢,随之影响细胞中绝大部分的代谢活动,从而使细胞生长表现出受抑制.
2.5 差异表达蛋白质相互作用网络及其功能
通过 STRING系统构建差异表达蛋白质的相互作用网络图(图4),包含 10个存在密切关系的相互作用蛋白质网络和位于整个网络周边的7个离散蛋白质.该蛋白质的相互作用网络以RpoA、RplM、RplL、Tuf、InfA 5个蛋白连接度较高,属于网络中的关键节点.
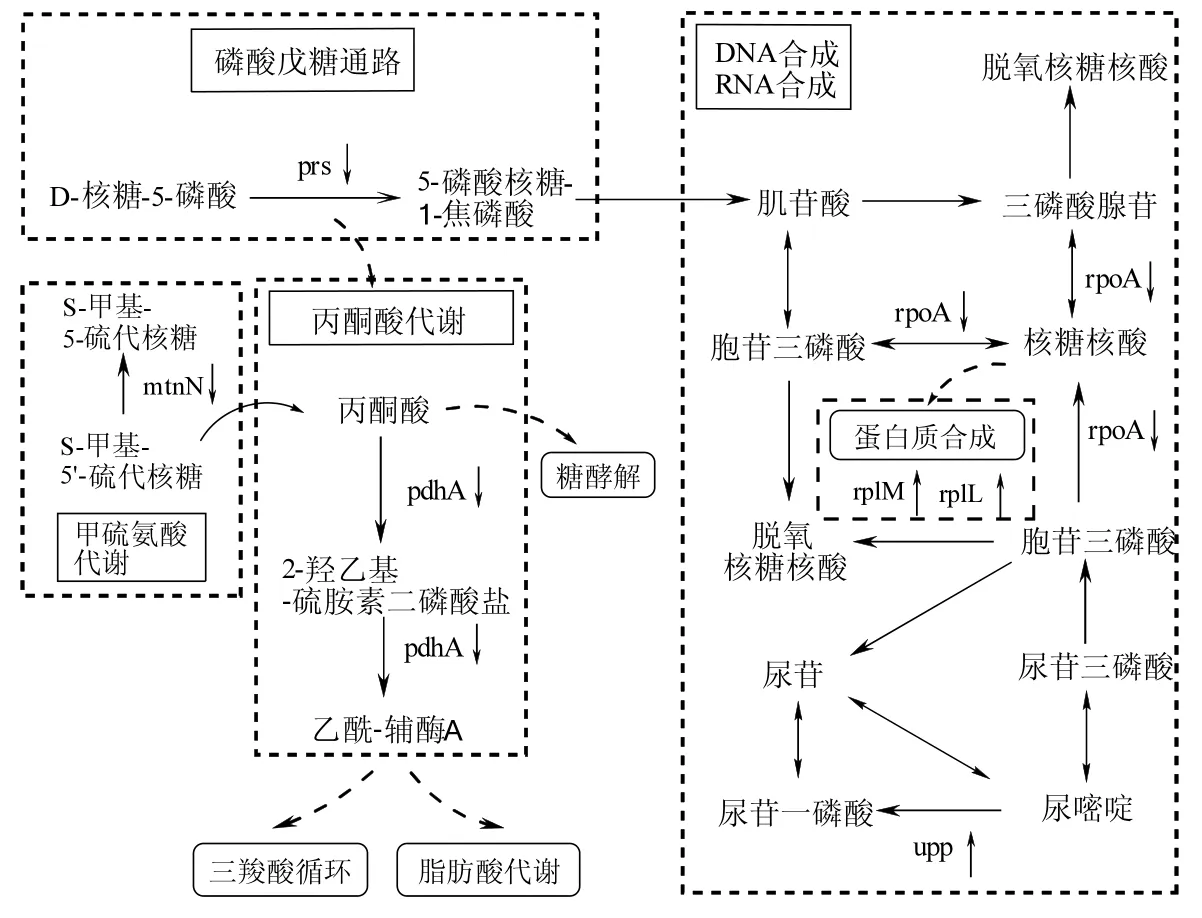
图3 差异蛋白质代谢通路Fig.3 Analysis of differential protein metabolism pathway

图4 差异表达蛋白的相互作用网络Fig.4 Interaction between differential expressed proteins
芽孢杆菌的一个重要特性是能够产生对不利条件具有特殊抵抗力的芽孢,而在营养丰富的环境中又能够重新萌发成营养细胞.SpoVR是芽孢形成过程中的关键酶[22-23],参与芽孢皮质的形成[24].此外,微生物在胁迫耐受中生物膜的形成会显著加强[25-26],5'-甲硫基腺苷/S-腺苷高半胱氨酸核苷酶(MtnN)间接减少生物膜的形成[27].本研究中,芽孢形成蛋白 SpoVR 上调,说明双氯芬酸钠可能诱导了苏云金芽孢杆菌的芽孢形成以抵抗不利环境.同时,菌体细胞通过下调MtnN加强了苏云金芽孢杆菌生物膜的形成,以此来降低双氯芬酸钠对菌体细胞的毒性.
在细胞核酸代谢中,核糖磷酸焦磷酸激酶(Prs)可将 ATP和核糖 5磷酸 R5P催化转化为AMP和磷酸核糖焦磷酸(PRPP),PRPP是众多生化反应的共同底物,作为关键的节点代谢物,对于嘌呤、嘧啶和吡啶核苷酸的合成具有重要意义[28-30].在嘧啶的合成途径中,PRPP是尿苷一磷酸合成酶 UMPS的重要辅因子.PRPP也为合成吡啶核苷酸 NAD+和 NADP+提供了核苷部分.除此之外,PRPP也在嘌呤的补救合成途径中确保了嘌呤碱基和核苷的有效重新利用,该补救途径合成PRPP仅需1mol ATP,而从头合成1mol核苷酸需消耗 7mol ATP[31].本研究中,双氯芬酸钠处理后,细胞中Prs的下调不仅对正常的DNA及RNA的合成代谢造成了负面影响,也可能对能量代谢产生影响.
在细胞内蛋白质合成过程中,核糖体蛋白质在蛋白质合成中起关键作用,包括催化肽键形成活性,提供G蛋白因子的结合位点(辅助起始、延伸和终止),帮助合成后的蛋白质正确折叠等[32-33]. RNA聚合酶亚基α(RpoA)是RNA聚合酶在细菌中高度保守并且是基因转录中的关键酶,它有助于稳定转录复合物,启动子识别,并调节启动基因转录.抑制RpoA会导致RNA聚合酶复合物不稳定,从而会直接干扰 mRNA的形成
[34-35].除此之外,核糖体蛋白RplM和RplL也参与蛋白质的合成.本研究中,菌体细胞中 RpoA、RplM和RplL均表现出下调,说明双氯芬酸钠胁迫在转录水平对功能蛋白质的合成造成了障碍.
三羧酸循环是生物细胞最重要的基础代谢过程之一.细胞丙酮酸脱氢酶(PDH)复合体(PdhA、PdhB、PdhC、PdhD)的 E1组分亚单位α(PdhA)将糖酵解/葡萄糖醛酸代谢与三羧酸循环连接,其功能是产生乙酰辅酶A,乙酰辅酶A不仅参与各种生物合成途径,如氨基酸、脂肪酸、聚酮化合物,并作为生长信号起到关键作用[36],还可以帮助细胞在三羧酸循环中产生 NAD+/NAD,作为细胞生长和营养供给的信号[37].PdhA是催化丙酮酸氧化脱羧反应的起始酶,在整个氧化脱羧反应过程中,其所催化的反应是唯一的不可逆步骤.PdhA的下调,使得苏云金芽孢杆菌的有氧代谢受阻,直接影响到乙酰辅酶A的形成,继而影响三羧酸循环和随之的能量代谢[34],随之影响细胞中绝大部分的代谢活动,从而使细胞生长表现出受抑制.
生物细胞的能量代谢是整个代谢网络的核心,在有氧呼吸中,能量代谢主要产生过程是三羧酸循环释放出的电子经过电子传递链而产生.NadK(NAD+激酶)催化NAD+进行磷酸化反应生成NADP+,在这个过程中与ATP的生成相偶联,在细胞能量代谢中扮演关键角色.FadE是酰基辅酶A脱氢酶(ACAD)的一种,酰基辅酶A脱氢酶是含有黄素腺嘌呤二核苷酸(FAD)的酶家族,黄素腺嘌呤二核苷酸(FAD)作为辅因子,催化酰基辅酶 A在脂肪酸和一些氨基酸分解代谢第一步中关键的酶[38],ACAD将电子从其相应的辅酶A酯底物转移到电子转运蛋白(ETF),伴随 ATP的生成[39].无论是NAD还是FAD介导的电子传递,最终ATP的生成需ATP合成酶的催化才能完成,而F0F1-ATP合成酶是与ATP合成相关的主要膜蛋白复合物,其中AtpH(ATP合成酶亚基delta)是 F0F1-ATP合成酶中 F1的主要组成部分[40],AtpH下调直接影响到 ATP的合成.有文献报道[11-13]真核细胞中,ATP合成损伤可能是双氯芬酸钠引起肝细胞毒性的关键原因.在本研究中,也观察到与ATP合成直接相关的AtpH及间接相关的NadK、PdhA均下调表达,说明双氯芬酸钠也抑制苏云金芽孢杆菌的 ATP合成.但同时也发现,FadE作为与 ATP合成相关蛋白,却表现出上调,意味着双氯芬酸钠对苏云金芽孢杆菌甚至对原核生物的ATP代谢的影响是非常复杂的,需要进一步深入研究.另外,在能量合成受损的情况下,催化反应需消耗ATP的Prs也相应下调,使得菌体细胞嘌呤、嘧啶和吡啶核苷酸合成代谢所需的PRPP转向消耗更少能量的补救途径进行,以适应受胁迫的环境.
3 结论
3.1 在双氯芬酸钠的处理下,苏云金芽孢杆菌的生长受到明显抑制.
3.2 蛋白质组分析共检测到108个蛋白,其中有17个差异表达蛋白,包括5个上调蛋白和12个下调蛋白,主要涉及到脂肪酸生物合成、DNA和RNA的合成、氧化磷酸化、丙酮酸代谢、糖酵解途径、磷酸戊糖途径和氨基酸代谢等代谢通路.
3.3 差异表达蛋白相互作用网络中,RpoA、RplM、RplL、Tuf、InfA 5个蛋白连接度较高,属于网络中的关键节点,可能起着重要的调控作用.
[1] Ji K, Liu X, Lee S, et al. Effects of non-steroidal antiinflammatory drugs on hormones and genes of the hypothalamicpituitary-gonad axis, and reproduction of zebrafish [J]. Journal of Hazardous Materials, 2013,254:242-251.
[2] Acuna V, Ginebreda A, Mor J R, et al. Balancing the health benefits and environmental risks of pharmaceuticals: diclofenac as an example [J]. Environment International, 2015,85:327-333.
[3] 史 瑞,张付申.碱催化水热氧化法处理废弃双氯芬酸钠类药物[J]. 中国环境科学, 2017,37(4):1386-1393.
[4] Yu H, Nie E, Xu J, et al. Degradation of diclofenac by advanced oxidation and reduction processes: kinetic studies, degradation pathways and toxicity assessments [J]. Water Research, 2013,47(5):1909-1918.
[5] Lepper P. Manual on the methodological framework to derive environmental quality standards for priority substances in accordance with Article 16of the Water Framework Directive(2000/60/EC) [C]//Schmallenberg, Germany: Fraunhofer-Institute Molecular Biology and Applied Ecology, 2005.
[6] 王 月,熊振湖,周建国.杯[4]芳烃修饰 Amberlite XAD-4树脂去除水中双氯芬酸 [J]. 中国环境科学, 2012,32(1):81-88.
[7] Oaks J L, Gilbert M, Virani M Z, et al. Diclofenac residues as the cause of vulture population decline in Pakistan [J]. Nature,2004,427(6975):630-633.
[8] Triebskorn R, Casper H, Heyd A, et al. Toxic effects of the nonsteroidal anti-inflammatory drug diclofenac: Part II.Cytological effects in liver, kidney, gills and intestine of rainbow trout (Oncorhynchus mykiss) [J]. Aquatic Toxicology, 2004,68(2):151-166.
[9] Cleuvers M. Aquatic ecotoxicity of pharmaceuticals including the assessment of combination effects [J]. Toxicology Letters, 2003,142(3):185-194.
[10] Cleuvers M. Mixture toxicity of the anti-inflammatory drugs diclofenac, ibuprofen, naproxen, and acetylsalicylic acid [J].Ecotoxicology and Environmental Safety, 2004,59(3):309-315.
[11] Ungprasert P, Cheungpasitporn W, Crowson C S, et al. Individual non-steroidal anti-inflammatory drugs and risk of acute kidney injury: a systematic review and meta-analysis of observational studies [J]. European Journal of Internal Medicine, 2015,26(4):285-291.
[12] Fattori V, Borghi S M, Guazelli C F S, et al. Vinpocetine reduces diclofenac-induced acute kidney injury through inhibition of oxidative stress, apoptosis, cytokine production, and NF-kappaB activation in mice [J]. Pharmacological research, 2017,120:10-22.
[13] Syed M, Skonberg C, Hansen S H. Mitochondrial toxicity of diclofenac and its metabolites via inhibition of oxidative phosphorylation (ATP synthesis) in rat liver mitochondria:possible role in drug induced liver injury (DILI) [J]. Toxicology in Vitro, 2016,31:93-102.
[14] Hickey E J, Raje R R, Redd V E, et al. Diclofenac induced in vivo nephrotoxicity may involve oxidative stress-mediated massive genomic DNA fragmentation and apoptotic cell death [J]. Free Radical Biology and Medicine, 2001,31(2):139-152.
[15] Ng L E, Vincent A S, Halliwell B, et al. Action of diclofenae on kidney mitochondria and cells [J]. Biochemical and Biophysical Research Communications, 2006,348(2):494-500.
[16] Fatta-Kassinos D, Hapeshi E, Achilleos A, et al. Existence of pharmaceutical compounds in tertiary treated urban wastewater that is utilized for reuse applications [J]. Water Resources Management, 2011,25(4):1183-1193.
[17] Zorita S, Martensson L, Mathiasson L. Occurrence and removal of pharmaceuticals in a municipal sewage treatment system in the south of Sweden [J]. Science of the Total Environment,2009,407(8):2760-2770.
[18] Kambiranda D, Katam R, Basha S M, et al. iTRAQ-based quantitative proteomics of developing and ripening muscadine grape berry [J]. Journal of Proteome Research, 2014,13(2):555-569.
[19] 张留辉.锌离子对肺炎链球菌影响的蛋白质组学研究 [D]. 广东:暨南大学, 2010.
[20] 黄莹莹,白 羽,王 燕,等.基于iTraq技术的加拿大一枝黄花提取物作用下铜绿微囊藻细胞差异表达蛋白 [J]. 中国环境科学,2015,35(6):1822-1830.
[21] McKenney P T, Driks A, Eichenberger P. The Bacillus subtilis endospore: assembly and functions of the multilayered coat [J].Nature Reviews Microbiology, 2013,11(1):33-44.
[22] Ozin A J, Henriques A O, Yi H, et al. Morphogenetic proteins SpoVID and SafA form a complex during assembly of the Bacillus subtilis spore coat [J]. Journal of Bacteriology, 2000,182(7):1828-1833.
[23] Chen F C, Shen L F, Tsai M C, et al. The IspA protease's involvement in the regulation of the sporulation process of Bacillus thuringiensis is revealed by proteomic analysis [J].Biochemical and Biophysical Research Communications, 2003,312(3):708-715.
[24] Huang J C, Wang X R, Cao Q, et al. ClpP participates in stress tolerance and negatively regulates biofilm formation in Haemophilus parasuis [J]. Veterinary Microbiology, 2016,182:141-149.
[25] Dorel C, Lejeune P, Rodrigue A. The Cpx system of Escherichia coli, a strategic signaling pathway for confronting adverse conditions and for settling biofilm communities [J]. Research in Microbiology, 2006,157(4):306-314.
[26] Silva A J, Parker W B, Allan P W, et al. Benitez. Role of methylthioadenosine/S-adenosylhomocysteine nucleosidase in Vibrio cholerae cellular communication and biofilm development[J]. Biochemical and Biophysical Research Communications,2015,461(1):65-69.
[27] Shimaoka M, Takenaka Y, Kurahashi O, et al. Effect of amplification of desensitized purF and prs on inosine accumulation in Escherichiacoli [J]. Journal of Bioscience and Bioengineering, 2007,103(3):255-261.
[28] Shi S B, Shen Z, Chen X, et al. Increased production of riboflavin by metabolic engineering of the purine pathway in Bacillus subtilis [J]. Biochemical Engineering Journal, 2009,46(1):28-33.
[29] Yin Y L, Ashihara H. Phosphate levels and expression of phosphoribosylpyrophosphate synthetase isozymes in suspension-cultured Arabidopsis thaliana cells [J].Phytochemistry Letters, 2009,2(3):126-129.
[30] 陈 朋.人类磷酸核糖焦磷酸合成酶 PRS1及其相关蛋白的结构与功能研究 [D]. 合肥:中国科学技术大学, 2013.
[31] Kovacs D, Rakacs M, Agoston B, et al. Janus chaperones:assistance of both RNA- and protein-folding by ribosomal proteins [J]. Febs Letters, 2009,583(1):88-92.
[32] Kim T Y, Ha C, Huh W K. Differential subcellular localization of ribosomal protein L7paralogs in Saccharomyces cerevisiae [J].Molecules and Cells, 2009,27(5):539-546.
[33] Husnain S I, Meng W M, Busby S J W, et al. Escherichia coli can tolerate insertions of up to 16amino acids in the RNA polymerase alpha subunit inter-domain linker [J]. Biochimica Et Biophysica Acta-Gene Structure and Expression, 2004,1678(1):47-56.
[34] Abushahba M F N, Mohammad H, Seleem M N. Targeting multidrug-resistant staphylococci with an anti-rpoA peptide nucleic acid conjugated to the HIV-1TAT cell penetrating peptide[J]. Molecular Therapy-Nucleic Acids, 2016,5:e339. https://doi.org/10.1038/mtna.2016.53.
[35] Cai L, Sutter B M, Li B, et al. Acetyl-CoA induces cell growth and proliferation by promoting the acetylation of histones at growth genes [J]. Molecular Cell, 2011,42(4):426-437.
[36] 李 艳.重组苏云金芽孢杆菌丙酮酸脱氢酶表达条件的优化及结构预测 [D]. 武汉:华中师范大学, 2008.
[37] Fleige T, Pfaff N, Gross U, et al. Localisation of gluconeogenesis and tricarboxylic acid (TCA)-cycle enzymes and first functional analysis of the TCA cycle in Toxoplasma gondii [J]. International Journal for Parasitology, 2008,38(10):1121-1132.
[38] Battaile K P, Molin-Case J, Paschke R, et al. Crystal structure of rat short chain acyl-CoA dehydrogenase complexed with acetoacetyl-CoA - Comparison with other acyl-CoA dehydrogenases [J]. Journal of Biological Chemistry, 2002,277(14):12200-12207.
[39] Le W P, Abbas A S, Sprecher H, et al. Long-chain acyl-CoA dehydrogenase is a key enzyme in the mitochondrial betaoxidation of unsaturated fatty acids [J]. Biochimica Et Biophysica Acta-Molecular and Cell Biology of Lipids, 2000,1485(2/3):121-128.
[40] Trchounian A. Escherichia coli proton-translocating F0F1-ATP synthase and its association with solute secondary transporters and/or enzymes of anaerobic oxidation-reduction under fermentation [J]. Biochemical and Biophysical Research Communications, 2004,315(4):1051-1057.

