尼古丁微生物降解代谢机制和应用的研究进展
李晓华,梅 枫,孔 雯,李 阳,马婷婷,皮 婷
(中南民族大学 生命科学学院 生物技术国家民委重点实验室,武汉 430074)
尼古丁微生物降解代谢机制和应用的研究进展
李晓华,梅 枫,孔 雯,李 阳,马婷婷,皮 婷
(中南民族大学 生命科学学院 生物技术国家民委重点实验室,武汉 430074)
为进一步了解利用生物降解消除环境中尼古丁污染的最有前景的方法,综述了国内外尼古丁降解微生物、尼古丁降解代谢途径(真菌的去甲基化途径、革兰氏阳性菌的吡啶和吡咯途径、革兰氏阴性菌吡啶与吡咯途径的混合途径)、降解基因簇及其在环保和药学领域的应用,旨在为微生物降解尼古丁的深入研究和应用提供参考.
尼古丁;微生物降解;代谢途径;应用
尼古丁[1-甲基-2-(3-吡啶基)-吡咯烷]是一种存在于茄科植物(茄属)中的生物碱,是烟草的重要成分,占烟叶干重的2%~8%[1]. 自然环境和人类生活环境中的尼古丁污染大部分来源于烟草工业废弃物,烟草工业每年生产约300274 t尼古丁废弃物[2]. 尼古丁易溶于水,导致地下水的污染,扰乱生态平衡. 因此,将尼古丁污染从土地和水中移除成为了亟待解决的问题.
尼古丁能引起严重的血管疾病、癌症、基因突变、畸形等,作为神经毒素能影响细胞凋亡、细胞的畸形生长、提高基因表达率、分泌激素和调节酶的活性等. 它还能快速地通过生物膜和血脑屏障,加快心率和升高动脉压,引起静脉功能障碍. 尼古丁还通过作用于位于中央神经系统的神经型尼古丁受体,释放如多巴胺和血清素的神经传导物质,激活大脑皮层中心多巴胺系统,加强生理依赖效应[3].
目前,降解尼古丁的主要方法有物理法、化学法和生物法. 物理法包括吸附法和萃取法;化学法则是利用强氧化剂改变尼古丁官能团或化学键的性质降解尼古丁. 但是物理或化学方法降解尼古丁耗时长、花费高、效率低、易造成二次污染,故利用生物降解是消除环境中尼古丁污染的最有前景的方法. 近年来,越来越多的尼古丁降解菌被分离,在尼古丁的微生物降解途径和应用方面取得进展. 本文对国内外尼古丁降解微生物、尼古丁降解代谢途径及其降解基因簇研究进展进行了综述.
1 降解尼古丁的微生物
降解尼古丁的微生物易适应被污染的环境,烟草环境中的细菌和真菌都有降解尼古丁的能力[4],通常将尼古丁作为生长所需的唯一碳源、氮源和能源. 自20世纪50年代起,尼古丁降解菌Arthrobacternicotinovorans已被广泛研究,近年来,越来越多的节杆菌被报道能够降解尼古丁(见表1).
从烟草种植地分离出的尼古丁降解菌ArthrobacternicotianaeK9,能在尼古丁浓度达到1.0 g/L的培养基中生长,尼古丁降解效率在24 h内达到85%[5]. Lei 等[6]用节杆菌K7和K3的细菌悬液处理收获的K326烟叶,烟叶的尼古丁含量减少. Ruan等[7]发现的Arthrobactersp. HF-2降解尼古丁的最适温度为30℃,最适pH为7.0. 在1953年,Wada和Yamasaki从土壤中分离出Pseudomonassp. 41,该菌株能在pH 6.4和30℃下降解尼古丁[8]. 假单胞菌属中的尼古丁降解菌能在高浓度尼古丁的培养基中生长,尼古丁降解效率随着细菌细胞的生长而增加,降解最适pH值为6.4~7.4,最适温度为30℃,而PseudomonasstutzeriZCJ为37℃[9].从土壤和烟叶中分离得到的降解尼古丁的细菌除了节杆菌和假单胞菌,还包括Rhodococcussp. Y22,Ensifersp. N7,AgrobacteriumtumefaciensS33,Acinetobactersp. TW和Ochrobactrumsp. SJY1等细菌. 大多数细菌能够在28~30℃、pH 7.0的条件下降解尼古丁,还有少数真菌被分离鉴定能够降解尼古丁.
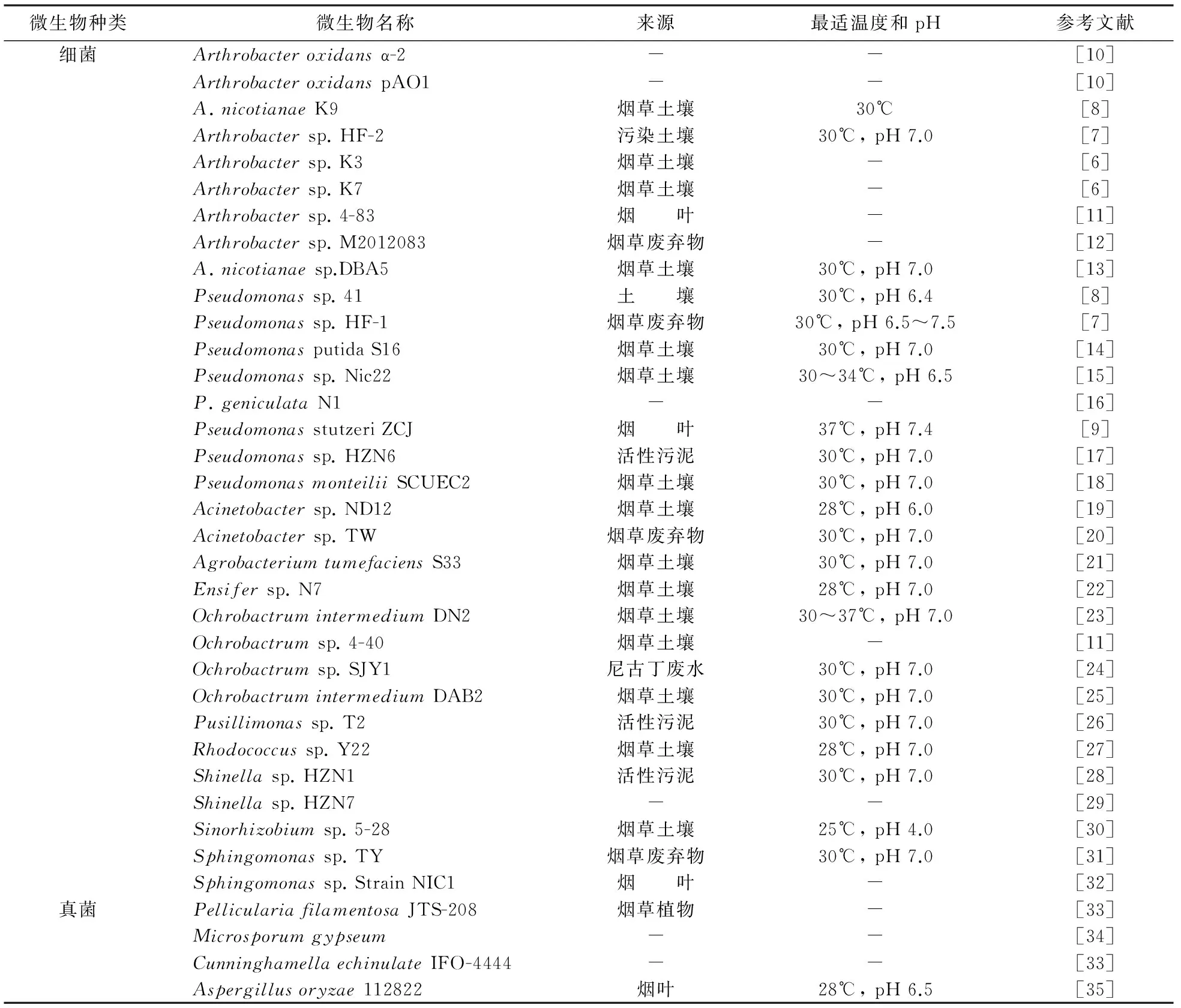
表1 尼古丁降解微生物及其降解最适条件Tab.1 Nicotine-degrading microorganisms and their optimal condition for degradation
注:-表示文献中未报道
2 微生物中尼古丁降解基因
编码尼古丁降解的基因不仅位于细菌染色体上,也存在于质粒上P.convex菌株尼古丁降解基因,除在染色体之外,由NIC质粒介导.A.nicotinovorans中大小为160 kb的质粒pAO1(见图1,参考文献[10、14、24、29])包含负责尼古丁降解的基因簇. Brandsch等[4]对pAO1进行亚克隆,利用表达载体pBR322和pUR222将6hdno基因在大肠杆菌进行表达. Grether等[36]测序分析发现,ndh基因与6hdno基因紧密相邻,3个不同大小的亚基组成了ndh基因,ndhA基因在ORFX(1个小的开放阅读框)之后,6hlno基因则在ndhC基因之后. Schenk[37]等利用噬菌体构建了质粒pAO1的DNA文库,筛选到基因6hlno和基因kdh. Brandsch等[4]将pAO1进行了全序列测定,推测尼古丁转运载体与6hdno形成一个基因簇. Chiribau等[38]从pAO1中克隆了包含purU-mabO-folD操纵子的基因簇,该操纵子仅在有尼古丁的条件下转录,并被转录激活因子pmfR调控.
通过基因组挖掘和代谢分析,Wang等[14]在Pseudomonassp. S16中发现了nic基因簇,4879 bp的nic基因簇编码3个ORF,ORF1编码nicA基因,ORF2编码hspA基因,ORF3功能未知. 新验证的hspB基因位于远离nic基因簇30 kb的基因簇上. 在Pseudomonassp. HZN6中,已验证了4种主要的中间代谢产物,分别为pseudooxynicotine(假氧化尼古丁)、SP(3-琥珀酰吡啶)、HSP(6-羟基-3-琥珀酰吡啶)和不稳定的复合物SAP(3-琥珀酰半醛吡啶),并外源表达了3种尼古丁降解基因(SirA2、pna、sap). 基因SirA2编码SirA2蛋白,是巯基转移酶,负责SP的降解. 假氧化尼古丁在基因pna编码的假氧化尼古丁胺氧化酶(PNAO)的催化作用下转化生成SAP. 基因sap编码依赖NADP+的3-琥珀酰半醛脱氢酶(SAPD),使SAP脱氢生成SP[39]. Wang等[40]研究表明Pseudomonassp. HF-1的尼古丁降解基因位于质粒pMF1(21 kb),并通过非染色体DNA的质粒调节.
通过生物信息学、转录分析和异源表达实验,许平等定位了Ochrobactrumsp. Strain SJY1降解尼古丁的基因簇. VPP基因簇包含6个基因,基因vppB与基因6hlno有24.4%的氨基酸同源性,基因vppD与基因hspB有61.8%的氨基酸同源性,基因vppH到基因vppG作为一个连续的转录本转录,可催化2,5-DHP(2,5-二羟基吡啶)到延胡索酸的转化过程[24].
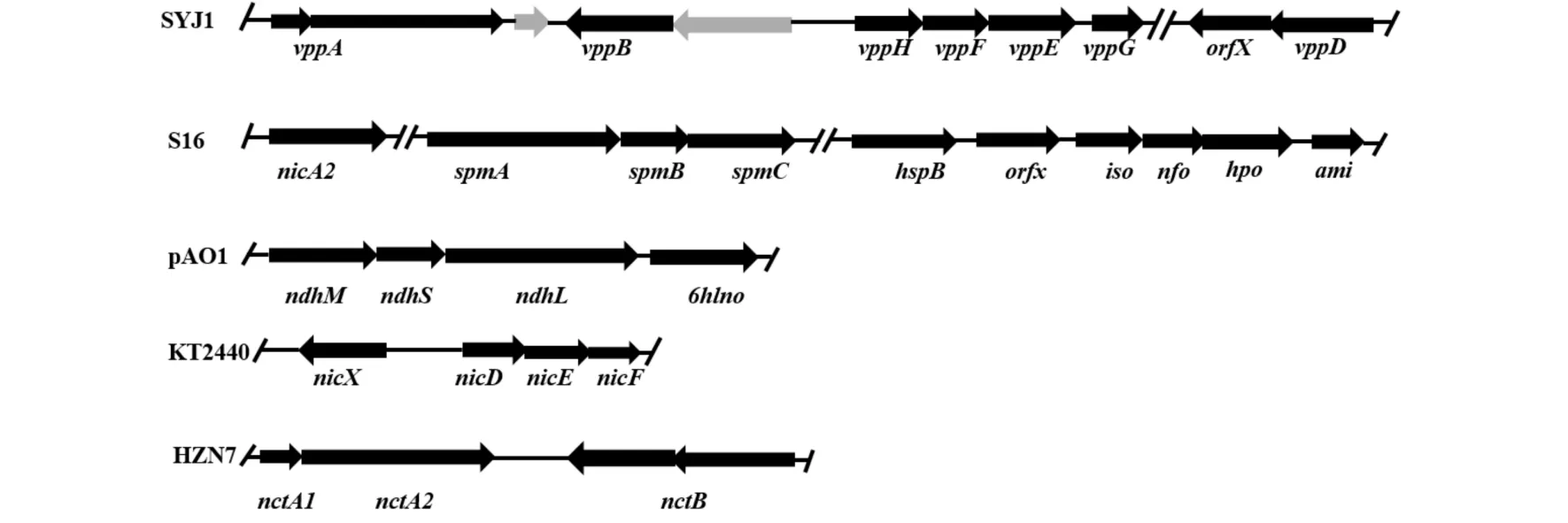
SYJ1: Ochrobactrum sp. SJY1; S16: Pseudomonas putida S16; pAO1: Arthrobacter nicotinovorans pAO1;KT2440: Pseudomonas putida KT2440; HZN7: Shinella sp. HZN7图1 已报道的尼古丁降解基因簇Fig.1 Gene cluster of reported nicotine degradation
3 微生物降解尼古丁的代谢途径
微生物降解尼古丁的途径因不同的菌种而有所差异. 革兰氏阳性菌Arthrobactersp.的尼古丁降解途径是吡啶途径,降解过程中相关的酶首先作用于吡啶环. 革兰氏阴性菌Pseudomonassp.的尼古丁降解途径是吡咯途径,降解过程中相关的酶首先作用于吡咯环. 尼古丁降解真菌一般是去甲基化途径,即去掉尼古丁吡咯环的甲基基团. 最近有研究表明根癌土壤杆菌和苍白杆菌的尼古丁降解途径是吡啶途径与吡咯途径的混合途径,即该途径起始于吡啶途径,之后转变为吡咯途径.
3.1 去甲基化途径
与尼古丁降解细菌相比,只有少数真菌被分离鉴定为能够降解尼古丁,包括Microsporumgypseum,PelliculariafilamentosaJTS-208和Cunninghamellaechinulata. 这些真菌降解尼古丁的步骤都起始于尼古丁的去甲基化,生成去甲尼古丁(nornicotine).
近期,从烟叶中分离出一株降解尼古丁的霉菌,鉴定为A.oryzae112822. 采用TLC、GC-MS、NMR和FT-IR等方法分析该菌株在尼古丁降解过程中产生的中间代谢产物,并推测尼古丁降解途径(见图2,参考文献[35]). 该途径起始于尼古丁吡咯烷的脱甲基化,先生成去甲尼古丁,再通过形成吡咯环的双键转化为麦斯明,随后吡咯烷裂解生成尚不明确的中间代谢产物,该中间代谢产物进一步被水解生成N-甲基烟酰胺和乙酸.N-甲基烟酰胺羟基化生成2-羟基-N-甲基烟酰胺,通过分解代谢生成2,3-DHP(2,3-二羟基吡啶)和CH3NHCOOH,CH3NHCOOH进一步转化生成碳酸,2,3-DHP开环生成琥珀酸进入三羧酸循环[22]. 但真菌的尼古丁降解途径中参与的酶尚未得到验证.
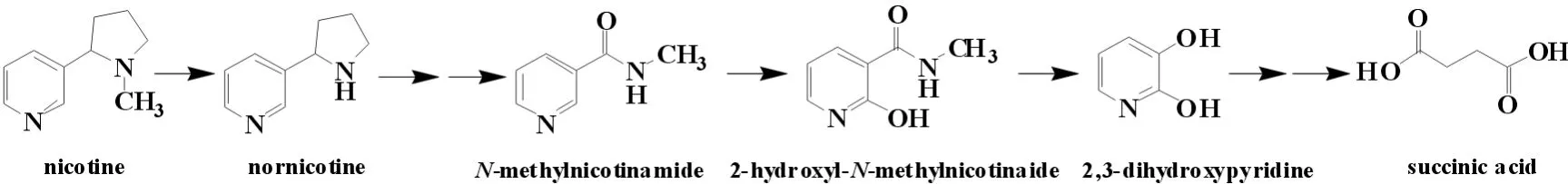
图2 Aspergillus oryzae 112822 降解尼古丁的去甲基化途径Fig.2 Demethylation pathway of nicotine degradation by Aspergillus oryzae 112822
3.2 吡啶途径
吡啶途径主要是节杆菌属降解尼古丁的途径,自20世纪50年代起,Arthrobacteroxidans作为尼古丁降解菌被广泛研究,Brandsch等[4]阐明了A.nicotinovorans尼古丁代谢途径和降解途径.
吡啶途径降解尼古丁起始于尼古丁吡咯环的6位碳羟基化,引入羟基基团生成6-HN(6-羟基尼古丁). 该步反应由NDH(尼古丁脱氢酶)催化,随后6-HN(6-羟基尼古丁)吡咯环的2位碳被氧化,生成6-HMM(6-羟基-N-甲基麦斯明),该步反应由6HLNO(6-羟基-L-尼古丁氧化酶)和6HDNO(6-羟基-D-尼古丁氧化酶)催化. 吡啶途径的第三步是6-HMM(6-羟基-N-甲基麦斯明)的水解反应,自发打开吡咯环,生成6-HPON(6-羟基假氧化尼古丁). 6-HPON吡啶环的2位碳被与尼古丁脱氢酶相似的KDH(酮脱氢酶)进一步羟基化,生成2,6-DHPON(2,6-羟基假氧化尼古丁),在2,6-DHPONH(2,6-二羟基假氧化尼古丁水解酶)的作用下,2,6-DHPON的侧链裂解生成MGABA(γ-N-甲基丁酸辛酯)和2,6-DHP(2,6-二羟基吡啶). 2,6-DHP进一步在吡啶环3位碳上羟基化生成2,3,6-THP (2,3,6-三羟基吡啶),由2,6-DHPH (2,6-二羟基吡啶-3-羟化酶)催化. 在有氧的条件下,2,3,6-THP自发氧化,并二聚体化该羟基化的吡啶环,部分形成尼古丁蓝. Chiribau等[38]的研究表明A.nicotinovorans的mobA基因编码负责生成尼古丁蓝的MobA(含钼辅因子胞嘧啶核苷酸转移酶).
MGABA进一步的降解有两种途径:第一种途径起始于MABO(γ-N-甲基丁酸辛酯氧化酶)的催化,生成GABA(γ-氨基丁酸)和亚甲基四氢叶酸. 亚甲基四氢叶酸进一步被FoLD(亚甲基-四氢叶酸脱氢酶)和PurU(甲酰四氢叶酸脱甲酰酶)催化转化生成甲醛,甲醛可能进入糖酵解途径. 而另外的代谢产物GABA则被MAO(单胺氧化酶)分解成NH3和Ssa(琥珀酸半醛). Ssa进一步被依赖于NADP+的SsaDH(琥珀酸半醛脱氢酶)氧化生成琥珀酸,琥珀酸可能进入三羧酸循环. MGABA的第二种降解途径是MGABA通过AO(单胺氧化酶)脱氨基生成Ssa和甲胺,并伴随着FAD还原成FADH2,最后生成琥珀酸,进入三羧酸循环(见图3,参考文献[10]).
3.3 吡咯途径
吡咯途径是假单胞菌属降解尼古丁的主要途径,有大量的代谢多样性,Ruan等[7]发现Pseudomonassp. HF-1有其他3种与Pseudomonassp. S16不完全相同的吡咯途径,例如尼古丁转化为可替宁,或者转化为二烯尼古丁、去甲尼古丁,而Pseudomonassp. Nic22则是经过可替宁和去甲尼古丁的吡咯途径降解尼古丁.
假单胞菌降解尼古丁的吡咯途径已被广泛研究,Chen等[15]阐明PseudomonasputidaS16的吡咯途径起始于尼古丁的吡咯环形成双键转化为N-甲基麦斯明,nicA基因编码的NicA(尼古丁氧化还原酶)催化该步脱氢反应,NicA还参与尼古丁转化为SP(3-琥珀酰吡啶)的过程. 第二步反应是可逆反应,N-甲基麦斯明的双键自发水解生成假氧化尼古丁,后者是一种烟草特异性的肺部致癌物质的前体物质[17],该物质进一步通过NicA脱氢生成甲胺和SP.
Qiu等[17]推测在假氧化尼古丁转化为SP的过程中产生了两种不稳定的复合物. SP的6位碳进一步被尚不明确的酶催化水解生成HSP(6-羟基-3-琥珀酰吡啶). 基因hsp编码HSP羟化酶,作用于HSP的3位碳,生成2,5-DHP(2,5-二羟基吡啶)和Ssa(琥珀酸半醛). 2,5-DHP进一步降解生成顺丁烯酰胺酸,通过水解作用脱氨基,生成顺丁烯. 顺丁烯的5、6位碳裂解生成丙酮酸,进入三羧酸循环(见图4,参考文献[14]). Ssa则被SsaDH(琥珀酸半醛脱氢酶)转化为琥珀酸,该酶在Pseudomonassp.中常见. 编码HSP羟化酶的基因hspA和hspB参与HSP到2,5-DHP的转化,该氧化反应中2分子氧中1个氧结合到底物,另1个氧则用于氧化NADH或NADPH. HspA是NADH依赖的,HspB则需要FAD作为辅基.
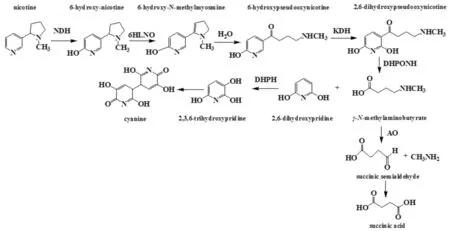
图3 A. nicotinovorans降解尼古丁的吡啶途径Fig.3 Pyridine pathway of nicotine degradation by A. nicotinovorans
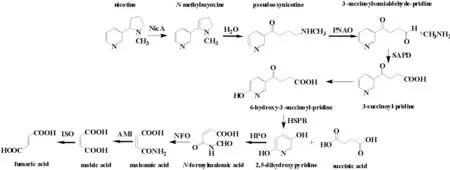
图4 Pseudomonas putida S16降解尼古丁的吡咯途径Fig.4 Pyrrolidine pathway of nicotine degradation by Pseudomonas putida S16
然而,Pseudomonassp. Nic22降解尼古丁的途径属于另外的吡咯途径,在尼古丁降解过程中产生myosmine(麦斯明),2,3-dipyridyl(2,3-二吡啶)和cotinine(可替宁)等中间代谢产物.Shinellasp. HZN1在尼古丁降解过程中产生了3种中间代谢产物,通过气质联用分析,验证确定为cotinine、myosmine、nicotyrine(二烯尼古丁)[41].PseudomonasCS3产生3种新型的中间代谢产物[36],尼古丁脱甲基生成3-(3,4-dihydro-2H-pyrrol-5-yl) pyridine[3-(3,4-二羟基-2H-5-吡咯)吡啶],尼古丁吡咯环的2位碳羟基化,生成1-methyl-5-(3-pyridyl) pyrroline-2-ol[1-甲基-5-(3-吡啶)-2-吡咯],进而生成cotinine.PseudomonasputidaJ5的基因panB编码酮泛解酸羟甲基转移酶,参与尼古丁的分解代谢. 吡咯途径的终产物丙酮酸是酮异戊酸的前体物质,该物质通过酮泛解酸羟甲基转移酶催化反应生成维生素、泛酸盐[42].
Raman等称PseudomonasplecoglossicidaTND35的尼古丁降解途径是一种吡咯途径的变异途径,不同于其他细菌和真菌,TND35菌株氧化尼古丁的部分吡咯环生成N-methylmyosmine(N-甲基麦斯明),该中间代谢产物的吡咯2位进一步羟基化,生成一种新型的类似于可替宁的代谢产物IM2 [2,3-二羟基-1-甲基-5-(3-吡啶)-1H-2-吡咯][43]. IM2经过脱甲基和羟基化,进一步氧化生成另一种类似于可替宁的代谢产物IM3 [5-(3-吡啶)-1H-吡咯],同时IM2被氧化并生成甲胺. 最后吡咯环开环生成终产物IM4[4-羟基-1-(3-吡啶)-1-丁酮] . 但是,在尼古丁降解过程中还观察到IM5(1-甲基2-吡咯烷)的存在.
3.4 吡啶途径与吡咯途径的混合途径
越来越多的细菌被鉴定能够降解尼古丁,如Rhodococcussp. Y22、Ensifersp. N7、AgrobacteriumtumefaciensS33、Acinetobactersp. TW和Ochrobactrumsp. SJY1. 其中AgrobacteriumtumefaciensS33、Ochrobactrumsp. SJY1和Shinellasp. HZN7,已有研究表明尼古丁降解途径是吡啶途径与吡咯途径的混合途径[24]. 通过TLC、HPLC、GC-MS等分析了Ochrobactrumsp. Strain SJY1的中间代谢产物. 在SYJ1菌株中,尼古丁在含钼的尼古丁羟化酶VPPA的作用下转化为6HN(6-羟基-尼古丁),该酶由两个亚基组成,小亚基有两个[2Fe-2S]簇结合域,大亚基有一个MoCo-结合域. 然后,VPPB(6HN氧化酶)催化6HN转化为6HMM(6-羟基-N-甲基麦斯明),VPPB以FAD作为辅酶. 6HMM自发水解生成6HPON(6-羟基假氧化尼古丁). 至此,VPP途径与吡啶途径的前半部分相同,随后经过6HPON转化为4-(6-羟基吡啶)-4-氧代丁醛作为过渡的中间代谢产物,再经酶转化为HSP (6-羟基-3-琥珀酰吡啶),进入VPP途径与吡咯途径后半部分相同的阶段. HSP在VPPD(HSP-3-单氧酶)的催化作用下生成2,5-DHP (2,5-二羟基吡啶),该步是吡咯途径的关键步骤,并且VPPD是需FAD作为辅酶、NAD(P)H依赖的酶. 2,5-DHP在VPPE(2,5-DHP双加氧酶)的催化作用下打开吡啶环,再在脱甲酰酶VPPF的作用下生成顺丁烯酰胺酸,顺丁烯酰胺酸经去酰胺作用转化为顺丁烯,在顺丁烯二酸异构酶的作用下生成延胡索酸(见图5,参考文献[24]).
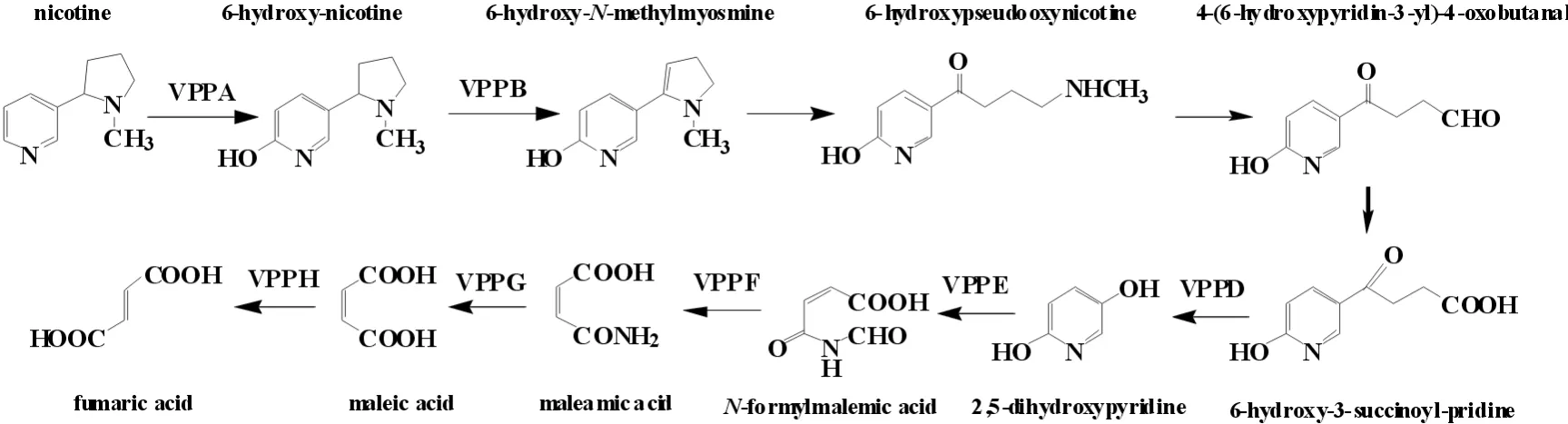
图5 Ochrobactrum sp. SJY1降解尼古丁的吡啶与吡咯的混合途径Fig.5 A composited pathway of pyridine and pyrrolidine of nicotine degradated by Ochrobactrum sp. SJY1
4 尼古丁降解微生物的应用
4.1 提高烟叶和烟草产品的质量
尼古丁降解菌已被用于减少烟叶中的尼古丁含量和提高香烟产品的质量. 如Cellulomonassp.和Pseudomonasputida在含尼古丁和硝酸盐的培养基中培养后,在含氧或不含氧的条件下用含菌的培养基处理白肋烟,降低了烟草的硝酸盐和尼古丁含量,产出降低了氧化氮、氰化氢和尼古丁含量的、温和的、而没有损失理想风味和特性的烟草制品[44,45].
4.2 处理烟草废弃物
许多微生物具有降解尼古丁和其他烟草废弃物中有害的化合物的能力,SBR(序批式反应器)技术已经成为处理烟草废水的一个好的选择,如Pseudomonassp. HF-1加入SBR系统中,与非自动化的系统相比,基础自动化系统表现出强有力的污水降解能力,在12 h能降解100%的尼古丁,并减少了84%的化学需氧量[21]. 利用尼古丁降解菌Acinetobactersp. TW也可以构建基础自动化系统,该系统中尼古丁的降解率达到95%,有机物的去除率达80% ~ 90%. 而且在TW菌株和土著菌株中未见明显的竞争关系. TW菌株在系统中的分布可能有助于尼古丁的降解,使活性污泥中的土著菌株免于尼古丁毒性的伤害,并增加了基础自动化系统中的生物多样性. 微生物产甲烷作用和堆肥也被应用于降解烟草废弃物中的尼古丁.
4.3 检测环境中的尼古丁含量
通过基因工程改造微生物作为生物传感器,用于原位环境检测,根据已知微生物的尼古丁降解酶的相关信息,在尼古丁降解微生物中合成转录调节因子. 利用大肠杆菌表达调节因子等相关基因,产生的调节蛋白作为环境中存在尼古丁时的效应物分子,可发展为尼古丁敏感探测器. 利用6HDNO(6-羟基-D-尼古丁氧化酶)操纵子的启动子和阻遏蛋白构建响应6HN的可诱导哺乳动物表达系统,为尼古丁敏感探测器研究提供基础[10].
4.4 产生有医用价值的中间代谢产物
尼古丁降解微生物能将尼古丁转化为多种中间代谢产物,其中一些中间代谢产物被用于制药工业. 如吡啶衍生物被广泛用于抗癌、镇痛、治疗帕金森、缓解高血压和中央神经系统的紊乱. DHP可用于合成植物生长调节剂、除草剂、抗癌药、亚铁血红素和叶绿素的卟啉类类似物. HSP是合成地棘蛙素类似物药物的前体,地棘蛙素是一种极其有效的止痛剂分子. Wang等[46]发现一种利用假单胞菌S16的细胞将烟草废弃物中的(S′)-尼古丁转化为HSP的技术,HSP易于从反应物中分离,避免了复杂的分离步骤. Yu等[24]构建了一株工程菌P.putidaP-HSP,该菌株能够从含有高浓度尼古丁的烟草废弃物中产出HSP.
4.5 为戒烟提供新方法
目前,用于戒烟有显著临床疗效的典型药理方法如尼古丁替代疗法、抗抑郁药物安非他酮,最新的瓦伦尼克林均表现出了较高的禁烟率. 但仅有15%~30%的吸烟者能在治疗后维持戒烟超过1年. 由于来自香烟的尼古丁能很快被人体吸收并进入动脉循坏,经过口腔黏膜和肺部快速分布到人体组织约20 s通过大脑. Xue等[47]提出将尼古丁降解酶NicA2作为戒烟的候选药物,该法主要通过破坏或减少尼古丁的循环,使大脑中尼古丁的浓度不能达到或维持在致瘾浓度.
5 结语
环境污染是当今世界面临的主要问题之一,烟草产生了大量的含尼古丁的废弃物,引起环境污染和人体健康问题.研究微生物参与降解尼古丁的代谢途径和基因,可将生物降解作为一种新的转化有毒复合物为有价值的复合物的工具,尼古丁降解微生物可用于降解烟草废弃物,大量产生的中间代谢产物可被用于制药业.
目前,用于烟草发酵和烟草废弃物的尼古丁降解微生物仍处于小范围或实验室水平,未来研究将集中在如何将其应用于大规模提高烟叶和香烟产品的质量、烟草废弃物的降解、生产有价值的中间代谢产物. 虽然某些特定的尼古丁降解菌的中间代谢产物和尼古丁降解途径已取得一定进展,但减少人体和环境中的尼古丁的毒害及其代谢过程,如何进行尼古丁降解微生物的基因修饰和代谢过程以提高尼古丁降解活性并减少有害代谢产物的积累,仍是未来重要的研究主题.
[1] Gurusamy R, Natarajan S. Current status on biochemistry and molecular biology of microbial degradation of nicotine[J]. The Scientific World J, 2012, 2013(1): 1653-1656.
[2] Liu J, Ma G H, Chen T, et al. Nicotine-degrading microorganisms and their potential applications[J]. Appl Microbiol Biotechnol, 2015, 99(9): 3775-3785.
[3] Civilini M, Domenis C, Sebastianutto N, et al. Nicotine decontamination of tobacco agro-industrial waste and its degradation by micro-organisms[J]. Waste Manage Res, 1997, 15(4): 349-358.
[4] Brandsch R. Microbiology and biochemistry of nicotine degradation[J]. Appl Microbiol Biotechnol, 2006, 69(5): 493-498.
[5] Xia Z Y, Lei L P, Wu Y P, et al. Isolation and identification of degrading nicotine bacteria-Arthrobacternicotianaestrain K9[J]. Chin Tob Sci, 2006, 2: 1-4.
[6] Lei L P, Xia Z Y, Guo R J. Reduction of nicotine in tobacco leaf treated withArthrobacterspp.[J]. Tob Sci Technol, 2008, 3: 56-58.
[7] Ruan A D, Min H, Peng X, et al. Isolation and characterization ofPseudomonassp. strain HF-1, capable of degrading nicotine[J]. Res Microbiol, 2005, 156 (5/6): 700-706.
[8] Wada E, Yamasaki K. Degradation of nicotine by soil bacteria[J]. JACS, 2002, 76(1):155-157.
[9] Zhao L, Zhu C J, Gao Y, et al. Nicotine degradation enhancement byPseudomonasstutzeriZCJ during aging process of tobacco leaves[J]. World J Microbiol Biotechnol, 2012, 28(5): 2077-2086.
[10] Kostrzynska M, Leung K T, Lee H, et al. Green fluorescent protein-based biosensor for detecting SOS-inducing activity of genotoxic compounds[J]. Microbiol Methods, 2002, 48(1): 43-51.
[11] Ma G H, Lei L P, Xia Z Y, et al. Diversity and phylogenetic analyses of nicotine degrading bacteria isolated from tobacco plantation soils[J]. Afr Microbiol Res, 2012, 6(34): 6392-6398.
[12] Yao Y X, Tang H Z, Su F, et al. Comparative genome analysis reveals the molecular basis of nicotine degradation and survival capacities ofArthrobacter[J]. Sci Rep, 2015, 5: 8642-8652.
[13] 孔 雯, 先 锋, 李长影, 等. 1株烟碱降解菌的筛选、鉴定及其降解性能的初步研究[J]. 华中农业大学学报, 2011, 30(1): 30-33.
[14] Wang S N, Xu P, Tang H Z, et al. Biodegradation and detoxification of nicotine in tobacco solid waste by aPseudomonassp.[J]. Biotechnol Lett, 2004, 26(19): 1493-1496.
[15] Chen C M, Li X M, Yang J K, et al. Isolation of nicotine-degrading bacteriumPseudomonassp. Nic22, and its potential application in tobacco processing[J]. Int Biodeterior Biodegrad, 2008, 62(3): 226-231.
[16] Tang H, Yao Y, Wang L, et al. Genomic analysis ofPseudomonasputida: genes in a genome island are crucial for nicotine degradation[J]. Sci Rep, 2012, 2(4): 377.
[17] Qiu J, Ma Y, Wen Y, et al. Functional identification of two novel genes fromPseudomonassp. strain HZN6 involved in the catabolism of nicotine[J]. Appl Environ Microbiol, 2012, 78(7): 2154-2160.
[18] 李 阳, 杨振飞, 马婷婷, 等. 烟碱降解菌SCUEC2菌株的分离及其降解特性[J]. 湖北农业科学, 2015, 54(7): 1586-1589.
[19] Li H J, Duan Y Q, Ma G H, et al. Isolation and characterization ofAcinetobactersp. ND12 capable of degrading nicotine[J]. Afr Microbiol Res, 2011, 5(11): 1335-1341.
[20] Wang H J, He H Z, Wang M Z, et al. Bioaugmentation of activated sludge withAcinetobactersp.TW enhances nicotine degradation in a synthetic tobacco wastewater treatment system[J]. Bioresour Technol, 2013, 142(8): 445-453.
[21] Wang S N, Liu Z, Xu P. Biodegradation of nicotine by a newly isolatedAgrobacteriumsp. strain S33[J]. Appl Microbiol, 2009, 107(3): 838-847.
[22] Lei L P, Zhang W, Wei H L, et al. Characterization of a novel nicotine-degradingEnsifersp. strain N7 isolated from tobacco rhizosphere[J]. Ann Microbiol, 2009, 59(2): 247-252.
[23] Yuan Y J, Lu Z X, Huang L J, et al. Biodegradation of nicotine from tobacco waste extract byOchrobactrumintermediumDN2[J]. Ind Microbiol Biotechnol, 2007, 34(8): 567-570.
[24] Yu H, Tang H, Zhu X, et al. Molecular mechanism of nicotine degradation by a newly isolated strainOchrobactrumsp.SJY1[J]. Appl Environ Microbiol, 2015, 81(1): 272-281.
[25] 李晓华, 杨晓潼, 翟贵发, 等. 烟碱降解菌DAB2菌株的筛选、鉴定和降解特性[J]. 中南民族大学学报(自然科学版), 2013, 32(3): 18-21.
[26] Ma Y, Wen R T, Qiu J G, et al. Biodegradation of nicotine by a novel strainPusillimonas[J]. Res Microbiol, 2015, 166(2): 67-71.
[27] Gong X W, Yang J K, Duan Y Q, et al. Isolation and characterization ofRhodococcussp. Y22 and its potential application to tobacco processing[J]. Res Microbiol, 2009, 160(3): 200-204.
[28] Jiang H J, Ma Y, Qiu G J, et al. Biodegradation of nicotine by a novel StrainShinellasp. HZN1 isolated from activated sludge[J]. J Environ Sci Health, 2011, 46(8): 703-708.
[29] Ma Y, Wei Y, Qiu J G, et al. Isolation, transposon mutagenesis, and characterization of the novel nicotine-degrading strainShinellasp. HZN7[J]. Appl Microbiol Biotechnol, 2014, 98(6): 2625-2636.
[30] 陈 晨, 麻广慧, 雷丽萍, 等. 尼古丁降解细菌5-28菌株的分离鉴定和降解特性[J]. 烟草科技, 2012, 298(5): 74-78.
[31] Wang M Z, Yang G Q, Wang X, et al. Nicotine degradation by two novel bacterial isolates ofAcinetobacter sp.TW andSphingomonassp. TY and their responses in the presence of neonicotinoid insecticides[J]. World J Microbiol Biotechnol, 2011, 27(7): 1633-1640.
[32] Zhu X Y, Wang W W, Xu P, et al. Complete genome sequence ofSphingomonassp. strain NIC1, an efficient nicotine-degrading bacterium[J]. Genome Announc, 2016, 4(4): e00666-16.
[33] Uchida S, Maeda S, Kisaki T. Conversion of nicotine into nornicotine andN-methylmysomine by fungi[J]. Agric Biol Chem, 1983, 47(9): 1949-1953.
[34] Sindelar R D, Rosasza J P, Barfknecht C F.N-demethylation of nicotine and reduction of nicotine-1′-N-oxide byMicrosporumgypseum[J]. Appl Environ Microbiol, 1979, 38(5): 836-839.
[35] Meng X J, Lu L L, Gu G F, et al. A novel pathway for nicotine degradation byAspergillusoryzae112822 isolated from tobacco leaves[J]. Res Microbiol, 2010, 161(7): 626-633.
[36] Grether-Beck S, Igloi G L, Pust S, et al. Structural analysis and molybdenum-dependent expression of the pAO1-encoded nicotine dehydrogenase genes ofArthrobacternicotinovorans[J]. Mol Microbiol, 1994, 13(5): 929-36.
[37] Schenk S, Hoelz A, Krass B, et al. Gene structures and properties of enzymes of the plasmid-encoded nicotine catabolism ofArthrobacternicotinovorans[J]. J Mol Biol, 1998, 284(5): 1323-1329.
[38] Chiribau B C, Sandu C, Fraaije M, et al. A novelγ-N-methylaminobutyrate demethylating oxidase involved in catabolism of the tobacco alkaloid nicotine byArthrobacternicotinovoranspAO1[J]. FEBS J, 2004, 271(23/24): 4677-4684.
[39] Qiu J, Ma Y, Wen Y, et al. Functional identification of two novel genes fromPseudomonassp. strain HZN6 involved in the catabolism of nicotine[J]. Appl Environ Microbiol, 2012, 78(7): 2154-2160.
[40] Wang M, Yang G, Min H, et al. A novel nicotine catabolic plasmid pMH1 inPseudomonassp. strain HF-1 [J]. Can J Microbio, 2009, 55(3): 228-233.
[41] Jiang H J, Ma Y, Qiu G J, et al. Biodegradation of nicotine by a novel StrainShinellasp. HZN1 isolated from activated sludge[J]. J Environ Sci Health B, 2011, 46(8): 703-708.
[42] Wei H, Lei L, Liu S, et al. PanB is involved in nicotine metabolism inPseudomonasputida[J]. Int Biodeter Biodegr, 2009, 63(8): 988-99.
[43] Raman G, Mohan K N, Manohar V, et al. Biodegradation of nicotine by a novel nicotine-degrading bacterium,PseudomonasplecoglossicidaTND35 and its new biotransformation intermediates[J]. Biodegradation, 2014, 25(1): 95-107.
[44] Geiss V L, Gregory C F, Newton R P, et al. Process for reduction of nicotine content of tobacco by microbial treatment: US, US4140136[P]. 1979-02-20.
[45] Gravely L E, Geiss V L, Gregory C F. Process for reduction of nitrate and nicotine content of tobacco by microbial treatment: US, US4557280[P]. 1985-12-10.
[46] Wang S N, Xu P, Tang H Z, et al. ″Green″ route to 6-hydroxy-3-succinoyl-pyridine from (S)-nicotine of tobacco waste by whole cells of aPseudomonassp.[J]. Environ Sci Technol, 2005, 39(17): 6877-6880.
[47] Xue S, Schlosburg J E, Janda K D. A new strategy for smoking cessation: characterization of a bacterial enzyme for the degradation of nicotine[J]. J Am Che Soc, 2015, 137(32): 10136-10139.
ResearchDevelopmentofNicotineDegradingMechanisminMicroorganismsandTheirApplications
LiXiaohua,MeiFeng,KongWen,LiYang,MaTingting,PiTing
(Key Lab for Biotechnology of the State Ethnic Affairs Commission,College of Life Sciences, South-Central University for Nationalities, Wuhan 430074, China)
To learn about the most promising way of biodegradation to eliminate environmental nicotine pollution,this article gave an overview about the development of nicotine degrading microorganisms, degradation pathways (demethylation pathway in fungi, pyridine and pyrrolidine pathway in gram-positive bacteria, and variant pathway of pyridine and pyrrolidine in gram-negative bacteria), degradation gene clusters and their application in environmental protection and pharmacology, which may serve as a reference to guide the further study and application of nicotine degrading microorganisms.
nicotine; microbial degradation; metabolic pathway; application
2016-10-25
李晓华(1968-),男,博士,教授,研究方向:微生物学,E-mail:xhli4451@sina.com
国家自然科学基金资助项目(31070087,30570046);湖北省自然科学基金重点资助项目(2011CDA079,2008CDB076) ;中央高校基本科研业务费专项资金资助项目(YCZW15104,CZW16005)
Q939
A
1672-4321(2017)04-0027-09

