贵州省3个地理种群大鲵的遗传多样性及遗传结构
吴俣学,谢巧雄,姚俊杰,崔 巍,周红霞,李晓林
( 1.贵州大学 动物科学学院 水产科学系,高原山地动物遗传育种与繁殖教育部重点实验室, 贵州 贵阳 550025; 2.贵州省水产技术推广站,贵州 贵阳 550025 )
贵州省3个地理种群大鲵的遗传多样性及遗传结构
吴俣学1,谢巧雄2,姚俊杰1,崔 巍2,周红霞1,李晓林1
( 1.贵州大学 动物科学学院 水产科学系,高原山地动物遗传育种与繁殖教育部重点实验室, 贵州 贵阳 550025; 2.贵州省水产技术推广站,贵州 贵阳 550025 )
测定了贵州省贵定县、松桃县和江口县共36尾大鲵的mtDNA D-loop区部分序列,探究贵州省内不同地理种群大鲵的遗传多样性及遗传结构。结果显示,36个序列的碱基平均组成为T(33.8%)、C(22.2%)、A(30.0%)、G(14.0%),其中T的含量最高,C的含量最低;A+T含量(63.8%)显著高于G+C含量(36.2%)。江口种群核苷酸多样性指数为0.00551,贵定种群及松桃种群的核苷酸多样性指数均为0。3个地理种群相比,江口种群大鲵遗传多样性水平较高。3个种群的平均遗传距离为0.00436,其中贵定种群与江口种群达到了种群的分化水平(P<0.01)。遗传结构分析结果表明,贵定种群先与松桃种群聚为一支,再与江口种群聚为一支。3个种群大鲵的整体遗传多样性水平低,遗传分化程度也低。
贵州省;大鲵;遗传多样性;遗传结构
大鲵(Andriasdauidianus)曾在中国广泛分布,主要分布区包括长江、黄河及珠江流域的中上游地区及其支流的山溪河流,遍及华中、华北、华南和西南等17个省市地区[1]。随着中国大鲵资源锐减,其分布区极大缩小,总体上带状和片状的分布区已不复存在,转变为不连续的点状分布格局,大鲵生境岛屿化严重[2-3]。中国大鲵的濒危处境引起了众多学者的关注。2003年,Peng等[4]对中国大鲵线粒体的基因组进行了测序,确定其长度为16 503 bp。同年,林茂等[5]对广东珠海某养殖场的大鲵亲本及其子二代进行了RAPD分析,亲本与子二代的平均遗传距离分别为0.0497和0.0166,Shannon多样性指数分别为0.237和0.078,表明子二代遗传多样性水平低于亲代。2005年,陶峰勇等[6]通过对广西、湖南、陕西、河南等地的大鲵进行遗传学研究,测得大鲵的mtDNA D-loop基因全序列长度为771 bp。2006年,方耀林等[7]对汉水流域大鲵的研究显示,单倍型多样性指数和核苷酸多样性指数分别为0.8230和0.00446,结论是该种群大鲵的遗传多样性水平低;之后,Murphy等[8]对安徽等地大鲵的mtDNA Cytb和ATP-6基因序列的分析也得到相同结果。2008年,方耀林等[9]对长江中上游和汉水流域野生大鲵驯养群体及其人工繁殖子代mtDNA D-loop区进行了研究,结果显示野生亲本的单倍型多样性及核苷酸多样性均略高于子代,子代的遗传多样性有所下降。孟彦等[10]发现人工养殖群体存在较大的等位基因丢失现象,且遗传多样性水平低于野生群体,也说明人工养殖导致大鲵群体的遗传多样性水平下降。2011年,杨丽萍等[11]采用AFLP技术对四川、贵州、湖北、陕西及河南5个野生大鲵种群的遗传多样性和遗传分化水平进行评估,认为中国大鲵遗传多样性绝大部分位于种群内,种群间遗传分化较弱。
贵州是我国大鲵的主要优生区之一,全省各市、州均有大鲵分布。黔东武陵山区曾是中国大鲵的主要产区,各处溪河中经常可见大鲵。1980年以前铜仁市的年捕获量3000~3500 kg;1986年全区捕获量仅有1000~1500 kg[12]。由于生态环境的破坏和人为影响,贵州大鲵资源也急剧下降。贵州省开展大鲵的人工养殖二十多年来,在人工繁殖及规模化繁育技术方面取得突破性进展,种群数量也得到一定的恢复。由于对大鲵的遗传背景缺乏了解,本研究对贵州省江口县、松桃县及贵定县3个地理种群大鲵的遗传多样性进行研究,测定和分析线粒体DNA控制区序列,检测其遗传多样性水平,分析其遗传结构,旨在为贵州大鲵种质资源的保护、种群复壮以及大鲵养殖业发展提供一定的基础资料。
1 材料与方法
1.1 材料
共采集到野生大鲵36尾,其中来自贵州省贵定县(N 26°23′34.35″、E 107°18′26.33″)6尾、江口县(N 27°46′27.51″、E 108°47′20.74″)20尾和松桃县(N 28°08′35.75″、E 108°52′9.84″)10尾。
采集大鲵新鲜的蜕皮,置于1.5 mL离心管中-20 ℃保存备用。用康为通用型柱式基因组提取试剂盒提取DNA,得到的DNA于-20 ℃保存备用。
1.2 PCR扩增
PCR引物根据大鲵mtDNA控制区(Genbank登录号:NC-004926)两端的tRNA基因序列设计,DUP:5′-TTTGGTGCCCTTCTGATT-3′,DDN:5′-CGTTGGGTAGGAAGAGTTATTGT-3′。
PCR反应在PTC-200型PCR仪(TECHNE公司)上进行。反应体系为40 μL:上下游引物各2 μL;模板DNA 4 μL,MIX 20 μL,加无菌去离子水至40 μL。反应程序为:95 ℃预变性3 min;94 ℃变性45 s,56 ℃复性45 s,72 ℃延伸1 min,并进行35个循环;72 ℃延伸10 min。
1.3 目的条带检测及测序
PCR产物用1.5%琼脂糖凝胶电泳检测,在DYY-Ⅲ32型平板电泳槽上进行。利用未加模板DNA的反应液作为空白对照,以检查是否有污染存在。点样量为5 μL,电压为100 V,时间35~40 min。PCR所得产物送上海英潍捷基公司进行正反双向测序。
1.4 数据分析
采用ClustalX软件将测序结果进行对位排序。用Mega 4.0软件中的Kimura双参数法计算各种群间的遗传距离;并使用邻近距离法及最大简约法构建系统进化树,同时应用自举检验估计系统树中节点的置信度。用DnaSP软件计算各地理种群的核苷酸多样性、单倍型多样性。
2 结 果
2.1 贵州3个地理种群mtDNA D-loop的序列特征
对贵州36尾大鲵的D-loop区532 bp序列分析显示,36个序列的碱基组成平均为T(33.8%)、C(22.2%)、A(30.0%)、G(14.0%),其中T的含量最高,C的含量最低;A+T含量63.8%,显著高于G+C含量(36.2%),表现出碱基组成偏向性(表1)。保守位点519个,约占核苷酸总数的97.56%;变异位点12个;简约信息位点5个;单态突变位点7个;其中转换位点8个,颠换位点4个,插入或缺失位点2个。
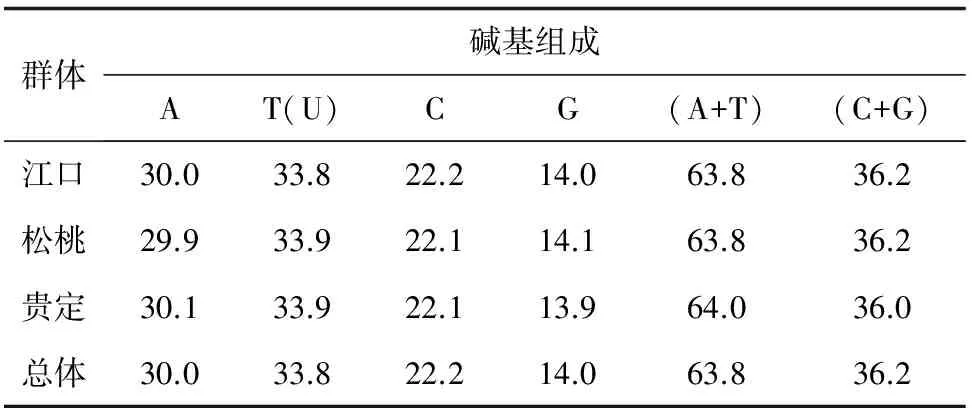
表1 大鲵各群体线粒体D-Loop区碱基组成 %
2.2 贵州3个地理种群mtDNA D-loop的遗传多样性
3个种群的总体平均核苷酸多样性指数为0.00435;单倍型多样性指数为0.7365(表2)。36尾个体分6种单倍型,3个群体间没有共享的单倍型,Hap 1、Hap 2、Hap 5、Hap 6为江口种群特有(表3)。其中单倍型频率最大的是Hap 4,有10个个体,占33.33%,其次是Hap 1,有10个个体,占27.78%。贵州3个地理种群各单倍型的平均遗传距离为0.00809。
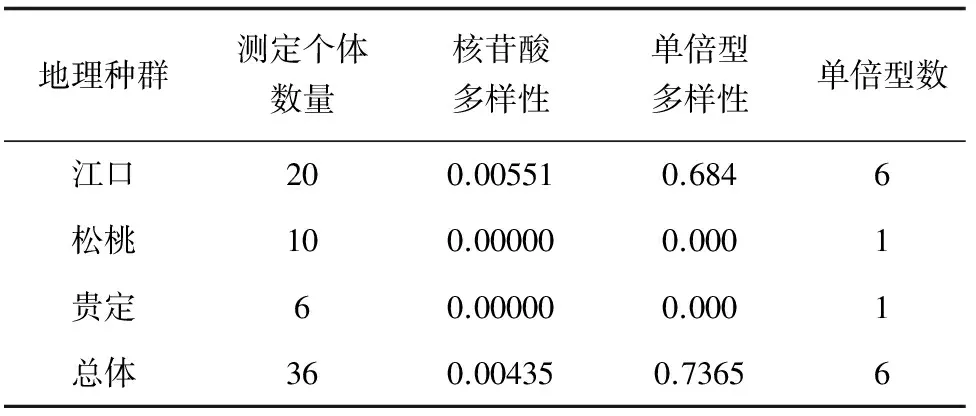
表2 贵州3个地理种群大鲵的核苷酸多样性、单倍型多样性和单倍型数
2.3 贵州3个地理种群的遗传结构
3个种群间的遗传距离见表4,江口种群与松桃种群间的遗传距离达到0.00473,贵定种群与江口种群间的遗传距离达到0.00531,贵定种群与松桃种群间的遗传距离为0.00189。以隐鳃鲵(Cryptobranchusalleganiensis)的同源序列(GenBank登录号:AB445803.1)为外群序列,构建了邻近距离树(图1)和最大简约树(图2)。两图中各节点的布展值均在50%以上,最高98%。观察发现,两图聚类趋势一致,Hap 4、Hap 5聚为一支,Hap 1、Hap 2聚为一支,Hap 3与Hap 4、Hap 5的距离较近。对所有36个大鲵个体进行邻近距离法聚类分析(图3),贵定、松桃群体能各自聚为一个群体,江口种群的个体则在各分支都有分布。
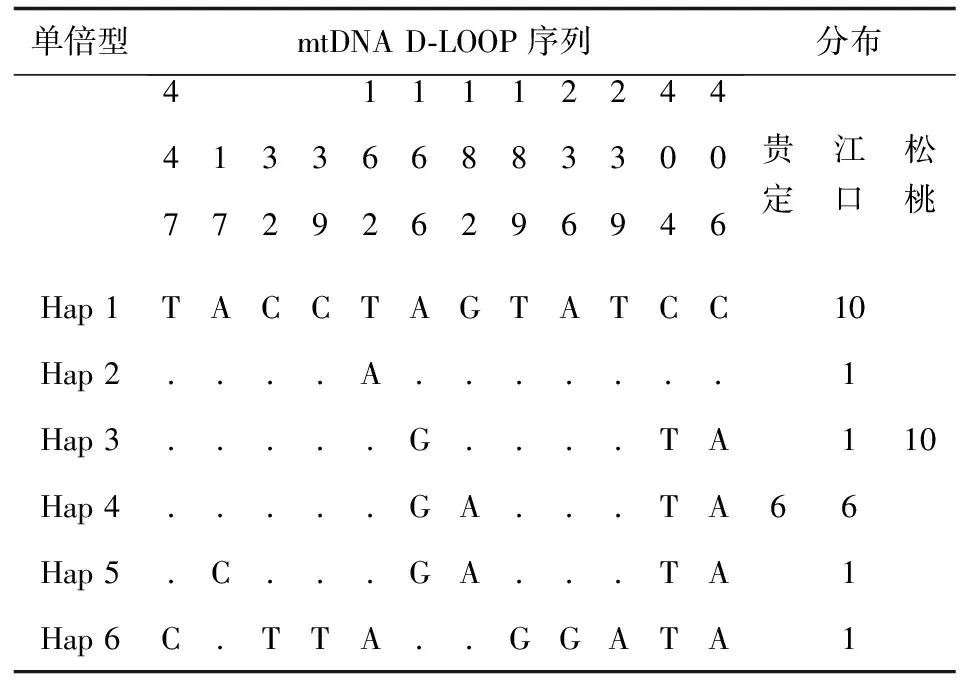
表3 各单倍型多态性位点分布及单倍型分布
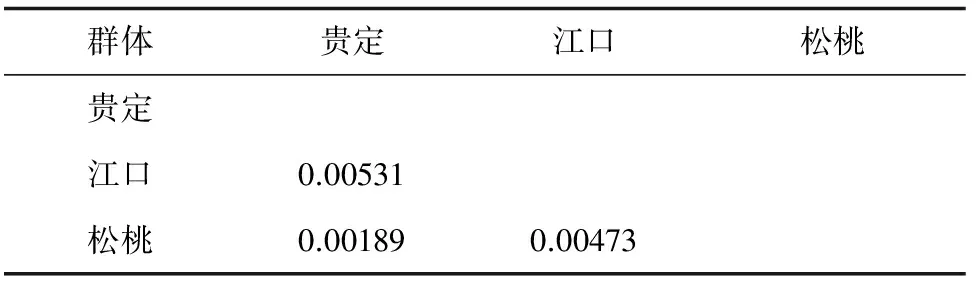
表4 各种群间遗传距离
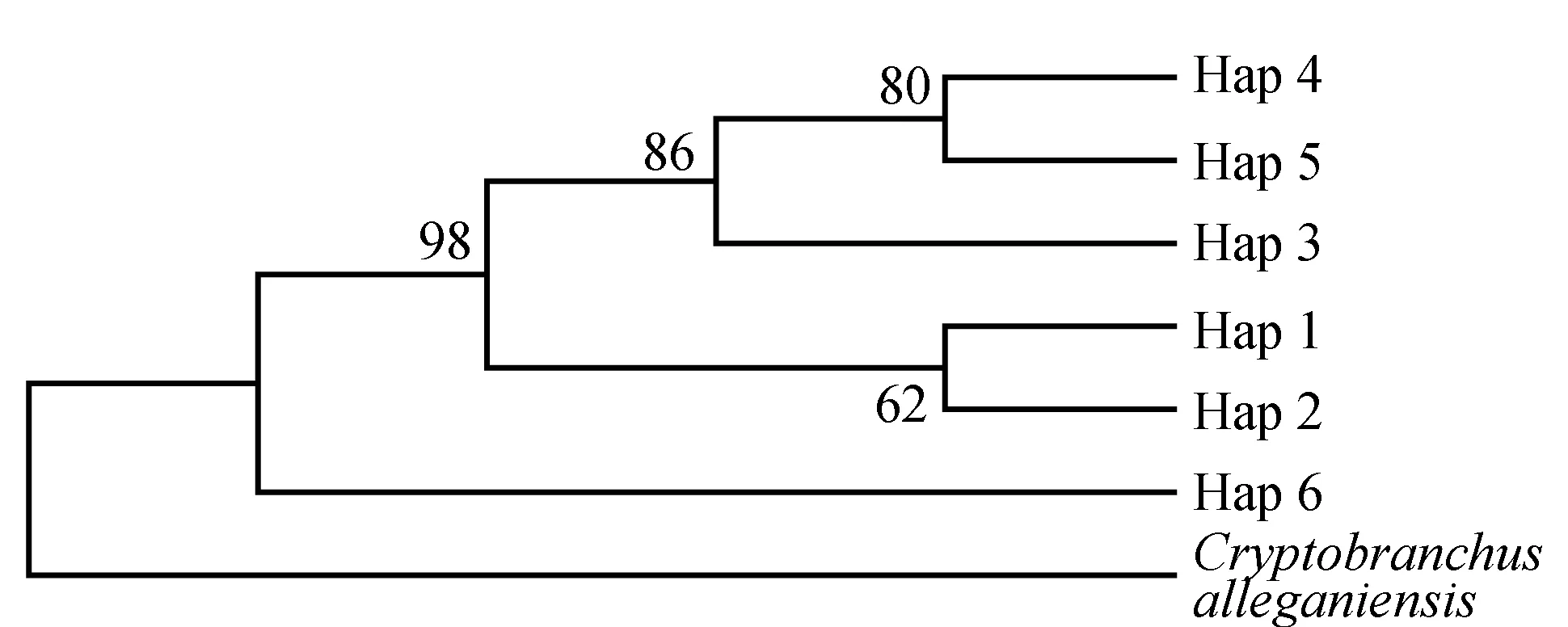
图1 大鲵单倍型邻近距离树以隐鳃鲵(GenBank登录号:AB445803.1)作外群.
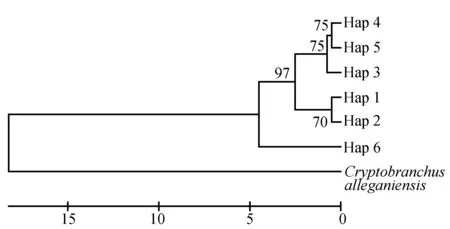
图2 大鲵单倍型最大简约树以隐鳃鲵(GenBank登录号:AB445803.1)作外群.
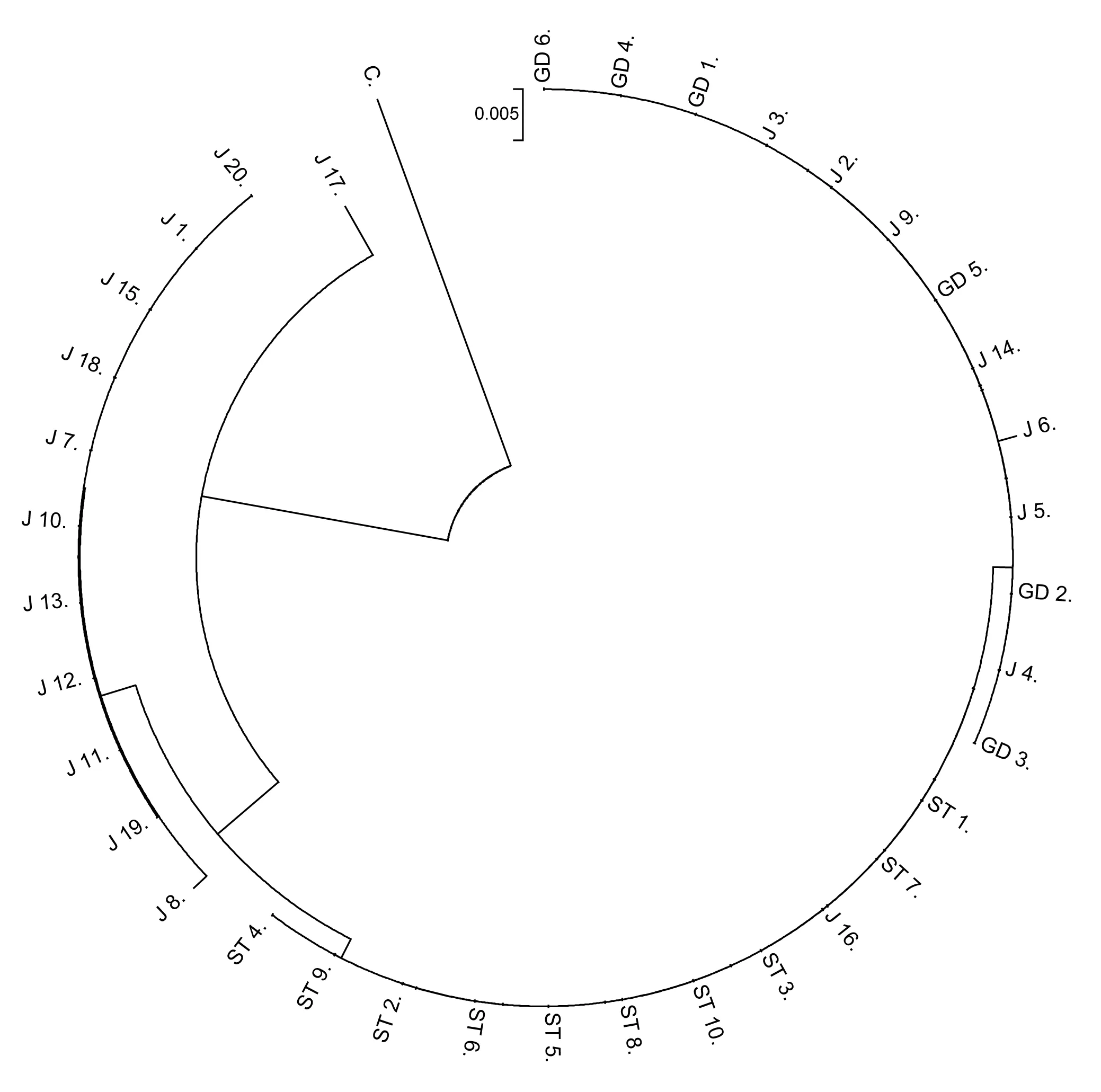
图3 所有个体邻近距离聚类C(隐鳃鲵,GenBank登录号:AB445803.1)作外群.
3 讨 论
3.1 贵州大鲵mtDNA D-loop区的碱基组成
贵州3个种群共36尾大鲵的线粒体DNA D-loop区的平均碱基组成为T(33.8%)、C(22.2%)、A(30.0%)、G(14.0%),与脊椎动物mtDNA碱基组成相似[13-14],体现在(A+T)>(G+C);其中G含量显著低于其他碱基含量,表现出明显的反G偏倚,体现了线粒体DNA的碱基组成特点[15]。两栖类线粒体DNA D-loop区已有的研究结果显示,挂榜山小鲵(Hynobiusguabangshanensis)G+C(33.4%)[16]、新疆北鲵(Ranodonsibiricus)G+C(33.4%)[17]、尾斑瘰螈(Paramesotritoncaudopunclatus)G+C(36.4%)[18]、贵州疣螈(Tylototritonkweichowensis)G+C(37.9%)[19],本试验所测的贵州大鲵线粒体DNA D-loop区G+C含量是36.2%,高于小鲵科的小鲵和新疆北鲵,略低于蝾螈科的尾斑瘰螈和疣螈,但大致符合两栖类线粒体DNA D-loop序列的碱基组成特点。
贵州大鲵的碱基组成中,G+C(36.2%)含量比汉水流域大鲵(35.2%)高[7],这可能与所分析的D-loop区的片段长度有关,本研究分析了大鲵D-loop区的532 bp序列,另一研究中则分析了汉水流域大鲵D-loop区的757 bp序列。除此之外,碱基的替换和变异速率也会影响碱基组成的分析。
3.2 贵州3个种群大鲵的遗传多样性
已有对长江流域、黄河流域、珠江流域和汉水流域大鲵种群的研究表明,遗传多样性均处于较低水平[6-7]。本研究中贵州省3个地理种群大鲵整体遗传多样性(核昔酸多样性=0.00435,具6种单倍型)也处于较低水平相比,只是相对来说江口种群大鲵的遗传多样性较丰富(核昔酸多样性=0.00551,具6种单倍型),贵定和松桃种群大鲵的遗传多样性则极度贫乏(核昔酸多样性=0,只1种单倍型)。而杨丽萍等[10]采用AFLP标记对四川、贵州、湖北、陕西及河南5个野生大鲵种群的遗传多样性和遗传分化水平进行的评估显示大鲵的遗传多样性高,孟彦等[10,21]采用微卫星技术对不同群体大鲵的分析显示其遗传多样性比较丰富。这与所用的遗传多样性分析方法及分析的DNA片段有一定关系,AFLP标记可获得丰富的多态位点[21],微卫星标记同样具有较丰富的多态性[20],且其研究分析的是大鲵整个基因组,本研究则只对mtDNA D-loop区进行分析,所获的多样性信息含量相对少很多。大鲵是两栖类动物,其线粒体DNA为母性遗传。相对来说,线粒体DNA呈母系遗传,几乎无重组,比核基因有更高的变异[22],较二者更适合作为大鲵遗传多样性分析的标记。而D-loop区为非编码区,保留突变的可能性大,且是mtDNA中进化速率最快的区域[23],适合种以下遗传多样性的研究。mtDNA D-loop区作为遗传学分子标记已广泛应用于研究许多物种的遗传管理、濒危物种的保护、种群结构和进化史等方面。
另一方面,研究的样品数量也可能影响分析的结果,本研究中贵定种群与松桃种群的样本量不到江口种群的一半。目前贵州境内能找到纯种野生大鲵的地方少之又少,有条件应增加研究对象,更进一步地探究贵州大鲵的遗传多样性。
3.3 贵州3个种群大鲵的遗传分化及遗传结构
根据Shaklee等[24]提出的属、种和种群三级水平上的遗传距离分别为0.90、0.30及0.05的分类依据,本研究中3个大鲵种群间均未达到种群分化的水平。贵州3个种群的6种单倍型间的平均遗传距离为0.00809(P<0.01),也说明3个种群间的遗传分化程度低。贵州省这3个种群的亲缘关系表现为贵定种群先与松桃种群聚为一支,然后再与江口种群聚为一支。对所有个体的邻近距离聚类结果同样显示贵定种群与松桃种群亲缘关系较近,与江口种群的亲缘关系较远。对3个种群间遗传距离的分析也得出相同结论。
贵州3个种群的地理分布中,江口种群与松桃种群较近,与贵定种群较远。分析结果却出现了地理位置近,遗传距离相对却远的情况,且地理位置较远的松桃种群和贵定种群亲缘关系却较近。大鲵对水的依赖性强,而2006年贵州省贵定县岩下大鲵资源的调查显示,岩下野生大鲵种群栖息地已经由地下(洞穴)与地面水域广布缩小到以地下水域为主[25]。贵州独特的喀斯特地形对省内不同地域的大鲵种群形成了小范围的地理隔离,使江口种群与松桃种群形成实际上的空间隔离,而贵定种群与松桃种群可能通过地下暗河还有一定基因交流。另一方面,在长期的进化过程中,3个种群的大鲵种群分化受遗传多样性影响,江口种群因遗传多样性较丰富,使其积累了一定的分化水平,而贵定种群和松桃种群低水平的遗传多样性也限制了2个种群分化可能,亲缘关系也就相对较近。
[1] 叶昌媛. 中国珍稀及经济两栖动物[M]. 成都:四川科学技术出版社, 1993.
[2] 章克家, 王小明, 吴巍,等. 大鲵保护生物学及其研究进展[J]. 生物多样性, 2002(3):291-297.
[3] 罗庆华, 刘英, 张立云,等. 湖南张家界市大鲵资源调查[J]. 四川动物, 2009, 28(3):422-426.
[4] Peng Z, Chen Y Q, Liu Y F, et al. The complete mitochondrial genome of the Chinese giant salamander,Andriasdavidianus(Amphibia: Caudata)[J]. Gene,2003,311(1):93-98.
[5] 林茂, 黄景, 李正,等. 大鲵野生亲代与人工繁育子二代的随机扩增多态DNA分析[J]. 上海水产大学学报, 2003,12(增刊):20-23.
[6] 陶峰勇, 王小明, 郑合勋,等. 中国大鲵四种群的遗传结构和地理分化[J]. 动物学研究, 2005, 26(2):162-167.
[7] 方耀林, 张燕, 杨焱清,等. 大鲵遗传多样性分析[J]. 淡水渔业, 2006, 36(6):8-11.
[8] Murphy R W, Fu J Z, Upton D E. Genetic variability among endangered Chinese giant salamander,Andriasdavidianus[J].Mol Ecol, 2000, 9(10):1539-1547.
[9] 方耀林, 张燕, 肖汉兵,等. 野生大鲵及其人工繁殖后代的遗传多样性分析[J]. 水生生物学报, 2008, 32(5):783-786.
[10] 孟彦, 杨焱清, 张燕,等. 野生和养殖大鲵群体遗传多样性的微卫星分析[J]. 生物多样性, 2008, 16(6):533-538.
[11] 杨丽萍, 蒙子宁, 刘晓春,等. 中国大鲵5个野生种群的AFLP分析[J]. 中山大学学报:自然科学版, 2011, 50(2):99-104.
[12] 陈广城. 黔东武陵山区大鲵的资源现状[J]. 淡水渔业, 1988(1):33-33.
[13] Broughton R E, Milam J E, Roe B A. The complete sequence of the zebrafish (Daniorerio) mitochondrial genome and evolutionary patterns in vertebrate mitochondrial DNA[J]. Genome Research, 2001, 11(11):1958- 1967.
[14] 冯慧, 黄原, 任轶,等. 陕西省林麝mtDNA D-loop区序列结构和种群遗传多样性[J]. 生态学报, 2014, 34(20):5887-5895.
[15] Wolstenholme D R. Animal mitochondrial DNA: structure and evolution[J]. International Review of Cytology, 1992, 141(6):173-216.
[16] 任巍, 牛艳东, 周毅,等. 挂榜山小鲵线粒体DNA COⅠ基因及D-loop区序列研究[J]. 生命科学研究, 2010(4):327-330.
[17] 袁亮, 李伟业, 叶小芳,等. 基于线粒体DNA控制区的新疆北鲵种群遗传多样性分析[J]. 厦门大学学报:自然科学版, 2015, 54(2):194-198.
[18] 陈光照, 肖羽, 陈学平,等. 尾斑瘰螈种群的D-loop区遗传多样性分析[J]. 动物学杂志, 2011, 46(3):55-63.
[19] 肖羽, 陈光照, 谷晓明. 贵州疣螈D-loop区和tRNAPhe序列变异及群体遗传多样性[J]. 动物学杂志, 2010, 45(3):21-29.
[20] 郭文韬. 大鲵遗传多样性及皮肤附属物特性研究[D]. 武汉:华中科技大学, 2013.
[21] Prior K A, Gibbs H L, Weatherhead P J. Population genetic structure in the black rat snake: implications for management[J]. Conservation Biology, 1997, 11(5):1147-1158.
[22] Cann R L, Brown W M, Wilson A C. Polymorphic sites and the mechanism of evolution in human mitochondrial DNA[J]. Genetics, 1984, 106(3):479-499.
[23] Sbisà E, Tanzariello F, Reyes A, et al. Mammalian mitochondrial D-loop region structural analysis: identification of new conserved sequences and their functional and evolutionary implications[J]. Gene, 1997, 205(1/2):209.
[24] Shaklee J B, Tamaru C S, Waples R S. Speciation and evolution of marine fishes studied by the electrophoretic analysis of proteins[J]. Pacificence, 1982, 36(2):141-157.
[25] 粟海军, 喻理飞, 马建章. 贵州岩下自然保护区的野生大鲵资源现状及历史动态[J]. 长江流域资源与环境, 2009, 18(7):652-657.
GeneticDiversityandGeneticStructureofGiantSalamanderfromThreeGeographicalPopulationsinGuizhouProvince
WU Yuxue1, XIE Qiaoxiong2, YAO Junjie1, CUI Wei2, ZHOU Hongxia1, LI Xiaolin1
( 1.Breeding and Reproduction in the Plateau Mountainous Region, Ministry of Education, Key Laboratory of Animal Genetics, Department of Aquatic Science, Guizhou University,Guiyang 550025,China;2.Aquaculture Technology Extending Stations in Guizhou Province,Guiyang 550025,China )
A research on mtDNA D-loop region partial sequence was studied in 36 giant salamandersAndriasdaudianussamples collected from Guiding county, Songtao county and Jiangkou county in Guizhou province. The results showed that the average contents of the four bases were 33.8% in T,22.2% in C,30.0% in A and G in 14.0%, with the maximal T content, and the minimal C content; A+T content was 63.8%, significantly higher than that in G+C content (36.2%). The nucleotide diversity(π) of the Jiangkou populations were 0.00551, and the nucleotide diversity(π) of the Guiding and Songtao population was 0. Compared with the three geographical populations, the genetic diversity level of giant salamander in Jiangkou county population was higher. The average genetic distance of three populations was 0.00436, and the Guiding and the Jiangkou population reached the population differentiation level(P<0.01). Analysis of the genetic structure indicated that Guiding and Songtao populations were clustered for first, and then clustered with Jiangkou popultion. The overall genetic diversity of these populations of giant salamander was low, and the genetic differentiation of these populations was low.
Guizhou province;Andriasdaudianus; genetic diversity; genetic structure
10.16378/j.cnki.1003-1111.2017.02.016
2016-03-31;
2016-06-29.
贵州省农委资助项目(黔财农【2014】233);贵州省水产养殖特色专业项目(80113610201);贵州大学创新基金资助项目(研农2016023).
吴俣学(1992-),男,硕士研究生;研究方向:水生动物繁殖与发育生物学. E-mail:745893505@qq.com. 通讯作者: 姚俊杰(1968-),男,教授,博士,硕士生导师;研究方向:水生动物繁殖与发育生物学. E-mail:junjieyao@163.com.
S917.4
A
1003-1111(2017)02-0207-05

