一种面向临床领域时序知识图谱的链接预测模型
陈德华 殷苏娜 乐嘉锦 王 梅 潘 乔 朱立峰
1(东华大学计算机科学与技术学院 上海 201600) 2(上海交通大学医学院附属瑞金医院 上海 200025)
一种面向临床领域时序知识图谱的链接预测模型
陈德华1殷苏娜1乐嘉锦1王 梅1潘 乔1朱立峰2
1(东华大学计算机科学与技术学院 上海 201600)2(上海交通大学医学院附属瑞金医院 上海 200025)
(chendehua@dhu.edu.cn)
知识图谱(knowledge graph)链接预测可以解决知识图谱中缺失信息的发现和还原,是目前知识图谱领域的研究热点.传统的知识图谱链接预测方法大多面向静态的数据,并不适用于具有动态变化特性的时序知识图谱.时序知识图谱广泛存在于不同领域中,以临床医学领域为例,糖尿病作为一种典型的慢性病,其病程是一个疾病缓慢发展演化的过程.因此,在临床医学时序知识图谱上进行临床意义的链接预测,比如预测糖尿病的并发症,则需要考虑糖尿病病程发展随时间变化的时序特性,这也为传统的知识图谱链接预测方法带来巨大挑战.为此,结合临床医学事实知识的时序特性,提出一种基于LSTM序列增量学习的临床领域时序知识图谱链接预测模型.该模型结合LSTM长短期记忆单元递归神经网络在序列学习上的优势,通过构建基于LSTM的序列增量学习层,以端到端的方式提取时序知识图谱中的三元组时序特征,从而实现对时序知识图谱的链接预测.通过在糖尿病时序知识图谱上的实验,验证了模型的高效性、可用性及稳定性.
时序知识图谱;知识图谱链接预测;转换模型TransR;长短期记忆网络;增量学习
知识图谱(knowledge graph)是表示知识的一种新方法,属于语义网络范畴,用于描述真实世界中存在的各种实体和概念以及这些实体、概念之间的关联关系,捕捉并呈现特定领域概念之间的语义关系[1].近年来知识图谱在医学领域也逐渐得到重视和关注,国内外均开展了医学领域知识图谱相关研究.国外有牛津大学创建的用于药学的LynxKB知识图谱[2]以及由日本东北大学将传统关系型数据库融合于知识图谱中进行基因研究[3];与此同时,国内医疗信息学领域也提出了多种医学知识图谱,包括中国中医科学院中医药信息研究所构建的中医药知识图谱[4]、基于知识图谱的基因组流行病学可视化分析[5]等.然而,这些医学知识图谱的知识来源主要是公开的医学文献,较少涉及医院的实际电子病历(electronic medical record, EMR)数据.
众所周知,EMR电子病历[6]记录着患者在医疗活动中产生的各种临床事实数据,蕴含着丰富的临床事实知识,主要体现为各种医学实体如患者实体、药物实体、诊断实体等,以及医学实体之间存在的各种联系.本文利用知识图谱表示EMR中临床事实知识,构建基于EMR的临床领域时序知识图谱,刻画临床数据中存在的实体和概念,提供具体且丰富的语义和时序信息,以便更准确地揭示实体之间的内在联系,从而避免来自不同数据源信息的语义异构.
知识图谱链接预测[7]是知识图谱学习与推理的重要应用,其主要任务是对知识图谱中实体间可能存在的关系进行预测,实现知识图谱中缺失信息的发现和还原[8].由于实际电子病历数据普遍存在数据质量不高的特点,使得基于EMR的临床领域知识图谱中可能存在着一些医学实体以及实体间关系的缺失,或者实体间存在错误的关系.通过对临床领域知识图谱的链接预测,能够将这些关系所补全或者纠正出错误的关系,从而获得更为完整和真实的知识图谱.
目前成熟的知识图谱链接预测包含张量分解模型[9]、NTN神经网络[10]、转换模型[11]等.然而这些预测模型都只在通用知识图谱上取得了不错的效果.通用知识图谱中大多为常识性知识,并不随时间而改变.与此相反,在临床领域中一般疾病的病程发展是一个缓慢演变的过程,可见临床事实知识具有时效性,包含大量时序知识.以糖尿病为例,在糖尿病患者临床诊治过程中,每次的血糖检查、糖化血红蛋白检查、用药情况、并发症诊断结果等均有时间的标记.这些临床事实知识可按照时间前后顺序,转换形成具有时序特性的临床领域时序知识图谱.但是,现有的知识图谱链接预测模型大多针对静态的数据,而未考虑到时序知识图谱中蕴含大量时序信息,无法对时序知识图谱做出准确的预测.
为解决上述问题,本文从医院实际的EMR数据出发,结合临床医生的经验与知识,建立临床领域时序知识图谱,并且提出了一种基于LSTM序列增量学习的临床领域时序知识图谱链接预测模型.该模型采取LSTM长短期记忆单元的递归神经网络的序列学习能力,并创建序列增量学习层对临床事实知识时序特征进行提取,同时通过端到端(end-to-end)的方式进行知识图谱三元组序列的增量学习过程,从而实现对临床领域时序知识图谱的链接预测.本文通过多种对比实验,从准确度、召回率和精准度等方面对增量LSTM新模型进行了评估验证;同时分析了新模型的时间复杂度,最终验证了新模型在时序知识图谱链接预测上具有较好的性能.
1 相关工作
近年来,业界陆续提出了多种不同的通用知识图谱,比如2012年Google公司推出Google Knowledge Graph[12],之后又提出了多种通用知识图谱,如FreeBase[13],DBPedia[14],WordNet[15]等.而对临床领域,国外较为流行的临床领域知识库有MorphoCol[16],Nursing KB[17]等,并基于此进行了临床决策诊断支持工作[18].国内探索了中医药知识图谱构建[4]方法以及基于医药知识图谱推理的辅助开药[19].之后,有关时间信息在知识图谱中的重要性被逐渐关注,比如对含有时间的知识进行知识图谱模型建立[20].
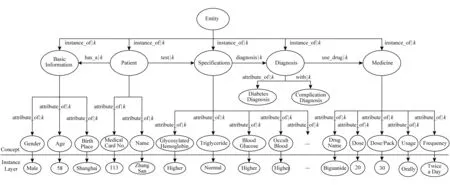
Fig. 1 Clinical domain temporal knowledge graph model图1 临床领域时序知识图谱模型
链接预测一直是知识图谱学习和推理的热点问题,许多研究者提出了不同的链接预测模型,用于学习和预测实体间存在的关系.现有成熟的知识图谱链接预测方法可分为3类:1)基于张量分解的知识图谱链接预测方法,包括Rescal、神经张量网络(NTN)等;2)基于向量转换模型的知识图谱链接预测方法,比如说TransE[21],TransH[22],TransR[23]等;3)以深度学习[24]为代表的知识图谱链接预测方法异军突起.比如文献[25]尝试使用深度神经网络进行通用知识图谱的链接预测.然而这些方法目前只适用于知识图谱的静态数据,还不适用于具有动态变化特性的时序知识图谱链接预测.
关于时间信息在知识图谱链接预测中的应用,文献[26]提出了基于TransE转换模型改进的TransE-TAE模型,通过对知识图谱中的时间信息分析,对关系作出了预测;文献[27]进而研究由时间导致的不确定性知识图谱上的预测模型.然而,大多数工作都集中在具有时效性的通用知识图谱中,缺乏对临床领域知识图谱中尤为突出的时序特征做研究.
2 临床领域时序知识图谱及其链接预测
2.1 临床领域时序知识图谱相关定义
临床领域时序知识图谱基于实际EMR数据构建而成,其形式化定义如下.
定义1. 临床领域时序知识图谱G.临床领域时序知识图谱为一张有向标签图Gt=(t0,te,E,R,τ),其中E为知识图谱的顶点集,用于表示实体集合;R为知识图谱的边集,用于表示事实关系集合;τ为E×E→R|k的函数,表示知识图谱中的所有元组.k表示在时间段[t0,te]按照时间前后排序的知识图谱三元组列表中,两实体之间存在第k次的关系R.举例来说,现有一个2015—2016年的临床领域时序知识图谱Gt=(2015-01-01,2016-01-01,E,R,τ),则病人张三的血糖检测三元组序列L(t0,te,τ)={(张三,血糖检查|第1次检查,正常),(张三,血糖检查|第2次检查,异常偏高),(张三,血糖检查|第3次检查,异常偏高),…}.
图1所示为临床领域时序知识图谱.由图1可见,临床领域时序知识图谱由概念层和实例层两部分组成.
其中概念层包括患者、疾病、检验指标、并发症及药品等实体类型.概念层中,不同实体类型之间存在概念层之间的关系.在图1所示的知识图谱中,实体概念层次可分为3个层级:一级实体概念包括基本信息、患者、检验报告、诊断和用药等;二级实体概念包括糖尿病诊断和并发症诊断;其他为三级实体概念.
在实例层中,每个实体都含有自己的属性以及属性值.例如“张三”是属于患者实体类,因此,将“张三”实例化为患者实体的姓名属性值,张三患者实体的医疗卡号“113”作为实例则对应于患者中的医疗卡号属性值.
2.2 时序链接预测相关定义
定义2. 时序链接预测.时序链接预测是指在临床领域时序知识图谱G中,通过对已知信息的分析,对图谱中2个实体E1和E2,预测出二者之间可能存在临床意义的关系R.
例1. 以图1所示的时序知识图谱为例,给出时序链接预测实例:由于糖尿病患者在就诊过程中有多次指标检测等,因此有如表1所示的糖尿病时序知识图谱中的三元组序列X(i)作为输入,经过链接预测,可以预测出该患者实体与眼病实体之间是否具有患有关系Y,即为输出.整个预测过程可以表示为
P={X,Y}={X(1),X(2),…,X(n),Y},
(1)
其中,X(i)描述有关该病人实体的各类关系所对应的三元组序列,可视为病人各方面的属性:
(2)
3 基于LSTM序列增量学习的时序链接预测
本节首先阐述临床领域时序知识图谱链接预测模型整体框架,然后具体阐述模型的细节,最后给出模型的训练过程.
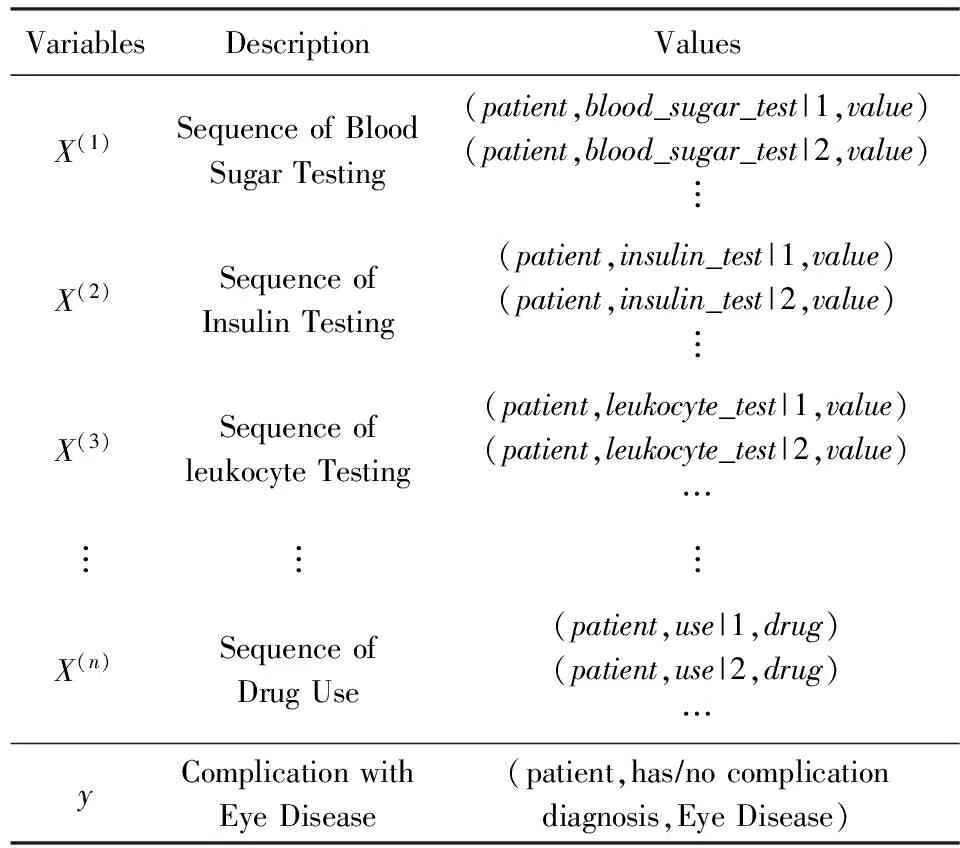
Table 1 Temporal Knowledge Base Link Prediction表1 临床领域时序知识图谱链接预测
3.1 模型的整体框架
本文提出基于LSTM序列增量学习的临床领域时序知识图谱链接预测模型,用来推理预测带时间的临床领域知识图谱中各个实体之间的链接.图2所示为模型结构图,一共包括4层:三元组向量化层(输入层)、LSTM序列增量学习层、序列特征组合层以及分类层(输出层).
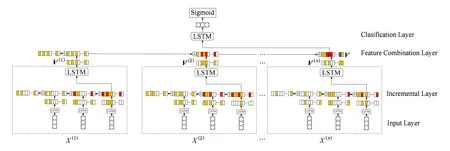
Fig. 2 Temporal link prediction model based on incremental LSTM sequence learning图2 基于LSTM序列增量学习的时序链接预测模型
模型的输入是临床领域时序知识图谱中的三元组序列,输出是实体之间的关系预测结果.模型训练过程主要是:在得到对应的序列化三元组x(n)之后,首先用TransR进行向量化作为输入层;其次输入到LSTM增量学习层,将得到的向量做增量计算;再将增量之后的向量V(n)输入到LSTM序列特征组合层计算得到V,最后进入分类层.
3.2 基于TransR的多语义三元组向量化
基于TransR的多语义三元组向量化层为时序链接预测模型的第1个层次.本层主要是采用TransR转换模型将临床领域时序知识图谱G中的三元组Ei,R,Ej嵌入到低维空间内.在临床领域时序知识图谱中存在多对多且语义不同的关系,比如多个患者实体和多种不同的检查指标实体之间都是检查关系,同时这些检查关系里有的是超声检查关系,有的是穿刺检查关系.TransR模型支持对不同实体拥有不同语义空间的处理,这符合临床数据中关系来自不同语义空间的特点;与此同时,在TransR中,首先将各个实体向量向关系空间中做投影,因此原来在实体空间中相似的实体就被区分开来了,从而在临床事实知识图谱中实现了对多对多关系两边不同实体的区分,并将实体和关系嵌入到低维向量.
TransR模型为了考虑不同语义空间,对于多对多关系有更精确的向量化表示,在TransE模型的基础上对实体向量向关系空间中进行投影,然后建立从头实体到尾实体的转换关系.
图3为TransR翻译模型运用在本文临床事实知识图谱上的一个简单例子:张三和上海2个实体通过在_born_in关系空间上做投影,从而嵌入到向量坐标中,两者间建立起被映射的_born_in向量转换关系.
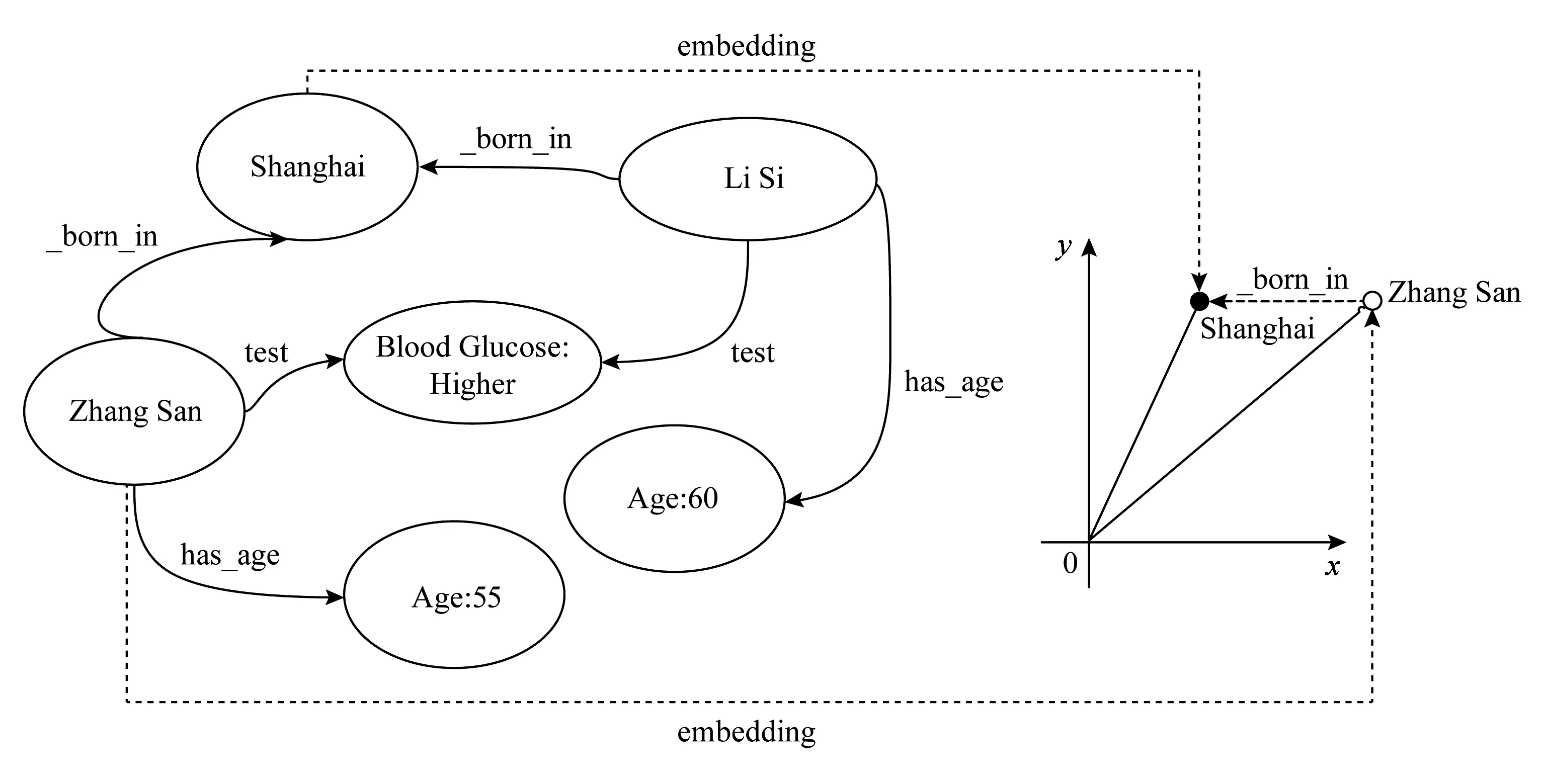
Fig. 3 Embeddings from Knowledge Graph图3 知识嵌入图
TransR中对于每个关系,都定义了一个投影矩阵Mr,将实体向量从实体空间投影到关系r的子空间,lh r和lh t表示为
lh r=lhMr,
lt r=ltMr.
(3)
对应的损失函数为
fr(h,t)=‖lhr+lr-ltr‖L1/L2.
(4)
因此,在TransR模型中,将每个实体都看作不同属性构成的,对于不同的关系,关注的是实体的不同属性;并且将有着不同语义空间的关系作出投影,从而区分多种语义,由此对临床领域知识图谱实现高效的向量化.
3.3 基于LSTM的三元组序列增量学习
基于LSTM的三元组序列增量学习层为时序链接预测模型的第2个层次.递归神经网络模型LSTM[28]是一种可以学习长期依赖信息的神经网络,其输入数据的形式记为f={Xn},n=t1,t2,…,tk该输入数据是带序列性质的数据向量.临床领域时序知识图谱通过TransR向量化后,输出为具有2个特点的三元组向量:1)临床数据在向量化后尽量不丢失原有的语义信息;2)向量化后输出的三元组按照时序排列,具有时序性.因此,本文将TransR后的三元组向量送入LSTM中,不仅保持了原有语义,同时也具有LSTM输入数据f的时序特征,从而适用于LSTM.所以,经过TransR后的语义三元组与LSTM的叠加增强作用,记忆单元可利用序列中的历史信息,从而能较充分且准确地挖掘序列之间的依赖信息.
目前,使用最广泛的LSTM单元有3个门:输入门、输出门和遗忘门,以保存历史信息.其中,输入门用于控制当前数据输入对记忆单元状态值的影响,遗忘门用于控制历史信息对当前记忆单元状态值的影响,通过计算得到记忆单元状态;输出门用于控制记忆单元状态值的输出.
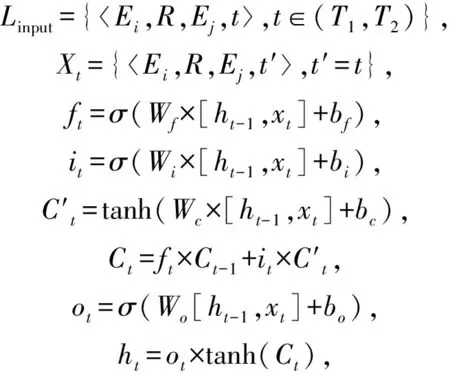
(5)
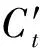
对于带时间序列的临床领域知识图谱,本文将LSTM改进为增量LSTM.该模型采用交叉熵函数作为损失函数,通过反向传播,对LSTM中的参数进行调整.
3.4 LSTM序列特征增量组合
LSTM序列特征增量组合为本文时序链接预测模型的第3个层次,在上下三元组序列中,采用增量形式代替简单的前后连接过程:当后一个向量与前一个向量在同一位置上有值时,即增量相加来强化特征,并将强化后的向量作为下一个的输入.图4所示为LSTM序列特征增量组合示意图.其中,每个长方形框对应向量中的每一位,颜色不同意味着每位的值不同.如果是白色,代表此向量在该位上无数据;若是黄色,代表此位置上有数据;若为红色,代表该位置是经过了增量相加后的数据.例如在时序数据中上一时间点的向量V(1)和目前向量V(2)的第1位都有数据,则需要对其进行增量相加,从而强化其特征,则该位置由黄色变成了红色.算法1所示为具体的特征增量组合过程描述.
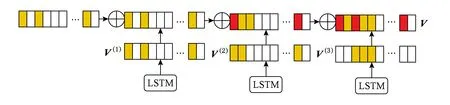
Fig. 4 Feature combination by incremental learning图4 通过增量进行特征组合
算法1. LSTM序列特征增量组合过程.
输入:LSTM的输出向量V(m);
输出:序列增量组合后的特征向量V.
V:特征组合层后的向量,V(m):序列增量层中第m个LSTM产生的向量.
①V(0)=V(1);
② fori=0 ton
③V(i)=V(i-1)+V(i);
④ endfor
⑤V=V(n).
3.5 时序链接预测模型的训练算法
经过上述3个层次处理之后,对每一类的三元组都通过增量计算进行上述的特征组合提取,直到得到最终的特征向量作为分类器的输入,整个训练过程采取反向传播进行调参.具体的时序链接预测模型的训练算法如算法2所示.
算法2. 时序链接预测模型训练过程.
输入:按照时间从前往后排序的三元组序列;
输出:0或1,1代表该三元组为正确事实,0则反之.
Ni t:使用批数据集训练的次数;
Dt r/Dt e:训练集/测试集;
M:序列增量层中LSTM的数量;
E:分类层中的损失值;
AEm:序列增量层中第m个LSTM的损失值;
S:批数据集的数量;
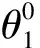
② fori=1 toNi t
③ forj=1 toS
④Dtr←GetMiniBatch(D);
⑤ form=1 toM
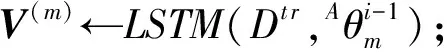
⑦V←Incremental(V,V(m));
⑧ endfor
⑨E←CLASSIFICATION_LSTM(V,
Cθi-1);
⑩ (Cθi,AE)←BACKWARD(E,Cθi-1);
4 实验结果与分析
4.1 实验数据
本文所用的数据来源于上海交通大学医学院附属瑞金医院实际EMR数据,该知识图谱由该院内分泌科近10年的EMR数据抽取而成,共有61 000个实体数、53种不同的关系类型、训练集规模为345 549个三元组.
为了验证本文所提的三元组序列增量学习模型预测的效果,本文在上述临床领域时序知识图谱数据集上采用10折交叉验证方法进行评估,故验证集和测试集规模均随机选取34 554个三元组.
4.2 对比模型
本文采取知识图谱中常用的6种推理预测模型作为对比模型:
1) 张量分解Rescal模型.Luo等人[29]采用张量分解的方法对临床健康数据进行分析,挖掘其隐藏知识.主要是将知识图谱中的三元组转换成张量Y,如果三元组h,R,t存在,则Yh rt=1,否则Yh rt=0.Rescal算法将分解为实体和关系表示,以此得到低维向量表示,通过矩阵分解计算对张量进行分解得到预测结果.
2) 张量神经网络(neural tensor network, NTN)模型.采用双线性张量取代传统神经网络中的线性变换层,通过将头、尾实体向量在不同的维度下的联系进行实体间预测.其中,对于每组三元组h,R,t,NTN对其都有一个评分函数:
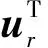
(6)
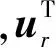
3) 转换模型TransE.TransE模型为转换模型的代表.将知识库中的关系看作是实体之间的某种平移向量,对于三元组h,R,t,TransE模型将lr表示关系r的向量,将lh和lt分别作为h和t的向量,lr可以作为lh和lt向量之间的平移,即将lr看作是lh和lt的转换.关系向量可以作为实体向量之间的平移,由此推测三元组的正确性.
4) 转换模型TransH.TransE模型在处理1-N,N-1,N-N复杂关系时,有着一定的局限性,TransH模型提出来对复杂关系局限性的解决方案.TransH模型将采用平移向量lr和超平面的法向量Wr来表示,从而进行复杂关系的推理预测.
5) 转换模型TransR.对不同语义空间中实体与关系的推理预测模型.具体请参见3.2节中的详细介绍.
6) 深度神经网络模型DNN.目前由Taheri等人提出的深度学习模型[25]成熟应用于ConceptNet知识库[30],该模型通过Word2Vec对知识库做向量化,接着通过双向LSTM联系上下文对向量做修正,最后通过深度神经网络(deep neural network, DNN)模型做分类预测.
4.3 评价指标
本文采用准确度(accuracy,AUC)、召回率(recall,R)和精确度(precision,P)作为模型评估的指标.
准确率指的是对于给定的测试数据集,模型正确预测的样本数与总样本数之比:
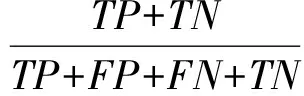
(7)
其中,TP是指被模型预测为正的正样本,TN是被模型预测为负的负样本,FP是被模型预测为正的负样本,FN是被模型预测为负的正样本.
同时,为了反映被正确判定的正例占总正例的比重,本文采取了召回率R作为评价指标,体现了模型对正样本的预测能力,召回率R越高,说明模型对正样本识别能力越强:
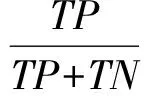
(8)
此外,精确度P体现了模型对负样本的区分能力,P越高,说明对负样本的区分能力越强.P指正确预测的正样本数占总正样本的比例:
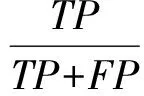
(9)
因此,本文还采用Precision-Recall(PR)图评估正负样本区分能力.同时,F1分数可以看作是准确率和召回率的加权平均.
本文分别对6种参考模型和新模型做AUC,R,F1-Score进行分类性能对比,对ROC,PR图进行图对比,同时也分析了本文增量LSTM模型中的参数选择对比以及各模型的时间复杂度.
4.4 结果分析与讨论
本节阐述对比实验结果以及对结果的分析.实验中将本文增量LSTM模型与其他参考模型相比较.
4.4.1 模型分类性能对比
首先,本文将本文模型与其他6种参考模型方法运用在临床领域时序知识图谱中,表2为这些方法的性能对比.
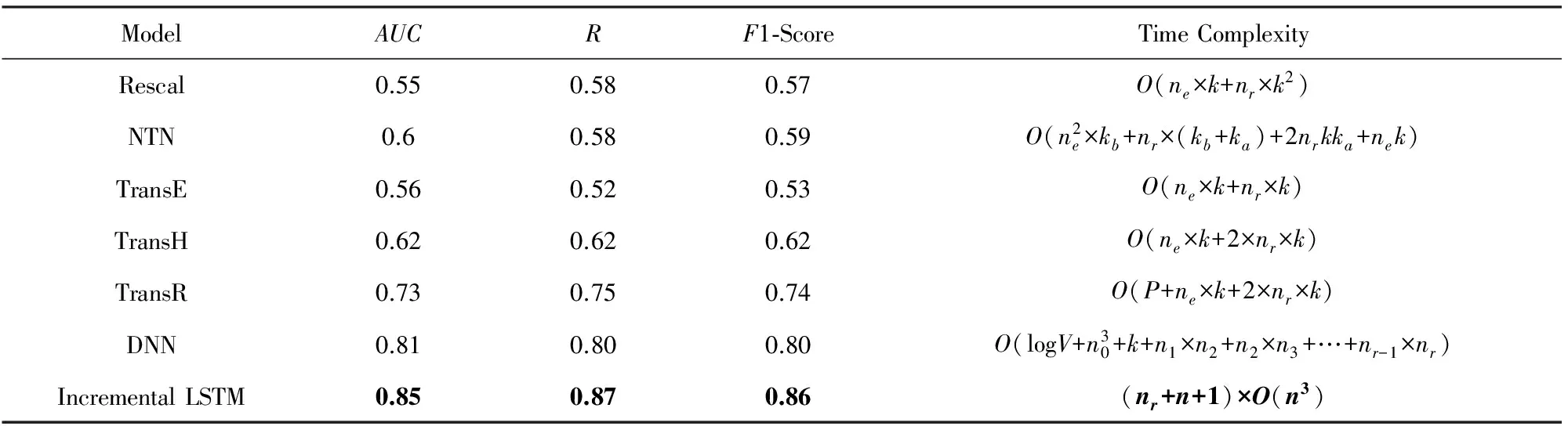
Table 2 Performance Comparison on Different Models表2 不同模型的性能对比
Note: The bold words mean best choice.
从表2中可以看出,通过该实验,翻译模型中的TransR模型,在本文的临床事实知识图谱中,准确度要比Rescal算法和NTN算法高.翻译模型中TransE和TransH模型,与Rescal和NTN相比,并无明显优势.而深度学习模型DNN通过对隐藏层的控制表达,对比传统链接预测方法取得了较大的优势.可见,针对临床领域时序知识图谱中语义丰富、时序性等特征,本文提出的增量LSTM模型取得了最高的准确度、召回率和F1-Score,分类性能有了明显提升.
4.4.2 模型ROC图对比
ROC曲线图以真阳性率TP作为纵轴,以假阳性率FP作为横轴.由图5可知,本文提出的增量LSTM模型ROC曲线下的面积最大,同时最凸,最靠近左上点,表明在这3类模型中新模型的诊断价值最大,准确性最高,利用价值大.
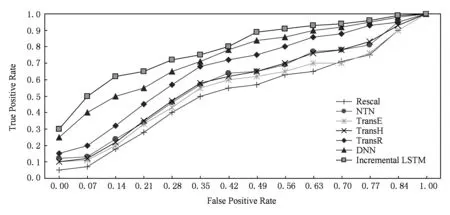
Fig. 5 ROC results on different models图5 ROC对比图
4.4.3 模型PR图对比
本文对这3个模型还做出了PR曲线图的对比.在PR曲线中,以召回率R为横坐标,P为纵坐标.召回率表明的是查全率,精确度表明的是查准率,两者不可同时兼得,一般R高,P就低,反之R低,P就高.因此,往往通过PR曲线去看它们之间的关系和权衡点.与ROC曲线左上凸不同的是,PR曲线中,越右上凸的曲线,说明该模型的效果越好.
由图6的各个模型的PR图对比,可以看出,相比于其他模型,增量LSTM模型在PR图上的表现有了比较明显的提高,右凸程度比原先大了很多,说明该模型的效果有了提升,是7种模型中右凸程度最明显的,因此,本文提出的增量LSTM效果最佳.
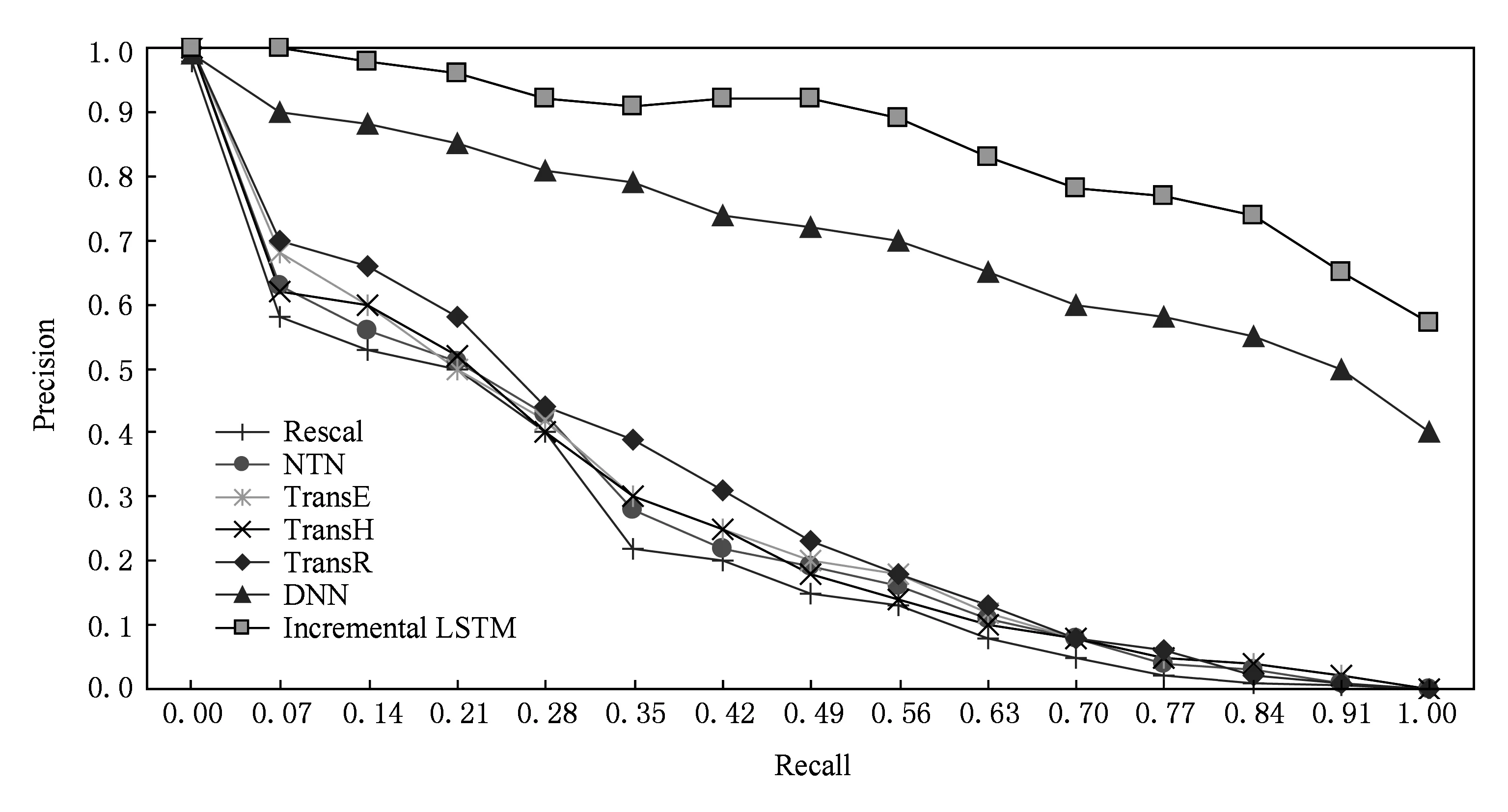
Fig. 6 PR results on different models图6 PR对比图
通过从以上准确度AUC、召回率R以及ROC曲线和PR曲线对比图方面进行分析,综合得出本文提出的增量模型相比原先的模型,在各个方面都有了显著的提升.
4.4.4 模型中网络结构对比
本文对增量LSTM模型中LSTM神经元个数进行了不同参数对比,表3为不同参数对最终性能的影响对比表.
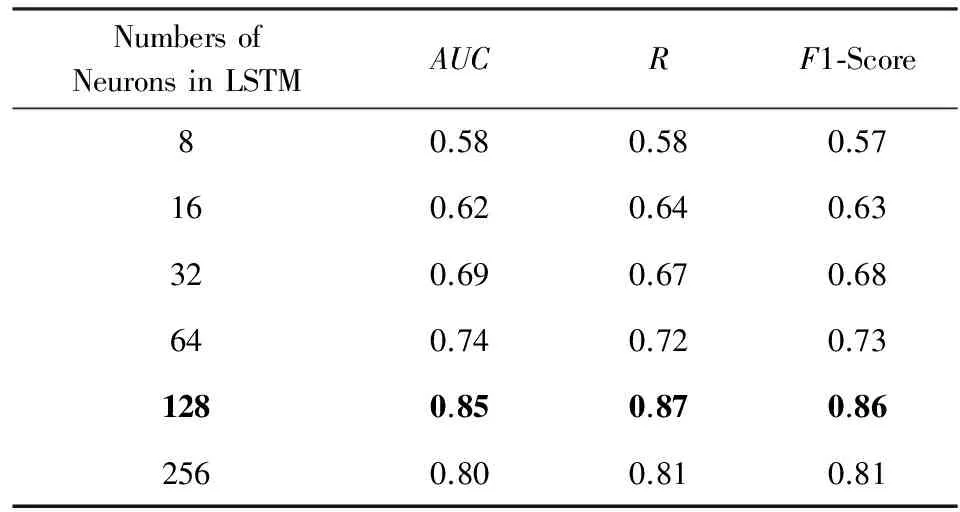
Table 3 Comparison on Different Network Parameters表3 不同网络参数的对比
Note: The bold words mean best choice.
随着LSTM中神经元数量的增加,增量LSTM模型在准确率、召回率以及F1分数上有了明显提高.然而,当神经元增加到256时,性能反而有所下降,其中的原因可能是因为过拟合.因此,本文在进行了模型参数对比实验后,最终选取128个LSTM神经元个数.
4.4.5 模型时间复杂度分析
表2中包含本文提出模型和其他模型的复杂度对比.其中ne指实体的数量,nr指关系的数量,k指向量化中向量的维度,Ka,kb,kc分别对于NTN中的第a,b,c层的大小,nr是第r层神经网络中的神经元个数,P代表TransR中做投影的时间,在DNN模型中,V是通过One-hot方式初始化稀疏向量的维度,n0是双向LSTM中输入层的序列数,nr是第r层神经网络中的神经元个数,n表示增量LSTM中输入的三元组序列数.
从表2中可见,Rescal的复杂度最高,需要的时间消耗最大.NTN方法的时间消耗也过长,若运行在更大的数据量中,时间限制则会加剧.而基于转换模型的时间复杂度大大降低,适于大数据量.增量LSTM模型的时间与输入量联系紧密,知识图谱中关系种类越多,输入的三元组序列数越大,时间复杂度即越高.
5 总 结
本文提出了一种基于深度学习的临床领域时序知识图谱链接预测模型.该模型用于以医院内部实际的EMR记录为基础,所创建具有时序特性的临床领域时序知识图谱.该模型选取适合大规模数据的TransR转换模型,在包含不同语义的关系空间中做实体投影,从而对图谱中的实体和复杂语义关系向量化.然后,采用LSTM递归神经网络,加入了图谱中的上下关联信息,进行序列化学习.接着对时序信息做增量计算,对时序信息提取更精准的特征向量.最后,不断通过增量计算和LSTM递归网络进行深层学习,提高预测准确度.实验表明:增量LSTM模型突出临床事实中隐含的语义和时序信息,有效地利用序列化学习挖掘其前后依赖信息,弥补了传统链接预测模型导致对时效性知识图谱预测准确度较低的不足.在未来的工作中,考虑将本文所提框架下的LSTM替换为其他LSTM变种方法,进一步集成其他深度学习方法,从而优化增量LSTM模型.
[1]Sequeda J F. Integrating relational databases with the semantic Web: A reflection[C] //Proc of the 13th Int Summer School. Berlin: Springer, 2017: 68-120
[2] Sulakhe D, Balasubramanian S, Xie Bingqing, et al. Lynx: A database and knowledge extraction engine for integrative medicine [J]. Nucleic Acids Research, 2014, 42(D1): 1007-1012
[3] Ogishima S, Takai T, Shimokawa K. Integrated database and knowledge base for genomic prospective cohort study in tohoku medical megabank toward personalized prevention and medicine[C] //Proc of the 15th World Congress on Health and Biomedical Informatics. Amsterdam: IOS Press, 2015: 1057-1057
[4] Jia Lirong, Liu Jing, Yu Tong, et al. Construction of traditional Chinese medicine knowledge graph [J]. Journal of Medical Informatics, 2015, 36(8): 51-55 (in Chinese) (贾李蓉, 刘静, 于彤, 等. 中医药知识图谱构建[J]. 医学信息学杂志, 2015, 36(8): 51-55)
[5] Wang Qiao, Wang Wei. Papers on genome epidemiology in the world: A knowledge map-based visual analysis [J]. Chinese Journal of Medical Library and Information Science, 2013, 22(4): 2-9 (in Chinese)(王俏, 王伟. 基于知识图谱的国际基因组流行病学可视化分析[J]. 中华医学图书情报杂志, 2013, 22 (4): 2-9)
[6] Liu Danhong, Luo Xiaonan, Xu Yongyong. Overview of electronic medical records and its application [J]. Chinese Health Quality Management, 2010, 17(4): 1-5 (in Chinese)(刘丹红, 罗小楠, 徐勇勇. 电子病历及其应用概述 [J]. 中国卫生质量管理, 2010, 17(4): 1-5)
[7] Taskar B, Fai W M, Abbeel P, et al. Link prediction in relational data[C] //Proc of the 17th Int Conf on Neural Information Processing Systems. Cambridge, MA: MIT Press, 2003: 659-666
[8] Baader F, Sertkaya B. Usability issues in description logic knowledge base completion[C] //Proc of the 7th Int Conf on Formal Concept Analysis. Berlin: Springer, 2009: 1-21
[9] Sahebi S, Yu-Ru L, Brusilovsky P. Tensor factorization for student modeling and performance prediction in unstructured domain[C] //Proc of the 9th Int Conf on Educational Data Mining. Berlin: Springer, 2016: 502-506
[10] Chang Kaiwei, Yih W, Yang Bishan, et al. Typed tensor decomposition of knowledge bases for relation extraction[C]//Proc of the 2014 Conf on Empirical Methods in Natural Language Processing. Stroundsburg, PA: ACL, 2014: 1568-1579
[11] Morales C, Collarana D, Vidal D E, et al. MateTee: A semantic similarity metric based on translation embeddings for knowledge graphs[C] //Proc of the 17th Int Conf on Web Engineering. Berlin: Springer, 2017: 246-263
[12] Vang K J. Ethics of Google’s knowledge graph: Some considerations [J]. Journal of Information, Communication and Ethics in Society, 2013, 11(4): 245-260
[13] Bollacker K D, Evans C, Paritosh P. Freebase: A collaboratively created graph database for structuring human knowledge[C] //Proc of the 39th ACM SIGMOD Int Conf on Management of Data. New York: ACM, 2008: 1247-1250
[14] Bizer C, Lehmann J, Kobilarov G, et al. DBpedia—A crystallization point for the Web of data [J]. Journal of Web Semantics, 2009, 7(3): 154-165
[15] Miller A, WordNet G. A lexical database for English [J]. Communications of the ACM, 1995, 38(11): 39-41
[16] Sousa A M, Pereira O M, et al. MorphoCol: An ontology-based knowledgebase for the characterisation of clinically significant bacterial colony morphologies [J]. Journal of Biomedical Informatics, 2015, 55: 55-63
[17] Abraham I L, Buckwalter K C. Geropsychiatric nursing: A clinical knowledge base in community and institutional settings [J]. Journal of Psychosocial Nursing and Mental Health Services, 1994, 32(4): 20-26
[18] Vives-Boix V, Fernández D R, et al. A knowledge-based clinical decision support system for monitoring chronic patients[C] //Proc of the 7th Int Work-Conf on the Interplay Between Natural and Artificial Computation. Berlin: Springer, 2017: 435-443
[19] Ruan Tong, Sun Chenglin, Wang Haofeng, et al. Construction of traditional Chinese medicine knowledge graph and its application [J]. Journal of Medical Informatics, 2016, 37(4): 8-13 (in Chinese)(阮彤, 孙程琳, 王昊奋, 等. 中医药知识图谱构建与应用 [J]. 医学信息学杂志, 2016, 37(4): 8-13)
[20] Xiang Y, Poh K L. A knowledge-based modeling system for time-critical dynamic decision-making[C] //Proc of the 9th Pacific Rim Int Conf on Artificial Intelligence. Berlin: Springer, 2006: 212-221
[21] Bordes A, Usunier N, García-Durán A, et al. Translating embeddings for modeling multi-relational data[C] //Proc of the 27th Annual Conf on Neural Information Processing Systems. Cambridge, MA: MIT Press, 2013: 2787-2795
[22] Wang Zhen, Zhang Jianwen, Feng Jianlin, et al. Knowledge graph embedding by translating on hyperplanes[C] //Proc of the 28th AAAI Conf on Artificial Intelligence. Menlo Park, CA: AAAI, 2014: 1112-1119
[23] Lin Yankai, Liu Zhiyuan, Sun Maosong, et al. Learning entity and relation embeddings for knowledge graph completion[C] //Proc of the 29th AAAI Conf on Artificial Intelligence Learning. Menlo Park, CA: AAAI, 2015: 2181-2187
[24] Xie Danfeng, Zhang Lei, Bai Li. Deep learning in visual computing and signal Pprocessing [J]. Applied Computational Intelligence and Soft Computing, 2017, 2017: Article ID 1320780
[25] Li Xiang, Taheri A, Tu Lifu. Commonsense knowledge base completion[C] //Proc of the 54th Annual Meeting of the Association for Computational Linguistics. Stroudsburg, PA: ACL, 2016: 1445-1455
[26] Jiang Tingsong, Liu Tianyu, Ge Tao, et al. Towards time-aware knowledge graph completion[C] //Proc of the 26th Int Conf on Computational Linguistics. Stroudsburg, PA: ACL, 2016: 1715-1724
[27] Melisachew W C, Giuseppe P, Joerg S, et al. Marrying uncertainty and time in knowledge graphs[C] //Proc of the 31th AAAI Conf on Artificial Intelligence. Menlo Park, CA: AAAI, 2017: 88-94
[28] Sepp H, Jürgen S. LSTM can solve hard long time lag problems[C] //Proc of the 10th Neural Information Processing Systems. Cambridge, MA: MIT Press, 1996: 473-479
[29] Luo Yuan, Ahmad F S, Shah J S. Tensor factorization for precision medicine in heart failure with preserved ejection fraction [J]. Journal of Cardiovascular Translational Research, 2017, 10(3): 305-312
[30] Robert S, Catherine H. Representing general relational knowledge in conceptNet 5[C] //Proc of the 8th Int Conf on Language Resources and Evaluation. Marrakech, Morocco: ELRA, 2012: 3679-3686
ALinkPredictionModelforClinicalTemporalKnowledgeGraph
Chen Dehua1, Yin Suna1, Le Jiajin1, Wang Mei1, Pan Qiao1, Zhu Lifeng2
1(CollegeofComputerScienceandTechnology,DonghuaUniversity,Shanghai201600)2(RuijinHospital,SchoolofMedicine,ShanghaiJiaoTongUniversity,Shanghai200025)
Link prediction on knowledge graph is the main task of knowledge base completion, predicting whether a relationship existing in the knowledge base is likely to be true. However, traditional knowledge link prediction models are only appropriate for static data rather than temporal knowledge base. Temporal knowledge base exists on various fields. Take medical medicine field as example, diabetes is a typical chronic disease which evolves slowly. Thus, link prediction on clinical knowledge base such as diabetic complication requires the analysis on temporal characteristic of temporal knowledge base, which is a great challenge for traditional link prediction models. Thus, to address the prediction of temporal knowledge base, this paper proposes a long short-term memory (LSTM) based model for temporal knowledge base. The proposed model adopts memory cells of LSTM for sequential learning, and then builds incremental learning layer. Afterwards, timing characteristics can be extracted by the way of end-to-end, which realizes the prediction on temporal knowledge base. In experiments, the proposed model in clinical temporal knowledge base shows significant improvements compared with baselines including Rescal, NTN, TransE, TransH, TransR and DNN.
temporal knowledge graph; knowledge graph link prediction; translation model TransR; long short term memory (LSTM) networks; incremental learning
2017-09-01;
2017-10-06
上海市科技创新行动计划项目(15511106900);上海市科技发展基金项目(16JC1400802);上海市信息化发展专项基金项目(XX-XXFZ-01-14-6349)
This work was supported by the Shanghai Innovation Action Project of Science and Technology (15511106900), the Science and Technology Development Foundation of Shanghai (16JC1400802), and the Shanghai Specific Fund Project for Information Development (XX-XXFZ-01-14-6349).
TP391

ChenDehua, born in 1976. PhD and associate professor. His main research interests include database, data warehouse, big data and deep learning.

YinSuna, born in 1994. Master candidate. Her main research interest is data mining (yinsuna312@126.com).

LeJiajin, born in 1951. Professor and PhD supervisor. Member of CCF. His main research interests include database and data warehouse, software engineering theory and practice (lejiajin@dhu.edu.cn).

WangMei, born in 1980. PhD and professor. Member of CCF. Her main research interests include database, image semantic analysis and information retrieval (wangmei@dhu.edu.cn).

PanQiao, born in 1977. Associate professor and deputy director of department. His main research interests include big data and cloud computing, machine learning (panqiao@dhu.edu.cn).

ZhuLifeng, born in 1976. PhD candidate at Donghua University, and senior engineer at Ruijin Hospital. His main research interests include medical information management and medicaldata (zlf@rjh.com.cn).

