p53蛋白性质与结构分析
*刘严木
(四川师范大学附属中学 四川 610000)
p53蛋白性质与结构分析
*刘严木
(四川师范大学附属中学 四川 610000)
p53作为关键肿瘤抑制蛋白参与了50%以上肿瘤的发生,本文对p53基本特性进行了初步地分析,通过分析p53氨基酸组成、疏水性表明p53蛋白具有酸性蛋白质、胶原蛋白、核蛋白、一些RNA结合模体等特征,整体呈现亲水性.利用MEGA软件进行p53系统发育分析,结果表明人p53与日本猕猴,食蟹猴,猕猴的p53亲缘关系高度相近.使用PSORT II分析工具对p53亚细胞定位进行预测,表明p53蛋白主要分布在细胞核中.使用PRABI-GERLAND算法对p53二级结构进行预测,结果表明18.07%为α螺旋,18.07%为β折叠,63.87%为无规则卷曲.最后,采用同源建模(比较建模)方法构建了p53蛋白的三级结构,从建模质量评估表明预测结果准确.
p53蛋白;系统发育分析;亚细胞定位预测;二级结构预测;三级结构预测
p53基因是迄今为止发现的与人类肿瘤相关性最高的基因之一.自1979年伦敦癌症研究所工作的David Lane和Lionel Crawford,美国普林斯顿大学(Princeton University)的Daniel Linzer和Arnold Levine等研究小组发现p53基因后,对其相关研究一直在持续进行.人们对p53基因的研究经历了癌蛋白抗原,癌基因到抑癌基因的认识转变.p53蛋白的实际分子量只有43.7kDa,因为该蛋白富含脯氨酸,所以在SDS聚丙烯酰胺凝胶电泳试验中的迁移率偏慢,于SDS凝胶电泳中测得分子量约53kDa.p53蛋白主要通过以下机制来避免癌症发生:(1)当DNA受损失,p53蛋白活化DNA修复蛋白以修复DNA.(2)p53蛋白能抑制细胞周期于G1/S节律点上,检查细胞基因组的完整性,并使DNA修复蛋白有更充足时间修复DNA损伤部位.(3)若细胞DNA损伤,p53蛋白开启细胞凋亡程序,避免细胞不正常增值.突变型p53蛋白使细胞无限生长,与肿瘤发生密切相关.p53与大部分人类肿瘤有关,如肝癌,乳腺癌,膀胱癌,胃癌等.在癌症高发的当今社会,研究p53基因及其基因产物无疑具有重要意义.本研究通过氨基酸组成分析,亚细胞定位预测,p53系统发育分析以及p53的二、三级结构预测对p53蛋白的序列特性与蛋白结果做了初步分析,以期对p53蛋白有更进一步地认识.
1.材料与方法
(1)材料
人与其它物种p53蛋白氨基酸序列
从uniprot上下载人或其它物种p53蛋白氨基酸序列(FASTA格式),以人p53蛋白序列为例:
gt;sp|P04637|P53_HUMAN Cellular tumor antigen p53 OS=Homo sapiens GN=TP53 PE=1 SV=4
MEEPQSDPSVEPPLSQETFSDLWKLLPENNVLSPLPSQAMDDLMLSPD DIEQWFTEDPGP
DEAPRMPEAAPPVAPAPAAPTPAAPAPAPSWPLSSSVPSQKTYQGSYG FRLGFLHSGTAK
SVTCTYSPALNKMFCQLAKTCPVQLWVDSTPPPGTRVRAMAIYKQSQH MTEVVRRCPHHERCSDSDGLAPPQHLIRVEGNLRVEYLDDRNTFRHSVVVPY EPPEVGSDCTTIHYNYMCNSSCMGGMNRRPILTIITLEDSSGNLLGRNSFEV RVCACPGRDRRTEEENLRKKGEPHHELPPGSTKRALPNNTSSSPQPKKKPLD GEYFTLQIRGRERFEMFRELNEALELKDAQAGKEPG
GSRAHSSHLKSKKGQSTSRHKKLMFKTEGPDSD
(2)方法
①p53氨基酸组成分析
利用ProtParam进行p53氨基酸组成分析.ProtParam允许各种物理和化学的一个给定的存储蛋白参数计算Swiss-Prot和TrEMBL或用户输入的蛋白序列.计算参数包括分子量,理论等电点,氨基酸组成,原子组成,消光系数,估计的半衰期,不稳定指数、脂肪总平均指数和亲水性.
②系统发育分析(分子进化分析)
使用MEGA(版本5.2)软件进行p53系统发育分析.MEGA是一款可以根据所提供的氨基酸序列,分析在物种进化过程中物种的亲缘关系的一种算法,并将结果以进化树的形式呈现,是目前最为常用的系统发育分析软件之一.
③亚细胞定位预测
利用PSORT II分析工具对p53亚细胞定位进行预测.PSORT II从氨基酸序列预测亚细胞定位.它将动物蛋白质定位于细胞质,细胞骨架,细胞核,细胞膜,内质网,高尔基体,溶酶体,线粒体,等细胞结构.
④p53蛋白二级结构预测
⑤p53蛋白三级结构预测
进行p53三级结构预测将利用SWISS-MODEL.SWISSMODEL是一个完全自动化的蛋白质结构同源建模服务器,采用同源建模(比较建模)方法构建蛋白质结果.该方法包括:A.搜索与目标蛋白序列相似的模板蛋白;B.BLAST搜索;C.目标序列与模板序列比对;D.ClustalW/X;E.建立骨架(将模板结构叠加起来,找结构保守区域).
2.结果与分析
(1)氨基酸组成分析
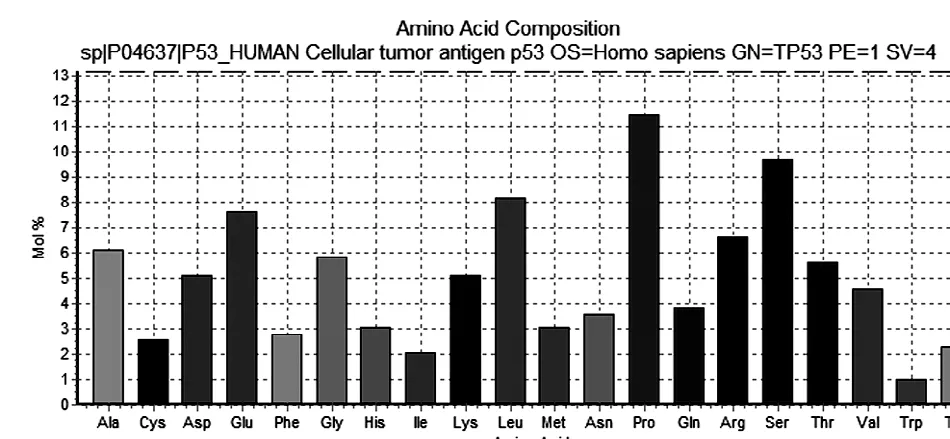
图1 p53蛋白氨基酸组成分析结果
氨基酸组成分析:占比大于5.0%,即含量高于平均值的有丙氨酸(Ala,A),天冬氨酸(Asp,D),谷氨酸(Glu,E),甘氨酸(Gly,G),赖氨酸(Lys,K),亮氨酸(Leu,L),脯氨酸(Pro,P),精氨酸(Arg,R),丝氨酸(Ser,S),苏氨酸(Thr,T).最高为脯氨酸(Pro,P),占比11.45%,最低为色氨酸(Trp,W),占比1.02%.
根据特定高含量氨基酸对应蛋白质的类型,推断该蛋白质具有以下特点:
①酸性蛋白质.因为D,E含量较高;
②胶原蛋白.因为G含量较高;
③核蛋白.因为K,R含量较高;
差异化教学是根据学生的特点、兴趣和天赋的不同,有针对性的制定人才培养计划,每个学生都有符合自身特点的教学安排。通过这些安排,教师有目的、有计划、有组织地引导学生积极自觉地学习,使得学生在自己最适合的方向快速成长,才能迅速提高,成为能够很好适应社会的人才。
④一些RNA结合模体,因为S,R含量较高;
⑤黏蛋白,寡聚糖结合位点,因为S,T含量较高.
(2)疏水性分析
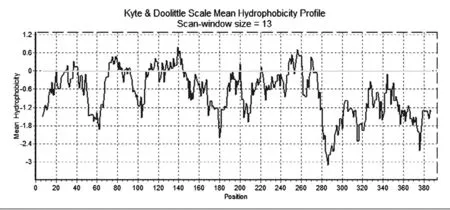
图2 p53蛋白疏水性曲线
横轴表示氨基酸位点,纵轴表示疏水性值.
蓝色线条表示疏水性参考线,蓝线上方表示疏水,下方表示亲水.
据图可知,p53蛋白约有4个疏水性区域,氨基酸呈疏水性对应区域分别约为20-40位,70-90位,110-150位,250-260位.其余均为亲水性区域,其中280-310区间呈现显著的亲水性.总体分析来看,因为p53蛋白多数曲线位于蓝线下方,即p53蛋白总体呈现亲水性.
(3)系统发育分析(进化分析)
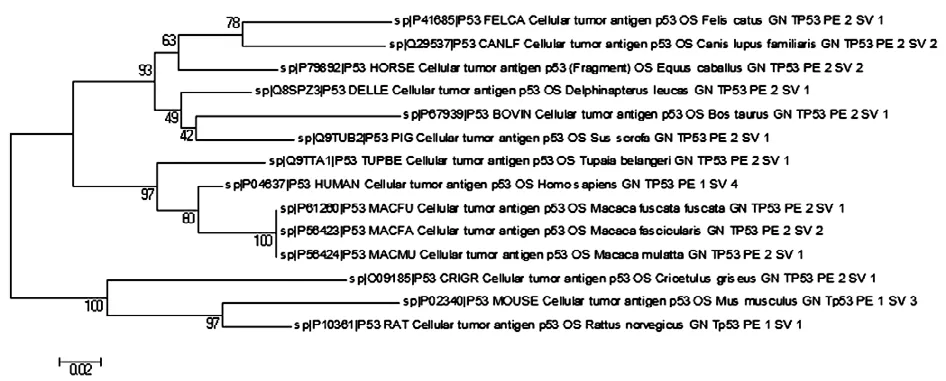
图3 系统发育分析结果
接着,我们想了解p53蛋白在不同物种之间的进化关系是怎样的,为此我们做了p53系统发育分析.最大进化树根据亲缘关系将不同物种进行分类,每个大类下还有相应小类.数值越大表明预测的亲缘关系越可靠.本次的材料选用了家猫(Felis catus)、家犬(Canis lupus familiaris)、马(Equus caballus)、白鲸(Delphinapterus leucas)、牛(Bos taurus)、野猪(Sus scrofa)、树鼩(Tupaia belangeri)、智人(Homo sapiens)、日本猕猴(Macaca fuscata)、食蟹猴(Macaca fascicularis)、猕猴(Macaca mulatta)、灰仓鼠(Cricetulus griseus)、小鼠(Mus musculus)以及大鼠(Rattus norvegicus)的p53蛋白序列.
据上图分析,最大进化树一共有11种分类(包括大分类和小分类).其中灰仓鼠,小鼠,大鼠的亲缘关系高度相近.日本猕猴,食蟹猴,猕猴的亲缘关系高度相近.人与日本猕猴,食蟹猴,猕猴的亲缘关系高度相近,推测是因为人是由灵长类动物进化而来所有保留高度的相似性.白鲸,牛,猪的值小于50,证明三者的亲缘关系相距较远.
(4)亚细胞定位预测
PSORT II部分运行结果显示p53蛋白有91.3%为细胞核,4.3%为细胞骨架,4.3%为囊泡分泌系统.这说明p53蛋白绝大多数在细胞核中,这与多数情况p53与DNA结合从而发挥生物学功能有关.
(5)二级结构预测
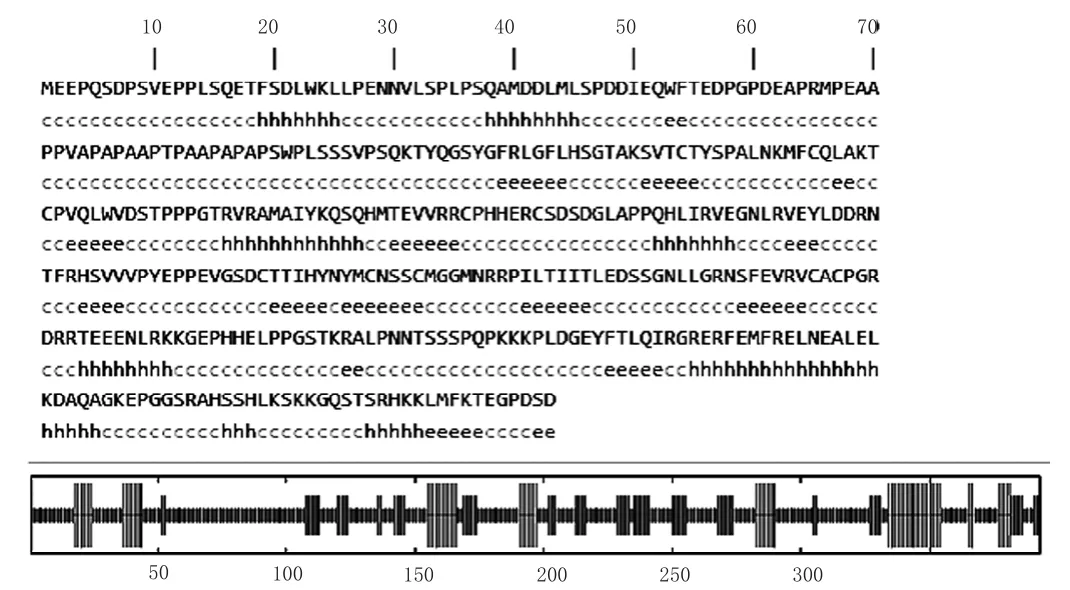
图4 二级结构类型及含量.蓝色表示α螺旋,红色表示扩展链,紫色表示无规则卷曲
从上述结果可以知晓,p53蛋白18.07%为α螺旋,18.07%为β折叠,63.87%为无规则卷曲.其中无规则卷曲占绝大多数.
图5 各位点氨基酸为α螺旋,扩展链,无规则卷曲的概率.
(6)三级结构预测
选用SWISS-MODEL提供的MODEL 01(此模板为p53-DNA结合的三级结构数据),通过同源模建方法对p53蛋白结构进行预测.3D结构如下:

图6 p53蛋白用SWISS-MODEL分析后不同模型类型以及不同显示方式展示
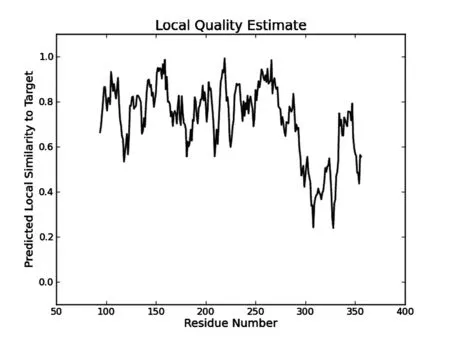
图7 建模质量评估.横坐标表示氨基酸位点,纵坐标表示预测局部与靶标的相似性.
从图7可以看出,SWISS-MODEL对p53蛋白的约90-280个氨基酸与模板的相似性较高,从而说明这一预测区间较为准确,300-320区间由于与模板的序列差异较大,从而相对预测准确性偏低.从全局模型质量评估(GMQE)数值来看(值为0.60),说明整体模型预测分析较准确.
[1]Arnold J. Levine and Moshe Oren. (2009) The first 30 years of p53: growing ever more complex. Nature Reviews Cancer, 9:749-757.
[2]李文娟,潘庆杰,李美玉.p53基因及其功能研究进展.生物技术通讯,2014,282-285.
[3]贾春平.抑癌基因p53与肿瘤研究的最新进展.生命科学,2008,450-453.
[4]The UniProt C: Uniprot: The universal protein knowledgebase. Nucleic acids research 2017,45:D158-D169.
[5]Tamura K, Peterson D, Peterson N, Stecher G, Nei M,Kumar S: Mega5: Molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods. Molecular biology and evolution 2011,28:2731-2739.
[6]Biasini M, Bienert S, Waterhouse A, Arnold K, Studer G, Schmidt T, Kiefer F, Gallo Cassarino T, Bertoni M, Bordoli L, Schwede T: Swiss-model: Modelling protein tertiary and quaternary structure using evolutionary information.Nucleic acids research 2014,42:W252-258.
刘严木(2000-),男,四川师范大学附属中学;研究方向:生物科学.
Properties and Structure Analysis of p53 Protein
Liu Yanmu
(Affiliated High School of Sichuan Normal University, Sichuan, 610000)
p53, as a key tumor suppressor protein, is involved in the occurrence of more than 50 % of tumor. This paper makes a preliminary analysis of the basic characteristics of p53 and through the analysis of p53 amino acid composition and hydrophobicity, the results indicate that p53 protein has the feature of acidic protein, collagen, nuclear protein, some RNA binding motifs and it presents hydrophilic as a whole. Using the MEGA software to make the phylogenetic analysis of p53, the results show that it has a close genetic relationship with the p53 of Japanese macaques, crab monkeys and rhesus monkeys. Using PSORT II analysis tool to predict p53 subcellular localization, it showed that p53 protein was mainly distributed in the nucleus. Using the PRABI-GERLAND algorithm to predict p53 secondary structure, the results show that 18.07% present alpha helix, 18.07%present beta folding, and 63.87% present random coil. Finally, the three-level structure of p53 protein was constructed by homologous modeling(comparative modeling) method, and the prediction results were indicated to be accurate from the modeling quality assessment.
p53 protein;phylogenetic analysis;subcellular localization prediction;secondary structure prediction;tertiary structure prediction
O
A

