DNA分子标记在不结球白菜遗传育种中的应用进展
李蒙蒙, 崔丽洁, 钱忠英, 开国银, 潘静娴
(上海师范大学 生命与科学学院 植物种质资源开发协同创新中心,上海 200234)
DNA分子标记在不结球白菜遗传育种中的应用进展
李蒙蒙, 崔丽洁, 钱忠英, 开国银*, 潘静娴*
(上海师范大学 生命与科学学院 植物种质资源开发协同创新中心,上海 200234)
概述了分子标记的分类和特点,以及在不结球白菜遗传多样性分析、遗传图谱构建、数量性状座位(quantitative trait locus,QTLs)定位和分子标记辅助育种等领域中的最新研究进展.总结了目前分子标记在不结球白菜遗传改良和辅助育种中存在的若干问题,并对该研究领域提出了一些展望和建议.
分子标记; 遗传育种; 不结球白菜
不结球白菜(Brassicacampestrisssp.chinensisMakino),为十字花科(Brassicaceae)芸薹属(Brassica)芸薹种(B.campestiris)蔬菜.原产于中国的江淮流域,在长江中下游及南方地区广泛栽培,常年在蔬菜供应中占较大比重.由于其适应性强,生长周期短,营养丰富等特点,近几年,不结球白菜逐渐被引种至中国北方,并开始在东南亚及欧美地区广泛栽培,逐渐成为一种全球性的蔬菜[1].随着市场需求的不断增大,中国育种学家对不结球白菜进行了大量的遗传基础研究,并培育了许多具有优良性状的新品种[2-3].传统的选育工作主要以农作物的表现型作为选育指标,通过对杂交后代的优化选择,最终得到能稳定遗传优良性状的个体.因此,传统的选育方法并不能排除环境因素的影响,给选育工作带来了许多不确定的因素.分子标记则是直接检测DNA序列的多态性,避免了外界环境对表型性状的影响,提升了遗传分析及育种工作的准确性.除此之外,分子标记遍布整个基因组,多态性高,且由于其大多分布于非编码区,不会影响目的性状的表达,所以在农作物的遗传育种研究中发挥着不可替代的作用.
本文作者重点阐述了分子标记在不结球白菜遗传多样性检测,遗传图谱构建,重要性状的数量性状座位(QTL)定位,及分子标记辅助育种等方面的应用.
1 DNA分子标记的分类
遗传标记指在生物体内受基因调控的、可遗传、可通过技术手段加以检测和识别的任何变异性状,在动植物的遗传育种工作中发挥着重要作用.根据检测方法的不同,遗传标记大致可分为形态标记、细胞标记、生化标记和分子标记.前三种是在个体、细胞和蛋白水平对基因的表达产物进行检测,是对核苷酸序列的间接反映,因此表现出多态性差、标记数目少、易受外界干扰等缺点.分子标记则是一类以个体间遗传物质内核苷酸序列变异为基础的遗传标记,能直接反映DNA遗传多态性.作为一种较为理想的遗传标记,由于其不受季节、生长时期等条件的限制,并且具有数量无限、多态性高、共显性遗传等优势,已经广泛应用于不结球白菜的遗传育种研究中.
经过四十几年的发展,DNA分子标记的种类已经达到了数十种之多.每一种DNA分子标记在多态性检测、重复性、经费开支及应用领域等方面的优势不尽相同(表1).根据技术原理的不同,可以将广泛应用于不结球白菜遗传育种工作中的DNA分子标记大致分为三类:第一类是以DNA分子之间杂交为基础的分子标记,主要代表是限制性片段长度多态性(RFLP).在生物进化过程中,分类地位相近物种间的同源序列由于发生部分DNA片段的插入、缺失、易位和倒位,从而使得该区域限制性内切酶的识别位点增加或者减少.RFLP就是利用限制性内切酶对DNA进行消化,通过比较产生DNA的多态性来检测两个体之间的亲缘关系[4].第二类是以聚合酶链反应(PCR)扩增技术为基础的分子标记,比较常用的包括随机扩增多态性DNA (RAPD),简单序列重复 (SSR)和扩增片段长度多态性(AFLP).RAPD是利用10bp的随机引物对基因组DNA进行扩增,通过比较扩增产物的多态性来揭示基因组中相应区域的DNA多态性[5].在真核生物的基因组序列中存在由1~6个碱基组成的重复单位,SSR就是利用该重复单位在不同等位基因间重复次数不同的特性,根据序列两端设计特异性引物对SSR序列进行PCR扩增并比较产物片段的多态性[6].AFLP则是利用限制性内切酶对基因组DNA进行消化,DNA的粘性末端加上人工接头后,利用引物与接头3′端的特异性识别的特点对酶切片段进行PCR扩增,最后通过比较产物的长度来检测多态性[7].第三类是基于单核苷酸多态性的DNA标记,即单核苷酸多态性标记 (SNP).SNP是指同一位点的不同等位基因之间存在个别核苷酸的差异或者小的插入和缺失,可通过基因芯片技术进行检测[8].
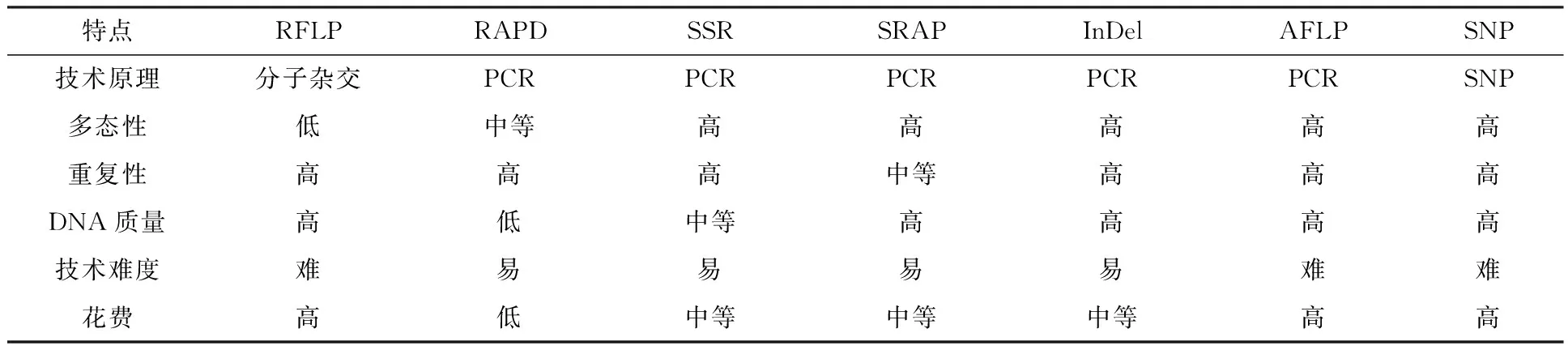
表1 常用的分子标记及其特点比较
2 DNA分子标记在不结球白菜的遗传多样性分析中的应用
2.1不结球白菜的起源与演化
中国作为芸薹属作物的起源中心,早在三国时期就有关于不结球白菜的描述和记载.但是,现阶段由于没有发现野生品种与描述起源的相关文献,所以对于不结球白菜具体的起源地还存在着争议.学者们主要通过查阅古典书籍,对不结球白菜的起源地提出了各种假说,由于其喜欢温暖湿润的气候,且现阶段大多在南方地区广泛栽培,所以大多数学者认为不结球白菜起源于中国的南方并慢慢演化到全国各地[9-10].近几年,随着分子标记技术的在遗传多样性研究中的广泛应用,学者们陆续使用各种分子标记对不结球白菜的起源与演化进行了研究.Wang等[11]收集了来自于中国、日本和泰国三个国家,总共68份普通白菜种质资源,ISSR分子标记检测结果表明中国的遗传多样性最高,并且来源相同地理分区的种质,往往亲缘关系较近.韩建明等[12-13]通过SRAP和RAPD分子标记技术,从分子层面对不结球白菜的发源地进行了研究,在64份来源不同的种质中,普通白菜的遗传丰富度要高于其他品种,且中国的种质资源比亚洲其他国家丰富.就中国而言,江淮流域的遗传多样性明显高于中国其他地域.刘冬媛等[14]利用SSR分子标记对不结球白菜的遗传分化进行了分析,结果也发现中国的种质资源要比日韩等国丰富,且以江淮流域的遗传多样性最高.肖禾等[15]结合表型特征和RAPD标记技术对中国不同地域普通白菜的遗传多样性进行了分析,统计显示普通白菜在江淮地区的遗传多样性最丰富.以上研究表明,作为不结球白菜的原产地,中国的种质资源丰富,遗传多样性要明显高于周边国家,且从分子层面说明中国的江淮地区可能是起源地.
2.2亲缘关系及其分类
不结球白菜作为芸薹属芸薹中的一个亚种,中国的植物学家们基于形态学及园艺学特征,对不结球白菜的不同品系进行了大量的分类工作.吴耕民最早在《中国蔬菜栽培学》一书中将不结球白菜分为长梗白菜类、油冬菜类、塌菜类和芸苔类4个大类[16].中国农科院则依据植物学特征与栽培特点,在《中国蔬菜优良品种》一书中将其分为白梗白菜类、青梗白菜类或油冬菜类、塌地白菜类和菜苔类.曹寿椿等[17]在外部形态特征的基础上,进一步结合其生物学特性,将不结球白菜分为普通白菜、菜薹、薹菜、塌菜、分蘖菜和白菜型油菜6类.曹家树等[18]根据种皮形态、叶部特征等外部形态对不结球白菜进行了系统分类,大致分为普通白菜变种、塌菜变种、紫菜墓变种、薹菜变种、菜心变种、分蘖菜变种.
由于中国不结球白菜的起源及演化过程较为复杂,且不断引种及不同品种间杂交选育,仅仅根据植物的外部形态及生物学特征,并不能将不同类型的不结球白菜进行准确地区分.曹家树等[18-19]对不同品种白菜类蔬菜进行了RAPD分析,结果显示DNA扩增片段在不结球白菜的不同品系中表现出明显的差异,为不同品系之间的分类提供了重要的分子证据.孙德岭等[20]利用AFLP分子标记技术对不结球白菜品种之间的的亲缘关系进行了分析,两个薹菜品种虽然聚类于不结球白菜亚种中,但与其他常见不结球白菜品种亲缘关系较远.郭晶心等[21]对来自6个国家的白菜种质进行了亲缘关系的研究,小白菜的聚类分析结果较为混乱,各品种之间的相似性较低,与表型多样性分析结果有较大差异.单晓政等[22]用优化后的SRAP体系对不结球白菜的各个品种进行了分类与鉴定,基于分子标记多态性分析的聚类结果与园艺性状的分类结果基本相似,普通白菜、乌塌菜与薹菜分别聚为三大类.轩淑欣等[23]结合SSR标记技术与UPGMA聚类分析法对78份不结球白菜种质进行分类,其中51份普通白菜聚为一类,并且4份乌塌菜聚在I-2亚类群,说明两者是不同的品种,该结果为不同栽培品种的分类提供了依据.以上研究通过分子标记检测不同品系之间DNA的多态性,为明确和细化不结球白菜的分类提供了重要的分子证据.
3 DNA分子标记在不结球白菜辅助育种中的应用
3.1不结球白菜遗传图谱构建
遗传图谱又称遗传连锁图谱,是以染色体重组交换率为相对长度单位,并将遗传标记按其遗传距离以线性排列的方式依次标定在染色体上所得到的连锁图谱[24].构建遗传图谱是遗传学研究中的重要环节,高密度的连锁图谱在进行数量性状定位、开展分子标记辅助育种以及功能基因克隆等研究领域发挥着重要作用.
不结球白菜遗传图谱构建工作起步较晚,耿建峰等[25]将不结球白菜“暑绿”进行游离小孢子培养,并以获得的112个双单倍体(DH)株系为作图群体成功构建了第一张不结球白菜遗传连锁图谱.该图谱总覆盖长度为1 116.90 cM,包含了14条连锁群和186个多态性标记.而后,Geng等[26]使用了一种考虑偏分离和缺失标记的新方法,对原图谱进行了进一步的改进和纠正,新图谱总长为1 923.75 cM,总共包含138个分子标记,总平均图距为15.52 cM.到2011年,国内外学者已经成功构建了大量的遗传图谱(表2).为了缩短时间并提高效率,构建图谱过程中大多选择了F2代和DH群体作为作图群体,而没有选择用构建年限较长的重组自交系.其中以杂种后代F2作为作图群体时,大多选择了使用种内的不同品种作为杂交亲本,以此来保证亲本间的DNA多态性.而在图谱构建过程中常用的分子标记种类包括了RAPD,AFLP,ISSR,SSR,SRAP和InDel,多种标记技术的混合使用,有利于实现各种分子标记的优势互补,提高了图谱的长度和密度.

表2 已报道的不结球白菜遗传图谱
3.2不结球白菜数量性状的标记和定位
农作物的大多数经济和农艺性状都是数量性状.不同于受主效基因控制的质量性状,数量性状是多对微效基因联合效应的结果,因此在遗传变异上呈现出一定的连续性,且表现型与基因型之间并不存在明显的对应关系[34].再者数量性状比较容易受环境条件的影响,因此基于个体表型的传统育种方法并不能对作物株高、品质和产量和抗病性等数量性状进行有效的选育.控制数量性状的微效多基因往往相对集中地存在于一个染色体或多个染色体中的某一区段,QTL定位就是指借助DNA分子遗传标记来确定与之连锁的数量性状基因在染色体上的位置,从而使某些经济性状的定向改良成为可能.
3.2.1 农艺性状QTL定位与分析
耿建峰[35]在构建遗传图谱的同时,在8个不同生长时期中,检测到了不结球白菜控制株高、开展度及叶型等重要农艺性状的多个QTL位点.成妍[36]采用复合区间作图法,一共检测到分布在10个遗传群上的47个与不结球白菜经济性状相关的QTL位点,并且控制相关表型性状的QTL聚集分布.王倩[30]在其所建的不结球白菜遗传图谱上一共检测到19个与抽薹开花等性状相关的QTL位点,其中5个与控制现蕾天数相关,6个与抽薹天数相关,6个与开花天数相关,2个与抽薹指数相关.班青宇[29]利用不结球白菜不同品种间杂交得到的F2群体,通过对抽薹性状的记录和观察,成功检测和定位了4个与抽薹日数相关的QTL位点,5个与开花日数相关的QTL位点.
3.2.2 品质性状QTL定位与分析
耿建峰[35]在连锁图谱上成功定位到了多个与不结球白菜品质相关的QTL位点,其中包含了控制可溶性蛋白QTL位点2个,控制干物质的QTL位点3个,控制叶片、叶柄重比的QTL位点4个.单晓政[37]在不结球白菜遗传图谱的第3和第4连锁群上筛选出两个与叶片维生素C性状相关的QTL位点.班青宇等[38]在遗传图谱中的第5连锁群成功定位到一个控制脯氨酸含量的QTL位点,在第3连锁群定位到一个与可溶性蛋白含量相关的增效QTL位点,前者的加性效应和贡献率为0.370%和8.320%,后者的加性效应和贡献率为0.004%和8.120%.Wang等[39]在遗传图谱的A05,A08和A10连锁群上检测到了11个与光合色素含量相关的QTL位点,其中位点的最大贡献率为38.000%.
3.2.3 抗病性状QTL定位与分析
有关不结球白菜抗病性状QTL定位的研究较少,陆鹏[31]利用不结球白菜抗虫种质杂交所得的两张遗传图谱,对不同白菜抗源抗小菜蛾基因进行了QTL定位,在‘599×114’组合遗传图谱的LG7、LG8和LG9连锁群上检测到3个与抗虫相关的QTL位点,在‘508×114’组合遗传图谱中也成功定位了3个与抗虫相关的QTL位点,分别位于LG1和LG10连锁群上.
3.3 不结球白菜分子标记辅助育种
分子标记辅助育种(MAS)是指利用分子标记与决定目标性状基因紧密连锁的特点,通过检测DNA分子标记而选育出具有优良性状的新品种.不同于直接对个体表型进行性状筛选的传统育种方法,DNA分子标记辅助育种是在分子水平上直接对决定性状的基因型进行筛选,由此避免了环境等外界因素对个体表型性状的间接影响,从而实现了对选育性状的定向改良.此外,在幼体时期就能够通过分子标记对性状进行准确的选择,从而可以大大提高育种进程并削减成本.
自交不亲和性作为一种防止近亲繁殖的遗传机制,能够有效地避免自交衰退,从而保持品种的遗传多样性[40].在不结球白菜的选育工作中,也常常把自交不亲和性作为一个重要的选育目标.Shi等[41]以自交不亲和品种SC104-1与自交亲和品种SI13-2杂交F2群体为材料,结合混合分组分析法(BSA)获得一个与自交不亲和基因连锁距离为7.53±0.11 cM的RAPD分子标记S1107-(900).葛婷婷等[42]以不结球白菜的近等基因系为群体材料,筛选得到3个与自交不亲和基因连锁的SSR分子标记sR6688,KBRH138G23和Na12E02,遗传距离分别为12.91、5.35和3.08 cM,SSR引物验证后,最终在Na12E02附近得到一个与自交不亲和基因紧密连锁的标记BrSS15,遗传距离为0.49 cM.Li等[43]对不结球白菜隐性核雄性不育系WS24-3和雄性系WS135杂交产生的F2群体进行分析,在A2连锁群上定位一个隐形核基因Bra2Ms,并在该区域发现了两个共分离的分子标记SSRa2-951和SSRa2-960.贾永鹏等[44]利用自交亲和系WS-199和自交不亲和系WS-85构建了F2群体,采用BSA法和SNP芯片技术成功筛选出100个差异SNP位点,在相应区域设计引物并成功筛选到3个与自交不亲和性相关的SSR分子标记BrA1-2,BrA1-3和BrA1-14.
抽薹开花是指在低温且长日照等环境因素的诱导下,植物由营养生长向生殖生长转变的过程.因此,农作物的过早抽薹甚至未熟抽薹将会对生产造成巨大损失.张波[45]在遗传图谱中成功筛选到一个与晚抽薹基因紧密连锁的SSR分子标记LB.李向[32]在对不结球白菜DH群体进行连锁分析时,在A02连锁群上得到一个开花时间特异标记.Du等[46]利用BSA法从普通白菜中筛选到一个与晚抽薹基因遗传距离为3.14 cM的RAPD分子标记S265(750).
抗病育种作为分子标记辅助育种的重要方向,目前已经筛选出多个与抗病性状紧密连锁的分子标记,为选育高产抗病的不结球白菜新品种提供了依据.冷月强[47]利用RAPD分子标记并结合BSA法,获得了一个不结球白菜抗霜霉病基因紧密连锁的分子标记AY121238.Wang等[33]将抗芜菁花叶病毒基因TuRBCH01定位在R6连锁群上,两侧的AFLP标记为E36M62-3和E44M48-1,间距为7.9和21 cM.彭海涛等[48]通过BSA法在不结球白菜的F2群体中筛选得到一个与芜菁花叶病毒抗病基因连锁距离为8.7 cM的SSR标记Ra3E05,而后转化为SCAR标记SCRa2.张慧等[49]利用感病品种T青和抗病品种‘CR’杂交产生的F2群体,采用BSA法成功筛选到一个与根肿病抗病基因遗传距离为9.72 cM的ISSR分子标记.
4 问题与展望
分子标记作为遗传育种工作的重要工具,已经在不结球白菜遗传多样性评价、遗传图谱构建、QTL定位及分子标记辅助育种等研究中展现了巨大潜力,大大地提高了选育的准确性和有效性.与此同时,分子标记在不结球白菜遗传育种研究中也存在以下几点不足.1)不结球白菜的高饱和遗传图谱有待建成.已报道不结球白菜的遗传图谱较少,且半数以上的作图群体都是选用不易保存的杂交后代,导致图谱不能通用并且无法继续饱和原图谱,而多种分子标记混合使用虽然在一定程度上增加了标记的数量,但是并不能满足高密度图谱的需求;2)经济性状相关的QTL位点与分子标记辅助育种之间缺少联系.由于缺少高密度遗传图谱,在很多时候并不能将与经济性状相关的QTL位点进行精确定位,使得筛选得到的分子标记并不能应用于辅助育种;3)与经济性状相关的QTL位点之间以及QTL位点与环境之间存在互做效应,在进行标记辅助育种过程中,由于图谱密度及QTL位点的缺陷,使得分子标记筛选后的个体内QTL位点之间失去互做效应;4)应该充分发挥分子标记在农作物苗期对经济性状的预测作用,在苗期对不结球白菜的抗病性、晚抽薹等重要经济性状进行筛选,能有效提高选育进程并节约资金.随着生物技术的发展和高通量检测的普及,以SNP为代表的第三代分子标记逐渐兴起,将为不结球白菜的遗传育种研究提供新的契机.由于其在稳定性及多态性等方面具有第一代和第二代分子标记无法比拟的优势,使得高密度图谱的构建及功能相关分子标记的开发成为可能.随着检测及分析技术的不断完善,SNP分子标记必将在遗传改良和辅助育种工作中发挥重要作用.
[1] 侯喜林.不结球白菜育种研究新进展 [J].南京农业大学学报,2003,26(4):111-115.
Hou X L.Advances in breeding of non-heading Chinese cabbage [J].Journal of Nanjing Agricultural University,2003,26(4):111-115.
[2] 曹寿椿,朱月林,黄保健,等.不结球白菜抗病育种的研究——Ⅲ白矮抗一号新品种的选育 [J].南京农业大学学报,1991,14(3):25-30.
Cao S C,Zhu Y L,Huang B J,et al.Studies on breeding for disease resistance in non-heading Chinese cabbageⅢ.Breeding of a new cultivar dwarf resistant NO.1 [J].Journal of Nanjing Agricultural University,1991,14(3):25-30.
[3] 侯喜林,曹寿椿,张蜀宁,等.优质不结球白菜新品种暑绿的选育 [J].南京农业大学学报,2005,28(3):30-33.
Hou X L,Cao S C,Zhang S N,et al.Selection of non-heading Chinese cabbage cultivar Shulv with the high-quality [J].Journal of Nanjing Agricultural University,2005,28(3):30-33.
[4] Bostin D,White R L,Skolnick M,et al.Construction of genetic linkage map in man using restriction fragment length polymorphisms [J].American Journal of Human Genetics,1980,32(3):314-331.
[5] Williams J G,Kubelik A R,Livak K J,et al.DNA polymorphisms amplified by arbitrary primers are useful as genetic markers [J].Nucleic acids research,1990,18:6531-6535.
[6] Moore S S,Sargeant L L,King T J,et al.The conservation of dinucleotide microsatellites among mammalian genomes allows the use of heterologous PCR primer pairs in closely related species [J].Genomics,1991,10(3):654-660
[7] Zabeau M,Vos P.Selective restriction fragment amplification :A general method for DNA fingerprinting:EP19910402542 [P].1993-04-01.
[8] Wang D G,Fan J B,Siao C J,et al.Large-scale identification,mapping,and genotyping of single-nucleotide polymorphisms in the human genome [J].Science,1998,280:1077-1082.
[9] 李家文.白菜起源和进化问题的探讨[J].园艺学报,1962,1(3-4):297-304.
Li J W.Discussion on the origin and evolution of cabbage [J].Horticultural Plant Journal,1962,1(3-4):297-304.
[10] 谭俊杰.试论芥(芸苔)属蔬菜的起源与分类 [J].河北农业大学学报,1980,1:104-114.
Tan J J.Discussionon the origin and classification of Brassica [J].Journal of Agricultural University of Hebei,1980,1:104-114.
[11] Wang A,Zhou G,Lin C,et al.Genetic diversity study ofBrassicacampestrisL.ssp chinensis Makino based on ISSR markers [J].Caryologia,2017,70:48-54.
[12] 韩建明,侯喜林,徐海明,等.不结球白菜(Brassicacampestrisssp.chinensisMakino)种质资源SRAP遗传分化分析 [J].作物学报,2007,33(11):1862-1868.
Han J M,Hou X L,Xu H M,et al.Genetic differentiation of non-heading Chinese cabbage (Brassicacampestrisssp.chinensisMakino) germplasm based on SRAP markers [J].Acta Agronomica Sinica,2007,33(11):1862-1868.
[13] 韩建明,侯喜林,徐海明,等.不结球白菜种质资源遗传多样性RAPD分析 [J].南京农业大学学报,2008,31(3):31-36.
Han J M,Hou X L,Xu H M,et al.RAPD analysis of genetic diversity of non-heading Chinese cabbage (Brassicacampestrisssp.chinensisMakino) germplasm [J].Journal of Nanjing Agricultural University,2008,31(3):31-36.
[14] 刘冬媛,王新华,刘杨,等.不结球白菜品种(系)遗传多样性与亲缘关系的SSR分析 [J].分子植物育种,2014,12(3):499-508.
Liu D Y,Wang X H,Liu Y,et al.Analysis of genetic diversity and relationship of pakchoi accessions baased on SSR markers [J].Molecular Plant Breeding,2014,12(3):499-508.
[15] 肖禾,王萌,崔丽洁,等.24份普通白菜遗传多样性的形态特征和RAPD分析 [J].上海师范大学学报(自然科学版),2015,44(6):585-592.
Xiao H,Wang M,Cui L J,et al.Morphological character and RAPD analysis of genetic diversity of different types ofBrassicacampestrisL.ssp.chinensis(L.) Makino var.communisTsee et Lee.pakchoi [J].Journal of Shanghai Normal University (Natural Sciences),2015,44(6):585-592.
[16] 吴耕民.中国蔬菜栽培学 [M].北京:科学出版社,1957.
Wu G M.Olericulture in China [M].Beijing:Science Press,1957.
[17] 曹寿椿,李式军.白菜地方品种的初步研究——Ⅰ方形态学的观察与研究 [J].南京农业大学学报,1980 (2):32-38.
Cao S C,Li S J.A preliminary study on the local variety of Chinese cabbage (BrassicacampestrisL) Ⅰ.Morphological observation and study [J].Journal of Nanjing Agricultural College,1980(2):32-38.
[18] 曹家树,曹寿椿,缪颖,等.中国白菜各类群的分支分析和演化关系研究 [J].园艺学报,1997,24(1):36-43.
Cao J S,Cao S C,Miao Y,et al.Cldistic operational analysis and study on the evolution of Chinese cabbage groups (BrassicacampestrisL) [J].Acta Horticulturae Sinica,1997,24(1):36-43.
[19] 曹家树,曹寿椿,易清明.白菜及其相邻类群基因组DNA的RAPD分析 [J].园艺学报,1995,22(1):47-52.
Cao J S,Cao S C,Yi Q M.RAPD analysis of genomic DNA in Chinese cabbage and its adjacent taxa [J].Horticultural Plant Journal,1995,22(1):47-52.
[20] 孙德岭,赵前程,宋文芹,等.白菜类蔬菜亲缘关系的AFLP分析 [J].园艺学报,2001,28(4):331-335.
Sun D L,Zhao Q C,Song W Q,et al.Relationships analysis of Chinese cabbage species by AFLP [J].Acta Horticulturae Sinica,2001,28(4):331-335.
[21] 郭晶心,周乃元,马荣才,等.白菜类蔬菜遗传多样性的AFLP分子标记研究 [J].农业生物技术学报,2002,10(2):138-143.
Guo J X,Zhou N Y,Ma R C,et al.Genetic diversity inBrassicaraparevealed by AFLP molecular markers [J].Journal of Agricultural Biotechnology,2002,10(2):138-143.
[22] 单晓政,侯喜林,李英,等.不结球白菜SRAP体系优化与品种聚类分析 [J].江苏农业学报,2009,25(3):610-615.
Shan X Z,Hou X L,Li Y,et al.Optimization of SRAP system and cluster analysis of non-heading Chinese cabbage (BrassicacampestrisL.ssp.chinensis) [J].Jiangsu Journal of Agricultural Sciences,2009,25(3):610-615.
[23] 轩淑欣,李向,顾爱侠,等.基于SSR标记的不结球白菜品种资源的聚类分析 [J].北方园艺,2014(3):83-87.
Xuan S X,Li X,Gu A X,et al.Cluster analysis of non-heading Chinese cabbage cultivars based on SSR marker [J].Northern Horticulture,2014(3):83-87.
[24] Staub J E,Serquen F C,Gupta M.Genetic markers,map construction,and their application in plant breeding [J].Hortscience,1996,31:729-741.
[25] 耿建峰,侯喜林,张晓伟,等.利用DH群体构建不结球白菜遗传连锁图谱 [J].南京农业大学学报,2007,30(2):23-28.
Geng J F,Hou X L,Zhang X W,et al.Construction of a genetic linkage map in non-heading Chinese cabbage (Brassicacampestrisssp.chinensis) using a doubled haploid (DH) population [J].Journal of Nanjing Agricultural University,2007,30(2):23-28.
[26] Geng J F,Zhu C S,Zhang X W,et al.A genetic linkage map of nonheading Chinese cabbage [J].Journal of the American Society for Horticultural Science,2007,132:816-823.
[27] 单晓政,李英,高素燕,等.不结球白菜分子遗传图谱的构建及分析 [J].西北植物学报,2009,29(6):1116-1121.
Shan X Z,Li Y,Gao S Y,et al.Molecular genetic map ofBrassicacampestrisssp.chinensis[J].Acta Botanica Boreali-Occidentalia Sinica,2009,29(6):1116-1121.
[28] Cheng Y,Geng J F,Zhang J Y,et al.The Construction of agenetic linkage map of non-heading Chinese cabbage (BrassicacampestrissspchinensisMakino) [J].Journal of Genetics and Genomics,2009,36:501-508.
[29] 班青宇.利用F_2群体构建不结球白菜遗传连锁图谱及抽薹性QTL定位 [D].南京:南京农业大学,2009.
Ban Q Y.Construction of a genetic linkage map and mapping QTL of bolting character using F2population in non-heading Chinese cabbage (Brassicacampestrisssp.Chinensis) [D].Nanjing:Journal of Nanjing Agricultural University,2009.
[30] 王倩.不结球白菜遗传图谱构建及重要农艺性状的QTL定位 [D].南京:南京农业大学,2011.
Wang Q.Construction of a genetic linkage map and mapping QTL of important agronomic traits in non-heading Chinese cabbage [D].Nanjing:Journal of Nanjing Agricultural University,2011.
[31] 陆鹏.不结球白菜优异种质对小菜蛾抗性的遗传分析及QTL定位 [D].北京:中国农业科学院,2010.
Lu P.Genetic analysis and QTL mapping of the resistance to diamondback moth (Plutella xylostella L.) in elite non-heading Chinese cabbage (Brassicacampestrisssp.Chinensis) Germplasms [M].Beijing:Chinese Academy of Agricultural Sciences,2010.
[32] 李向.不结球白菜的群体结构分析及分子遗传图谱的构建 [D].保定:河北农业大学,2010.
Li X.Analysis of population structure and construction genetic linkage map in non-heading Chinese cabbage [D].Baoding:Agricultural University of Hebei,2010.
[33] Wang X H,Liu Y,Chen H Y.A linkage map of pak-choi (Brassicarapasspchinensis) based on AFLP and SSR markers and identification of AFLP markers for resistance to TuMV [J].Plant Breeding,2011,130:275-277.
[34] 朱玉英,张素琴,凌超,等.不结球白菜高代自交系杂交育种的效应及其利用 [J].上海农业学报,1994,10(2):33-36.
Zhu Y Y,Zhang S Q,Lin C,et al.Study on effects and utilization of cross-breeding of generations self-lines in nonheading Chinese cabbage [J].Acta Agricul Turae Shanghai,1994,11:3120-3123.
[35] 耿建峰.利用DH群体构建不结球白菜遗传连锁图谱及重要农艺性状QTL定位 [D].南京:南京农业大学,2007.
Geng J F.Construction of a genetic linkage map and mapping QTL of important agronomic traits using DH population in non-heading Chinese cabbage (Brassicacampestrisssp.chinensis) [D].Nanjing:Nanjing Agricultural University,2007.
[36] 成妍.不结球白菜分子遗传图谱构建及数量性状位点分析 [D].南京:南京农业大学,2009.
Cheng Y.Construction of molecular genetic MAP and analysis of QTL in non-heading Chinese cabbage [D].Nanjing:Nanjing Agricultural University,2009.
[37] 单晓政.不结球白菜遗传图谱的构建及Vc性状的QTL定位 [D].南京:南京农业大学,2009.
Dan X Z.Construction of a genetic linkage map and mapping QTL of Vc trait in non-heading Chinese cabbage [M].Nanjing:Nanjing Agricultural University,2009.
[38] 班青宇,耿建峰,侯喜林,等.不结球白菜叶片脯氨酸与可溶性蛋白含量的QTL分析 [J].南京农业大学学报,2010,33(2):35-38.
Ban Q Y,Geng J F,Hou X L,et al.QTL mapping for proline and soluble protein content of leaves in non-heading Chinese cabbage [J].Journal of Nanjing Agricultural University,2010,33(2):35-38.
[39] Wang Q,Hou X L,Geng J F,et al.Quantitative trait loci for photosynthetic pigment concentration in a doubled-haploid population of Pak Choi (Brassicarapasspchinensisvar.communis Tsen et Lee) [J].Journal of Horticultural Science & Biotechnology,2010,85:421-426.
[41] Shi G J,Hou X L.Identification of a RAPD marker tightly linked to a self-incompatibility gene in non-heading Chinese cabbage [J].Acta Horticulturae,2004,1:279-283.
[42] 葛婷婷.不结球白菜‘矮脚黄’自交不亲和性的SSR分子标记 [D].南京:南京农业大学,2013.
Ge T T.SSR molecular marker of self-incompatibility gene in ′AIJIAOHUANG′ a cultivar of Non-heading chinese cabbage [D].Nanjing:Nanjing Agricultural University,2013.
[43] Li X,Wang A H,Zu F,et al.Identification of a nuclear-recessive gene locus for male sterility on A2 chromosome using the Brassica 60 K SNP array in non-heading Chinese cabbage [J].Genes & Genomics,2016,38:1151-1157.
[44] 贾永鹏,李霞,周国林,等.小白菜自交不亲和性相关基因的分子标记开发 [J].长江蔬菜,2016,8:38-41.
Jia Y P,Li X,Zhou G L,et al.Molecular marker development of self-incompatibility related genes in Chinese cabbage [J].Journal of Changjiang Vegetables,2016,8:38-41.
[45] 张波.不结球白菜晚抽薹分子标记及抽薹性遗传分析 [D].南京:南京农业大学,2007.
Zhang B.Molecular markers of late-bolting and inheritance for date of bolting in non-heading Chinese [D].Nanjing:Nanjing Agricultural University,2007.
[46] Du Z X,Hou R X,Zhu Y Y,et al.A random amplified polymorphic DNA (RAPD) molecular marker linked to late-bolting gene in pak-choi (Brassica campestris ssp chinensis Makino L.) [J].African Journal of Biotechnology,2011,10:7962-7968.
[47] 冷月强.不结球白菜霜霉病抗性机制及其分子标记 [D].南京:南京农业大学,2006.
Leng Y Q.Study on mechanisms and molecular markers resistance to downy mildew innon-heading Chinese cabbage [D].Nanjing:Nanjing Agricultural University,2006.
[48] 彭海涛,宋小明,侯喜林,等.不结球白菜抗芜菁花叶病毒基因的遗传分析及SSR标记 [J].江苏农业学报,2012,28(2):396-401.
Peng H T,Song X M,Hou X L,et al.Inheritance and SSR marker of resistance to turnip mosaic virus in nonheading Chinese cabbage [J].Jiangsu Journal of Agricultural Sciences,2012,28(2):396-401.
[49] 张慧,徐海,况媛媛,等.不结球白菜抗根肿病基因的分子标记 [J].江苏农业学报,2013,29(3):633-636.
Zhang H,Xu H,Kuang Y Y,et al.Molecular marker of clubroot resistance genes inBrassicacampestrisssp.chinensis[J].Jiangsu Journal of Agricultural Sciences,2013,29(3):633-636.
(责任编辑:顾浩然,冯珍珍)
ApplicationprogressofDNAmolecularmarkersinnon-headingChinesecabbagegeneticbreeding
Li Mengmeng, Cui Lijie, Qian Zhongying, Kai Guoyin*, Pan Jingxian*
(Development Center of Plant Germplasm Resources,College of Life and Environmental Sciences,Shanghai Normal University,Shanghai 200234,China)
This paper reviews the classification and characteristics of DNA molecular markers in non-heading Chinese cabbage (Brassicacampestrisssp.chinensisMakino) genetic breeding,and its application in genetics diversity analysis,genetic map construction,quantitative trait locus (QTLs) mapping and molecular marker-assisted selection.Finally,some limitations of DNA molecular markers on genetic improvement and assistant breeding of non-heading Chinese cabbage are pointed out,and some suggestions are put forward,aiming to provide valuable information for genetic breeding of non-heading Chinese cabbage.
molecular marker; genetic breeding; non-heading Chinese cabbage
S 634.3
A
1000-5137(2017)05-0720-09
2017-08-31
上海市科委项目(17JC1404300,15430502700);上海植物种质资源工程技术研究中心项目(17DZ2252700)
李蒙蒙(1986-),女,硕士研究生,主要从事植物次生代谢产物分子方面的研究.E-mail:1562708056@qq.com
*
开国银(1977-),男,博士,教授,主要从事植物分子生物学及次生代谢工程等方面的研究.E-mail:kaiguoyin@163.com;潘静娴(1963-),女,博士,副教授,主要从事功能基因组学方面的研究.E-mail:panjingx@shnu.edu.cn
——庆祝南京农业大学建校120周年

