面向社交网络的潜在药物不良反应发现
赵明珍,林鸿飞,徐 博,郝辉辉
(大连理工大学 信息检索实验室,辽宁 大连 116024)
面向社交网络的潜在药物不良反应发现
赵明珍,林鸿飞,徐 博,郝辉辉
(大连理工大学 信息检索实验室,辽宁 大连 116024)
随着互联网的发展,社交网络中积累了大量的医疗健康领域的文本数据。该文利用基于信息熵的方法,从健康社交网络中的用药者评论数据中识别药物的潜在不良反应;同时,对于潜在药物不良反应,该文提出了基于Word2vec和Skip-gram模型的蛋白质关联紧密度函数,尽最大努力发现药物引起其“潜在”不良反应的证据链。实验证明,该方法用来寻求潜在药物不良反应证据链是有效的。
社交网络;药物不良反应;信息熵;Word2vec;Skip-gram
1 引言
目前,药物不良反应(adverse drug reactions,ADRs)已经成为医学界和民众关注的热点,用药安全问题日益得到全社会的重视。由于药物开发时试验人群的数量及试验周期等限制,会造成具有潜在药物不良反应的新药流入市场的可能。因此,如何判断和预测药物的不良反应具有重大的理论价值和实用价值。
随着互联网的迅速普及,出现了很多关于医疗健康的社交网站,例如,DailyStrength, MedHelp, Healthamp;Wellness, Yahoo!Group, Ask a Patient等。用户可以在健康社交网站上建立自己的“好友圈”,讨论各种与健康相关的话题,例如对药品或疾病发表自己用药的体验和评价。这些社交网站积聚了大量来自用户的医疗健康文本数据。与传统的报告制度相比,这些信息是来自用药者的第一手资料,更为充分、及时,传播更快。
一方面,社交网络中积累的医疗健康数据隐藏了丰富的有待挖掘的“知识”;另一方面,在社交网络中,用户的用语很随意,经常出现拼写错误和语法错误,这些弊端给社交网络中的文本挖掘带来很大的挑战。到现在为止,从健康社交网站中提取药物不良反应的研究还相对较少。
Leaman[1]使用基于滑动窗口的字典匹配方式识别用药者评论数据中的不良反应,从结果中可以看出,从用药者评论中识别潜在不良反应是可行的。Azadeh Nikfarjam[2]从Leaman使用的标注数据集中提取用户表达不良反应的语言模式,在这些模式的基础上利用关联规则的方法从用药者评论中提取药物不良反应,在同一数据集上,他们的结果同Leaman的结果相比,略有下降,但优点是无需使用词典。Andrew Yates[3]标注了250条关于乳腺癌药物的用药者评论,并在此基础上构建ADRTrace系统,从训练数据集中提取不良反应出现的模式来识别不良反应。但该系统训练数据少,并且只是针对乳腺癌相关的药物,泛化性较差。Jiang Bian[4]利用自然语言处理和支持向量机等技术从Twitter挖掘药物不良反应,但效果不是很好。因为Twitter是开放性的社交网站,不是专门面向“医疗卫生”领域,所以噪声很多,对分类器的影响很大。
基于社交网络可以很快地收集到药物的潜在不良反应信息,但是由于用户的报告是根据个人的意愿和表现,这些潜在不良反应并没有经过严格医学意义上的检验。所以能否成为药物在医学意义上的药物不良反应还需要进一步检验和证实,也需要一定的解释机制。如果可以为潜在药物不良反应找到某些原因,例如某种蛋白质,那么就可以大大地减少医学专业人士用来确定药物不良反应的时间,这对不良反应的实时监测是非常重要的。
本文首先利用基于信息熵的非监督不良反应识别方法从健康社交网络的用药者评论数据中识别潜在的药物不良反应。然后在生物医学文献数据库MEDLINE的基础上,使用修改的Skip-gram[5]模型,寻求可以把药物和不良反应联系起来的蛋白质,尽最大努力发现药物引起不良反应的内部机制,减少领域专家最终确定药物不良反应的时间和经济代价。
2 研究框架
本文的研究目的是从健康社交网站的用药者评论数据中识别潜在药物不良反应,并尽最大努力为发现的潜在药物不良反应提供蛋白质级别的证据。因此,本文系统主要包括三部分: 数据获取模块,潜在不良反应识别模块和寻求关联蛋白质模块,如图1 所示。
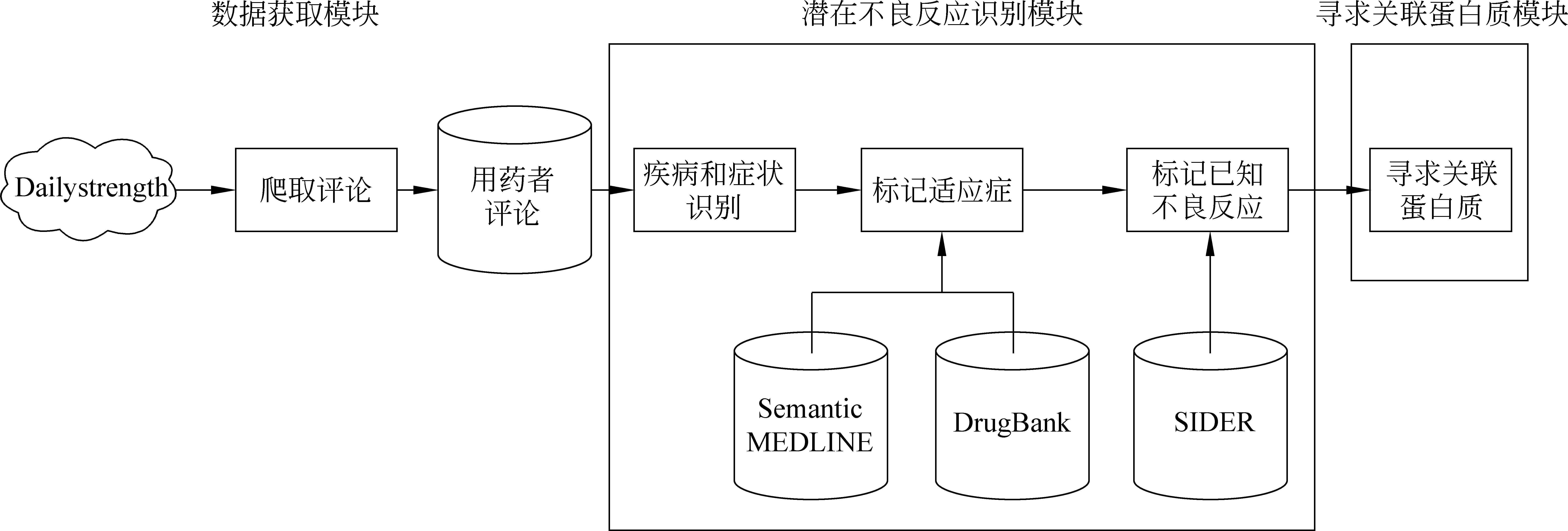
图1 系统流程图
(1) 数据获取模块
本文利用scrapy*http://scrapy.org/程序包搭建网络爬虫,从社交网站中获取相应的用药者评论。
(2) 潜在不良反应识别模块
该模块首先利用信息熵的原理从用药者的评论数据中识别疾病和不良反应实体,然后使用DrugBank[6-9]和Semantic MEDLINE[10-12]对药物的适应症进行过滤,并利用SIDER[13]对已登录的药物不良反应进行过滤,最终得到潜在药物不良反应。
(3) 寻求关联蛋白质模块
在本文中,关联蛋白质是指可以把药物与不良反应联系起来的蛋白质,某种程度上,关联蛋白质可以解释药物引起不良反应的原因。如果能够为潜在药物不良反应找到关联蛋白质,对于潜在药物不良反应在医学意义上的最终确定具有重要的意义。本文提出了基于Skip-gram[5]模型的生物实体关联度计算方法,并在此基础上定义了关联蛋白质的关联紧密度函数,以此来寻求潜在药物不良反应的关联蛋白质。
3 识别潜在药物不良反应
疾病名称和不良反应名称具有很大的重叠性,如headache可以是疾病的名称,也可以是不良反应的名称。本文的主要研究目的是识别用户评论中的潜在不良反应,所以对于药物的适应症(疾病)和药物的不良反应应加以区分,实现过滤适应症、识别不良反应的目标。在本文中,识别潜在药物不良反应在整体上包括三部分: 识别“疾病和不良反应”名称,过滤药物适应症(疾病名称),以及过滤已知的不良反应。
识别“疾病和不良反应”名称,包括构建词典和名称识别两部分;对于药物适应症过滤,本文使用已有的生物医学资源DrugBank和Semantic MEDLINE进行药物适应症过滤。同时,为了实现识别药物的“潜在”不良反应,需要对已知的不良反应加以标记。具体的,本文使用药物不良反应数据库SIDER标记已知的药物不良反应并加以过滤,从而得到药物的“潜在”不良反应列表。
3.1 生成疾病和不良反应词典
本文使用的疾病和不良反应词典IndSyn是基于SIDER[13]数据库生成的,该数据库中包含5 719种不良反应名称和2 669种适应症的名称。通过合并,得到“疾病和药物”词典IndSyn。由于不良反应和疾病有交集,词典IndSyn包含6 315种疾病和不良反应实体名称。
3.2 基于信息熵的疾病和不良反应实体识别
从用药者评论中识别疾病和不良反应实体,可以理解为: 从用药者评论中提取表达疾病和不良反应的文本片段,这些文本片段应该具有高频率和高信息熵的特点。一个文本片段的信息熵越高,说明这个片段是一个“词”的概率就越大。
信息熵被广泛地用于微博数据中的新词发现和关键词提取[14-15],在本文中我们使用信息熵来识别候选疾病和不良反应实体。具体地,假设s表示一个文本片段,L表示s在评论数据中的左邻接词集合,R表示s在评论数据中的右邻接词集合。
s的左信息熵定义如式(1)所示。
其中p(w)表示w是s的左邻接词的概率。
同理,s的右信息熵定义如式(2)所示。
其中p(w)表示w是s的右邻接词的概率。
如果s的左信息熵和右信息熵都比较高,那么s表示一个词的概率就很大。但是本文的目的是识别用药者评论中的疾病和不良反应实体,而不是对用药者评论进行分词,所以对于信息熵较高的s要进行过滤,如果s可以映射到“疾病和不良反应”词典IndSyn中的某一项,则保留s,否则去除s。
本文利用Jaccard相似性系数作为文本重叠度函数,将文本s映射到词典IndSyn中。具体的,设t∈IndSyn,表示一种疾病或者症状,定义文本片段s和词典项t的重叠度如式(3)所示。
其中Ws和Wt表示对文本s和t进行分词和去停用词后包含的单词集合。|W|表示集合W所包含的元素个数。令map(s)表示s映射到词典IndSyn中的项,则map(s)定义如式(4)所示。
如果map(s)!=NULL,则表示文本s可以映射到词典IndSyn中,即s是疾病名称或者不良反应名称。
3.3基于DrugBank和SemanticMEDLINE的“适
应症”标记
在“疾病和不良反应”名称识别的基础上,需要过滤药物的适应症。用药者在分享用药经历或者评论某种药物时,不可避免地会提到该药物的适应症或者用药的原因。比如药物trazodone的一条评论: “Iusethisprimarilyformysleeplessness”,明确地说明sleeplessness是用药的原因,不是trazodone的不良反应。所以,应当从识别出的“疾病和不良反应”实体中标记药物的适应症,并将其过滤掉。
药物的适应症可以从DrugBank数据库中得到。DrugBank数据库中药物的适应症描述是非结构化的,例如:trazodone的适应症为 “Forthetreatmentofdepression”。在本文中,我们使用MetaMap*http://mmtx.nlm.nih.gov/从DrugBank的适应症描述中识别出相关的疾病实体,并使用词典IndSyn去除非疾病和不良反应实体,从而得到药物的适应症。
除了药物说明书上的适应症,药物经常还有其他的适应症。比如从上述评论中我们还可以看出trazodone除了治疗depression之外,还可以用于治疗sleeplessness。所以本文还用SemanticMEDLINE对药物适应症做进一步过滤。SemanticMEDLINE是SemRep[16]从MEDLINE引用中识别出的三元语义关系(subject-predicate-object)知识库,这些三元组表示subject和object之间的语义关系为predicate。例如,如果在SemanticMEDLINE中存在三元语义关系: (trazodone-TREATS-sleeplessness),我们就可以断定trazodone可以用来治疗sleeplessness,从而说明sleeplessness也是trazodone的适应症。
综上,本文使用DrugBank和SemanticMEDLINE相结合的方法来标记药物的适应症。
3.4基于SIDER的已知药物不良反应标记
经过“适应症”过滤,就可以得到与药物有关的不良反应。然而这些不良反应有很多在药物的说明书中已有记录,本文称其为已知的药物不良反应。SIDER[13]是从药物说明书中提取的药物不良反应数据库。本文的目的是识别“潜在”的药物不良反应,所以本文使用SIDER数据库对已知的药物不良反应进行标记,并将其过滤掉。
3.5“潜在”药物不良反应的标记
经过适应症和已知不良反应的标记,剩下未标记的“疾病和不良反应”实体就可以作为“潜在”的药物不良反应。图2是识别潜在药物不良反应的详细算法。

输入:药品di的用药者评论数据集Ci输出:“疾病和不良反应”实体名称,以及每个实体与药品di的关系1:在集合Ci中生成文本片段集合Si={s|LE(s)gt;0,RE(s)gt;0}2:计算平均频率:fre=1Si∑s∈Sifre(s),其中fre(s)表示s出现的次数。3:计算平均左右信息熵:LE=1Si∑s∈SiLE(s)和LR=1Si∑s∈SiLR(s)4:生成疾病和不良反应集合:Di={l=map(s)|fre(s)gt;fre,LE(s)gt;LE,RE(s)gt;RE,map(s)!=NULL,s∈Si}5: forem∈Di6: ifem是药品di的适应症,则将em标记为”-”7: elseifem是药品di的已知不良反应,则将em标记为”+”8: else将em标记为”*”图2 识别潜在不良反应的算法
4 寻求潜在不良反应的证据
不良反应识别完成之后,可以得到药物的潜在不良反应列表。然而所识别出的潜在不良反应尚未经过严格医学意义上的检验,所以能否成为临床意义上的不良反应还需要进一步的检验和证实。检测潜在不良反应的真实性需要进行大量的临床试验和观察,这是一个耗时耗力的过程,不利于及时发现药物的安全隐患。如果可以利用文本挖掘的相关技术,挖掘药物导致潜在不良反应的内部机制,并推荐给医学领域的专家作参考,对于检验潜在不良反应的真实性,改善用药安全,具有重要意义。
蛋白质是生命活动的主要承担者,是生命的物质基础,因此药物的不良反应也多与蛋白质有关。为了便于研究,本文假定药物通过蛋白质引起不良反应。因此,对于上一步发现的“潜在”药物不良反应,本文尽最大努力找到可以把药物和其“潜在”不良反应联系起来的蛋白质,把这些蛋白质作为药物导致不良反应的“证据”,并把这些(药物,蛋白质,不良反应)作为三元组关系,推荐给医学领域的专家作参考,为他们最终确定药物和不良反应的关系减少时间和经济代价。
为了寻求关联蛋白质,本文首先利用Skip-gram模型生成生物医学实体的分布式向量,根据实体向量计算实体之间的关联度,在此基础上,根据药物-蛋白质-不良反应三者之间的关联度函数挖掘关联蛋白质。
4.1基于Skip-gram模型的生物实体关联度
传统计算实体x和y的关联度的方法(如点间互信息PMI)直接基于x和y的“共现”情况来计算,如果x和y经常共现,那么它们之间的关联度也越高。这种方法过于简单,会带来很多噪声。为了更好地计算实体之间的关联度,本文采用基于Word2vec的Skip-gram[5]模型生成生物医学实体的分布式向量,通过实体向量计算实体之间的关联度。
为了训练Skip-gram模型,本文选择的数据是MEDLINE文献引用的MeSH词域。生物医学专家为MEDLINE中每篇文献都使用某些MeSH词进行标注,这些MeSH词能很好地描述论文的主题内容,同时这些MeSH词也可以作为共现信息来使用。
原始的Skip-gram模型是一种语言模型,所需要的训练数据是“有序”的自然语言文本,然而MEDLINE文献引用的MeSH词域是MeSH词集合,是“无序”的。所以,需要对Skip-gram进行修改,使其可以利用MEDLINE文献引用的MeSH词域进行训练,生成MeSH词的词向量。
具体的,令pi表示第i篇包含MeSH词的MEDLINE引用,Si表示pi的MeSH词集合,mij表示pi中第j个MeSH词,j=1,2,…,|Si|,|Si|表示集合Si的大小。修改后的Skip-gram模型的目标函数如式(5)所示。
其中N表示包含MeSH词的所有MEDLINE文献总数。
通过上述修改后的Skip-gram模型,就可以得到每个MeSH词的词向量。MeSH词的关联度定义为余弦相似度,如式(6)所示。
4.2关联度
对于蛋白质p,本文利用关联度函数f(d,p,a)来衡量其作为药物d和“潜在”不良反应a的“证据”的可信度。f(d,p,a)越大,表示蛋白质p越能把药物d和“潜在”不良反应a联系起来,也就表示蛋白质p作为“证据”越可信,从而蛋白质p越能解释药物d和“潜在”不良反应a的内部机制。本文将这种蛋白质称为关联蛋白质。
具体的,f(d,p,a)的定义如式(7)所示。
其中sim(x,y)表示实体x和y的关联度。直观上,如果sim(d,p)+sim(p,a)越高,那么关联紧密度f(d,p,a)也越高。但是为了防止因sim(d,p)或者sim(p,a)单方过高而导致的f(d,p,a)过高,这里对其使用1+|sim(d,p)-sim(p,a)|进行“平滑”。即: 如果sim(d,p)+sim(p,a)很高,并且sim(d,p)和sim(p,a)差异很小,f(d,p,a)才会高。
5 实验结果分析
针对本文提出的方法,本文共进行了三个实验: 实验一主要用于说明基于信息熵的方法可以有效地进行不良反应名称识别,并分析了在社交网络中潜在不良反应发现的结果;实验二用于说明修改后的Skip-gram模型在MEDLINE数据集上可以捕捉生物医学实体之间的关联度,从而可以用于发现药物和不良反应之间的关联蛋白质;实验三给出了为潜在药物不良反应寻找“证据链”的结果,说明基于MeSH词向量的关联度函数可以有效地发现药物和不良反应的关联蛋白质,为领域专家尽早确定潜在药物不良反应的真实性提供参考依据。
5.1不良反应识别结果
由于在“社交网络中识别药物不良反应名称”这一领域缺乏权威的数据集,为了便于比较,本文选择Leaman[1]所用的健康社交网站Dailystrength*http://www.dailystrength.org/作为用户评论数据的来源。
具体的,在本文中,使用基于Python的scrapy爬虫框架,以2014年6月2日为截止日期,从Dailystrength中爬取600237条用药者评论。这些评论中,总共涉及1075个健康话题,其中绝大数是关于药物的话题。本文选取评论数最多的50种药物作为研究对象。
表1是与Leaman[1]结果的对比情况。从中可以看出,本文的结果与Leaman[1]的结果相似性很高,说明本文使用的基于信息熵和词典匹配的不良反应名称识别方法是有效的。其中识别错误主要源于词典IndSyn中某些名称包含很多停用词。如表1中的“notaseffective”,去停用词后变为“effective”,从而导致包含effective的用户评论都会识别出该不良反应。另一主要的识别错误是由于词典IndSyn中某些名称是由常用词组成的,而且相对较短。如“feelinghigh”和“effectincrease”,在词典映射过程中,feeling和feelings等都会映射到“feelinghigh”,“increase”和“increased”等都会映射到“effectincrease”,从而导致识别错误。
表2是基于信息熵的方法对于上述50种药物的不良反应识别结果。本实验从50种药物的评论数据中抽取出993个(药物,疾病或症状)关系。总共识别出265个适应症关系,其中DrugBank标记出34个,SemanticMEDLINE标记出234个,所占的百分比分别为3.4%和23.3%。对于药物不良反应关系,240个在SIDER中有相应的记录,而488个在SIDER中并没有记录,所占的百分比分别为24.2%和49.1%。这488个未记录的药物不良反应就是“潜在”的药物不良反应。
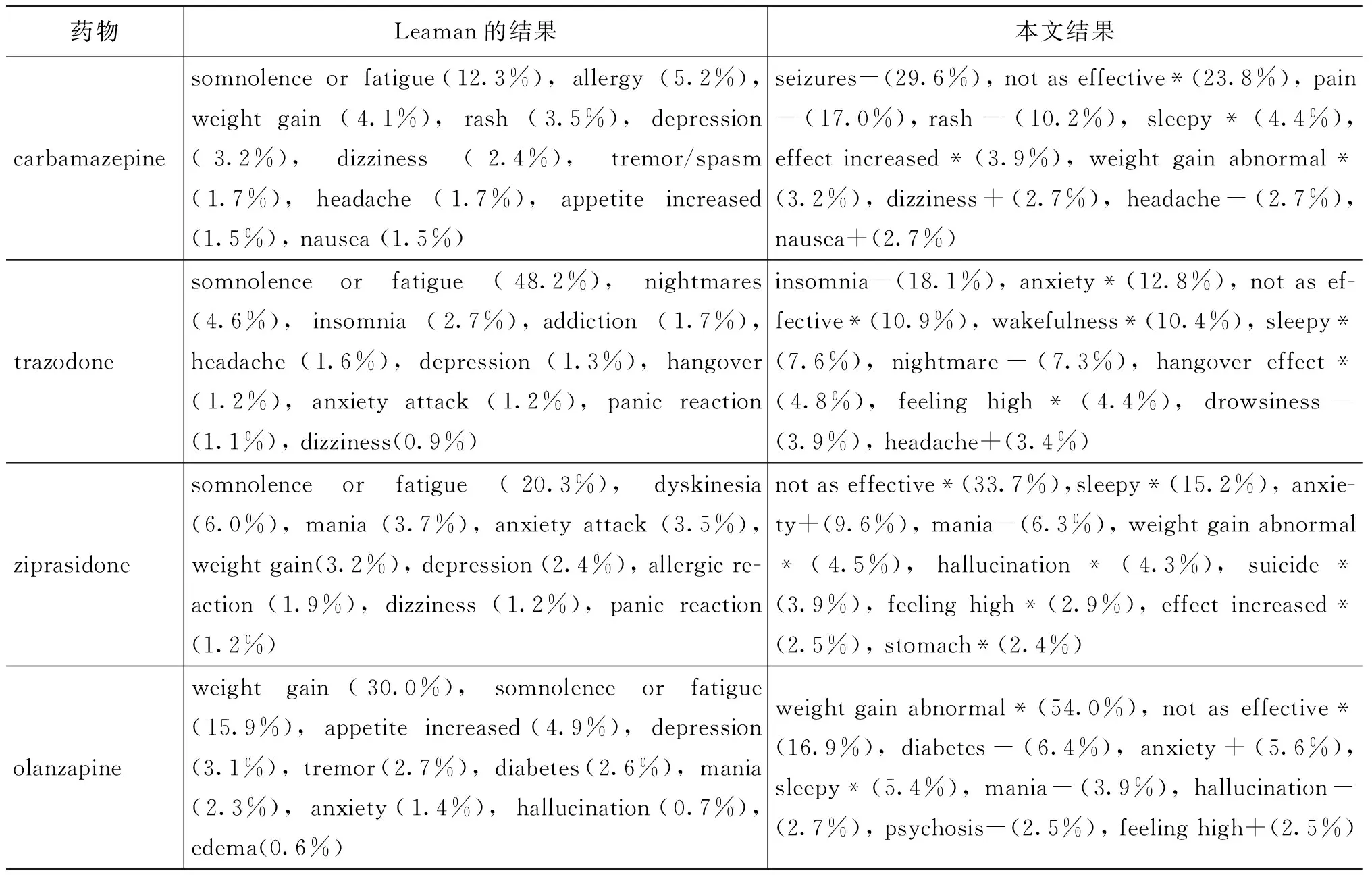
表 1 与Leaman[1]的结果对比
注: 在本文方法的识别结果中,疾病和不良反应名称使用“+”,“-”,“*”来标记。“-”表示适应症。“+”表示已知的药物不良反应;“*”表示“潜在”的不良反应。

表 2 用户评论中“疾病和不良反应”的分布
从表2的结果可以看出,在社交网络中,用药者更倾向于“陈述”药品说明书中未记录的药物不良反应,这也符合实际情况。如果药品说明书中已经说明了某种不良反应,则用户就不会过分“担心”这种不良发应,在心理上甚至认为这种不良反应在某种程度上是“正常”的。相反,如果药品说明书中没有出现某种不良反应,而用药者自身出现了该不良反应,则其更倾向于“寻求”帮助和分享自己的经历。
对于“潜在”不良反应(d,a),首先在Semantic MEDLINE中检索是否存在三元组(d,CAUSES,a),如果存在,则说明药物d会引起不良反应a。从Semantic MEDLINE中总共可以为10个“潜在”不良反应找到上述三元组。由于篇幅限制,表3展示了其中三个不良反应关系。
5.2 MeSH词向量
本实验以点间互信息(point mutual information,PMI)方法作为对比,用来说明修改后的Skip-gram模型可以有效地捕捉MeSH词之间的关联度。

表3 Semantic MEDLINE中寻求(d,CAUSES,a)的部分结果
注: drowsiness和somnolence都是“睡意,困倦”的意思
MEDLINE通过人工标注的方式为每篇文献赋予一些足以描述论文主题的MeSH词,每篇引用的MeSH词组成一个共现集合。在本实验中,选取2013年(含2013年)之前的含有MeSH域的2 200万篇MEDLINE引用作为基础,并从中抽取相应的MeSH词共现集合组成训练数据集。在训练修改的Skip-gram模型时,本文采用Word2vec的Hierarchical Softmax算法,生成的MeSH词向量为100维。表4给出了两个模型分别用于求Headache和Snake venoms最相近的10个MeSH词的结果。
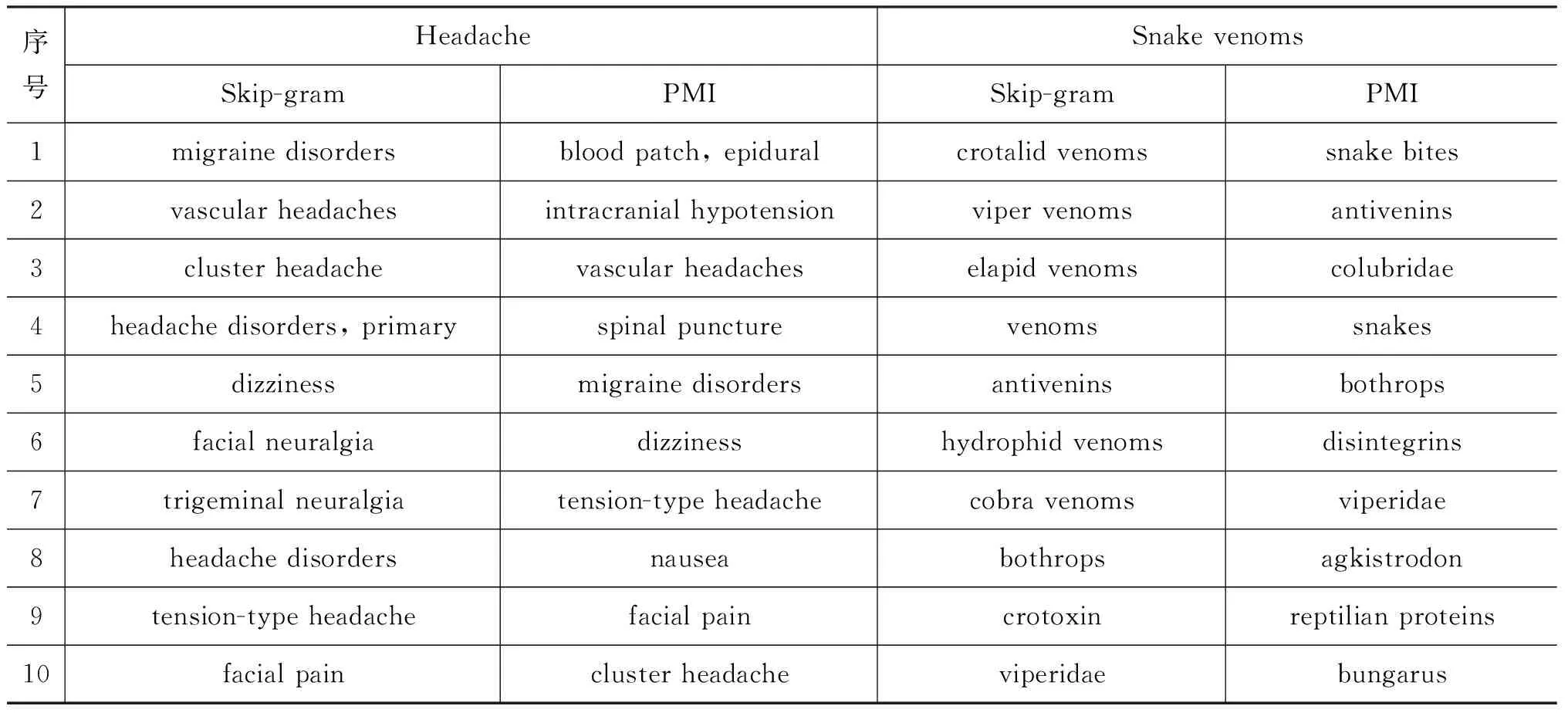
表4 修改的Skip-gram模型与PMI模型对比
在Headache、Skip-gram得到的词中,六个是具体的头痛类型,还得到facial neuralgia和trigeminal neuralgia等与Headache语义很相似的词。而PMI中只有三个是具体的头痛类型,而且PMI模型得到的词中,nausea、spinal puncture(脊髓穿刺)等词跟headache是无关的,可见相较于Skip-gram,PMI会引入更大的“噪声”。
对于Snake venoms(蛇毒),两个模型所得到的MeSH词都是相关的。Skip-gram模型得到四个是具体的蛇毒类型,一个毒液的总称,并得到了抗蛇毒素和响尾蛇毒蛋白。只有蝮蛇科、具窍蝮蛇属和竹叶青蛇属与蛇毒的“相似性”差些。而PMI模型得到的只有抗蛇毒素、单链蛇毒多肽和爬虫类蛋白质与蛇毒相近,其他的词更大程度上跟“蛇”相近。
通过这两个简单的例子可以看出,经过修改的Skip-gram模型可以有效地计算MeSH词之间的关联度,并且引入的噪声相对较少。
5.3 寻求“潜在”不良反应的“证据”
在潜在药物不良反应识别和MeSH词向量的基础上,本文尽最大努力挖掘药物和其潜在不良反应之间的关联蛋白质。本实验用于展示关联蛋白质的挖掘结果。
为了使用上述修改的Skip-gram模型生成的MeSH词向量来计算实体之间的关联度,对于药物d和不良反应a,需要对其使用Restrict to MeSH算法将其映射为MeSH词。对于上述488个“潜在”不良反应关系,其中10个已经在Semantic MEDLINE中找到依据,对于剩余的478个“潜在”不良反应关系,Restrict to MeSH算法将其中的160个关系成功地使用MeSH词来表示。
对于每个“潜在”的药物不良反应关系(d,a),本文选取f(d,p,a)最高的五个蛋白质作为药物d和不良反应a的关联蛋白质。表5是trazodone和anxiety关联蛋白质的提取结果。

表 5 Trazodone和anxiety的关联蛋白质提取结果
Trazodone是一种抗抑郁药,属于5-hydroxytryptophan受体拮抗剂和再摄取抑制剂,也是serotonin摄取抑制剂。此外,trazodone也会阻塞alpha-adrenergic,对alpha2-adrenergic有一定的阻塞作用。
下面主要对不良反应anxiety进行论述。
Gingrich[17]指出“To date several inactivation mutations of specific serotonin receptors have been generated producing interesting behavioral phenotypes related to anxiety, depression, drug abuse, psychosis, and cognition”,可以看出serotonin receptors与anxiety是相关的。
Goldman[18]指出“HTTLPR (minor allele frequency 0.40) alters serotonin transporter function to affect anxiety, dysphoria and obsessional behavior, which are assessed in COMBINE and may be related to relapse and addictive behavior”。可以看出,serotonin transporter也会影响anxiety。可以推断,作为特殊的serotonin transporter,serotonin plasma membrane transport proteins跟anxiety也是相关的。
Shishkina[19]指出“Brain alpha2-adrenergic receptors (alpha2-ARs) have been implicated in the regulation of anxiety, which is associated with stress”,说明adrenergic receptors跟anxiety是有联系的。
通过以上简要分析,本文找到了trazodone和anxiety之间的三个三元关系组: (trazodone,“serotonin receptors”,anxiety),(trazodone,“serotonin plasma membrane transport proteins”,anxiety),(trazodone,“adrenergic receptors”,anxiety),这些三元关系组为trazodone和anxiety关系的确定提供了参考。
6 结论
本文旨在从社交网络中提取药物的不良反应,并为“潜在”的不良反应寻求蛋白质级别的“证据”,尽最大努力解释药物和其“潜在”不良反应的关系。
本文首先使用基于信息熵的方法提取用药者评论中的不良反应,并加以词典的辅助,良好地完成了不良反应名称的识别工作。本方法是非监督的方法,具有较好的泛化能力。但由于本方法是基于统计的方法,需要的用户评论数应尽可能地多。
然后,本文利用修改的Skip-gram模型生成的MeSH词向量,尽最大努力地为“潜在”不良反应寻求蛋白质证据,尝试找到可以把药物和其不良反应关联起来的蛋白质,从而为最终确定药物和不良反应的关系推荐线索。不足之处在于,药名和不良反应名称是UMLS超级叙词表中的概念,而修改的Skip-gram模型使用的是MeSH词,restrict to mesh算法并不能实现完全映射。在未来的工作中,我们致力于解决这一问题。
综上,由社交网络启动,融合生物信息资源的药物不良反应发现研究,可以及时发现潜在药物不良反应,并尽最大努力寻求可以把药物和不良反应联系起来的蛋白质,使潜在药物不良反应的检测具有更加实用的价值,对改善人类健康水平、减少经济损失具有重大的意义。
[1] Leaman R, Wojtulewicz L, Sullivan R, et al. Towards internet-age pharmacovigilance: extracting adverse drug reactions from user posts to health-related social networks[C]//Proceedings of the 2010 workshop on biomedical natural language processing. Association for Computational Linguistics, 2010: 117-125.
[2] Nikfarjam A, Gonzalez G H. Pattern mining for extraction of mentions of adverse drug reactions from user comments[C]// Proceedings of AMIA Annual Symposium American Medical Informatics Association, 2011: 1019.
[3] Yates A, Goharian N. ADRTrace: detecting expected and unexpected adverse drug reactions from user reviews on social media sites[C]//Proceedings of European Conference on Information Retrieval. Springer, Berlin, Heidelberg, 2013: 816-819.
[4] Bian J, Topaloglu U, Yu F. Towards large-scale twitter mining for drug-related adverse events[C]//Proceedings of the 2012 international workshop on Smart health and wellbeing. ACM, 2012: 25-32.
[5] Mikolov T, Sutskever I, Chen K, et al. Distributed representations of words and phrases and their compositionality[C]//Proceedings of advances in neural information processing systems. 2013: 3111-3119.
[6] Law V, Knox C, Djoumbou Y, et al. DrugBank 4.0: shedding new light on drug metabolism[J]. Nucleic Acids Research. 2014, 42(D1): D1091-D1097.
[7] Knox C, Law V, Jewison T, et al. DrugBank 3.0: a comprehensive resource for ‘omics’ research on drugs[J]. Nucleic Acids Research. 2011, 39(suppl 1): D1035-D1041.
[8] Wishart D S, Knox C, Guo A C, et al. DrugBank: a knowledgebase for drugs, drug actions and drug targets[J]. Nucleic Acids Research. 2008, 36(suppl 1): D901-D906.
[9] Wishart D S, Knox C, Guo A C, et al. DrugBank: a comprehensive resource for in silico drug discovery and exploration[J]. Nucleic Acids Research. 2006, 34(suppl 1): D668-D672.
[10] Kilicoglu H, Fiszman M, Rodriguez A, et al. Semantic MEDLINE: a web application for managing the results of PubMed Searches[C]//Proceedings of the 3rd international symposium for semantic mining in biomedicine. 2008: 69-76.
[11] Rindflesch T C, Kilicoglu H, Fiszman M, et al. Semantic MEDLINE: An advanced information management application for biomedicine[J]. Information Services and Use. 2011, 31(1): 15-21.
[12] Kilicoglu H, Shin D, Fiszman M, et al. SemMedDB: a PubMed-scale repository of biomedical semantic predications[J]. Bioinformatics. 2012, 28(23): 3158-3160.
[13] Kuhn M, Campillos M, Letunic I, et al. A side effect resource to capture phenotypic effects of drugs[J]. Molecular Systems Biology, 2010, 6(1).
[14] 任禾,曾隽芳. 一种基于信息熵的中文高频词抽取算法[J]. 中文信息学报, 2006,20(5): 40-43.
[15] 闫兴龙,刘奕群,马少平,等. 面向浏览推荐的网页关键词提取[J]. 智能系统学报, 2012,07(5): 398-403.
[16] Rindflesch T C, Fiszman M. The interaction of domain knowledge and linguistic structure in natural language processing: interpreting hypernymic propositions in biomedical text[J]. Journal of Biomedical Informatics, 2003, 36(6): 462-477.
[17] Gingrich J A. Mutational analysis of the serotonergic system: recent findings using knockout mice[J]. Current Drug Targets-CNS amp; Neurological Disorders, 2002, 1(5): 449-465.
[18] Goldman D, Oroszi G, O’Malley S, et al. COMBINE genetics study: the pharmacogenetics of alcoholism treatment response: genes and mechanisms[J]. Journal of Studies on Alcohol and Drugs. 2005(15): 56.
[19] Shishkina G T, Kalinina T S, Dygalo N N. Attenuation of αlt; subgt; 2A-adrenergic receptor expression in neonatal rat brain by RNA interference or antisense oligonucleotide reduced anxiety in adulthood[J]. Neuroscience, 2004, 129(3): 521-528.

赵明珍(1989—),硕士研究生,主要研究领域为文本挖掘、机器学习、自然语言处理。
E-mail: 1064328629@qq.com

林鸿飞(1962—),通信作者,博士、教授、博导,主要研究领域为搜索引擎、文本挖掘、情感计算和自然语言处理。
E-mail: hflin@dlut.edu.cn

徐博(1984—),博士,讲师,主要研究领域为面向生物医学领域的文本挖掘。
E-mail: boxu@dlut.edu.cn
PotentialAdverseDrugReactionsDiscoveryfromSocialNetworks
ZHAO Mingzhen, LIN Hongfei, XU Bo, HAO Huihui
(Information Retrieval Laboratory, Dalian University of Technology, Dalian, Liaoning 116024, China)
With the development of the Internet, social networks have accumulated large amounts of text data about health care. This paper presents an information entropy based method to recognize potential adverse drug reactions from user comments in health related social networks. Meanwhile, to recognize the potential adverse drug reactions, this paper proposes a protein association function based on Word2vec and Skip-gram. Following this functions, this paper tries to detect the evidences between drugs and their potential adverse drug reactions. The results show that this method is promising in providing evidence chain for potential adverse drug reactions.
social networks; adverse drug reactions; information entropy; Word2vec; Skip-gram
1003-0077(2017)05-0194-09
TP391
A
2015-04-07定稿日期2016-10-20
国家自然科学基金(61572102,61632011,61772103);中央高校基本科研业务费(DUT16ZD216)
— 多适应症药物准入评估方法比较研究

