利用rBE3和rBE4系统进行水稻基因单碱基编辑的特异性和遗传稳定性分析
任斌,严芳,旷永洁,李娜,张大伟,林宏辉,周焕斌
利用rBE3和rBE4系统进行水稻基因单碱基编辑的特异性和遗传稳定性分析
任斌1,2*,严芳1*,旷永洁1,李娜1,张大伟2,林宏辉2,周焕斌1
1中国农业科学院植物保护研究所植物病虫害生物学国家重点实验室,北京 100193 2四川大学生命科学学院生物资源与生态环境教育部重点实验室,四川成都 610065

任斌, 严芳, 旷永洁, 等. 利用rBE3和rBE4系统进行水稻基因单碱基编辑的特异性和遗传稳定性分析. 生物工程学报, 2017, 33(10): 1776–1785.Ren B, Yan F, Kuang YJ, et al. Specificity and inheritance of rBE3 and rBE4 endonuclease-induced gene modifications in rice. Chin J Biotech, 2017, 33(10): 1776–1785.
对基于rBE3 (Rice base editor) 和rBE4碱基编辑系统创制获得和等基因的单碱基编辑突变体材料进行编辑特异性和遗传稳定性分析,旨在全面了解和更好地利用该碱基编辑系统。首先对和sgRNA的潜在脱靶位点进行预测,并对T0代材料中的各脱靶位点进行PCR扩增和Sanger测序检测;同时对该两个基因的突变体材料自交获得的T1代植株的靶位点序列和外源T-DNA分离进行检测。结果显示各T0代材料均未检测到潜在脱靶位点发生单碱基编辑;此外,和含有相同的sgRNA位点,且两个位点均能发生单碱基编辑;rBE3或rBE4系统介导产生的碱基编辑可稳定遗传至T1代,并在T1代可获得无外源T-DNA的纯合突变体。上述结果表明由rBE3或rBE4介导的碱基编辑具有较高的特异性,可进行多位点编辑,引入的碱基替换可稳定遗传至后代。
CRISPR/Cas9n,大鼠胞苷脱氨酶,脱靶效率,T-DNA分离,水稻
近年来,基因组定点编辑技术作为一种重要技术手段在植物学研究和作物品种改良中得到了广泛应用。目前,植物基因组定点编辑技术主要是利用CRISPR/Cas9等系统在目标基因组靶位点处引入DNA双链断裂,再经非同源末端连接 (Non-homologous end Joining,NHEJ) 机制修复后在切割位点产生数个核苷酸的缺失或插入,进而导致所在靶位点基因的阅读框移码使得靶基因功能丧失[1-2]。除了常见的在特定基因组靶位点处引入核苷酸缺失或插入,对基因组靶位点特定碱基进行定向编辑也已成为基因编辑领域的难点和热点。对基因组靶位点特定碱基进行定向编辑,可实现靶基因定向功能激活或者丧失,这对于研究基因功能研究和作物品种改良具有重要作用。利用植物细胞内基因组DNA修复途径的同源重组 (Homologous recombination,HR) 或非同源末端连接机制,研究人员已经成功地在水稻中实现了靶基因的定点插入和基因矫正[3-4]。然而,较低的基因编辑效率限制了其广泛的使用。因此,对植物基因组特定碱基进行定向改造以得到基因功能获得性突变体仍然具有一定的挑战性。2016年,在哺乳动物细胞中,基于由Cas9n切口酶、大鼠胞苷脱氨酶APOBEC1和尿嘧啶DNA糖基化酶抑制剂UGI组成的融合蛋白 (即碱基编辑器,Base editor 3,BE3) 和sgRNA的引导成功对靶标基因实现了单碱基编辑[5],这为单碱基编辑技术的发展提供了全新的思路。其原理为sgRNA引导碱基编辑器结合到基因组靶位点,胞嘧啶脱氨酶将活性窗口内的胞嘧啶碱基(C) 向胸腺嘧啶碱基(T) 或鸟嘌呤碱基(G)、腺嘌呤碱基(A) 转换,尤其是当UGI存在时,胞嘧啶碱基对向胸腺嘧啶转换具有一定的偏好性。此后,科研人员很快将此技术应用到植物中,利用APOBEC1-Cas9n-UGI组成的编辑编辑器BE3相继在水稻、小麦、玉米、拟南芥和番茄中实现了单碱基编辑,获得了相应突变体材料[6-11]。然而,对BE3介导的植物单碱基编辑的脱靶效应、遗传稳定性等尚未见报道。因此,本课题组对前期基于rBE3 (APOBEC1-Cas9n-UGI,PAM序列为NGG) 和rBE4 (APOBEC1-Cas9n (VQR)-UGI,PAM序列为NGA[12-13]等) 而创制的水稻单碱基编辑突变体进行了脱靶效应和遗传稳定性等的检测,旨在全面地了解并更好地应用该技术。
1 材料与方法
1.1 材料
和等基因的单碱基编辑T0代和T1代突变体材料,均由本实验室以水稻品种Kitaake为背景材料分别利用rBE3或rBE4碱基编辑系统创制获得[8],均种植在玻璃温室中。CTAB购自北京睿恒均安科技有限公司,无水乙醇、氯仿和异丙醇等常规化学试剂 (分析纯) 均购自国药集团,I-5TM2× High-Fidelity Master Mix购自克劳宁 (北京) 生物科技有限公司,2× Tsingke Master Mix购自北京擎科新业生物技术有限公司。试验所用引物由北京擎科新业生物技术有限公司合成。
1.2 方法
1.2.1 潜在脱靶位点的预测分析
将各基因的靶位点sgRNA序列在http://rice. plantbiology.msu.edu/standalone_blast.shtml网站上进行Blastn比对 (Expect Threshold设为1 000,其余参数为默认设置),在比对获得相似序列中,选择其3′端含有PAM序列 (NGG或NGA) 且PAM序列前20个左右的碱基与靶位点sgRNA序列相似性较高的位点作为潜在的脱靶位点。
1.2.2 水稻基因组DNA的提取
参考Porebski等的方法[14],采用CTAB法提取水稻叶片的基因组DNA。具体操作为:剪取的水稻幼苗叶片经全自动样品快速研磨仪研磨 (60 Hz,60 s) 后,加入600 μL 2× CTAB提取液 (含0.1% β-巯基乙醇) 后振荡混匀,65 ℃水浴裂解45 min;加入500 μL氯仿抽提,14 000 r/min室温离心10 min;离心后取上清液400 μL于离心管,加入400 μL异丙醇,颠倒混匀后置于–20 ℃中沉淀30 min;14 000 r/min离心10 min后弃上清液,白色DNA沉淀经70%乙醇洗涤风干后加入30 μL ddH2O溶解。DNA溶液置于–20 ℃保存备用。
1.2.3 碱基编辑位点及潜在脱靶位点的检测
以水稻突变体DNA为模板,利用特异引物 (见表1) 对各编辑位点及潜在脱靶位点的两侧序列进行PCR扩增。PCR扩增体系为:I-5TM2× High-Fidelity Master Mix 25 μL,引物F/R (10 μmol/L) 各2 μL,模板DNA 1 μL,ddH2O 20 μL。PCR扩增程序为:98 ℃预变性2 min;98 ℃变性10 s,55 ℃退火10 s,72 ℃延伸10 s,循环34次;72 ℃延伸5 min。PCR产物经1.5%琼脂糖凝胶电泳检测后直接委托北京擎科新业生物技术有限公司进行Sanger测序,分析其碱基编辑类型。

表1 文中所用到的引物及其序列
1.2.4 无外源T-DNA元件突变体的鉴定
选择发生碱基编辑事件的T0代植株单株自交结实获得T1种子,并将其播种成苗后提取叶片基因组DNA。以T1代植株DNA为模板,分别利用T-DNA上的潮霉素特异引物-F1/R1和Cas9特异引物-F1/R1 (见表1) 对T-DNA的有无进行两轮PCR检测。PCR扩增体系为:2× Tsingke Master Mix 5 μL,引物F/R (10 μmol/L)各0.2 μL,模板DNA 0.5 μL,ddH2O补齐至10 μL。PCR扩增程序为:95 ℃预变性5 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,循环25次;72 ℃延伸5 min。PCR产物利用1.5%琼脂糖凝胶进行电泳检测。
2 结果与分析
2.1 rBE3介导的多位点单碱基编辑
在创制突变体材料过程中,对其sgRNA (AAGATTATCCATCGTGA TGT,下划线为PAM序列NGG) 的特异性进行了评估,通过在水稻基因组数据库中进行Blastn比对分析,检索到基因中也含有该sgRNA序列,即通过该sgRNA可能同时对这两个基因的靶位点碱基进行编辑。因此,通过设计特异引物对突变体材料中基因的靶位点进行了PCR扩增和Sanger测序。结果显示在12株T0代突变体材料中,检测到了5株材料在基因的靶位点处发生了预期的碱基编辑事件。其中,突变体#419的和的靶位点碱基编辑方式均为G-16>A/WT (杂合突变,WT为野生型);突变体#417的靶位点发生杂合突变 (G-16>A/WT),而靶位点则为纯合突变 (G-14TG-16>ATA) (见图1和表2)。此外在靶位点处还检测到2株材料发生Indel (插入或缺失) 现象 (见表2)。
2.2 rBE3介导的单碱基编辑脱靶分析
将的靶位点sgRNA序列 (AAGATTATCCATCG TGATGT,下划线为PAM序列NGG) 对水稻全基因组序列进行比对,获得5个潜在脱靶位点,其中中的相似位点其5′端 (GCA) 不存在PAM序列NGG,故排除其为潜在脱靶位点的可能性;而其余4个位点均可能为脱靶位点 (图2)。因此分别对这4个潜在脱靶位点设计特异引物,并以T0代突变体材料的DNA为模板进行PCR扩增和测序,在检测的12株突变体材料中,均未检测到潜在脱靶位点发生碱基编辑 (表2)。将的靶位点sgRNA序列 (CTTCTATGCATCTTCAACC,下划线为PAM序列NGAC) 与水稻全基因组序列进行比对分析,结果显示未检索出与其靶位点sgRNA相似的序列,即该sgRNA将特异地引导的靶位点碱基编辑,不存在潜在脱靶位点。
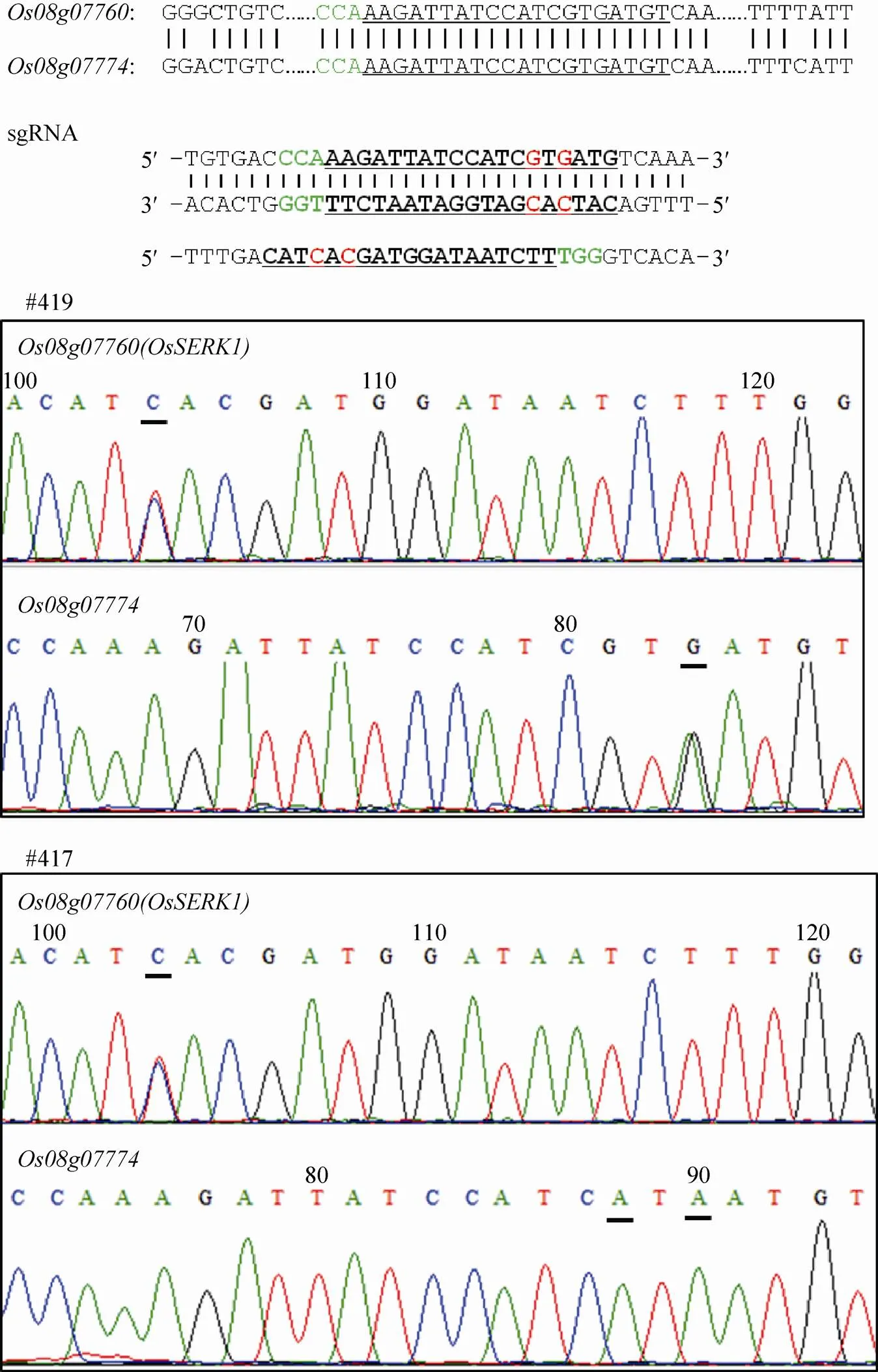
图1 OsSERK1(D428N) T0代突变体#419和#417的两个靶位点的测序结果
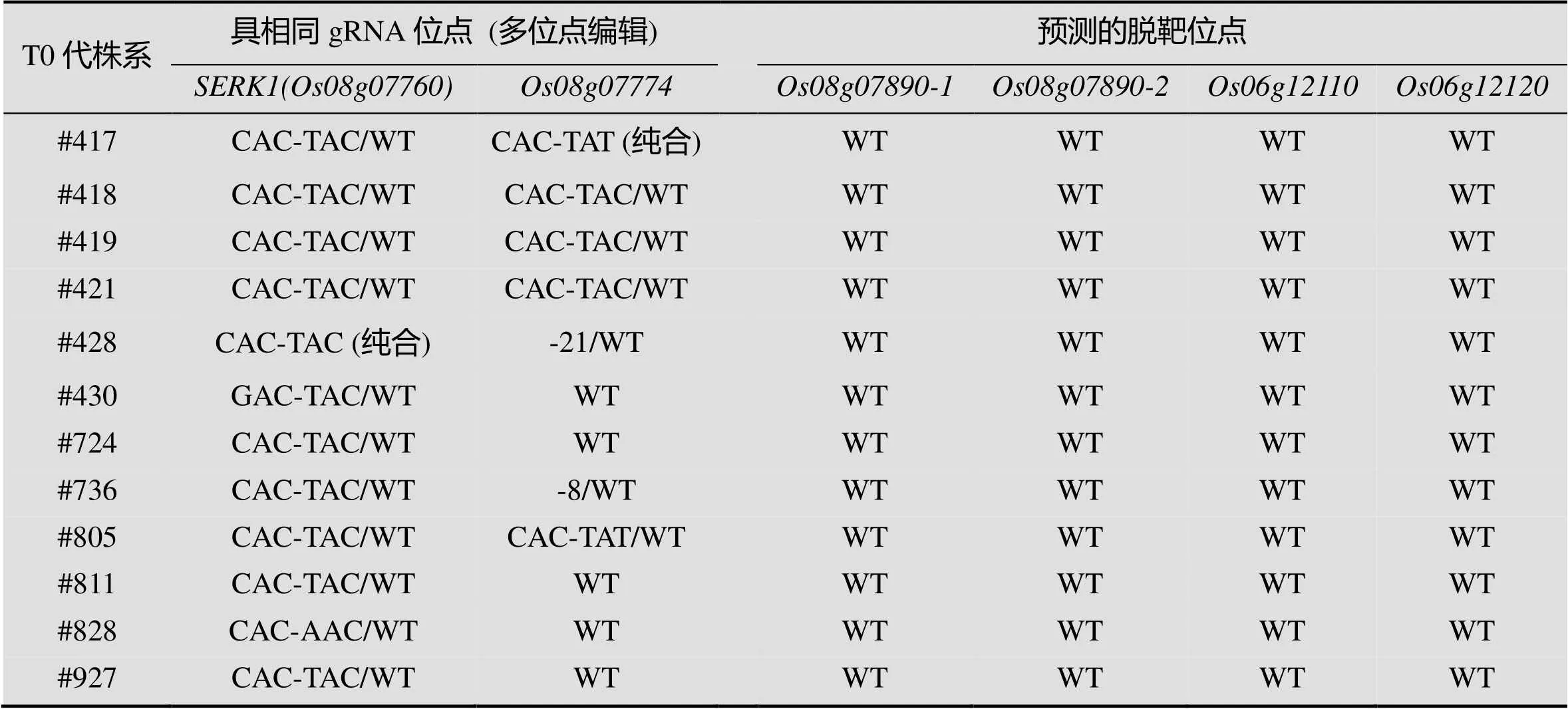
表2 OsSERK1(D428N) T0代突变体多位点碱基编辑和脱靶检测

图2 预测的潜在脱靶位点序列和靶位点序列比对分析
2.3 单碱基编辑突变体的遗传稳定性分析
2.4 单碱基编辑突变体后代外源T-DNA元件分离
为进一步获得无外源T-DNA插入元件的单碱基编辑突变体,利用引物-F1/R1和-F1/R1对#805和#22的T1植株进行两轮PCR检测。结果显示在T1代植株中筛选获得了无外源T-DNA的材料 (图4)。在22株#805 T1代植株中检测出9株不含外源T-DNA,其中包含3株纯合突变体和4株杂合突变体。在36株#22 T1代植株中检测出9株不含外源T-DNA,其中仅包含2株杂合突变体。这表明在通过农杆菌导入的外源T-DNA元件可以从突变体T1代中分离出去并可获得无外源DNA片段的单碱基编辑突变体材料。

图3 OsSERK1(D428N) #805 (左) 和pi-ta(S918F) #22 (右)突变体的T1代植株的靶位点测序结果
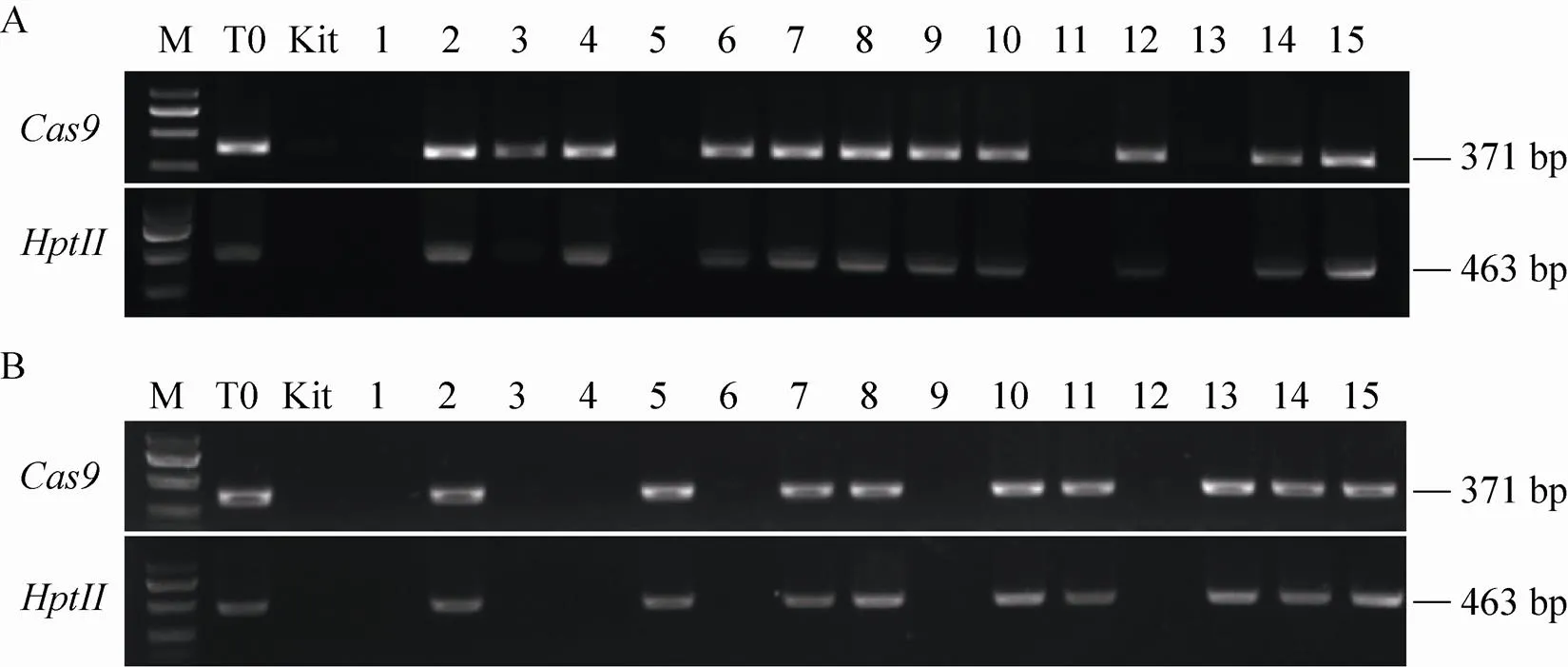
图4 OsSERK1(D428N) #805 (A)和pi-ta(S918F) #22 (B) 突变体的T1代植株中潮霉素HptII和Cas9基因检测
3 讨论
CRISPR/Cas9技术是近年来发展起来的一种简单、快速、高效的对基因定点编辑技术,广泛应用于微生物和动植物中[15-20],其常见编辑类型有基因定点插入[21]、DNA片段缺失[22-23]、多位点突变[23-24]等,但对于实现单碱基编辑仍是难点且对其编辑的特异性和稳定性缺乏了解。本课题组前期研究中将水稻密码子优化的APOBEC1和UGI与Cas9n、Cas9n (VQR) 融合构建了一套新的rBE3、rBE4系统,实现了在水稻基因组DNA中的单碱基编辑[8],本研究对该系统单碱基编辑的脱靶现象和稳定遗传性进行了研究。
本研究选择了(rBE3) 和(rBE4) 两个突变体材料进行脱靶分析,通过在水稻基因组数据库中将sgRNA进行Blastn比对分析发现的sgRNA具有较好的特异性,并没有发现潜在的脱靶位点;而的sgRNA序列不仅与一致,而且还具有其他的潜在脱靶位点,我们通过Sanger测序发现,在的12株T0代突变体植株中,有41.67%的植株实现了双位点的单碱基编辑,但并没有检测到脱靶现象的发生。由于碱基编辑系统主要用来编辑一些具有重要功能的关键碱基,在设计sgRNA引物时的可选区域很受限制,因此可能会导致所用sgRNA的特异性差和编辑效率低的问题。同时rBE系统引入的碱基编辑多为杂合突变,即基因组仍会存在野生型序列。因此对于本研究中突变体在非目的区域处同时发生的碱基编辑的情况,经T0代突变体自交分离后,在其后代中已成功筛选获得仅含有突变的植株 (数据未显示)。本研究中通过将sgRNA序列在水稻基因组数据库中进行Blastn检索潜在脱靶位点、PCR扩增和Sanger测序对T0代突变体材料的潜在脱靶位点进行分析,结果虽未检测出脱靶现象的发生,但由于仅进行Blastn检索可能会遗漏其他可能相似的潜在脱靶位点和PCR扩增会使以低频率嵌合体存在的脱靶突变进一步稀释导致检测不到脱靶发生,因此后期如果该材料进一步用于遗传育种时将会在全基因组层面进行脱靶检测。Kim等[25]利用Digenome-Seq技术对单碱基编辑系统 (BE3) 在人HEK293T细胞系中的脱靶效应进行了研究,发现BE3系统的脱靶位点远少于CRISPR/Cas9系统,具有很高的特异性。此外,对于相同sgRNA和脱靶导致的非目的位点突变还可通过与亲本多次回交的方法进行去除。这些结果表明rBE系统可以利用特异性的sgRNA产生特定碱基编辑的突变体材料。有趣的是,仅有2株T0代突变体材料在基因中检测到了Indel事件的发生,这与Yuan等[9]研究类似,同时Lu等[6]研究表明BE3在植物出现Indel事件的几率 (10%) 高于动物细胞。Indel事件可能是由于细胞内基因组DNA修复途径造成。Cas9n可以切割基因组DNA使其出现nicks,而sgRNA通常编辑DNA的一条链,而另一条链并没有参与到基因编辑,但是在后续的DNA修复机制会同时对两条DNA链进行修复,这个修复过程可能会造成Indel事件的发生。这一事件的发生机制是否与靶位点区域内外的序列有关暂时还不清楚。
相对于目前广泛的基因功能缺失突变体材料的定制应用,基因功能获得性突变体材料的创制也具有重要意义。rBE系统所利用的单碱基编辑为植物基因功能获得性突变体材料的定制提供了新的方向。因此,rBE系统的编辑事件是否能够稳定遗传尤其重要。研究发现,和T0代植株自交后可以在T1代中分离出无T-DNA的纯合突变体植株,这也表明利用rBE单碱基编辑系统产生的突变性状可以稳定遗传给后代。
本研究结果显示rBE系统可以利用特异性的sgRNA产生特定碱基突变的突变体材料,同时还可实现多位点突变,产生的碱基突变也能稳定地遗传给后代,表明rBE系统可以被用于定制基因功能获得型水稻材料,对水稻基因的功能学研究具有巨大推动作用。
[1] Xiao A, Zhang B. Progress of new-generation genome editing mediated by engineered endonucleases. Chin J Biotech, 2015, 31(6): 917–928 (in Chinese). 肖安, 张博. 人工核酸内切酶介导的新一代基因组编辑技术进展. 生物工程学报, 2015, 31(6): 917–928.
[2] Yan F, Zhou HB. Overviews and applications of the CRISPR/Cas9 system in plant functional genomics and creation of new plant germplasm. Sci Sin Vitae, 2016, 46(5): 498–513 (in Chinese). 严芳, 周焕斌. CRISPR/Cas9技术在植物基因功能研究和新种质创制中的应用与展望. 中国科学: 生命科学, 2016, 46(5): 498–513.
[3] Sun YW, Zhang X, Wu CY, et al. Engineering herbicide-resistant rice plants through CRISPR/Cas9-mediated homologous recombination of acetolactate synthase. Mol Plant, 2016, 9(4): 628–631.
[4] Li J, Meng XB, Zong Y, et al. Gene replacements and insertions in rice by intron targeting using CRISPR-Cas9. Nat Plants, 2016, 2(10): 16139.
[5] Komor AC, Kim YB, Packer MS, et al. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature, 2016, 533(7603): 420–424.
[6] Lu YM, Zhu JK. Precise editing of a target base in the rice genome using a modified CRISPR/Cas9 system. Mol Plant, 2017, 10(3): 523–525.
[7] Li JY, Sun YW, Du JL, et al. Generation of targeted point mutations in rice by a modified CRISPR/Cas9 system. Mol Plant, 2017, 10(3): 526–529.
[8] Ren B, Yan F, Kuang YJ, et al. A CRISPR/Cas9 toolkit for efficient targeted base editing to induce genetic variations in rice. Sci China Life Sci, 2017, 60(5): 516–519.
[9] Zong Y, Wang YP, Li C, et al. Precise base editing in rice, wheat and maize with a Cas9-cytidine deaminase fusion. Nat Biotechnol, 2017, 35(5): 438–440, doi: 10.1038/nbt.3811.
[10] Chen YY, Wang ZP, Ni HW, et al. CRISPR/Cas9-mediated base-editing system efficiently generates gain-of-function mutations in. Sci China Life Sci, 2017, 60(5): 520–523, doi: 10.1007/s11427-017-9021-5.
[11] Shimatani Z, Kashojiya S, Takayama M, et al. Targeted base editing in rice and tomato using a CRISPR-Cas9 cytidine deaminase fusion. Nat Biotechnol, 2017, 35(5): 441–443, doi: 10.1038/nbt.3833.
[12] Kleinstiver BP, Prew M, Tsai SQ, et al. Engineered CRISPR-Cas9 nucleases with altered PAM specificities. Nature, 2015, 523(7561): 481–485.
[13] Hu XX, Wang C, Fu YP, et al. Expanding the range of CRISPR/Cas9 genome editing in rice. Mol plant, 2016, 9(6): 943–945.
[14] Porebski S, Bailey LG, Baum BR. Modification of a CTAB DNA extraction protocol for plants containing high polysaccharide and polyphenol components. Plant Mol Biol Rep, 1997, 15(1): 8–15.
[15] Jiang WY, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nat Biotechnol, 2013, 31(3): 233–239.
[16] Fang YF, Tyler BM. Efficient disruption and replacement of an effector gene in the oomyceteusing CRISPR/Cas9. Mol Plant Pathol, 2016, 17(1): 127–139.
[17] Arazoe T, Miyoshi K, Yamato T, et al. Tailor-made CRISPR/Cas system for highly efficient targeted gene replacement in the rice blast fungus. Biotechnol Bioeng, 2015, 112(12): 2543–2549.
[18] Chang NN, Sun CH, Gao L, et al. Genome editing with RNA-guided Cas9 nuclease in zebrafish embryos. Cell Res, 2013, 23(4): 465–472.
[19] Mali P, Yang LH, Esvelt KM, et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823–826.
[20] Shan QW, Wang YP, Li J, et al. Targeted genome modification of crop plants using a CRISPR-Cas system. Nat Biotechnol, 2013, 31(8): 686–688.
[21] Auer TO, del Bene F. CRISPR/Cas9 and TALEN-mediated knock-in approaches in zebrafish. Methods, 2014, 69(2): 142–150.
[22] Essletzbichler P, Konopka T, Santoro F, et al. Megabase-scale deletion using CRISPR/Cas9 to generate a fully haploid human cell line. Genome Res, 2014, 24(12): 2059–2065.
[23] Xie KB, Minkenberg B, Yang YN. Boosting CRISPR/Cas9 multiplex editing capability with the endogenous tRNA-processing system. Proc Natl Acad Sci USA, 2015, 112(11): 3570–3575.
[24] Shen L, Hua YF, Fu YP, et al. Rapid generation of genetic diversity by multiplex CRISPR/Cas9 genome editing in rice. Sci China Life Sci, 2017, 60(5): 506–515.
[25] Kim D, Lim K, Kim ST, et al. Genome-wide target specificities of CRISPR RNA-guided programmable deaminases. Nat Biotechnol, 2017, 35(5): 475–480.
(本文责编 郝丽芳)
Specificity and inheritance of rBE3 and rBE4 endonuclease-induced gene modifications in rice
Bin Ren1,2*, Fang Yan1*, Yongjie Kuang1, Na Li1, Dawei Zhang2, Honghui Lin2, and Huanbin Zhou1
1 State Key Laboratory for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection, Chinese Academy of Agricultural Sciences, Beijing 100193, China 2 Ministry of Education Key Laboratory of Bio-Resource and Eco-Environment, College of Life Sciences, Sichuan University, Chengdu 610065, Sichuan, China
To gain more insights into the rice base editor (rBE3 and rBE4), we evaluated the mutation efficiency, off-target and inheritance ofandgenes modified with rBE endonucleases. We predicted and analyzed the putative off-target sites of the sgRNA designed forandby PCR amplification and Sanger sequencing. Then we further characterized the inheritance and stability of targeted base mutations and T-DNA segregation in the progeny of the self-fertilized T0 plants. Analysis of the DNA sequencing data of T0 plantsof) revealed no nucleotide change at any of the four potential off-target sites. Forandcarry the same sgRNA targeting sites, base substitution at both two loci were detected at a frequency of 41.67%. The targeted base mutations could be transmitted readily to T1 progeny. Furthermore, genetic segregation caused the loss of T-DNA at a frequency between 25.0% and 40.9% in the T1 transgenic plants ofand. These results demonstrated that the rBE3 and rBE4 systems could mediate specifically targeted base editing in one- or multi-site, and the targeted base editing could be stably inherited to next generation.
CRISPR/Cas9n, rat APOBEC1, off-target, T-DNA segregation, rice
April 25, 2017;
August 28, 2017
Honghui Lin. Tel: +86-28-85411792; Fax: +86-28-85415300; E-mail: hhlin@scu.edu.cn Huanbin Zhou. Tel: +86-10-62815914; Fax: +86-10-62894642; E-mail: hbzhou@ippcaas.cn
Supported by:The Agricultural Science and Technology Innovation Program of The Chinese Academy of Agricultural Sciences and Open Project of State Key Laboratory for Biology of Plant Diseases and Insect Pests, Institute of Plant Protection.
中国农业科学院科技创新工程和植物病虫害国家重点实验室开放基金资助。
2017-09-06
*These authors contributed equally to this work.
网络出版地址:http://kns.cnki.net/kcms/detail/11.1998.Q.20170906.1057.002.html
周焕斌 中国农业科学院植物保护研究所研究员,博士生导师。主要从事各种不同用途基因组定点编辑技术在水稻等主要农作物的研发与应用,借助其深度探讨水稻与病原菌互作的分子机制,发掘重要基因资源,并加以组合修饰,获得无生物安全风险的基因编辑材料,服务于农业生产。已在、、、等SCI杂志上发表文章10篇,被引用达600多次。


