南极长城湾海绵共附生可培养细菌多样性研究
刘 欢,俞 勇,廖 丽,刘 双,3,陈 波
南极长城湾海绵共附生可培养细菌多样性研究
刘 欢1,2,俞 勇2,廖 丽2,刘 双2,3,陈 波1,2
(1.上海海洋大学海洋科学学院,上海 201306;2.中国极地研究中心,国家海洋局极地科学重点实验室,上海 200136;3.华东理工大学生物工程学院,上海 200237)
研究了南极长城湾潮间带海绵共附生可培养细菌多样性及其疑似新种应用有利于放线菌生长的选择性培养基ISP2从长城湾海绵动物组织中分离出120株细菌,基于这些菌株的16SrRNA基因序列开展系统发育分析。结果显示,所分离菌株隶属于三个细菌门五大类群的11个目、22个属,表明长城湾海绵共附生细菌的种属分布较广、多样性非常高。其中红球菌Rhodococcus属占优势,另外包含α-变形菌纲Puniceibacterium属、放线菌门Aeromicrobium属、β-变形菌纲Limnobacter属等罕见种属。16SrRNA基因序列同源性比对分析表明,菌株HM145与Rhodococcus属模式种Rhodococcus qingshengii具有最高序列相似性97.0%,菌株HQ09与Puniceibacterium antarcticum有96.9%的最高相似性,为2株疑似新种,其分类地位和种水平上的分类学研究有待开展多相分类进行鉴定。
南极长城湾;潮间带;海绵共附生可培养细菌;多样性
海绵动物是一类多孔滤食性的底栖生物,可以过滤大量海水[1-2],一部分居住在海绵组织内的微生物可能是暂时性的周围海水的组成部分[3]。海绵共附生微生物主要包括:a)附着在海绵表面或内腔表面的附生微生物;b)进入了海绵细胞间中质层而躲过了海绵细胞吞噬作用的海绵细胞外微生物;c)被海绵细胞吞噬的可为海绵细胞提供营养的海绵细胞内微生物[4]。海绵的共附生微生物可以达到海绵生物量的40%,这些微生物参与到海绵的宿主防御、营养和代谢过程中[5]。温带和热带海域海绵共附生微生物群落已被广泛研究,基于可培养和非培养技术研究显示,海绵中的共附生微生物既有原核的古菌和细菌,也有大量的真菌,约有40个门的微生物物种存在于海绵体内,其中以变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、绿弯菌门(Chloroflexi)、拟杆菌门(Bacteriodetes)和蓝藻门(Cyanophyta)为主[6]。已有少量的文献报道海绵动物是南极半岛沿岸底栖生物群落常见的类群,其中寻常海绵纲(Demospongiae)占比最大,其次是六放海绵纲(Hexactinellida),其地理分布主要受地质类型、水温和深度的影响[7]。2004年LARS等[8]使用特异的 PCR引物构建了海绵Aplysina aerophoba共附生细菌的16S rRNA基因文库,通过系统发育分析发现了与已知门细菌16S rRNA基因同源性<75%的海绵菌门(Poribacteria),提示海绵动物存在丰富的共附生细菌新类群。
我国南极长城站及其毗邻的长城湾位于西南极乔治王岛的菲尔德斯半岛(Fildes Peninsula)南部,气候相对温暖。长城湾是菲尔德斯半岛的内湾,最大水深35 m,沿岸具有岩礁、沙质和泥沙等3种主要海岸类型,潮间带至潮下带动植物种类丰富。已有的调查表明,西南极的乔治王岛和南极半岛海域海水和沉积环境具有较多微生物,多样性比较丰富。迄今南极低温海洋环境的海绵动物共附生微生物的类群和多样性研究仍然少见。本文利用长城湾潮间带采集到的海绵,通过选择性培养基获得以放线菌为主的海绵共附生细菌,研究了这些菌株的多样性,以期为海绵共附生微生物的分类、代谢及其基础应用奠定基础。
1 材料与方法
1.1 样品来源
2013年中国第29次南极科学考察期间于长城湾潮间带的沙质表层沉积物中(62°12′41"S、58°57′08"W)采集到 3个未鉴定的海绵动物样品。
1.2 实验方法
1.2.1 菌株分离
将海绵动物组织样品剪碎,取1 g置于装有10 mL无菌海水中匀浆器中进行组织破碎,静置5 min后,取上清涂布添加终浓度为30μg·mL-1萘啶酮酸的ISP2分离培养基(ISP2:葡萄糖4 g,麦芽粉10 g,酵母粉4 g,琼脂15 g,海水定容至1 L,调 pH至7.2~7.3)。20℃培养4周以上,根据菌落形态挑取单菌落转接到ISP2培养基上进行纯化培养。
1.2.2 16SrRNA基因 PCR与测序
使用博大泰克(北京)的细菌基因组小量快速提取试剂盒提取细菌gDNA,按照其操作说明进 行。 采 用 通 用 引 物 27F: 5′-AGAGTTTGATCCTGGCTCAG-3′及 1492R:5′-GGTTACCTTGTTACGACTT-3′,进 行 细 菌 16S rRNA基因的扩增[9]。PCR程序为98℃预变性5 min;95℃变性30 s,55℃退火30 s,72℃延伸1 min 30 s,共30个循环;72℃延伸10 min;4℃低温保存。使用pMD18-T质粒,按照说明书步骤进行TA克隆。挑取3~6个阳性单克隆进行液体培养,之 后 采 用 通 用 引 物 M13F(-47):-CGCCAGGGTTTTCCCAGTCACGAC和 M13R(-48):-AGCGGATAACAATTTCACACAGGA-进行菌液PCR。PCR程序为95℃预变性5 min;94℃变性30 s,55℃退火30 s,68℃延伸2 min,共25个循环;68℃延伸10 min;4℃低温保存。用0.8%琼脂糖凝胶电泳检测PCR产物,在1.5 Kb左右检测到清晰的目的条带后,送PCR产物至上海桑尼生物科技有限公司或上海美吉生物医药科技有限公司进行测序。
1.2.3 序列分析与系统发育树构建
测序结果提交至生物信息学数据库GenBank(https://www.ncbi.nlm.nih.gov)及 EzTaxon-e[10](http://www.ezbiocloud.net)进行序列的相似性比较分析。选择 MEGA 6.0[11]软件里渐进的多序列比对方法Cluster W进行海绵共附生细菌16SrRNA基因序列比对,树的拓扑形状采用邻接法[12](Neighbor-Joining),分枝上的数字为 1 000次bootstrap[13]分析所得的值,自展值设定在50以上构建系统发育树。基于数据库EzTaxon-e的16S rRNA基因比对结果,寻找最高相似性在97%左右的菌株,选取比对结果中与其相似性较高的模式株并加入一个外群,采用MEGA 6.0软件的邻接法构建系统发育树,其它方法同上。
2 结果与分析
2.1 海绵共附生可培养细菌的种类
经过20℃培养4周以上,平板上出现形态多样的菌落,进一步在ISP2培养基上进行划线和纯化,共获得120株海绵共附生细菌。提取菌株的基因组DNA后,用16SrRNA基因通用引物进行PCR扩增,测序后获得的16S rRNA基因序列。通过与GenBank数据库中序列的比对分析,发现所得16SrRNA基因序列与数据库中很多已有序列的相似性较高(98%~100%),但这些序列多数来自未培养或者未鉴定的细菌,且有部分序列来自本实验室之前在中山站附近分离并提交的菌株,另有部分序列来自北极海洋环境的菌株。进一步将所得序列与EzTaxon-e数据库中序列进行比对,发现所分离菌株的16S rRNA基因序列与已描述模式菌株的16S rRNA基因序列的相似度为96.9%~100%,其中有2株菌株与已知细菌种的16SrRNA基因序列相似性低于97%,有5株菌株与已知细菌种的16S rRNA基因序列相似性低于98%。另有8株菌株与已知细菌种的16S rRNA基因序列相似性为100%。
基于16S rRNA基因序列的比对分析,所获得的120个菌株可归属为拟杆菌门(Bacteroidetes),变形菌门(Proteobacteria)的 α-变形 菌 纲 (α-Proteobacteria)、β-变 形 菌 纲 (β-Proteobacteria)和 γ-变形菌纲(γ-Proteobacteria),放线菌门(Actinobacteria)等三个门五大类群的11个目、22个属。属水平的菌株分布见图1,其中放线菌门各属占绝对优势,表明ISP2培养基作为选择性培养基有利于海绵动物共附生放线菌的分离,组份中萘啶酮酸的使用抑制了大多数革兰氏阴性细菌的生长,能够以此培养基分离获得的革兰氏阴性细菌在一定程度上能耐受萘啶酮酸的抑制作用。分离菌株数最多的属为Rhodococcus属,共获得57个菌株,占总分离菌数的47%。其它属所获得的菌株数量明显少于Rhodococcus属,占比分别在1%~8%,表现出了较高的多样性,并获得了一些比较罕见的属,例如α-变形菌纲 Puniceibacterium属、放线菌门Aeromicrobium属、β-变形菌纲 Limnobacter属等,这些属的细菌在GenBank数据库中已有的序列和信息并不多,目前针对其开展的分离培养也较少见。

图1 长城湾海绵共附生可培养细菌的属水平上的分布Fig.1 Distribution of sponge-associated culturable bacteria from the Great Wall Bay on genus level
2.2 海绵共附生可培养细菌的系统发育分析
进一步对获得菌株进行基于16SrRNA基因序列的系统发育分析,构建了进化树(图2)。放线菌门的菌株在系统发育树上体现了较高的多样性,有些属则表现了明显的优势性,Corynebacterium属的4个菌株和Mycetocola属的5个菌株其16S rRNA基因序列高度相似。其它属的菌株则表现了一定的差异,代表了多个不同种(图2-A)。Rhodococcus属占据了最大的比例,呈现了绝对的优势性(图2-B),由图2-B可见,该属可以归为4个主要的分支,且其中一个分支占据了主要优势。从16SrRNA基因序列的相似性比对结果分析,该属57个菌株中最相似的标准菌株只有5个,即很多菌株跟同一个标准菌株具有很高的相似性,可能为同一个种内的不同菌株。严立恩等[14]比较了南极潮间带和陆地土壤放线菌的分布和多样性,认为潮间带相较于陆地土壤,放线菌占比更大,但是多样性较低,本研究优势属Rhodococcus数量多但多样性不高的结果与其一致,而不同之处在于本研究选择性培养基的使用可从海绵组织样品中分离获得包括Rhodococcus属在内的12个属的放线菌菌株。
2.3 疑似新种的发现
16SrRNA基因序列相似性分析表明所分离的菌株HM145和HQ09与已知细菌种的序列相似性均低于97%。按照目前普遍接受的16S rRNA基因序列相似性97%为细菌新种划分的界限标准[15-16],这两株极地海绵共附生细菌可能代表新的细菌种,笔者进一步对这两株细菌菌株进行了基于16S rRNA基因序列的系统发育分析。
与菌株HM145的16S rRNA基因序列相似性在95%以上的已描述细菌种只分布在Nocardiaceae科的 Rhodococcus属和 Nocardia属。菌株 HM145与 Rhodococcus属的模式种Rhodococcus qingshengii[17]的 16S rRNA基因序列相似性最高,为 97.0%,其次为 Nocardia arthritidis[18],两者间有 96.6%的相似性。选择Escherichia coli作为外群,Nocardiaceae科中与菌株HM145的16SrRNA基因序列相似性>95%模式菌株作参比菌株,构建系统发育树。从系统发育树(图3)看,Rhodococcus属和Nocardia属的菌株形成两个分支,为两个进化簇,HM145位于Rhodococcus属进化簇的根部,难以归类为Rhodococcus属或Nocardia属,其准确的分类地位有待进一步通过多相分类研究确定。

图2 长城站潮间带120株海绵共附生细菌的系统发育树Fig.2 Phylogenetic tree of 120 sponge-associated bacterial strains isolated from sponge samples in the intertidal sand sediment,Great Wall Bay(bootstrap values,based on 1 000 replications,over 50%are shown at nodes)

图3 基于菌株HM145及其相应模式株16S r RNA基因序列,使用邻接法构建的系统发育树Fig.3 Neighbour-joining phylogenetic tree based on 16S r RNA gene sequences for the positions of strain HM145 and other related species(bootstrap values,based on 1000 replications,over 50%are shown at nodes.Bar,0.02 substitutions per nucleotide position.GenBank accession numbers are indicated in parentheses after the sequence name)
与菌株HM09比对相似性在96%以上的模式种有 Rhodobacteraceae科的 Puniceibacterium属、Pseudooceanicola属、Phaeobacter属和 Marivita属的模式菌。菌株 HQ09与 Puniceibacterium antarcticum[19]有 96.9%的最高相似性,其次是Pseudooceanicola antarcticus[20]的 96.8%,与新属Puniceibacterium的另一个模式种Puniceibacterium sediminis[21]有 96.8% 的 相 似 性。 选 择Rhodobacteraceae科的10个属和作为外群的一个Escherichia coli共17个16S rRNA基因序列构建系统发育树(图4)。由进化树可以看出,菌株HQ09与Puniceibacterium属所在支更接近,自展值为57。目前为止Puniceibacterium属只有2株已经鉴定的新种,HQ09的发现可能成为该属的第3个新种。
3 讨论与小结
本文应用选择性培养基ISP2,从南极长城站毗邻的长城湾潮间带沙质沉积物的海绵动物组织中分离获海绵共附生细菌120株,开展了基于16SrRNA基因序列的系统发育分析,结果表明,所获得的分离菌株可归属为拟杆菌门(Bacteroidetes),变形菌门(Proteobacteria)的 α-变形 菌 纲 (α-Proteobacteria)、β-变 形 菌 纲 (β-Proteobacteria)和 γ-变形菌纲(γ-Proteobacteria),放线菌门(Actinobacteria)等三个门五大类群的11个目、22个属,其中含放线菌12个属,显示了该海绵共附生细菌非常高的多样性,以Rhodococcus属菌株最多,呈现了明显的优势性。其中α-变形菌纲下的Puniceibacterium属、放线菌门下的 Aeromicrobium属、β-变形菌纲下的Limnobacter属菌株在GenBank数据库中已有的序列和信息并不多,目前针对其开展的分离培养也较少见,为罕见种属,表明了选择性培养基ISP2的应用有利于这些罕见属菌株的分离获得。
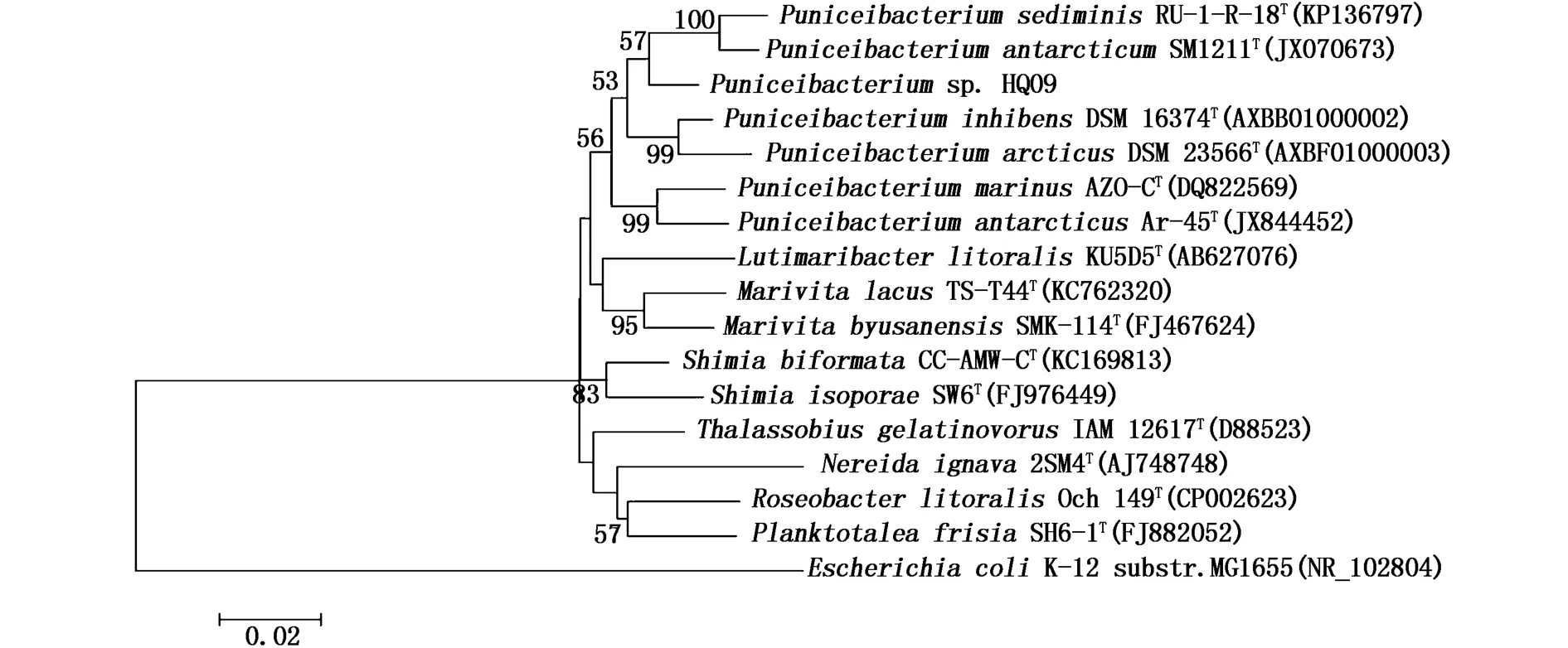
图4 基于菌株HQ09及其相应模式株16S r RNA基因序列,使用邻接法构建的系统发育树Fig.4 Neighbour-joining phylogenetic tree based on 16Sr RNA gene sequences for the positions of strain HQ09 and other related species(bootstrap values,based on 1000 replications,over 50%are shown at nodes.Bar,0.02 substitutions per nucleotide position.GenBank accession numbers are indicated in parentheses after the sequence name)
16SrRNA基因序列同源性比对分析发现,菌株HM145与Rhodococcus属模式种Rhodococcus qingshengii具有相似性最高为97.0%,与Nocardia arthritidis有96.6%的相似性。菌株HM145具体的分类地位和种水平上的分类学研究有待进一步开展多相分类进行鉴定。菌株 HQ09与Puniceibacterium antarcticum有96.9%的最高相似性,与 Pseudooceanicola antarcticus有96.8%的相似性,与 Puniceibacterium属的另一个模式种Puniceibacterium sediminis有96.8%的相似性。系统发育分析显示,菌株HQ09与Puniceibacterium属所在支更接近,目前Puniceibacterium属只有2株已经鉴定的新种,HQ09的发现可能成为该属的第3个新种。
海绵为一类多孔滤食性的底栖无脊椎动物,与多种微生物存在共生或附生关系,是微生物新分类类群的一个重要来源,也是今后深入研究获得多种天然产物的重要研究对象。海绵动物中分离的多数天然化合物均为其共附生的微生物所产生,例如张海涛[22]首次从海绵中大量分离获得放线菌株,其中约61%的菌株具有多种抗菌活性,且在抗肿瘤筛选中,20%的放线菌具有超过50%的抑制肿瘤细胞生长能力,显示了海绵共附生微生物为药源天然产物的重要潜在资源。本研究可为南极海洋环境海绵共附生微生物新分类群和药源微生物菌株资源的发现提供一个新的途径。
致谢:感谢中国第29次南极科学考察队和中国南极长城站为本研究提供南极现场考察的后勤装备和条件支持。本实验室孙茜和季青在菌株保藏和测序前的实验工作中给予了许多帮助和支持,在此一并由衷致谢。
[1] PILE A J,PATTERSON M R,WITMAN J D.In situ grazing on plankton<10 microns by the boreal sponge Mycale lingua[J].Marine Ecology Progress Series,1996(141):95-102.
[2] BELL A H,BERGQUIST P R,BATTERSHILL C N.Feeding biology of Polymastia croceus[J].Memoirs of the Queensland Museum,1999(44):51-56.
[3] WEBSTER N S,NEGRI A P,MUNRO M H G,et al.Diverse microbial communities inhabit Antarctic sponges[J].Environmental Microbiology,2004,6(3):288-300.
[4] 刘玲枝,黄惠琴,鲍时翔.海绵共附生微生物的研究新进展[J].生物技术通报,2007(3):93-96.
LIU Z L,HUANGH Q,BAOShi-xiang.Progress in studies on sponge-associated microorganism[J].Biotechnology Bulletin,2007(3):93-96.
[5] TAYLOR M W,RADAX R,STEGER D,et al.Sponge-associated microorganisms: evolution,ecology, and biotechnological potential[J].Microbiology and Molecular Biology Reviews,2007,71(2):295-347.
[6] MARCONI S R,IGLESIA R D l,DÍEZ B,et al.Characterization of bacterial,archaeal and eukaryote symbionts from Antarctic sponges reveals a high diversity at a three-domain level and a particular signature for this ecosystem[J].Plos One,2015,10(10):1-19.
[7] KERSKEN D,FELDMEYER B,JANUSSEN D.Sponge communities of the Antarctic Peninsula:influence of environmental variables on species composition and richness[J].Polar Biology,2016(5):851-862.
[8] LARS F,MATTHIAS H,MICHAEL W,et al.Discovery of the novel candidate phylum“Poribacteria”in marine sponges[J].Applied and Environmental Microbiology,2004,70(6):3724-3732.
[9] STACKEBRANDT E,GOODFELLOW M.Nucleic acid techniques in bacterial systematics[M].New Jersey:John Wiley&Sons,1991.
[10] KIM O S,CHO Y J,LEE K,et al.Introducing EzTaxone:A prokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species[J].International Journal of Systematic and Evolutionary Microbiology,2012,62(3):716-721.
[11] TAMURA K,STECHER G,PETERSON D,et al.MEGA6:Molecular evolutionary genetics analysis version 6.0[J].Molecular Biology and Evolution,2013,30(12):2725-2729.
[12] SAITOU N,NEI M.The neighbor-joining method:A new method for reconstructing phylogenetic trees[J].Molecular Biology and Evolution,1987(4):406-425.
[13] FELSENSTEIN J.Confidence limits on phylogenies:An approach using the bootstrap[J].Evolution,1985,39(4):783-791.
[14] 严立恩,陈志荣,王晶晶,等.南极乔治王岛陆地土壤和潮间带放线菌多样性及其抑菌活性研究[J].中国海洋药物,2016,35(1):19-28.
YAN L E, CHEN Z R, WANG J J, et al.Phylogenetic diversity analysis and antimicrobial activity of the terrestrial and intertidal zone of the King George Island at the Western Antarctic[J].Chinese Journal of Marine Drugs,2016,35(1):19-28.
[15] GOEBEL B M,STACKEBRANDT E.Cultural and phylogenetic analysis of mixed microbial populations found in natural and commercial bioleaching environments[J].Applied and Environmental Microbiology,1994,60(5):1614-1621.
[16] TINDALL B J,ROSSELLÓ-MÓRA R,BUSSE H J,et al.Notes on the characterization of prokaryote strains for taxonomic purposes[J].International Journal of Systematic and Evolutionary Microbiology,2010,60(1):249-266.
[17] XU J L,HE J,WANG Z C,et al.Rhodococcus qingshengii sp. nov., a carbendazim-degrading bacterium[J].International Journal of Systematic and Evolutionary Microbiology,2007,57(12):2754-2757.
[18] KAGEYAMA A,TORIKOE K,IWAMOTO M,et al.Nocardia arthritidis sp.nov.,a new pathogen isolated from a patient with rheumatoid arthritis in Japan[J].Journal of Clinical Microbiology,2004,42(6):2366-2371.
[19] LIU C,ZHANG X Y,SU H N,et al.Puniceibacterium antarcticum gen.nov.,sp.nov.,isolated from seawater[J].International Journal of Systematic and Evolutionary Microbiology,2014,64(5):1566-1572.
[20] LAI Q L,LI G Z,LIU X P,et al.Pseudooceanicola atlanticus gen.nov.sp.nov.,isolated from surface seawater of the Atlantic Ocean and reclassification of Oceanicola batsensis, Oceanicola marinus,Oceanicola nitratireducens,Oceanicola nanhaiensis,Oceanicola antarcticus and Oceanicola flagellatus,as Pseudooceanicola batsensis comb. nov.,Pseudooceanicola marinus comb. nov.,Pseudooceanicola nitratireducens comb. nov.,Pseudooceanicola nanhaiensis comb. nov.,Pseudooceanicola antarcticus comb. nov., and Pseudooceanicola flagellatus comb. nov.[J].Antonie van Leeuwenhoek,2015,107(4):1065-1074.
[21] ZHANGDC,NEUNERK,WUJ,et al.Puniceibacterium sediminis sp.nov.isolated from Sakhalin Island,Russia[J].International Journal of Systematic and Evolutionary Microbiology,2015,65(5):1462-1466.
[22] 张海涛.海绵放线菌的分离和多样性研究[D].北京:中国科学院研究生院,2006.
ZHANG H T.Isolation and diversity study of culturable actinobacteria from Chinese marine sponges[D].Beijing:Chinese Academy of Science,2006.
Diversity of sponge-associated culturable bacteria,Great Wall Bay,Antarctica
LIU Huan1,2,YU Yong2,LIAO Li2,LIU Shuang2,3,CHEN Bo1,2
(1.College of Marine Science,Shanghai Ocean University,Shanghai 201306,China;2.SOA Key Laboratory for Polar Science,Polar Research Institute of China,Shanghai 200136,China;3.College of Bioengineering,East China University of Science and Technology,Shanghai 200237,China)
This paper studied the diversity of intertidal sponge-associated culturable bacteria and suspected novel species in the Great Wall Bay,Antarctica.120 bacterial strains were isolated from three unidentified sponge samples in the intertidal sediment by using the selective medium ISP2 which was beneficial to the growth of actinomycetes.Phylogenetic analysis based on 16SrRNA gene sequences showed these strains could be classified into 11 orders,22 genera in 3 phyla and 5 taxa,indicating that these culturable spongeassociated bacteria had a wide genus distribution with high diversity.In addition,the Rhodococcus strains were dominant and contained the rare strains from genus Puniceibacterium,Limnobacter and Aeromicrobium.Analysis on the homology alignment of 16SrRNA gene sequences revealed that strain HM145 had the highest similarity of 97.0%with the type strain Rhodococcus qingshengii from genus Rhodococcus,and strain HQ09 had the highest similarity of 96.9%with the type strain Puniceibacterium antarcticum.These data indicated that strain HM145 and HQ09 would be the suspected novel species.However,their classification status and polyphasic taxonomy at the level of species need to be further studied.
Great Wall Bay;Antarctica;intertidal;sponge-associated culturable bacteria;diversity
S 932.8
A
1004-2490(2017)05-0554-08
2016-05-18
南北极环境综合考察与评估专项/南极周边海域海洋生物多样性和生态考察专题(CHINARE 2016-01-05);南极周边海域与大陆资源潜力综合评估专题(CHINARE 2016-04-02)
刘 欢(1989-),女,安徽阜阳人,硕士研究生,主要从事极地微生物多样性及其新种多相分类研究。
E-mail:buzhilh@hotmail.com
陈 波,研究员。Tel:021-58711026,E-mail:chenbo@pric.org.cn

