白念珠菌生物被膜形成的遗传调控机制
郭东东,岳慧珍,魏羽佳,黄广华
1 贵州医科大学附属医院,贵州 贵阳 550004
2 中国科学院微生物研究所 真菌学国家重点实验室,北京 100101
白念珠菌生物被膜形成的遗传调控机制
郭东东1*,岳慧珍2*,魏羽佳1,黄广华2
1 贵州医科大学附属医院,贵州 贵阳 550004
2 中国科学院微生物研究所 真菌学国家重点实验室,北京 100101
白念珠菌是人体重要的条件性致病真菌。形态的多样性和可塑性是白念珠菌典型的生物学特征,这与它的致病性、宿主适应能力以及有性生殖过程密切相关。白念珠菌生物被膜(Biofilm)是由不同形态细胞(包括酵母型、菌丝和假菌丝)以及胞外基质组成的致密结构,也是毒性和耐药性形成的重要因子。生物被膜对抗真菌药物、宿主免疫系统和环境胁迫因子等都表现出较强的抵抗力和耐受性,是临床上病原真菌感染防治的重大挑战。随着基因表达谱和遗传操作技术的发展,白念珠菌生物被膜的形成及其耐药性的获得所依赖的遗传调控通路和分子调控机制越来越清楚。主要包括MAPK和cAMP介导的信号途径以及Bcr1和Tec1等因子介导的转录调控。此外,白念珠菌生物被膜的形成与形态转换和有性生殖之间存在密切的联系。文中综述了白念珠菌生物被膜形成的遗传调控机制,重点介绍了细胞壁相关蛋白、转录因子和交配型对该过程的调控以及生物被膜的耐药机制。
白念珠菌,生物被膜,调控机制,MTL,耐药性
1 前言
生物被膜是由微生物细胞(细菌或真菌)及其胞外分泌物(Extracellular polymeric substances,EPS)聚集于生物或非生物体表面而形成的一种复杂群体结构。EPS主要包括多糖、蛋白、核酸和脂类等高分子化合物。生物被膜中的细胞与浮游型细胞具有明显不同的表型和基因表达特征。微生物形成生物被膜后能够增强对宿主免疫攻击、营养限制和抗菌药物等有害因子的耐受性[1-2]。人体病原菌可以粘附和定殖在惰性材料、有机聚合物和医疗器材表面,并逐步形成成熟的生物被膜;随后部分病原菌细胞脱离成熟的生物被膜,进行扩散和传播[3-4]。在临床上,患者内置的导尿管、心脏瓣膜、起搏器和假牙等医用材料都为病原菌生物被膜的形成提供了良好的环境和场所,同时为病原微生物的繁殖和传播创造了有利的条件[5-6]。病原菌生物被膜的形成对人类健康构成了严重的威胁,在浅表的粘膜感染和深度系统感染中都起着重要作用[7-9]。2012年美国国立卫生研究院(NIH)研究显示,生物被膜在所有病原微生物感染中所占的比例已超过80%[10]。因此,生物被膜的形成已成为临床病原感染中极具有挑战性的医疗健康问题[5-6]。
白念珠菌Candida albicans是人体中常见的共生菌,同时也是重要的机会性致病真菌[11]。它能够长期地定殖于口腔、胃肠道和泌尿生殖道等部位而不引发疾病;但当宿主免疫能力下降、体内微生物菌群结构失衡或体内环境发生重大改变时,则能够引发不同程度的念珠菌感染(例如皮肤感染、鹅口疮和阴道炎等浅表和粘膜感染,以及心内膜炎、脑膜炎和败血症等系统或深部器官感染)[12]。白念珠菌容易感染免疫功能低下的患者(如艾滋病患者或免疫抑制药物治疗者)及植入医疗器械的健康人群[12-13]。这种感染进一步增加了临床上疾病治疗的风险和成本。白念珠菌对宿主不同环境的适应能力和致病能力依赖于其独特的生物学特性,即形态多样性和可塑性。该菌能够在多种形态之间相互转换。酵母-菌丝型转换是白念珠菌常见的一种形态转换模式[14]。酵母型细胞呈游离态,菌落表面光滑;能够定殖在皮肤和黏膜的表面,并随血液循环进行传播引发宿主系统性感染[15-16]。菌丝是较长的多细胞结构丝状体,菌落表面易形成皱褶或侵入固体培养基内;它有利于白念珠菌侵入和穿透宿主组织细胞[15-16]。White-opaque转换是白念珠菌另一典型的形态转换系统[17]。White细胞较小,呈球形或椭球形,其菌落为白色,表面光滑。Opaque细胞较大,呈长椭球形或柱形,并常有大液泡结构;其菌落在含有荧光桃红染料(Phloxine B)的固体培养基上呈红色,表面较为粗糙。此外,white和opaque对宿主组织细胞具有不同的侵染和破坏能力;white细胞更容易粘附人体上皮细胞并侵入组织,opaque细胞则易于定殖和破坏皮肤和黏膜组织[18-19]。生物被膜是白念珠菌重要的毒力因子。在常规实验条件下,white细胞比 opaque细胞更容易形成生物被膜。因此,对白念珠菌形态转换和生物被膜形成机制的研究将有助于认识念珠菌病的发病机理。
白念珠菌生物被膜的形成与酵母和菌丝型细胞的发育存在密切的关系[20-21]。其中,细胞壁及粘附相关蛋白(例如Als蛋白、Hwp1、Eap1、Ywp1、Ece1和Csh1)不仅是白念珠菌酵母型或菌丝型细胞发育的重要因子,在细胞粘附和生物被膜形成调控中也发挥重要作用[21-22]。另外,由不同的转录因子(例如 Bcr1、Efg1、Tec1、Gat2、Ndt80 和 Rob1,以及 Zap1、Ume6、Adh1 和 Chk1)介导的遗传调控网络通过响应不同的环境信号分子对白念珠菌生物被膜形成的各阶段进行严格的调控,从而确保了生物被膜有条不紊地建成和发育[23-24]。White-opaque形态转换以及MTL(Mating type like locus,交配型基因座)也参与调控白念珠菌生物被膜的形成[25-29]。根据交配基因型的不同,白念珠菌能形成两种不同的生物被膜:一种是具有致病性特征的MTL-杂合型生物被膜,另一种是具有促进交配特性的MTL-纯合型生物被膜[30-31]。这两种不同类型的生物被膜分别受 cAMP/PKA信号通路和性信息素介导的MAPK信号途径的调控[28]。耐药性是生物被膜独特的生物学功能,同时也是白念珠菌研究的热点[32]。对白念珠菌生物被膜的遗传调控机制的深入研究,将有助于进一步揭示其耐药性的分子调控机制,并为临床上预防和治疗生物被膜引发的病原感染提供理论基础。
2 白念珠菌生物被膜的形成及遗传调控
白念珠菌生物被膜结构复杂,由多种不同形态的细胞(包括酵母、菌丝和假菌丝型细胞)和胞外基质组成[10,33-34]。生物被膜的形成是一个连续的过程,主要包括粘附、起始、成熟、脱离和扩散几个阶段[23](图1)。首先,酵母型细胞通过粘附作用定殖在生物或非生物体表面,粘附细胞进行分裂和增殖形成一个紧密的微菌落群体,即生物被膜的基层[21,23]。随后,酵母型细胞开始伸长并形成大量的菌丝或假菌丝,同时伴有胞外基质的产生[21,35]。胞外基质能够有效地保护白念珠菌逃避宿主免疫细胞的吞噬、阻止抗菌药及有害物质的杀伤,并维持生物被膜内部营养物质的稳定[36-37]。最后,酵母型细胞自发地脱离成熟的生物被膜菌体,扩散至其他部位并引发新生生物被膜的形成和发育[8]。白念珠菌生物被膜的形成、发育和扩散过程复杂多变,随着近年来基因组测序、分子生物学和遗传学技术的改进,相关调控机制的研究取得了很大的进展,大量研究表明很多细胞壁及粘附相关蛋白、转录调控因子、MTL基因座和激酶等都参与了这些过程的调控[38](表1)。
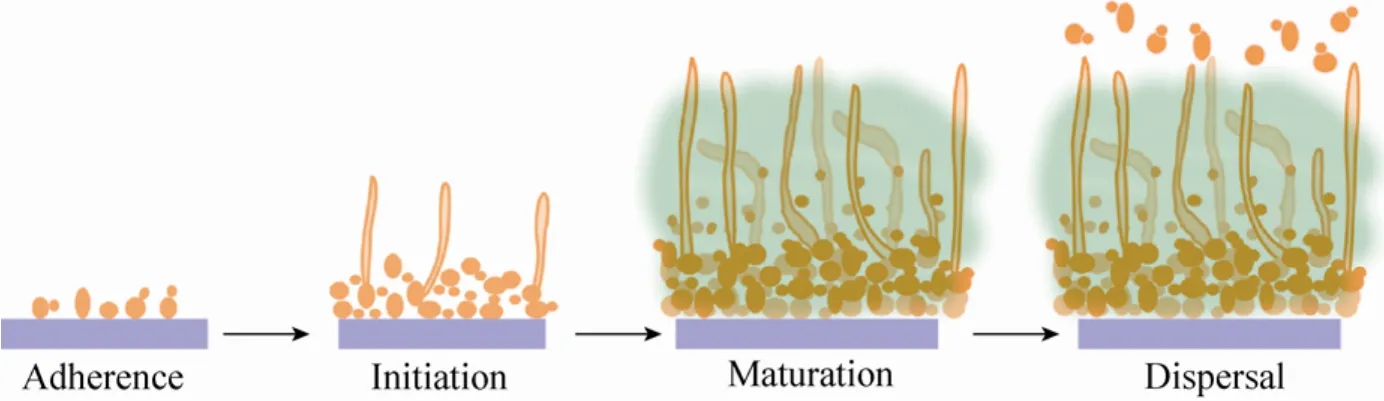
图1 白念珠菌生物被膜形成的四个阶段(粘附、起始、成熟和扩散)Fig.1 Four steps of biofilm development inC.albicans.
2.1 细胞壁及粘附相关蛋白
粘附是白念珠菌成功定殖在黏膜及其他物体表面的第一步,同时也是生物被膜形成并引发病原感染的开始[39]。粘附蛋白,即粘附素,是细胞粘附和定殖过程中的关键诱导因子,它们在生物被膜的形成过程中发挥着重要的作用[40]。粘附是病原菌细胞与宿主表面的一种相互作用,因此,病原菌细胞壁结构、宿主细胞表面受体以及外部环境等都能调控粘附过程的发生[38]。
2.1.1 Als(Agglutinin-like sequence,凝集素样序列)蛋白家族
Als蛋白家族是一类细胞壁糖蛋白,也是白念珠菌中研究最多的一类调控细胞粘附作用的蛋白分子。它由8个具有相同 N-端分泌性信号序列和 C-端 GPI锚定结构域的蛋白组成,即Als1-7和Als9[41]。Als蛋白家族中,Als1、Als3和Als5在白念珠菌与宿主细胞相互粘附过程中发挥着重要作用;其中,Als1和Als3蛋白主要与宿主内皮细胞和上皮细胞发生粘附作用,而 Als5蛋白主要与宿主细胞胞外基质蛋白相结合[42]。
ALS1是白念珠菌ALS基因家族中第一个被鉴定的基因,它与酿酒酵母细胞表面粘附蛋白 α-凝集素具有高度相似的氨基酸序列,并由此推测Als1是白念珠菌中重要的细胞粘附蛋白[43]。Als3蛋白在白念珠菌生物被膜形成过程中起着关键作用,当基因ALS3敲除后,突变株als3/als3在体外生物材料上无法形成正常的生物被膜[44-45]。有意思的是,格氏链球菌Streptococcus gordonnii还可以通过与 Als3蛋白结合促进口腔黏膜中白念珠菌生物被膜的形成[46]。此外,Als1和Als3在不同形态细胞中的蛋白表达水平存在差异;Als1在酵母和菌丝型细胞中均能够表达,而Als3是菌丝细胞特异性表达蛋白[47]。
2.1.2 Hwp1、Rbt1、Eap1和 Ywp1
Hwp1、Rbt1、Eap1和 Ywp1都是 GPI(Glycosyl phosphatidyll inositol,糖基磷脂酰肌醇)-锚定细胞壁蛋白。Hwp1是菌丝特异性细胞壁蛋白,它能调控生物被膜中细胞芽管和菌丝的形成,并促进白念珠菌与宿主细胞的接触和粘附;所以 Hwp1也是白念珠菌重要的致病因子[48-49]。另一重要的GPI-锚定型细胞壁蛋白 Rbt1与Hwp1具有相似的结构和功能,两者具有相同的C-端结构域[50]。RBT1也是菌丝特异性细胞壁蛋白编码基因,其表达水平在菌丝型细胞生长过程中明显上调。当HWP1或RBT1基因敲除以后,其突变株hwp1/hwp1和rbt1/rbt1生物被膜形成能力均受到抑制;而当两者同时敲除后,其双突变株对生物被膜形成的抑制作用明显增强,这说
明HWP1和RBT1在生物被膜形成过程中具有一定的协同作用[50]。此外,Hwp1和Rbt1还能通过响应性信息素来调控生物被膜与细胞交配的过程[51-52]。Eap1是较早发现的细胞壁粘附蛋白,它在酵母型和菌丝型细胞中均能够表达[53]。白念珠菌eap1/eap1突变株不仅大大减弱了对聚苯乙烯的粘附能力,它在宿主体内及体外的生物被膜形成能力也受到了抑制[54]。同样,白念珠菌Eap1在酿酒酵母中能够异源表达并赋予酿酒酵母粘附能力[53],这也进一步表明了Eap1是生物被膜形成过程中重要的细胞壁粘附蛋白。Ywp1是酵母特异性细胞壁蛋白,它是酵母型细胞生物被膜形成的抑制因子。过表达Ywp1使酵母细胞失去粘附能力;而ywp1/ywp1突变株能增强酵母细胞的粘附能力、增加生物被膜的厚度,并减少生物被膜细胞的脱离和扩散[55-56]。
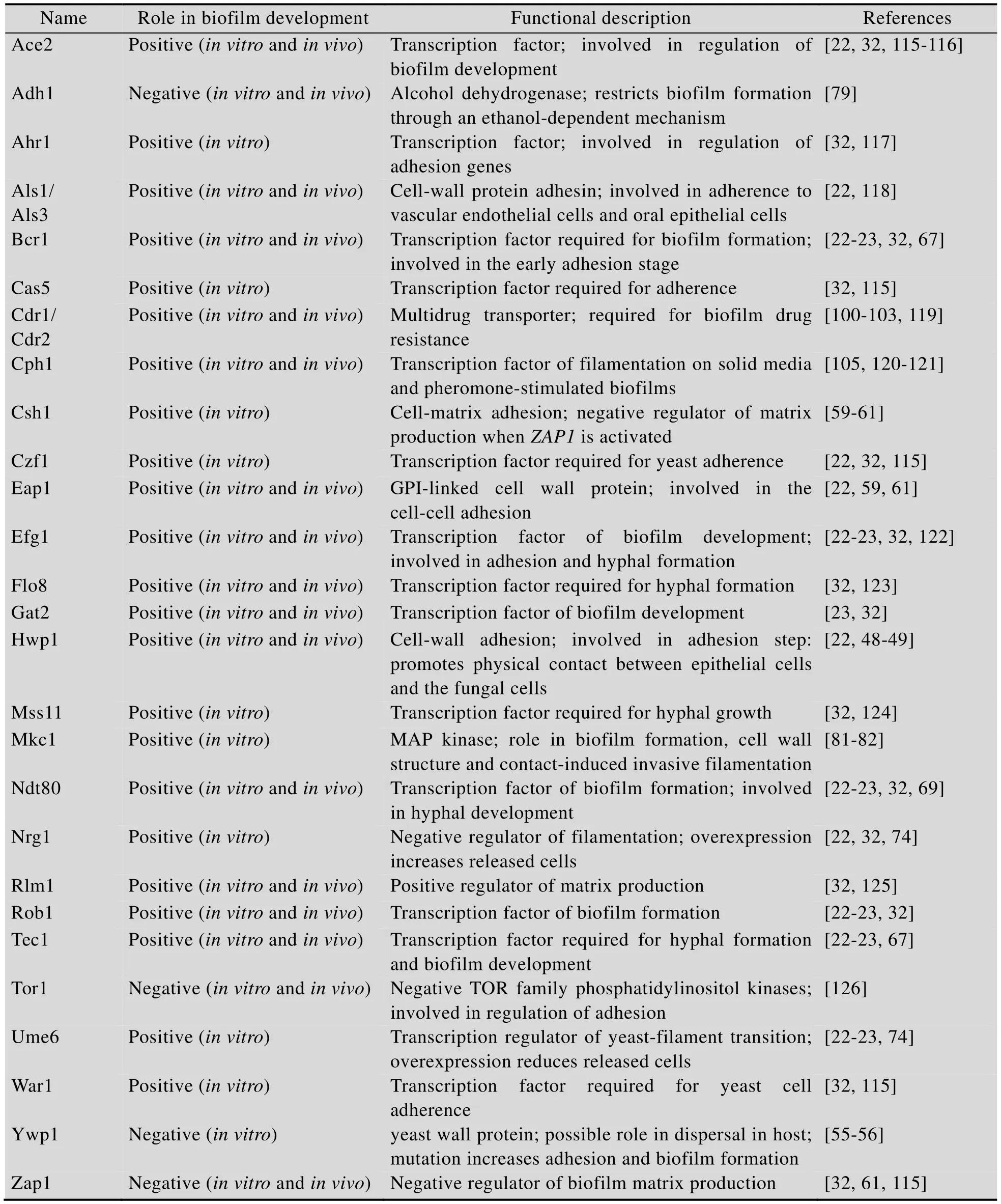
表1 参与白念珠菌生物被膜形成的相关蛋白Table1 Selected proteins involved in biofilm development inC.albicans
2.1.3 Ece1和Csh1
ECE1最早被鉴定为菌丝特异性表达基因,但其生物学功能却不清楚[57]。直到2006年,Nobile等发现Ece1可以促进生物被膜的形成,这可能是由于Ece1能使细胞表面粘附素暴露从而增强了细胞的粘附能力[45]。Csh1是一种细胞表面疏水性蛋白[58]。疏水蛋白与细胞的粘附性密切相关,Csh1能增强细胞粘附并促进生物被膜的形成[59-60]。此外,Csh1还能调控成熟生物被膜胞外基质的产生,而这一作用机制主要受控于转录因子Zap1的表达[61]。
2.2 转录调控因子和蛋白激酶
信号转导途径和转录因子对白念珠菌形态发生起着重要的调控作用,它们同样能调控白念珠菌生物被膜的形成。转录因子和蛋白激酶通过激活或抑制下游相关基因的表达以实现对生物被膜形成和发育的调控作用。
2.2.1 正调控因子
Bcr1、Efg1和Tec1在白念珠菌形态发生过程中起着重要的调控作用,同样也是生物被膜形成的关键转录调控因子。BCR1编码蛋白具有典型的C2H2型锌指结构,它参与调控细胞粘附和生物被膜的形成[45]。转录因子Bcr1的下游靶标基因主要为细胞壁粘附蛋白编码基因ALS1、ALS3和HWP1,但它却不参与调控菌丝的生长,这说明Bcr1主要在生物被膜形成的初始阶段起调控作用[45,48]。另外,Bcr1的下游靶标基因RBT5、PGA10和CSA1均为胞外基质蛋白编码基因,它们可通过表面受体或粘附作用参与调控生物被膜的形成[62-63]。Efg1是白念珠菌生物被膜形成的另一重要转录因子,它对细胞的粘附作用和菌丝生长均有调控作用[64]。Efg1通过调控下游基因Eap1的表达来参与调控细胞的粘附作用[53],此外它还参与转录因子 Tec1的调控通路[65]。Tec1属于TEA/ATTS转录因子家族[66],它对白念珠菌致病性和菌丝生长具有重要调控作用[45]。转录因子Efg1-Tec1共同参与调控生物被膜的菌丝发育过程[65]。有意思的是,转录因子Bcr1的表达也依赖于Tec1的调控作用。Bcr1位于 Efg1-Tec1调控的菌丝发育信号途径的下游,通过赋予菌丝型细胞粘附特性再次参与调控生物被膜的形成[67]。
Gat2(也称Brg1)、Ndt80和Rob1是最近发现的参与调控白念珠菌生物被膜形成的转录因子[23]。Gat2蛋白具有GAGA型锌指结构域,其突变株gat2/gat2表现出菌丝生长缺陷、粘附能力下降的特点,并失去生物被膜形成能力[68]。NDT80是白念珠菌菌丝发育和毒力表现的关键基因,同时也是生物被膜形成的重要转录因子[23,69-70]。它不仅能调控细胞壁蛋白Als3、Hwp1和Ece1的表达,同时对转录因子Tec1和Ume6的表达具有间接调控作用[69]。ROB1缺失株rob1/rob1不能形成生物被膜,但其调控机制却仍不清楚[23]。Bcr1、Efg1、Gat2、Ndt80、Rob1和Tec1六个主要转录因子形成一个相互调控的环路,并调控一系列生物被膜相关基因,是生物被膜形成的调控核心[65]。
2.2.2 负调控因子
除了以上正调控转录因子外,Zap1、Ume6、Adh1和 Chk1等在白念珠菌生物被膜的形成过程中起负调控作用。Zap1是锌感应型转录因子,抑制生物被膜胞外基质的产生,并在生物被膜成熟阶段发挥作用[61]。β-1,3葡聚糖是生物被膜胞外基质的重要成分,Zap1下游靶标基因CSH1、IFD6、GCA1和ADH5能调控β-1,3葡聚糖产生[61]。其中,CSH1和IFD6在Zap1调控下能抑制胞外基质的形成;而GCA1、GCA2和ADH5在Zap1调控下能够促进胞外基质的形成和积累[61]。
在生物被膜形成过程中,酵母型细胞的脱离和扩散是诱导新生生物被膜形成的关键因素[71]。Ume6是白念珠菌菌丝延伸和生长的转录调控因子,它能抑制酵母型细胞脱离生物被膜,并对新生生物被膜的形成和发展具有负调控作用[71-73]。此外,PES1和NRG1是调控菌丝-酵母形态转换和酵母型细胞生长的重要基因,它们能促进成熟生物被膜酵母型细胞的脱离和扩散[71,74]。
法尼醇(Farnesol)是白念珠菌重要的群感效应分子,它能抑制菌丝的生长和生物被膜的形成[75-77]。CHK1是组蛋白激酶编码基因,其突变株chk1/chk1能解除法尼醇对白念珠菌菌丝生长及生物被膜形成的抑制作用[78]。因此,转录因子Chk1可能参与调控法尼醇介导的生物被膜的形成。2006年,Mukherjee等发现乙醇能抑制白念珠菌生物被膜的形成,这一负调控作用与乙醇脱氢酶编码基因ADH1密切相关[79]。在乙醇存在条件下,adh1/adh1突变株具备生物被膜形成能力[79],但Adh1调控生物被膜形成的分子机制仍需进一步探索。
2.2.3 蛋白激酶Mkc1和Cek1
MAPK(Mitogen-activated protein kinase,丝裂原激活蛋白激酶)信号途径是第一个与白念珠菌形态发生相关的信号调控通路,并在不同物种的进化中具有高度保守性[80]。Mkc1是一种MAPK蛋白激酶,它是白念珠菌细胞壁完整结构的一部分,并参与调控细胞表面接触反应[81-82]。表面接触反应是细胞进行菌丝侵入性生长和生物被膜形成的前提条件[82]。MKC1基因敲除以后,mkc1/mkc1突变株形成大量具有菌丝缺陷的生物被膜,而且此异常生物被膜对氟康唑的敏感性增强[24,82]。Cek1是一种胞外信号调节激酶,它也是 MAPK家族的重要成员并在细胞表面接触反应过程起作用[83]。与MKC1一样,CEK1也是白念珠菌形成正常生物被膜的重要调节基因,但Mkc1和Cek1对生物被膜形成的具体调控机制却不清楚[83]。
3 交配型基因座MTL对生物被膜形成的调控作用
白念珠菌通常以二倍体形式存在,有3种不同交配型(MTLa/α,MTLa/a 或MTLα/α)。MTLa/α交配型细胞形成MTL-杂合型生物被膜,MTLa/a或MTLα/α则形成MTL-纯合型生物被膜。这两种生物被膜具有相似的形态和结构特征,但两者却受控于不同的信号传递通路,并在细胞交配和耐药能力方面存在明显差异[29-31]。
3.1 “病原性”和“交配性”生物被膜的形成
自然界中分离得到的白念珠菌大部分为MTLa/α型菌株,由此推测,宿主体内以及内置医用导管表面所形成的生物被膜主要为MTL-杂合型生物被膜。MTL-杂合型生物被膜似乎保留了病原菌固有的致病特性,它能够阻止1.6–112 kDa分子量的物质的渗透、抵御宿主多形核白细胞的侵袭,并对氟康唑表现出较强的耐药性,因此也称它为“病原性”生物被膜[84-86]。
MTLa/α型菌株第5号染色体经过纯合化后产生MTLa/a或MTLα/α型细胞,MTL-纯合型细胞能够从酵母型white细胞转变为具有高效交配能力的opaque细胞[25-26]。与MTLa/α型细胞一样,MTLa/a和MTLα/α型的white细胞也能形成生物被膜;然而,MTLa/a和MTLα/α型的opaque细胞却无法形成生物被膜[27,87]。少量的(<10%)MTL-纯合型 opaque细胞(MTLa/a和MTLα/α等比例混合)通过释放性信息素能够增强MTL-纯合型 white细胞生物被膜的厚度,这也进一步说明 white-opaque形态转换能够调控细胞生物被膜的形成;然而,过多(>40%)MTL-纯合型 opaque细胞反而会影响MTL-纯合型white细胞生物被膜的整体结构而不利于其厚度的增加[27]。MTL-纯合型 white细胞能够自发地低频率转变成opaque细胞,这些opaque细胞可以通过旁分泌系统产生与自身相反交配型的性信息素,从而刺激自身生物被膜的形成[28]。与MTL-杂合型生物被膜相比,MTL-纯合型生物被膜致病性的特征并不明显;低或高分子量的物质以及宿主免疫细胞均能渗入MTL-纯合型生物被膜内,并对抗菌药物表现出敏感性[29]。有意思的是,诱导MTL-纯合型 white细胞生物被膜形成的性信息素同样能促进其少量存在的opaque细胞进行交配[28];因此,MTL-纯合型 white生物被膜也被称为“交配性”生物被膜。
3.2 交配基因型MTL对生物被膜形成的调控
cAMP/PKA和MAPK信号途径是调控白念珠菌形态发生的主要信号传递途径[28,88-90]。MTL-杂合型生物被膜形成的信号调控通路是 Ras1-cAMP/PKA信号途径[28]。其中,cAMP依赖性蛋白激酶编码基因TPK2是MTL-杂合型生物被膜形成的必要基因[28,91-92],而Efg1、Tec1和Bcr1则是MTL-杂合型生物被膜维持正常生长所必需的转录因子[45]。转录因子Tec1不仅能直接调控Bcr1的表达,同时还直接参与调控MTL-杂合型生物被膜的菌丝形成过程[28,45,93]。然而,Tec1同样也是MTL-纯合型生物被膜菌丝形成的关键转录因子,这说明Tec1可能参与调控生物被膜基本构造的形成,而转录因子Bcr1则可能参与MTL-杂合型调控生物被膜胞外基质的产生。
MTL-纯合型 white细胞生物被膜的形成与细胞交配均受控于性信息素介导的MAPK信号途径,但 MAPK信号途径通过激活下游不同的转录因子来调控两种生物过程的发生[28,94-97]。其中,转录因子 Tec1能够与 white细胞特异的性信息素响应元件结合并触发与生物被膜形成相关基因的表达[93],而 Cph1是调控 opaque细胞交配的关键转录因子[98-99]。
4 生物被膜与耐药性形成的关系
白念珠菌生物被膜对抗真菌药物具有一定的耐药性[8,84-85]。生物被膜耐药性形成机制复杂多样,这与生物被膜的不同发展阶段密切相关。其中,药物外排泵(Drug efflux pumps)的正调控作用、胞外基质(Extracellular matrix)的产生以及持留细胞(Persister cells)的存在是造成白念珠菌生物被膜耐药性的重要原因[32]。
4.1 外排泵的正调控作用
药物的外排作用是导致耐药性产生的主要原因。在白念珠菌中,与药物外排泵相关的两大类基因分别为 ATP结合区转运子基因(ATP-binding cassette transporter,CDR1和CDR2)和主要易化子基因(Major faciliator,MDR1)[100-102]。当抗菌药物存在时,浮游型细胞外排泵的表达水平明显升高;有意思的是,生物被膜外排泵的表达却并不取决于抗菌药物,它在细胞发生粘附后几个小时便开始持续上调[23,102-106]。由此可见,外排泵的自主高表达作用促成了生物被膜高度耐药的生物学特性。
4.2 胞外基质与耐药性
胞外基质的分泌是生物被膜形成耐药性的另一关键原因。生物被膜在成熟阶段会释放出大量的胞外基质,它作为天然的物理屏障可以阻挡药物的渗入,并维持和保护生物被膜结构的完整性[35-36,107-108]。由于胞外基质的主要成分难以鉴定,这使得深入了解其耐药机制的研究更加具有挑战性。最近研究发现,胞外基质主要是由大分子物质蛋白和糖蛋白(55%)、碳水化合物(25%)、脂质(15%)和核酸(5%)构成[109]。其中,蛋白质已鉴定出500多种,大部分为酶类;由此推测,胞外基质可能通过酶解外源分子保护自身并为生物被膜的发展提供营养物质。多糖是胞外基质第二大组成成分,主要为甘露聚糖复合体[109]。目前已知,β-1,3葡聚糖是胞外基质发挥耐药作用的重要分子;当用β-1,3葡聚糖酶处理生物被膜时,生物被膜对氟康唑的敏感性明显增加;而且,外源添加β-1,3葡聚糖也能够增强浮游型细胞对氟康唑的抗药性[107,110]。另外,胞外DNA在生物被膜抵抗药物的杀伤过程中也表现出间接的促进作用[111]。有研究报道,脱氧核糖核酸酶能够增强卡泊芬净和两性霉素 B对成熟期生物被膜的破坏和杀伤能力[112]。然而,β-1,3葡聚糖和胞外DNA只是胞外基质组成的一小部分,这更充分说明胞外基质并不单单是生物被膜所释放的细胞壁组分,它还具备独特的生物学功能。
4.3 持留细胞
持留细胞(Persister cells)是生物被膜随机产生的少量处于休眠状态的酵母型细胞。它们也是白念珠菌生物被膜形成耐药性的关键因子,对抗真菌药物具有较强的耐受性[113]。虽然生物被膜中持留细胞的形成和作用机制尚不清楚,但它的耐药性却不依赖于细胞壁的构成和外排泵的表达,而与持留细胞所处的休眠代谢状态有关[113-114]。持留细胞对生物被膜耐药性的形成至关重要,但它的形成和调控机制却仍有待进一步深入研究。
5 结论与展望
生物被膜不仅是白念珠菌形态多样性的表现,更是对宿主环境变化的一种适应性策略。生物被膜的形成为白念珠菌的定殖和侵染提供了更加有利的条件,同时也为临床上预防和治疗白念珠菌病带来了困难和挑战。近年来,白念珠菌生物被膜的研究越来越深入,尤其在生物被膜的形成和分子调控机制、MTL依赖性生物被膜的信号调控网络以及耐药性形成的分子机制等方面取得了重大进展,但该领域仍存在很多问题亟待进一步研究。例如,菌丝发育和 white-opaque形态转换怎样调控生物被膜的形成?MTL-纯合型生物被膜胞外基质的产生是如何被调控的?其他病原真菌中是否存在类似于白念珠菌中不同功能的“病原性”和“交配性”生物被膜?生物被膜胞外基质的组成成分还有哪些?其生物学功能是否与耐药性有关?这些研究将有助于加深人们对白念珠菌生物被膜的生物学特性及分子调控机制的认识和了解,为临床上防治白念珠菌引起的感染提供理论依据,并为新型抗真菌药物的研发提供新思路。
[1]Whitchurch CB, Tolker-Nielsen T, Ragas PC, et al.Extracellular DNA required for bacterial biofilm formation.Science,2002,295(5559):1487.
[2]Flemming HC, Wingender J.The biofilm matrix.Nat Rev Microbiol,2010,8(9):623–633.
[3]Dongari-Bagtzoglou A.Pathogenesis of mucosal biofilm infections: challenges and progress.Expert Rev Anti Infect Ther,2008,6(2):201–208.
[4]Nikolaev I, Plakunov VK.Biofilm——“City of microbes” or an analogue of multicellular organisms? Mikrobiologiia,2007,76(2):149–163.
[5]Allegranzi B, Nejad SB, Combescure C, et al.Burden of endemic health-care-associated infection in developing countries: systematic review and meta-analysis.Lancet,2011,377(9761):228–241.
[6]Zarb P, Coignard B, Griskeviciene J, et al.The European Centre for Disease Prevention and Control(ECDC)pilot point prevalence survey of healthcare-associated infections and antimicrobial use.Euro Surveill,2012,17(46):20316.
[7]Donlan RM.Biofilm formation: a clinically relevant microbiological process.Clin Infect Dis,2001,33(8):1387–1392.
[8]Donlan RM, Costerton JW.Biofilms: survival mechanisms of clinically relevant microorganisms.Clin Microbiol Rev,2002,15(2):167–193.
[9]Kojic EM, Darouiche RO.Candidainfections of medical devices.Clin Microbiol Rev,2004,17(2):255–267.
[10]Fox EP, Nobile CJ.A sticky situation: untangling the transcriptional network controlling biofilm development inCandida albicans.Transcription,2012,3(6):315–322.
[11]Brown GD, Denning DW, Gow NAR, et al.Hidden killers: human fungal infections.Sci Transl Med,2012,4(165):165rv13.
[12]Pappas PG, Rex JH, Sobel JD, et al.Guidelines for treatment of candidiasis.Clin Infect Dis,2004,38(2):161–189.
[13]Calderone RA, Fonzi WA.Virulence factors ofCandida albicans.Trends Microbiol,2001,9(7):327–335.
[14]Odds FC.Morphogenesis inCandida albicans.Crit Rev Microbiol,1985,12(1):45–93.
[15]Sudbery PE.Growth ofCandida albicanshyphae.Nat Rev Microbiol,2011,9(10):737–748.
[16]Zhu WD, Filler SG.Interactions ofCandida albicanswith epithelial cells.Cell Microbiol,2010,12(3):273–282.
[17]Slutsky B, Staebell M, Anderson J, et al."White-opaque transition": a second high-frequency switching system inCandida albicans.J Bacteriol,1987,169(1):189–197.
[18]Lan CY, Newport G, Murillo LA, et al.Metabolic specialization associated with phenotypic switching inCandida albicans.Proc Natl Acad Sci USA,2002,99(23):14907–14912.
[19]Kennedy MJ, Rogers AL, Hanselmen LR, et al.Variation in adhesion and cell surface hydrophobicity inCandida albicanswhite and opaque phenotypes.Mycopathologia,1988,102(3):149–156.
[20]Fanning S, Mitchell AP.Fungal biofilms.PLoS Pathog,2012,8(4): e1002585.
[21]Finkel JS, Mitchell AP.Genetic control ofCandida albicansbiofilm development.Nat Rev Microbiol,2011,9(2):109–118.
[22]Araújo D, Henriques M, Silva S.Portrait ofCandidaspecies biofilm regulatory network genes.Trends Microbiol,2017,25(1):62–75.
[23]Nobile CJ, Fox EP, Nett JE, et al.A recently evolved transcriptional network controls biofilm development inCandida albicans.Cell,2012,148(1/2):126–138.
[24]Nobile CJ, Mitchell AP.Genetics and genomics ofCandidaalbicansbiofilm formation. Cell Microbiol,2006,8(9):1382–1391.
[25]Lockhart SR, Daniels KJ, Zhao R, et al.Cell biology of mating inCandida albicans.Eukaryot Cell,2003,2(1):49–61.
[26]Miller MG, Johnson AD.White-opaque switching inCandida albicans is controlled by mating-type locus homeodomain proteins and allows efficient mating.Cell,2002,110(3):293–302.
[27]Daniels KJ, Srikantha T, Lockhart SR, et al.Opaque cells signal white cells to form biofilms inCandida albicans.EMBO J,2006,25(10):2240–2252.
[28]Yi S, Sahni N, Daniels KJ, et al.Alternative mating type configurations(a/α versus a/a or α/α)ofCandida albicansresult in alternative biofilms regulated by different pathways.PLoS Biol,2011,9(8): e1001117.
[29]Soll DR, Daniels KJ.Plasticity ofCandida albicansbiofilms.Microbiol Mol Biol Rev,2016,80(3):565–595.
[30]Soll DR.Why doesCandida albicansswitch?FEMS Yeast Res,2009,9(7):973–989.
[31]Soll DR.The evolution of alternative biofilms in an opportunistic fungal pathogen: an explanation for how new signal transduction pathways may evolve.Infect Genet Evol,2014,22:235–243.
[32]Nobile CJ, Johnson AD.Candida albicansbiofilms and human disease.Annu Rev Microbiol,2015,69:71–92.
[33]Ramage G, Mowat E, Jones B, et al.Our current understanding of fungal biofilms.Crit Rev Microbiol,2009,35(4):340–355.
[34]Ramage G, Saville SP, Thomas DP, et al.Candidabiofilms: an update.Eukaryot Cell,2005,4(4):633–638.
[35]Al-Fattani MA, Douglas LJ.Biofilm matrix ofCandida albicansandCandida tropicalis: chemical composition and role in drug resistance.J Med Microbiol,2006,55(Pt8):999–1008.
[36]Baillie GS, Douglas LJ.Matrix polymers ofCandidabiofilms and their possible role in biofilm resistance to antifungal agents.J Antimicrob Chemother,2000,46(3):397–403.
[37]Chandra J, Kuhn DM, Mukherjee PK, et al.Biofilm formation by the fungal pathogenCandida albicans:development, architecture, and drug resistance.J Bacteriol,2001,183(18):5385–5394.
[38]Wang YC, Huang SH, Lan CY, et al.Prediction of phenotype-associated genes via a cellular network approach: aCandida albicansinfection case study.PLoS ONE,2012,7(4): e35339.
[39]Silva S, Negri M, Henriques M, et al.Candida glabrata,Candida parapsilosisandCandida tropicalis: biology, epidemiology, pathogenicity and antifungal resistance.FEMS Microbiol Rev,2012,36(2):288–305.
[40]Silva S, Negri M, Henriques M, et al.Adherence and biofilm formation of non-Candida albicansCandidaspecies.Trends Microbiol,2011,19(5):241–247.
[41]Mayer FL, Wilson D, Hube B.Candida albicans pathogenicity mechanisms.Virulence,2013,4(2):119–128.
[42]Tronchin G, Pihet M, Lopes-Bezerra LM, et al.Adherence mechanisms in human pathogenic fungi.Med Mycol,2008,46(8):749–772.
[43]Cota E, Hoyer LL.TheCandida albicansagglutinin-like sequence family of adhesins:functional insights gained from structural analysis.Future Microbiol,2015,10(10):1635–1648.
[44]Liu YP, Filler SG.Candida albicansAls3, a multifunctional adhesin and invasin.Eukaryot Cell,2011,10(2):168–173.
[45]Nobile CJ, Andes DR, Nett JE, et al.Critical role of Bcr1-dependent adhesins inC.albicansbiofilm formationin vitroandin vivo.PLoS Pathog,2006,2(7): e63.
[46]Bamford CV, d’Mello A, Nobbs AH, et al.Streptococcus gordonii modulatesCandida albicansbiofilm formation through intergeneric communication.Infect Immun,2009,77(9):3696–3704.
[47]Nobile CJ, Schneider HA, Nett JE, et al.Complementary adhesin function inC.albicansbiofilm formation.Curr Biol,2008,18(14):1017–1024.
[48]Henriques M, Azeredo J, Oliveira R.Candidaspecies adhesion to oral epithelium: factors involved and experimental methodology used.Crit Rev Microbiol,2006,32(4):217–226.
[49]Orsi CF, Borghi E, Colombari B, et al.Impact ofCandida albicanshyphal wall protein1(HWP1)genotype on biofilm production and fungal susceptibility to microglial cells.Microb Pathog,2014,(69/70):20–27.
[50]Ene IV, Bennett RJ.Hwp1 and related adhesins contribute to both mating and biofilm formation inCandida albicans.Eukaryot Cell,2009,8(12):1909–1913.
[51]Bennett RJ, Uhl MA, Miller MG, et al.Identification and characterization of aCandida albicansmating pheromone.Mol Cell Biol,2003,23(22):8189–8201.
[52]Daniels KJ, Lockhart SR, Staab JF, et al.The adhesin Hwp1 and the first daughter cell localize to the a/a portion of the conjugation bridge duringCandida albicansmating.Mol Biol Cell,2003,14(12):4920–4930.
[53]Li F, Palecek SP.EAP1, aCandida albicansgene involved in binding human epithelial cells.Eukaryot Cell,2003,2(6):1266–1273.
[54]Li F, Svarovsky MJ, Karlsson AJ, et al.EAP1p, an adhesin that mediatesCandida albicansbiofilm formationin vitroandin vivo.Eukaryot Cell,2007,6(6):931–939.
[55]Granger BL, Flenniken ML, Davis DA, et al.Yeast wall protein1 ofCandida albicans.Microbiology,2005,151(Pt5):1631–1644.
[56]Granger BL.Insight into the antiadhesive effect of yeast wall protein1 ofCandida albicans.Eukaryot Cell,2012,11(6):795–805.
[57]Birse CE, Irwin MY, Fonzi WA, et al.Cloning and characterization ofECE1, a gene expressed in association with cell elongation of the dimorphic pathogenCandida albicans.Infect Immun,1993,61(9):3648–3655.
[58]Singleton DR, Hazen KC.Differential surface localization and temperature-dependent expression of theCandidaalbicansCSH1 protein.Microbiology,2004,150(Pt2):285–292.
[59]Silva TM, Glee PM, Hazen KC.Influence of cell surface hydrophobicity on attachment ofCandida albicansto extracellular matrix proteins.J Med Vet Mycol,1995,33(2):117–122.
[60]Samaranayake YH, Wu PC, Samaranayake LP, et al. Relationship between the cell surface hydrophobicity and adherence ofCandida kruseiandCandida albicansto epithelial and denture acrylic surfaces.APMIS,1995,103(10):707–713.
[61]Nobile CJ, Nett JE, Hernday AD, et al.Biofilm matrix regulation byCandida albicansZap1.PLoS Biol,2009,7(6): e1000133.
[62]Ding C, Vidanes GM, Maguire SL, et al.Conserved and divergent roles of Bcr1 and CFEM proteins inCandida parapsilosisandCandida albicans.PLoS ONE,2011,6(12): e28151.
[63]Pérez A, Pedrós B, Murgui A, et al.Biofilm formation byCandida albicansmutants for genes coding fungal proteins exhibiting the eight-cysteine-containing CFEM domain.FEMS Yeast Res,2006,6(7):1074–1084.
[64]Connolly LA, Riccombeni A, Grózer Z, et al.The APSES transcription factor Efg1 is a global regulator that controls morphogenesis and biofilm formation inCandida parapsilosis.Mol Microbiol,2013,90(1):36–53.
[65]Berman J, Sudbery PE.Candida albicans: a molecular revolution built on lessons from budding yeast.Nat Rev Genet,2002,3(12):918–932.
[66]Schweizer A, Rupp S, Taylor BN, et al.The TEA/ATTS transcription factor CaTec1p regulates hyphal development and virulence inCandida albicans.Mol Microbiol,2000,38(3):435–445.
[67]Nobile CJ, Mitchell AP.Regulation of cell-surface genes and biofilm formation by theC.albicanstranscription factor Bcr1p.Curr Biol,2005,15(12):1150–1155.
[68]Du H, Guan GB, Xie J, et al.Roles ofCandida albicansGat2, a GATA-type zinc finger transcription factor, in biofilm formation,filamentous growth and virulence.PLoS ONE,2012,7(1): e29707.
[69]Sellam A, Askew C, Epp E, et al.Role of transcription factor CaNdt80p in cell separation,hyphal growth, and virulence inCandida albicans.Eukaryot Cell,2010,9(4):634–644.
[70]Chen CG, Yang YL, Shih HI, et al.CaNdt80 is involved in drug resistance inCandida albicans by regulatingCDR1.Antimicrob Agents Chemother,2004,48(12):4505–4512.
[71]Uppuluri P, Chaturvedi AK, Srinivasan A, et al.Dispersion as an important step in theCandida albicansbiofilm developmental cycle.PLoS Pathog,2010,6(3): e1000828.
[72]Banerjee M, Thompson DS, Lazzell A, et al.UME6,a novel filament-specific regulator ofCandida albicanshyphal extension and virulence.Mol Biol Cell,2008,19(4):1354–1365.
[73]Childers DS, Kadosh D.Filament condition-specific response elements control the expression ofNRG1andUME6, key transcriptional regulators of morphology and virulence inCandida albicans.PLoS ONE,2015,10(3): e0122775.
[74]Uppuluri P, Pierce CG, Thomas DP, et al.The transcriptional regulator Nrg1p controlsCandida albicansbiofilm formation and dispersion.EukaryotCell,2010,9(10):1531–1537.
[75]Hornby JM, Jensen EC, Lisec AD, et al.Quorum sensing in the dimorphic fungusCandida albicansis mediated by farnesol.Appl Environ Microbiol,2001,67(7):2982–2992.
[76]Oh KB, Miyazawa H, Naito T, et al.Purification and characterization of an autoregulatory substance capable of regulating the morphological transition inCandida albicans.Proc Natl Acad Sci USA,2001,98(8):4664–4668.
[77]Ramage G, Saville SP, Wickes BL, et al.Inhibition ofCandida albicansbiofilm formation by farnesol,a quorum-sensing molecule. Appl Environ Microbiol,2002,68(11):5459–5463.
[78]Kruppa M, Krom BP, Chauhan N, et al.The two-component signal transduction protein Chk1p regulates quorum sensing inCandida albicans.Eukaryot Cell,2004,3(4):1062–1065.
[79]Mukherjee PK, Mohamed S, Chandra J, et al.Alcohol dehydrogenase restricts the ability of the pathogenCandida albicansto form a biofilm on catheter surfaces through an ethanol-based mechanism.Infect Immun,2006,74(7):3804–3816.
[80]Moosa MYS, Alangaden GJ, Manavathu E, et al.Resistance to amphotericin B does not emerge during treatment for invasive aspergillosis.J Antimicrob Chemother,2002,49(1):209–213.
[81]Navarro-García F, Alonso-Monge R, Rico H, et al.A role for the MAP kinase geneMKC1in cell wall construction and morphological transitions inCandida albicans.Microbiology,1998,144(Pt2):411–424.
[82]Kumamoto CA.A contact-activated kinase signalsCandida albicansinvasive growth and biofilm development.Proc Natl Acad Sci USA,2005,102(15):5576–5581.
[83]Csank C, Schröppel K, Leberer E, et al.Roles of theCandida albicansmitogen-activated protein kinase homolog, Cek1p, in hyphal development and systemic candidiasis.Infect Immun,1998,66(6):2713–2721.
[84]Kumamoto CA.Candidabiofilms.Curr Opin Microbiol,2002,5(6):608–611.
[85]Douglas LJ.Candidabiofilms and their role in infection.Trends Microbiol,2003,11(1):30–36.
[86]Blankenship JR, Mitchell AP.How to build a biofilm: a fungal perspective.Curr Opin Microbiol,2006,9(6):588–594.
[87]Yi S, Sahni N, Daniels KJ, et al.Self-induction of a/a or α/α biofilms inCandida albicansis a pheromone-based paracrine system requiring switching.Eukaryot Cell,2011,10(6):753–760.
[88]Whiteway M, Bachewich C.Morphogenesis inCandida albicans.Annu Rev Microbiol,2007,61:529–553.
[89]Leberer E, Harcus D, Broadbent ID, et al.Signal transduction through homologs of the Ste20p and Ste7p protein kinases can trigger hyphal formation in the pathogenic fungusCandida albicans.Proc Natl Acad Sci USA,1996,93(23):13217–13222.
[90]Liu H, Kohler J, Fink GR.Suppression of hyphal formation inCandida albicansby mutation of a STE12 homolog.Science,1994,266(5191):1723–1726.
[91]Bockmuhl DP, Ernst JF. A potential phosphorylation site for an A-type kinase in the Efg1 regulator protein contributes to hyphal morphogenesis ofCandida albicans.Genetics,2001,157(4):1523–1530.
[92]Wang A, Raniga PP, Lane S, et al.Hyphal chain formation inCandida albicans: Cdc28-Hgc1 phosphorylation of Efg1 represses cell separation genes.Mol Cell Biol,2009,29(16):4406–4416.
[93]Sahni N, Yi S, Daniels KJ, et al.Tec1 mediates the pheromone response of the white phenotype ofCandida albicans: insights into the evolution of new signal transduction pathways.PLoS Biol,2010,8(5): e1000363.
[94]Sahni N, Yi S, Daniels KJ, et al.Genes selectively up-regulated by pheromone in white cells are involved in biofilm formation inCandida albicans.PLoS Pathog,2009,5(10): e1000601.
[95]Sahni N, Yi S, Pujol C, et al.The white cell response to pheromone is a general characteristic ofCandida albicansstrains.Eukaryot Cell,2009,8(2):251–256.
[96]Yi S, Sahni N, Daniels KJ, et al.The same receptor,G protein, and mitogen-activated protein kinase pathway activate different downstream regulators in the alternative white and opaque pheromone responses ofCandida albicans.Mol Biol Cell,2008,19(3):957–970.
[97]Yi S, Sahni N, Pujol C, et al.ACandida albicans-specific region of the alpha-pheromone receptor plays a selective role in the white cell pheromone response.Mol Microbiol,2009,71(4):925–947.
[98]Chen J, Chen J, Lane S, et al.A conservedmitogen-activated protein kinase pathway is required for mating inCandida albicans.Mol Microbiol,2002,46(5):1335–1344.
[99]Magee BB, Legrand M, Alarco AM, et al.Many of the genes required for mating inSaccharomyces cerevisiaeare also required for mating inCandida albicans.Mol Microbiol,2002,46(5):1345–1351.
[100]Anderson JB. Evolution of antifungal-drug resistance: mechanisms and pathogen fitness.Nat Rev Microbiol,2005,3(7):547–556.
[101]Cowen LE.The evolution of fungal drug resistance:modulating the trajectory from genotype to phenotype.Nat Rev Microbiol,2008,6(3):187–198.
[102]Ramage G, Bachmann S, Patterson TF, et al.Investigation of multidrug efflux pumps in relation to fluconazole resistance inCandida albicansbiofilms.J Antimicrob Chemother,2002,49(6):973–980.
[103]Mateus C, Crow SA Jr, Ahearn DG.Adherence ofCandida albicansto silicone induces immediate enhanced tolerance to fluconazole.Antimicrob Agents Chemother,2004,48(9):3358–3366.
[104]Mukherjee PK, Chandra J, Kuhn DM, et al.Mechanism of fluconazole resistance inCandida albicansbiofilms: phase-specific role of efflux pumps and membrane sterols.Infect Immun,2003,71(8):4333–4340.
[105]Nett JE, Lepak AJ, Marchillo K, et al.Time course global gene expression analysis of anin vivo Candidabiofilm.J Infect Dis,2009,200(2):307–313.
[106]Yeater KM, Chandra J, Cheng G, et al.Temporal analysis ofCandida albicansgene expression during biofilm development.Microbiology,2007,153(Pt8):2373–2385.
[107]Nett J, Lincoln L, Marchillo K, et al.Putative role of β-1,3 glucans inCandida albicansbiofilm resistance.Antimicrob Agents Chemother,2007,51(2):510–520.
[108]Nett JE, Sanchez H, Cain MT, et al.Genetic basis ofCandidabiofilm resistance due to drug sequestering matrix glucan.J Infect Dis,2010,202(1):171–175.
[109]Zarnowski R, Westler WM, Lacmbouh GA, et al.Novel entries in a fungal biofilm matrix encyclopedia.mBio,2014,5(4): e01333–14.
[110]Mitchell KF, Taff HT, Cuevas MA, et al.Role of matrix β-1,3 glucan in antifungal resistance of non-albicansCandidabiofilms.Antimicrob Agents Chemother,2013,57(4):1918–1920.
[111]Martins M, Uppuluri P, Thomas DP, et al.Presence of extracellular DNA in theCandida albicansbiofilm matrix and its contribution to biofilms.Mycopathologia,2010,169(5):323–331.
[112]Martins M, Henriques M, Lopez-Ribot JL, et al.Addition of DNase improves thein vitroactivity of antifungal drugs againstCandida albicansbiofilms.Mycoses,2012,55(1):80–85.
[113]LaFleur MD, Kumamoto CA, Lewis K.Candida albicansbiofilms produce antifungal-tolerant persister cells.Antimicrob Agents Chemother,2006,50(11):3839–3846.
[114]Khot PD, Suci PA, Miller RL, et al.A small subpopulation of blastospores inCandida albicansbiofilms exhibit resistance to amphotericin B associated with differential regulation of ergosterol andβ-1,6-glucan pathway genes.Antimicrob Agents Chemother,2006,50(11):3708–3716.
[115]Finkel JS, Xu WJ, Huang D, et al.Portrait ofCandida albicansadherence regulators.PLoS Pathog,2012,8(2): e1002525.
[116]Kelly MT, MacCallum DM, Clancy SD, et al.TheCandidaalbicansCaACE2gene affects morphogenesis, adherence and virulence.Mol Microbiol,2004,53(3):969–983.
[117]Askew C, Sellam A, Epp E, et al.The zinc cluster transcription factor Ahr1p directs Mcm1p regulation ofCandida albicansadhesion.Mol Microbiol,2011,79(4):940–953.
[118]Sheppard DC, Yeaman MR, Welch WH, et al.Functional and structural diversity in the Als protein family ofCandida albicans.J Biol Chem,2004,279(29):30480–30489.
[119]Watamoto T, Samaranayake LP, Egusa H, et al.Transcriptional regulation of drug-resistance genes inCandida albicansbiofilms in response to antifungals.J Med Microbiol,2011,60(Pt9):1241–1247.
[120]Lewis RE, Lo HJ, Raad II, et al.Lack of catheter infection by theefg1/efg1 cph1/cph1double-null mutant, aCandida albicansstrain that is defective in filamentous growth. Antimicrob Agents Chemother,2002,46(4):1153–1155.
[121]Lin CH, Kabrawala S, Fox EP, et al.Genetic control of conventional and pheromone-stimulated biofilm formation inCandida albicans.PLoS Pathog,2013,9(4): e1003305.
[122]Ramage G, VandeWalle K, López-Ribot JL, et al.The filamentation pathway controlled by the Efg1 regulator protein is required for normal biofilm formation and development inCandida albicans.FEMS Microbiol Lett,2002,214(1):95–100.
[123]Fox EP, Bui CK, Nett JE, et al.An expanded regulatory network temporally controlsCandida albicansbiofilm formation.Mol Microbiol,2015,96(6):1226–1239.
[124]Tsai PW, Chen YT, Yang CY, et al.The role of Mss11 inCandida albicansbiofilm formation.Mol Genet Genomics,2014,289(5):807–819.
[125]Nett JE, Sanchez H, Cain MT, et al.Interface ofCandida albicansbiofilm matrix-associated drug resistance and cell wall integrity regulation.Eukaryot Cell,2011,10(12):1660–1669.
[126]Bastidas RJ, Heitman J, Cardenas ME.The protein kinase Tor1 regulates adhesin gene expression inCandida albicans.PLoS Pathog,2009,5(2):e1000294.
(本文责编 郝丽芳)
Genetic regulatory mechanisms ofCandida albicansbiofilm formation
Dongdong Guo1*, Huizhen Yue2*, Yujia Wei1, and Guanghua Huang2
1Affiliated Hospital of Guizhou Medical University,Guiyang550004,Guizhou,China
2State Key Laboratory of Mycology,Institute of Microbiology,Chinese Academy of Sciences,Beijing100101,China
Candida albicansis an important opportunistic fungal pathogen of humans.Phenotypic plasticity is a typical biological feature ofC.albicans, which is associated with pathogenicity, host adaptation, and sexual reproduction.BiofilmofC.albicansis a complex community formed by different morphological types of cells(yeast, hyphae and pseudohyphae)and secreted extracellular matrix.C.albicansbiofilms are intrinsically resistant to antifungal drugs, the host immune system, and environmental stresses.Biofilm is an important virulence factor and a major clinical challenge.With the development of new technologies in global gene expression profiles and genetic manipulation, the regulatory mechanisms that governC.albicansbiofilm development and drug resistance become more and more clear.Major regulatory mechanisms involve the MAPK and cAMP signaling pathways and transcriptional regulators such as Bcr1 and Tec1.In addition, morphological transitions and sexual reproduction are also involved in the regulation of biofilm development.In this review, we focus on the genetic regulatory mechanisms of biofilm including the roles of cell-wall related proteins,transcription factors, and theMTLlocus.In the last section, we also summarize the mechanisms of drug resistance of biofilm inC.albicans.
Candida albicans, biofilm, regulatory mechanism,MTL, drug resistance
March28,2017;Accepted:July19,2017
s:Yujia Wei.Tel: +86-851-86774009, E-mail: weiyujia1@sina.com Guanghua Huang.Tel: +86-10-64806133, E-mail: huanggh@im.ac.cn


郭东东, 岳慧珍, 魏羽佳, 等.白念珠菌生物被膜形成的遗传调控机制.生物工程学报,2017,33(9):1567–1581.
Guo DD, Yue HZ, Wei YJ, et al.Genetic regulatory mechanisms ofCandida albicansbiofilm formation.Chin J Biotech,2017,33(9):1567–1581.
Supported by:the Special Foundation of Governor for Talents in Science and Technology and Education of Guizhou Province(No.[2011]28).
*These authors contributed equally to this work.
贵州省优秀科技教育人才省长专项资金(No.[2011]28号)资助。
时间:2017-08-24
http://kns.cnki.net/kcms/detail/11.1998.Q.20170824.0944.002.html
魏羽佳 贵州医科大学附属医院主任医师、皮肤科副主任。长期从事真菌性皮肤病和性病的中西医结合治疗研究,擅长各种性传播疾病、皮肤湿疹类皮肤病及癣菌病的诊治。近年来,致力于临床皮肤及侵袭性真菌致病机理及真菌耐药机理研究。
黄广华 中国科学院微生物研究所研究员、博士生导师。2012年入选中科院“百人计划”,2016年获国家自然科学基金委员会“杰出青年科学基金”资助。近年来主要从事人体致病真菌白念珠菌形态发生、有性生殖和分子致病机理等方面研究,在PLoS Biology、PLoS Genetics和Molecular Microbiology等国际著名杂志上发表了一系列文章。

