铜绿假单胞菌铁摄取与生物被膜形成研究进展
于珊,马旅雁
1中国科学院生物物理研究所,北京 100101
2中国科学院微生物研究所,北京 100101
铜绿假单胞菌铁摄取与生物被膜形成研究进展
于珊1,马旅雁2
1中国科学院生物物理研究所,北京 100101
2中国科学院微生物研究所,北京 100101
生物被膜是单细胞微生物通过其分泌的胞外多聚基质粘附于介质表面并将其自身包绕其中而成的膜样微生物细胞聚集物。生物被膜的形成使细菌具有更强的适应外界环境的能力,也是导致微生物产生耐药性及慢性感染性疾病难以治疗的重要原因之一。铜绿假单胞菌在肺部的定殖是肺囊性纤维化病患者发病和死亡主要原因,其造成的感染通常与形成抗生素抗性极强的生物被膜有关。铜绿假单胞菌生物被膜的形成受控于多种复杂的细菌调控体系之下,包括群体感应系统及参与调节胞外多聚基质合成的双组分调控系统等。此外,为了利用低浓度的环境铁来维持生存并完成各种生理功能,铜绿假单胞菌进化出了一系列铁摄取系统,这些系统对其毒力因子的释放和生物被膜的形成又起着重要的调控作用。本文主要对铜绿假单胞菌生物被膜的形成与调控机制及其铁摄取系统进行了综述,为进一步了解及清除铜绿假单胞菌引发的问题提供途径与思路。
铜绿假单胞菌,生物被膜,铁摄取,群体感应,调控因子
生物被膜(Biofilm)是指单细胞微生物通过粘附于介质表面,分泌胞外多聚基质(胞外多糖、蛋白、DNA等),将其自身包绕其中而成的膜样微生物细胞聚集物[1]。生物被膜能够保护微生物在不利环境中得以存活并在条件有利时散播种子细胞去占领新的地盘。生物被膜可以在多种介质表面形成,在自然界、工业环境和医院等场所都普遍存在。这种固着的聚集状态的微生物,其生理指标与游离的菌细胞相比存在很大的差异[2-3]。在临床环境中,细菌生物被膜形成后会对抗生素的抗性大大提高并产生多种选择性的表型突变,因而可引起多种难以清除的慢性感染。对生物被膜在基因和分子水平的形成机制上的深入了解可为防治生物被膜引起的临床感染和问题提供新的解决策略。
生物被膜的胞外多聚物(EPS)是由多糖、胞外 DNA(eDNA)和蛋白质等组成的混合物(图1A)。EPS可以形成网络,并像分子胶水一样将细菌细胞网罗在一起(图1B)。这种基质网对维持生物被膜的整体结构和抗生素的抗性起到很大作用[4-5]。揭示EPS在生物被膜形成中的作用可为设计对抗生物被膜的靶向分子提供有益的思路。
条件致病菌铜绿假单胞菌具有极强的生存能力,极易形成生物被膜,是研究生物被膜的模式生物之一。该菌极易感染免疫力较弱的患者,如患代谢性疾病、血液病和恶性肿瘤的患者,以及术后或某些治疗后的患者(如癌症、大面积烧伤、HIV等),导致极高的发病率和致死率。铜绿假单胞菌可在囊性纤维化(Cystic fibrosis,CF)患者的肺部定殖并导致难以根除的慢性感染。已证实这种慢性感染难以根除的原因之一就是因为铜绿假单胞菌可在患者的肺部形成生物被膜。铜绿假单胞菌在不同的营养条件下可形成不同形态的生物被膜[8]。例如,在以葡萄糖为碳源的限制性培养基中,铜绿假单胞菌PAO1的生物被膜发育周期可分为5个典型阶段(图2)。
本文介绍了铜绿假单胞菌生物被膜形成调控机制及其基质网的各组分的近期研究进展,并进一步详细阐述了其铁摄取的主要系统与策略。深入了解铜绿假单胞菌生物被膜的形成机制和生存策略,可为解决铜绿假单胞菌引起的问题与感染提供途径与思路。

图1 铜绿假单胞菌胞外基质网的主要组分[6](A)和纤维丝态的Psl多糖基质网(上)及其包被的铜绿假单胞菌生物膜(下)(B)[7]Fig.1 Mainly components of extracellular matrix ofPseudomonas aeruginosa[6](A)and Psl fiber matrix(upper)and encasedP.aeruginosabiofilm(lower)(B)[7].
1 铜绿假单胞菌生物被膜基质网
铜绿假单胞菌在肺部的定殖是肺囊性纤维化病(CF)患者发病和死亡主要原因。铜绿假单胞菌造成的感染通常与形成抗生素抗性极强的生物被膜有关,菌细胞被包裹在基质网中形成微菌落聚集体。即使长期使用大量的抗生素进行治疗,感染仍然会持续存在,非常难以清除[9-10]。EPS基质网所起的重要作用与菌株的遗传背景、营养条件和生物被膜所处的不同发展阶段相关[11]。普遍接受的观点认为,EPS可以作为生物被膜的结构骨架并在不利的环境下为菌细胞提供保护屏障[12-13]。在铜绿假单胞菌中,至少存在3种胞外多糖在维持生物被膜的结构和抗生素抗性方面起着重要作用[5,7,12,14-16]。与生物被膜形成、发展及功能相关的基质网组成部分主要有胞外多糖(Psl多糖、Pel多糖和褐藻多糖)、胞外DNA、胞外蛋白以及菌体表面的一些蛋白附属结构,如纤毛、四型菌毛(T4P)及鞭毛。
1.1 Psl多糖
2004年3个不同的研究组分别鉴定出了Psl的多糖合成位点[14-15,17]。Psl多糖是由15个共转录的基因(pslA-pslO, PA2231–PA2245)组成的基因簇所编码的一系列蛋白共同合成的。Psl多糖对于菌体的初始吸附、生物被膜结构的维持以及提供细胞与细胞间、细胞与介质表面的相互作用至关重要[7,18-20]。相关研究进一步表明psl合成基因簇的15个基因中的前11个是合成Psl多糖所必需的[21],铜绿假单胞菌PA14由于pslA-pslD基因的缺失导致其丧失了合成Psl多糖的能力[22]。早先有研究表明 Psl多糖富含半乳糖、甘露糖及少量的葡萄糖和木糖[18]。最近有研究进一步证实了Psl多糖是由D-甘露糖、D-葡萄糖和L-鼠李糖形成的四糖重复单位组成的[21]。此外,在基因及生化水平对单个的psl基因,如pslA、pslB和pslD的功能研究揭示出这些基因对 Psl多糖的合成及生物被膜的形成都是非常关键的[23-25]。

图2 铜绿假单胞菌生物被膜的典型发育周期[7].1:可逆性吸附期:浮游微生物附着至载体表面的初期,附着于载体的微生物也可以回到单细胞浮游状态(Ⅰ);2:不可逆吸附期(Ⅱ);3:生物被膜形成初期(Ⅲ);4:生物被膜成熟期(Ⅳ);5:“种子散播期”(Ⅴ).(Ⅰ–Ⅴ)在生物被膜发育的每个阶段,Psl多糖是如何包被生物被膜中的细菌及其在相应时期的变化(Psl多糖由红色荧光标记,菌体由绿色荧光标记)Fig.2 Scheme of biofilm development inP.aeruginosa[7].Selected images showed how the matrix of Psl polysaccharide(red fluorescence)enmeshes bacterial cells(green fluorescence)within bacterial communities during biofilm development.1: initial attachment(Ⅰ);2: irreversible attachment(Ⅱ);3: microcolony formation(Ⅲ);4:biofilm maturation(Ⅳ);5: biofilm dispersion(Ⅴ).
由于 Psl多糖在铜绿假单胞菌生物被膜的形成中起着非常重要的作用,因而对其在生物被膜形成中的具体功能和角色已经有了许多非常广泛深入的研究[20]。首先,Psl多糖的过量生产可以增强菌细胞之间、菌细胞与介质表面间的黏附作用,提示 Psl多糖在吸附中的重要功能对起始和维持生物被膜的结构非常关键[19,21]。后来 Ma及其同事用荧光标记的凝集素进一步直接观察到了 Psl多糖在生物被膜不同发育阶段的形态和结构(图2),而且发现了Psl多糖是以一种双螺旋的形态锚定在菌细胞表面并通过这种锚定方式有效地促进了菌细胞之间的相互作用,并以此完成早期生物被膜及其基质网的形成和组装。在生物被膜形成后期,Psl多糖主要聚集在立体的微菌落结构的外围。这种定位方式为生物被膜提供了结构支撑并便于之后的种子散播[7]。除此之外,对Psl多糖的染色观察表明 Psl可以形成一种纤维状的网络将菌细胞网罗在生物被膜中。近来,有研究发现Psl多糖基质网是通过依赖四型菌毛的迁移策略形成的,T4P介导的细菌迁移导致了Psl纤维基质网的形成,缺失了菌毛的突变株丧失了形成Psl纤维结构的能力并且导致生物被膜的生物量下降[26]。Zhao及其同事也同时证明铜绿假单胞菌通过在介质表面的迁移留下Psl多糖踪迹,进一步影响后续菌细胞的表面运动并最终导致生物被膜形成的起始[27]。以上这些最新发现都扩展了对Psl多糖的生物学理解。
令人惊讶的是,Psl多糖还具有信号功能,可通过刺激两个二鸟苷酸环化酶 SiaD和 SadC来升高胞间第二信使分子c-di-GMP的水平,形成一个独特的正反馈调节回路最终提高 Psl多糖的产量[28]。
此外,Psl多糖在赋予菌体抗原性及保护菌细胞免受宿主免疫系统攻击等方面也有作用。Psl多糖可通过增强菌细胞与上皮细胞的接触直接刺激NF-κB的活性并促进鞭毛介导的促炎信号[29]。Mishra等发现铜绿假单胞菌的Psl多糖可通过限制补体介导的调理作用减弱中性白细胞的吞噬以及氧化应激反应的发生[30]。此类研究清晰地表明 Psl多糖对宿主内环境中的菌体生存具有重要意义,并提供了将Psl多糖作为靶点清除生物被膜从而治疗相关慢性感染的直接依据。
有研究证实 Psl多糖在细菌抗生素耐药性的产生中也发挥作用。最近有研究揭示Psl多糖可以促进细菌对生物被膜抑制剂聚山梨醇酯80(Polysorbate80)的抗性[31]。Yang等报道生物被膜中依赖 Psl多糖形成的微菌落结构使铜绿假单胞菌对抗生素的处理变得不敏感[20]。通过用荧光标记的抗生素观察到胞外基质网可通过限制妥布霉素的渗透保护生物被膜中的菌细胞免遭抗生素的杀伤,且这种对抗生素的隔离作用主要发生在生物被膜的外周区域[32]。然而,没有明确证据能直接证实生物被膜基质网对这种保护和相互作用的关键功能。此外,另一个研究组报道在生物被膜的形成起始阶段 Psl多糖作为第一道防线来抵御阳离子或阴离子类抗生素的攻击,且这种由Psl多糖介导的保护作用可以扩展到混合培养生物被膜中的大肠杆菌和金黄色葡萄球菌[33]。
总之,Psl多糖不仅可作为生物被膜发育过程中的结构骨架,还可以作为一种信号分子存在最终导致生物被膜的形成。这种正反馈回路代表了一种铜绿假单胞菌在不同环境中生存定殖的进化策略。此外,Psl多糖还可作为屏障来抵御宿主免疫和抗生素的杀伤。
1.2 Pel多糖
Pel多糖是由pelA-pelF(PA3058-PA3064)组成的pel合成基因簇编码的蛋白合成。Pel多糖是一种富含葡萄糖且对纤维素处理敏感的胞外多聚物[34]。pel合成基因簇在革兰氏阴性菌中比较保守[35]。这部分基因的缺失会导致细菌在静置培养条件下无法形成气液交界面的菌膜,此外,Pel多糖在介质表面形成牢固的生物被膜过程中也发挥作用[14]。有趣的是,缺失菌毛的铜绿假单胞菌PAK的pel突变株,其在固体介质表面的初始吸附能力大大减弱了,提示 Pel多糖可能在其他一些黏附因子如四型菌毛缺失时可以提供代偿作用[7]。然而,Pel多糖在其他铜绿假单胞菌菌株的吸附过程中的明确作用仍需进一步深入的研究。此外,Pel多糖通过维持细胞间的相互粘连在菌株 PA14形成的生物被膜中为菌细胞提供主要的骨架支撑,并通过增强生物被膜对氨基糖甙类抗生素的抗性为细菌提供保护[16]。进一步对Pel功能的研究揭示Pel多糖可以和其他类型的EPS共同作用来促进铜绿假单胞菌的生物被膜发育[20]。
1.3 褐藻多糖
褐藻多糖(Alginate)是 CF患者肺部临床分离株产生的主要胞外多糖[36]。典型黏液型表型的产生原因是由于菌株生产了大量的褐藻多糖,这种多糖形成的生物被膜胞外基质网可以保护铜绿假单胞菌免受肺部不利环境的影响。然而,这种多糖对于体外非黏性生物被膜的形成并不是必需的[37]。褐藻多糖在保护和维持生物被膜结构稳定等方面起着重要作用——有保水及储存营养物质的功能[4]。有趣的是,近来的研究发现,黏性菌株生物被膜的形成也依赖Psl多糖的产生[38-39]。褐藻多糖还有维持细胞存活和免疫逃避的功能[40]。褐藻多糖的大量生产可以增强菌细胞对抗生素和调离吞噬作用的抵抗能力[41-42]。这种多糖在体内可以清除由中性粒细胞和巨噬细胞产生的自由基[43]。Bragonizi等也曾报道褐藻多糖的大量合成并没有增强铜绿假单胞菌在小鼠感染模型中长久生存能力,这也许是由于感染过程中菌株从黏液型到非黏液型的表型转变有关[44]。
1.4 胞外DNA
胞外DNA(eDNA)是铜绿假单胞菌生物被膜基质网中的另一重要组分[17,45-46]。eDNA可能源于基因组DNA,在生物被膜中提供细胞间的相互联结。对生物被膜中的eDNA进行染色发现其主要大量存在于蘑菇状微菌落的柄部[45]。此外,在生物被膜微菌落中也存在着菌细胞的自溶现象,但目前尚不明确 eDNA是否来源于菌细胞自溶释放出的基因组DNA[47]。关于eDNA的来源目前主要的几个假设有:来源于由前噬菌体裂解的亚群体、由菌细胞直接分泌及由小的外膜囊泡产生等[48]。
与生物被膜基质网中的其他组分相同,eDNA对生物被膜的形成也具有多重作用,有研究证实外源添加 DNaseⅠ可以降解较为“年轻”的生物被膜,此外 eDNA也与抗生素抗性的产生有关[49]。有趣的是,eDNA还可在饥饿状态时作为菌细胞的营养来源[50],近来有研究表明,铜绿假单胞菌产生一种胞外脱氧核糖核酸酶(PA3909)可利用eDNA作为营养来吸收利用,这进一步扩展了我们对于 eDNA在铜绿假单胞菌生物被膜形成过程中作用的理解[51]。此外,eDNA还可以与坏死的中性粒细胞释放出的F-肌动蛋白形成束状物,这些束状物可以促进铜绿假单胞菌生物被膜的形成起始[52]。eDNA作为铜绿假单胞菌生物被膜中的主要促炎因子,通过CpG和TLR9-依赖的机制激活中性粒细胞[53]。进一步的研究发现 eDNA可以通过维持细胞间的粘连和排列来调节生物被膜边缘部位先锋细胞群的运动,进而促进由颤动作用介导的生物被膜扩张[54]。
1.5 胞外蛋白及菌体表面蛋白附属结构
除胞外多糖和胞外DNA外,胞外蛋白与四型菌毛、鞭毛和纤毛等蛋白多聚体细胞附属物也是组成生物被膜基质网的成分。这些细胞附属物主要作为黏附因子和结构支撑为铜绿假单胞菌生物被膜的形成提供辅助作用[55]。
鞭毛可以介导铜绿假单胞菌的游动(Swimming)和丛集运动(Swarming),还可以作为黏附因子为细胞和介质表面的初始接触提供关键助力[56]。四型菌毛(T4P)是一种线状的运动器官,采用一种“伸展-握紧-撤回”的机制形成铜绿假单胞菌的颤动(Twitching)能力[57]。四型菌毛在铜绿假单胞菌微菌落的典型“蘑菇冠”结构的形成中起着重要作用[56]。如上所述,T4P介导的菌体运动可以帮助Psl多糖纤维丝状网的形成[26],而另有研究认为铜绿假单胞菌的鞭毛和菌毛并不是初始吸附或形成生物被膜所必需的[58],即鞭毛或菌毛介导的生物被膜形成可能是依条件和营养而异的。近来,有研究证实一个新鉴定的粘附素CdrA是铜绿假单胞菌EPS基质网中的关键组分蛋白,通过直接与Psl多糖作用介导菌体的自聚集并增强生物被膜的稳定性[59]。
铜绿假单胞菌的纤毛类附属物由“分子伴侣-引导蛋白(Chaperon-Usher)”机制组装而成,其对生物被膜的形成亦具有促进作用[60]。在铜绿假单胞菌PAO1中,鉴定出至少4个纤毛系统(CupA、B、C和 D)并分别具有不同的组织形式和功能[61]。有研究表明Cup纤毛有利于生物被膜的形成起始,特别是在介导细胞间互作和微菌落的形成过程中起着关键作用[18,62]。
2 铜绿假单胞菌生物被膜形成的调控
生物被膜的形成受控于多种复杂的细菌调控体系之下,包括群体感应系统(Qurum sensing,QS)及参与调节胞外多糖合成的双组分调控系统等[63]。这些调控系统的缺失会导致生物被膜的结构、形态及保护能力的变化。现对铜绿假单胞菌与生物被膜形成相关的关键调控系统进行简要概述。
2.1 群体感应系统与生物被膜形成
群体感应是细菌群体的一种细胞间的交流方式,通过生产及接受能扩散的信号分子感应菌细胞的群体密度,并通过这种机制调控毒力因子的产生、运动力的变化及生物被膜的形成[64-65]。铜绿假单胞菌具有两种主要的QS系统(las和rhl),分别通过合成酶LasI、RhlI合成两种自诱导信号分子:N-3-氧十二烷酰高丝氨酸内酯(3-oxo-C12-HSL)和 N-丁酰高丝氨酸内酯(C4-HSL)[65]。信号分子与相应的受体蛋白(转录因子 LasR和 RhlR)结合后激活一系列基因(包括lasI和rhlI在内)的转录。此外,铜绿假单胞菌还有第3个QS系统——PQS系统,依赖喹诺酮信号分子,与酰基高丝氨酸内酯(AHLs)信号分子之间存在着复杂的交互调控关系[65](图3)。
Davies等已证明了las系统在铜绿假单胞菌生物被膜形成和成熟过程中的作用[2]。与野生型相比,las系统缺失株形成更扁平、均一化的生物被膜,且受到十二烷基硫酸钠的处理后更易从介质表面剥离,但las系统在生物被膜形成中的具体功能尚不明确。而后 Gilbert等报道QS转录因子LasR可以结合在psl操纵子的启动子区——表明QS系统可以调控psl基因簇的表达[67]。rhl系统亦被证实可以通过促进Pel多糖的合成参与铜绿假单胞菌生物被膜形成:在rhl系统缺失株中pel操纵子的转录水平明显降低[68]。而PQS系统与生物被膜发育过程中eDNA的释放有关,pqsA突变株形成的生物被膜中的eDNA含量比野生型生物被膜中的更少[45,69]。综上所述,以上3种QS系统均在铜绿假单胞菌生物被膜的发育过程中起着较为重要的调控作用。

图3 铜绿假单胞菌的层级群体感应系统[66]Fig.3 The hierarchy quorum sensing network inP.aeruginosa[66].
不能忽略的是,QS系统还可通过调控铜绿假单胞菌的丛集运动、颤动及鼠李糖脂、凝集素的产量来间接影响生物被膜的形成过程。铜绿假单胞菌的丛集运动能力是由鞭毛、菌毛和胞间接触共同介导的一种复杂的表面运动能力[70-71]。rhl系统可以通过调节丛集运动参与早期铜绿假单胞菌生物被膜的形成[72]。铜绿假单胞菌在促进丛集运动的培养基(如以谷氨酸或琥珀酸为碳源的生长培养基)中形成扁平均一的生物被膜,而在丧失丛集运动的情况下形成融合样细胞聚集状态的生物被膜[8]。颤动能力是一种由极生四型菌毛介导的一种细菌运动[73],已知其在铁缺乏的限制性培养基中受到rhl系统的调控[74]。颤动能力是铜绿假单胞菌在生物被膜形成过程中将单层细胞组装成微菌落的立体结构所必需的[56]。
鼠李糖脂,除了作为一种表面活性剂和铜绿假单胞菌的毒力因子[75],可受到rhl系统的调控并在生物被膜的形成中起多种作用[76]:1)参与生物被膜微菌落的形成[77];2)通过干扰细胞-细胞、细胞-介质表面间的相互作用阻碍菌细胞定殖并维持生物被膜中通道结构的开放[78];3)通过促进3D蘑菇状结构的形成帮助铜绿假单胞菌生物被膜发育[77];4)高产鼠李糖脂的突变株更易从生物被膜中脱离,因此鼠李糖脂的过量表达可以促进铜绿假单胞菌生物被膜的瓦解[79]。最后,铜绿假单胞菌的细胞毒力因子——凝集素LecA和 LecB,也参与生物被膜的形成,LecA和LecB的突变株只能形成比野生型更薄的生物被膜[80-81],而LecA和LecB的表达也是受到rhl系统调控的[82]。
2.2 GacS/GacA和RetS/LadS双组分系统对生物被膜的调控
在铜绿假单胞菌基因组中的60个双组分调控系统中[83],GacS/GacA系统参与调控 QS系统并调节多种毒力因子的产生和生物被膜的形成[84]。Gac系统由一个跨膜感应激酶GacS和一个配对反应调节蛋白GacA组成,GacS可通过自磷酸化转移一个磷酸基给 GacA,磷酸化的GacA可上调两个小RNA——RsmZ和RsmY的表达,RsmZ和RsmY可结合在一种小RNA结合蛋白RsmA上,而RsmA在转录后水平抑制psl合成簇(pslA-L)的转录[85-86](图4)。GacS/GacA系统还对AHL系统有调控作用:可通过使游离的RsmA失活解除其对 C4-HSL和3-oxo-C12-HSL合成的抑制作用,从而通过las和rhl系统调控胞外毒力因子的产生[87-88]。

图4 铜绿假单胞菌GAC双组分系统[89]Fig.4 The GAC system network inP.aeruginosa[89].
有报道称混合传感组氨酸激酶RetS可抑制生物被膜的形成[90-91],而另一个组氨酸激酶LadS对 RetS有拮抗作用[92]。与野生型菌株PAO1相比,retS突变株可形成结构化程度更高的生物被膜[90];而天然缺乏ladS基因的菌株PA14与ladS过表达株相比,所形成的生物被膜更薄[93]。有研究表明,RetS和LadS与GacS/GacA系统之间存在着交互作用:即可通过调节 GacS的磷酸化状态来抑制或促进GacA的磷酸化[93-94]。有趣的是,GacS/GacA和RetS/LadS系统还参与调控铜绿假单胞菌从急性感染到慢性感染的表型转变过程[90]。
2.3 环二鸟苷酸(C-di-GMP)依赖的多糖合成与生物被膜的形成
铜绿假单胞菌胞外多糖的合成依赖于胞内环二鸟苷酸(c-di-GMP)的浓度[95-96],c-di-GMP是细菌中广泛存在的一种胞内第二信使[97]。在菌细胞内,两分子的鸟苷三磷酸(GTP)在二鸟苷酸环化酶的催化下合成一分子的c-di-GMP,后者又可在底物专一性的磷酸二酯酶的催化下降解为两分子的鸟苷酸(GMP)[97](图5)。
高浓度的c-di-GMP可通过与合成褐藻多糖和Pel多糖所必需的Alg44和PelD蛋白结合分别促进这两种多糖的合成[96-98]。然而,这种c-di-GMP调控糖前体多聚化的具体机制尚不明确。反过来,低浓度的c-di-GMP可通过加强鞭毛运动促进菌细胞的游动和解聚[96]。
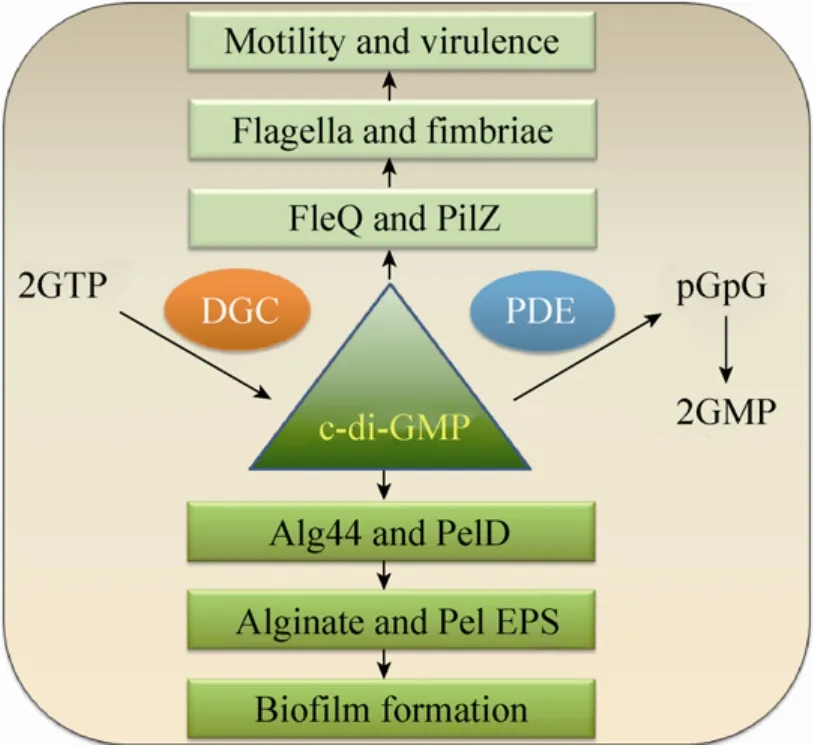
图5 铜绿假单胞菌c-di-GMP的信号作用[97]Fig.5 Physiological functions ofc-di-GMP[97].
3 铜绿假单胞菌的铁摄取系统
铁离子是地球上绝大多数生物生存必不可少的元素之一,生物体内的很多酶催化反应都需要铁离子的参与。然而,由于氧的存在,环境中的大部分铁离子都以三价铁的形式存在,很难溶解于水溶液中。因此,环境中的铁看起来很多,实际上大部分都是铁的化合物,自由铁的浓度很低,满足不了微生物的生长需求。此外,由于宿主铁大多被亚铁血红素分子及转铁蛋白、乳铁蛋白之类的循环蛋白所结合,因而病原微生物在宿主体内也面临着严峻的铁摄取问题[99-100],细菌为了利用铁来维持自身生长并完成各种生理功能,进化出了一系列能够螯合、摄取铁的系统,而这些系统对细菌毒力因子的释放和生物被膜的形成均起着重要的调控作用。
铜绿假单胞菌可以通过如下不同策略来获取铁元素:1)生产 Fe3+螯合分子,即铁载体:荧光嗜铁素(Pyoverdine)和鳌铁蛋白(Pyochelin),并通过 TonB依赖的受体(TonB-dependent receptors,TBDR)摄入三价铁-载体复合物。2)摄入外源铁载体复合物(非铜绿假单胞菌本身产生的铁载体)。3)从宿主产生的血红素蛋白摄入血红素分子。4)利用吩嗪(Phenazine)和Fe2+专一性摄取系统(Feo系统)在胞外将 Fe3+还原为 Fe2+并加以吸收利用。5)在宿主体内,依据造成感染的不同类型(急性感染或慢性感染),铜绿假单胞菌能够采取不同的铁摄取策略在不造成过多能量消耗的前提下满足自身的铁需求。
3.1 铜绿假单胞菌所产铁载体及其介导的铁摄取系统
铁载体是一类能够以高亲和力专一性螯合Fe3+的低分子量的分泌型分子,能够依赖由TonB周质膜蛋白所提供的能量被特异性受体识别[101-105]。铁载体依据其与铁所形成的复合物的形式,可分为不同的种类,如酚盐铁载体、苯酚铁载体、异羟肟酸铁载体、羧化铁载体及混合铁载体。
3.2 荧光嗜铁素:引发急性感染所必需的高亲和力铁载体
铜绿假单胞菌荧光嗜铁素是由一个肽链和一个发色团组成的混合型铁载体[106-108]。荧光嗜铁素是能发荧光的一些假单胞菌(如荧光假单胞菌、恶臭假单胞菌、丁香假单胞菌和铜绿假单胞菌)的标志性特点,其在环境铁浓度过低时被产生出来[106-108]。荧光嗜铁素与铁的亲和力极高,可将铁从转铁蛋白中螯合出来,且荧光嗜铁素的产生是引起小鼠烧伤模型和肺病模型的感染所必需的[106,109-113]。同样地,TonB蛋白的突变株丧失了在小鼠模型中的致病力[111]。在Imperi等的研究中,筛选到了一些通过FDA认证的抑制铜绿假单胞菌荧光嗜铁素合成的化合物,其中一个化合物——抗真菌药物氟胞嘧啶对多株铜绿假单胞菌产生的不同类型的荧光嗜铁素均有明显的抑制作用,且这种抑制作用是通过作用于σ因子PvdS来实现的,而PvdS是荧光嗜铁素合成基因簇转录所必需的调节因子[113]。荧光嗜铁素不仅是一种铁载体,还是一种信号分子,可诱导两种胞外毒力因子蛋白酶 PrpL和外毒素 A的产生[108,114-117](图6)。此外,尽管荧光嗜铁素是铜绿假单胞菌引起急性感染所必需的因素,其也在较厚的成熟生物被膜形成过程中发挥作用[74,118-119]。
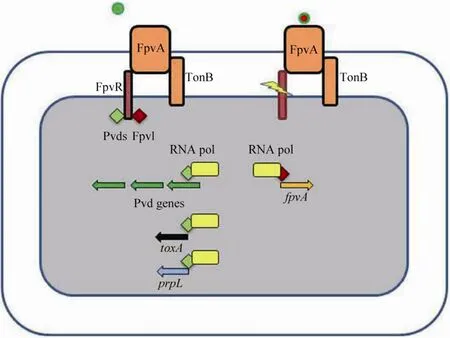
图6 荧光嗜铁素是一种信号分子[120]Fig.6 Ferripyoverdine is also a signal molecule[120].
3.3 鳌铁蛋白:一种低亲和力铁载体
鳌铁蛋白是所有铜绿单胞菌均产生的一种铁载体,但其对铁的亲和力较荧光嗜铁素低[121-123]。鳌铁蛋白合成所需的基因数比荧光嗜铁素所需基因数更少[124],且有研究表明铜绿假单胞菌优先合成鳌铁蛋白,在环境铁浓度非常低的情况下再切换为荧光嗜铁素的合成[125]。鳌铁蛋白-铁复合物可以进行氧化还原循环并引起宿主的氧化损伤和炎症反应,尤其是在铜绿假单胞菌的另一种胞外产物绿脓菌素也存在的情况下[126-128]。在慢性感染过程中,如在CF患者肺部,鳌铁蛋白的产生可引起持续的炎症反应并导致组织损伤[129]。在 CF痰培养基中,铜绿假单胞菌的鳌铁蛋白合成量增加[130]。
3.4 亚铁血红素摄取系统
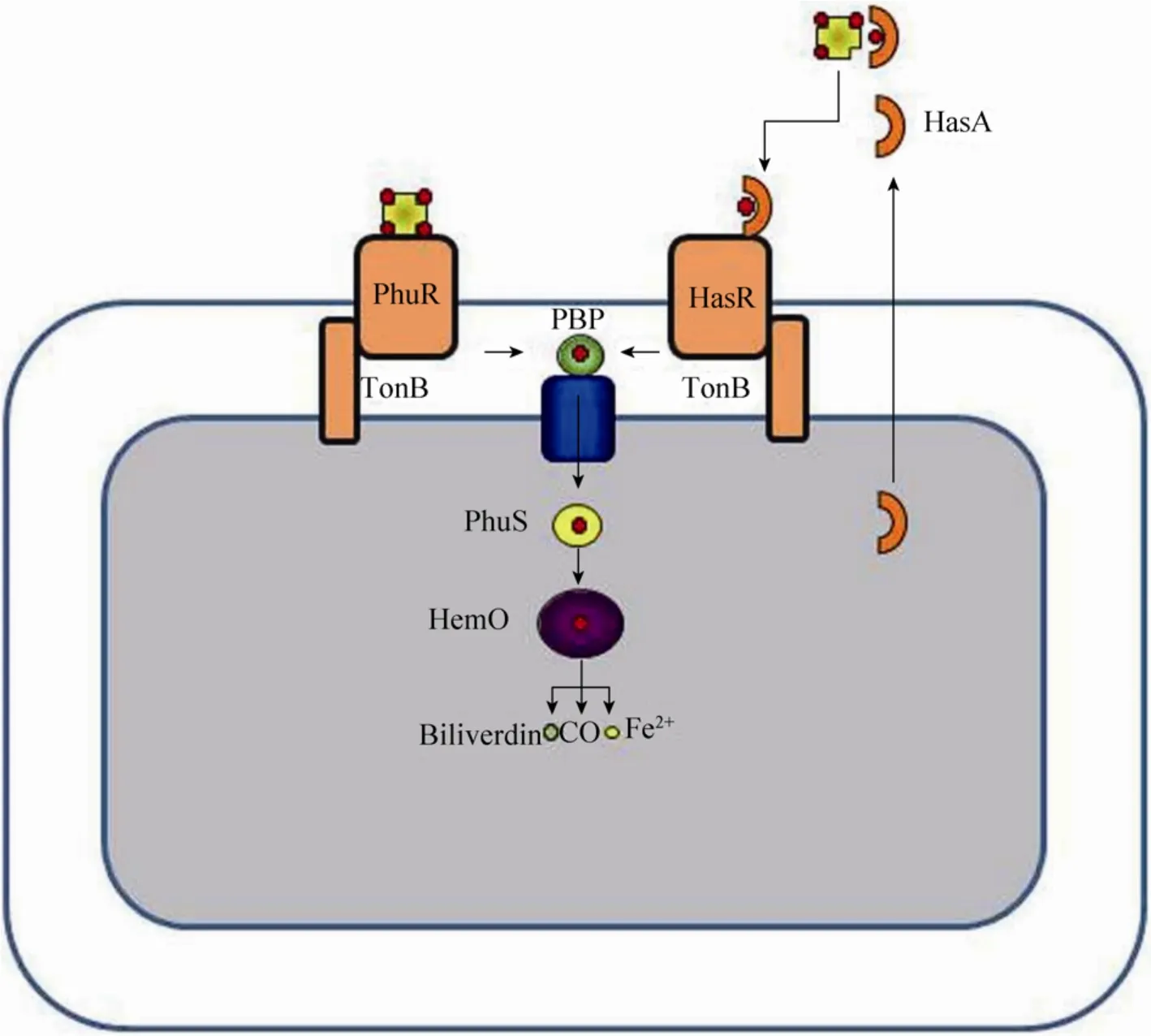
图7 铜绿假单胞菌的两个血红素摄取系统:Phu和Has[120]Fig.7 P.aeruginosahas two heme uptake systems, Phu and Has[120].
铜绿假单胞菌可以通过Has和Phu两套系统从血红蛋白中摄取亚铁血红素[131](图7)。亚铁血红素无法以自由游离形式存在,因为强疏水性使其与膜相结合并在膜上进行非酶促氧化还原反应[132],因此亚铁血红素必须从血红蛋白或血液结合素上提取得到。在铜绿假单胞菌的Phu系统中,亚铁血红素是从一种外膜TBDR中提取到的,而在 Has系统中,亚铁血红素先由血红蛋白摄取,然后这种“血红素载体-血红素”复合物再被另一种TBDR——HasR识别[133-135]。在细胞周质中,血红素与周质结合蛋白结合后通过ABC转运系统运输到胞内。而在ABC转运系统中,血红素先与血红素分子伴侣PhuS结合,然后被运输给血红素氧化酶 HemO,进一步将血红素降解为胆绿素、CO和Fe2+[136-138]。对Has或Phu系统的一个单突变不会影响血红素摄取,而对两个系统的双突变则会导致铜绿假单胞菌完全丧失将血红素作为铁源的摄取能力[131]。
3.5 外源铁载体的摄取
铜绿假单胞菌至少具有30个以上的基因用来编码 TBDRs,而其中大多数都参与铁与载体复合物的摄入[139-141]。不同的TBDRs可以分为两类:简单的TBDR和TonB依赖的转换子,即TBDT[142]。以TBDT系统中的“铁-荧光嗜铁素”复合物受体FpvA为例,FpvA可以通过与一个作为抗 σ因子的膜蛋白相互作用来感受“铁-荧光嗜铁素”复合物的存在[142]。这种受体识别一旦发生就会使抗 σ因子蛋白水解,从而释放出外ECFσ(Extracytoplasmic function sigma factor),ECFσ可与RNA聚合酶作用,从而引发下游基因转录自诱导反应[117,140,142-143]。大多数的铜绿假单胞菌(98%)都具有另一种“一型铁-荧光嗜铁素受体”——FpvB,这意味着几乎所有菌株都能利用这种荧光嗜铁素作为铁源[139,144]。铜绿假单胞菌还可以通过各种不同的受体来摄取各种各样由其他细菌产生的“铁-载体复合物”,这种能够“偷取”其他细菌铁源的能力,可能赋予其在混合感染中较强的竞争优势[145]。
3.6 通过Feo系统与吩嗪摄入Fe2+

图8 铜绿假单胞菌两种主要的吩嗪——PCA和绿脓菌素的结构(A)和还原态PCA分泌出胞外后将Fe3+还原为Fe2+[120](B)Fig.8 Structure of the two majorP.aeruginosaphenazines, PCA and pyocyanin(A).Reduced PCA is excreted out of the cell and is oxidizedresulting in the reduction of Fe3+to Fe2+[120](B).
与Fe3+不同,Fe2+具有水溶性,并存在于低pH值的厌氧或微厌氧环境中[146]。Fe2+可以扩散至外膜并通过在革兰氏阴性菌中广泛存在的FeoABC系统转运至细胞质[147](图8)。Fe2+通过由透性酶FeoB和FeoA、FeoC蛋白组成的转运系统继续被运输至细胞内部[147]。Fe2+的摄入可能与铜绿假单胞菌对微厌氧或厌氧环境的感受有关,如CF患者的肺部的生物被膜黏液环境[148-149]。
吩嗪类物质是一种由铜绿假单胞菌产生的次级代谢产物[150],1-羧基-吩嗪酸(PCA)是绿脓菌素的前体,绿脓菌素是铜绿假单胞菌所产生的一种蓝绿色特征性化合物,这两种吩嗪物质都具有氧化还原能力[150]。PCA及一些少量的绿脓菌素可将宿主蛋白结合的Fe3+还原为Fe2+,在生物被膜中Fe2+可经由Feo系统摄入[151]。近来有研究报道当病情恶化时,可在CF患者肺部检测到吩嗪和 Fe2+的大量积累[152-153],而 Fe2+和 Fe3+两种离子的螯合剂联用可以大大削弱铜绿假单胞菌的生物被膜形成能力[153]。

图9 铜绿假单胞菌的TonB依赖的铁摄取调控[158]Fig.9 Regulation of iron uptake via TonB-dependent receptors inP.aeruginosa[158].
3.7 铜绿假单胞菌铁平衡的调节机制
与大多数细菌一样,Fur是假单胞菌的一个主要的铁调节蛋白,且在铜绿假单胞菌中,fur是生存必需基因[154]。大部分基因参与“铁-载体复合物”摄取的基因受到Fur的间接调控(图9)。Fur调控一些ECFσ因子的表达,这些σ因子包括荧光嗜铁素生物合成基因pvdS、荧光嗜铁素复合物基因fpvA和其他一些编码TonB依赖型外源铁复合物受体的基因,它们是实现一些下游基因转录所必需的。铁浓度可作为信号通过ECFσ因子引发这些基因的表达。受体与“铁-载体复合物”的结合改变了受体蛋白 N端与膜抗 σ因子的相互作用,释放出 ECFσ因子,使后者能够与核RNA聚合酶结合。铜绿假单胞菌还具有另一种铁调节机制,即通过AraC调节子来控制鳌铁蛋白的合成与铁摄取[155]。最后,两个小RNA——PrrF1和PrrF2可以作为mRNA反义转录后调节子参与Fur的调节,主要通过参与上调氧化还原应激、铁储存和中间代谢等Fe-Fur基因的表达[156-157]。
综上所述,铜绿假单胞菌可以从合成较低铁亲和力的鳌铁蛋白转为合成较高铁亲和力但更耗能量的荧光嗜铁素来摄取足够量的Fe3+[125]。一个典型的例子即发生在铜绿假单胞菌对 CF患者肺部环境的适应过程中[129]。当铜绿假单胞菌开始侵染肺部时,可能产生荧光嗜铁素,但随其在肺部的进一步定殖,细菌开始由于某些原因合成鳌铁蛋白并引发强烈的炎症反应,进而导致组织损伤和包括血红蛋白和其他含铁蛋白等细胞内容物的释放[128]。尽管包括荧光嗜铁素在内的铁载体在 CF患者的痰液样本中被检测到[159],但不能产生荧光嗜铁素的突变株具有更久的定殖能力[160-161],这意味着铜绿假单胞菌可以利用其他代偿性机制来满足自身的铁需求,例如可以从炎症反应中释放出的血红蛋白中摄取血红素,或者摄取经由吩嗪类物质,特别是PCA介导的氧化还原反应产生的Fe2+[151,153,161-162]。
4 铁与生物被膜形成
有研究发现外源添加铁螯合剂乳铁蛋白可对铜绿假单胞菌生物被膜的形成产生明显的抑制作用[163],而适宜的高铁浓度环境则促进其生物被膜的形成[164-165]。这表明环境铁可作为一种信号调节铜绿假单胞菌生物被膜的发育。与上述研究结论相一致,研究者还发现丧失了螯合环境铁能力的铜绿假单胞菌突变株不能形成正常的生物被膜[74,118]。这种铁对铜绿假单胞菌生物被膜的调节效应可能是受到铁受体调节子 Fur的调控而产生[141,166]。近年来有研究报道高铁浓度可以抑制铜绿假单胞菌黏性菌株的主要胞外多糖——褐藻多糖的产生,使其生物被膜的形成能力降低[167]。最新研究发现,铜绿假单胞菌的胞外多糖Psl可直接结合环境中的自由铁,而高铁浓度信号通过抑制转录因子 AmrZ降低鼠李糖脂的合成量,从而通过解除其对Psl多糖的糖前体竞争抑制而产生更多的Psl多糖,形成更大生物量的生物被膜(图10),这是一种铜绿假单胞菌摄取铁的新策略[168]。
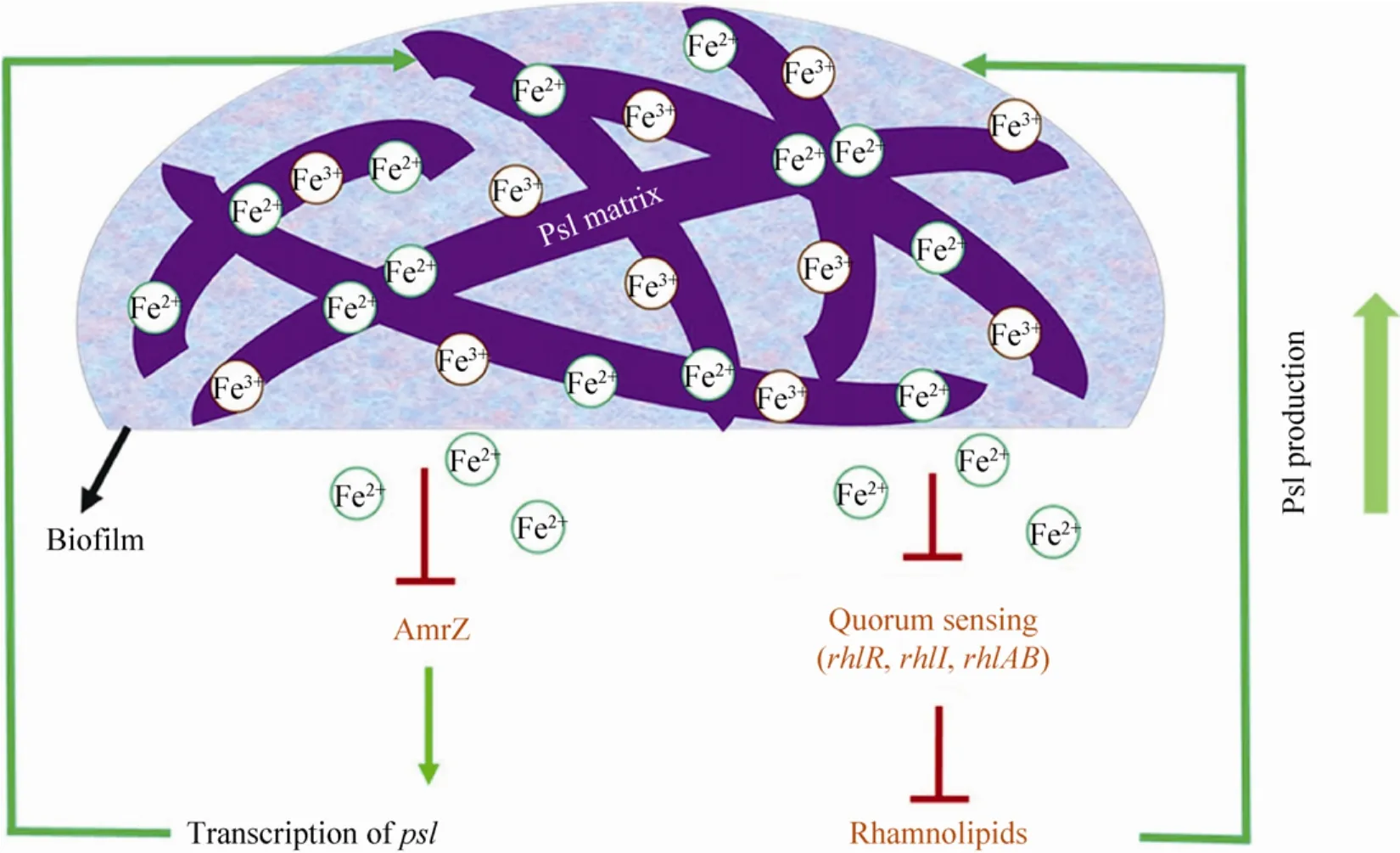
图10 铜绿假单胞菌Psl多糖与铁的相互作用模式图[168]Fig.10 A schematic shows the mechanisms of the iron-Psl interplay inP.aeruginosa[168].
5 结语
本文概述了铜绿假单胞菌生物被膜发展发育的主要过程及其系统调控网络,并特别介绍了铁摄取系统的工作原理及铁信号与其生物被膜形成之间的关系。进一步增强了人们对于微生物生物被膜发育过程以及环境适应性的认识,同时为生物被膜的防控提供了潜在的候选药物靶标。
[1]Karatan E, Watnick P.Signals, regulatory networks and materials that build and break bacterial biofilms.Microbiol Mol Biol Rev,2009,73(2):310–347.
[2]Davies DG, Parsek MR, Pearson JP, et al.The involvement of cell-to-cell signals in the development of a bacterial biofilm.Science,1998,280(5361):295–298.
[3]Hall-Stoodley L, Costerton JW, Stoodley P.Bacterial biofilms: from the natural environment to infectious diseases.Nat Rev Microbiol,2004,2(2):95–108.
[4]Sutherland IW.Biofilm exopolysaccharides: a strong and sticky framework.Microbiology,2001,147(1):3–9.
[5]Branda SS, Vik Å, Friedman L, et al.Biofilms: the matrix revisited.Trends Microbiol,2005,13(1):20–26.
[6]Flemming HC, Wingender J.The biofilm matrix.Nat Rev Microbiol,2010,8(9):623–633.
[7]Ma LY, Conover M, Lu HP, et al.Assembly and development of thePseudomonas aeruginosabiofilm matrix.PLoS Pathog,2009,5(3):e1000354.
[8]Shrout JD, Chopp DL, Just CL, et al.The impact of quorum sensing and swarming motility onPseudomonas aeruginosabiofilm formation is nutritionally conditional.Mol Microbiol,2006,62(5):1264–1277.
[9]Costerton JW, Stewart PS, Greenberg EP.Bacterial biofilms: a common cause of persistent infections.Science,1999,284(5418):1318–1322.
[10]Drenkard E, Ausubel FM.Pseudomonasbiofilm formation and antibiotic resistance are linked to phenotypic variation.Nature,2002,416(6882):740–743.
[11]Flemming HC, Neu TR, Wozniak DJ.The EPS matrix: the "house of biofilm cells".J Bacteriol,2007,189(22):7945–7947.
[12]Ryder C, Byrd M, Wozniak DJ.Role of polysaccharides inPseudomonas aeruginosabiofilm development.Curr Opin Microbiol,2007,10(6):644–648.
[13]Parsek MR, Singh PK.Bacterial biofilms: an emerging link to disease pathogenesis.Annu Rev Microbiol,2003,57:677–701.
[14]Friedman L, Kolter R.Two genetic loci produce distinct carbohydrate-rich structural components of thePseudomonas aeruginosabiofilm matrix.J Bacteriol,2004,186(14):4457–4465.
[15]Jackson KD, Starkey M, Kremer S, et al.Identification ofpsl, a locus encoding a potential exopolysaccharide that is essential forPseudomonas aeruginosaPAO1 biofilm formation.J Bacteriol,2004,186(14):4466–4475.
[16]Colvin KM, Gordon VD, Murakami K, et al.The pel polysaccharide can serve a structural and protective role in the biofilm matrix ofPseudomonas aeruginosa.PLoS Pathog,2011,7(1): e1001264.
[17]Matsukawa M, Greenberg EP. Putative exopolysaccharide synthesis genes influencePseudomonas aeruginosabiofilm development.J Bacteriol,2004,186(14):4449–4456.
[18]Ruer S, Stender S, Filloux A, et al.Assembly of fimbrial structures inPseudomonas aeruginosa:functionality and specificity of chaperone-usher machineries.J Bacteriol,2007,189(9):3547–3555.
[19]Ma LY, Jackson KD, Landry RM, et al.Analysis ofPseudomonas aeruginosaconditional Psl variantsreveals roles for the Psl polysaccharide in adhesion and maintaining biofilm structure postattachment.J Bacteriol,2006,188(23):8213–8221.
[20]Yang L, Hu YF, Liu Y, et al.Distinct roles of extracellular polymeric substances inPseudomonas aeruginosabiofilm development. Environ Microbiol,2011,13(7):1705–1717.
[21]Byrd MS, Sadovskaya I, Vinogradov E, et al.Genetic and biochemical analyses of thePseudomonas aeruginosaPsl exopolysaccharide reveal overlapping roles for polysaccharide synthesis enzymes in Psl and LPS production.Mol Microbiol,2009,73(4):622–638.
[22]Winsor GL, Lam DKW, Fleming L, et al.Pseudomonasgenome database: improved comparative analysis and population genomics capability forPseudomonasgenomes.Nucleic Acids Res,2011,39(S1): D596–D600.
[23]Campisano A, Schroeder C, Schemionek M, et al.PslD is a secreted protein required for biofilm formation byPseudomonas aeruginosa.Appl Environ Microbiol,2006,72(4):3066–3068.
[24]Overhage J, Schemionek M, Webb JS, et al.Expression of the psl operon inPseudomonas aeruginosaPAO1 biofilms: PslA performs an essential function in biofilm formation.Appl Environ Microbiol,2005,71(8):4407–4413.
[25]Lee HJ, Chang HY, Venkatesan N, et al.Identification of amino acid residues important for the phosphomannose isomerase activity of PslB inPseudomonas aeruginosaPAO1.FEBS Lett,2008,582(23/24):3479–3483.
[26]Wang SW, Parsek MR, Wozniak DJ, et al.A spider web strategy of type IV pili-mediated migration to build a fibre-like Psl polysaccharide matrix inPseudomonasaeruginosabiofilms. Environ Microbiol,2013,15(8):2238–2253.
[27]Zhao K, Tseng BS, Beckerman B, et al.Psl trails guide exploration and microcolony formation inPseudomonas aeruginosabiofilms.Nature,2013,497(7449):388–391.
[28]Irie Y, Borlee BR, O’Connor JR, et al.Self-produced exopolysaccharide is a signal that stimulates biofilm formation inPseudomonas aeruginosa.Proc Natl Acad Sci USA,2012,109(50):20632–20636.
[29]Byrd MS, Pang B, Mishra M, et al.ThePseudomonas aeruginosaexopolysaccharide Psl facilitates surface adherence and NF-κB activation in A549 cells.mBio,2010,1(3): e00140-10.
[30]Mishra M, Byrd MS, Sergeant S, et al.Pseudomonas aeruginosaPsl polysaccharide reduces neutrophil phagocytosis and the oxidative response by limiting complement-mediated opsonization.Cell Microbiol,2012,14(1):95–106.
[31]Zegans ME, Wozniak D, Griffin E, et al.Pseudomonas aeruginosaexopolysaccharide Psl promotes resistance to the biofilm inhibitor polysorbate80.Antimicrob Agents Chemother,2012,56(8):4112–4122.
[32]Tseng BS, Zhang W, Harrison JJ, et al.The extracellular matrix protectsPseudomonas aeruginosabiofilms by limiting the penetration of tobramycin.Environ Microbiol,2013,15(10):2865–2878.
[33]Billings N, Millan MR, Caldara M, et al.The extracellular matrix component Psl provides fast-acting antibiotic defense inPseudomonas aeruginosabiofilms.PLoS Pathog,2013,9(8):e1003526.
[34]Friedman L, Kolter R.Genes involved in matrix formation inPseudomonas aeruginosaPA14 biofilms.Mol Microbiol,2004,51(3):675–690.
[35]Vasseur P, Vallet-Gely I, Soscia C, et al.Thepelgenes of thePseudomonas aeruginosaPAK strain are involved at early and late stages of biofilm formation.Microbiology,2005,151(3):985–997.
[36]Govan JR, Deretic V.Microbial pathogenesis in cystic fibrosis: mucoidPseudomonas aeruginosaandBurkholderia cepacia.Microbiol Mol Biol Rev,1996,60(3):539–574.
[37]Wozniak DJ, Wyckoff TJO, Starkey M, et al.Alginate is not a significant component of the extracellular polysaccharide matrix of PA14 andPAO1Pseudomonas aeruginosabiofilms.Proc Natl Acad Sci USA,2003,100(13):7907–7912.
[38]Ma LY, Wang SW, Wang D, et al.The roles of biofilm matrix polysaccharide Psl in mucoidPseudomonasaeruginosabiofilms. FEMS Immunol Med Microbiol,2012,65(2):377–380.
[39]Yang L, Wang HZ, Wu H, et al.Polysaccharides serve as scaffold of biofilms formed by mucoidPseudomonas aeruginosa.FEMS Immunol Med Microbiol,2012,65(2):366–376.
[40]Leid JG, Willson CJ, Shirtliff ME, et al.The exopolysaccharide alginate protectsPseudomonas aeruginosabiofilm bacteria from IFN-γ-mediated macrophage killing.J Immunol,2005,175(11):7512–7518.
[41]Simpson JA, Smith SE, Dean RT.Alginate inhibition of the uptake ofPseudomonas aeruginosaby macrophages.Microbiology,1988,134(1):29–36.
[42]Simpson JA, Smith SE, Dean RT.Alginate may accumulate in cystic fibrosis lung because the enzymatic and free radical capacities of phagocytic cells are inadequate for its degradation.Biochem Mol Biol Int,1993,30(6):1021–1034.
[43]Simpson JA, Smith SE, Dean RT.Scavenging by alginate of free radicals released by macrophages.Free Radic Biol Med,1989,6(4):347–353.
[44]Bragonzi A, Paroni M, Nonis A, et al.Pseudomonas aeruginosamicroevolution during cystic fibrosis lung infection establishes clones with adapted virulence.Am J Respir Crit Care Med,2009,180(2):138–145.
[45]Allesen-Holm M, Barken KB, Yang L, et al.A characterization of DNA release inPseudomonas aeruginosacultures and biofilms.Mol Microbiol,2006,59(4):1114–1128.
[46]Whitchurch CB, Tolker-Nielsen T, Ragas PC, et al.Extracellular DNA required for bacterial biofilm formation.Science,2002,295(5559):1487.
[47]Webb JS, Thompson LS, James S, et al.Cell death inPseudomonas aeruginosabiofilm development.J Bacteriol,2003,185(15):4585–4592.
[48]Spoering AL, Gilmore MS.Quorum sensing and DNA release in bacterial biofilms.Curr Opin Microbiol,2006,9(2):133–137.
[49]Mulcahy H, Charron-Mazenod L, Lewenza S.Extracellular DNA chelates cations and induces antibiotic resistance inPseudomonas aeruginosabiofilms.PLoS Pathog,2008,4(11): e1000213.
[50]Finkel SE, Kolter R.DNA as a nutrient: novel role for bacterial competence gene homologs.J Bacteriol,2001,183(21):6288–6293.
[51]Mulcahy H, Charron-Mazenod L, Lewenza S.Pseudomonas aeruginosaproduces an extracellular deoxyribonuclease that is required for utilization of DNA as a nutrient source.Environ Microbiol,2010,12(6):1621–1629.
[52]Parks QM, Young RL, Poch KR, et al.Neutrophil enhancement ofPseudomonas aeruginosabiofilm development: human F-actin and DNA as targets for therapy.J Med Microbiol,2009,58(4):492–502.
[53]Fuxman Bass JI, Russo DM, Gabelloni ML, et al.Extracellular DNA: a major proinflammatory component ofPseudomonas aeruginosabiofilms.J Immunol,2010,184(11):6386–6395.
[54]Gloag ES, Turnbull L, Huang A, et al.Self-organization of bacterial biofilms is facilitated by extracellular DNA.Proc Natl Acad Sci USA,2013,110(28):11541–11546.
[55]Mann EE, Wozniak DJ.Pseudomonasbiofilm matrix composition and niche biology.FEMS Microbiol Rev,2012,36(4):893–916.
[56]O’Toole GA, Kolter R.Flagellar and twitching motility are necessary forPseudomonas aeruginosabiofilm development.Mol Microbiol,1998,30(2):295–304.
[57]Skerker JM, Berg HC.Direct observation of extension and retraction of type Ⅳ pili.Proc Natl Acad Sci USA,2001,98(12):6901–6904.
[58]Klausen M, Heydorn A, Ragas P, et al.Biofilm formation byPseudomonas aeruginosawild type,flagella and type Ⅳ pili mutants.Mol Microbiol,2003,48(6):1511–1524.
[59]Borlee BR, Goldman AD, Murakami K, et al.Pseudomonasaeruginosauses a cyclic-di-GMP-regulated adhesin to reinforce the biofilm extracellular matrix.Mol Microbiol,2010,75(4):827–842.
[60]Vallet I, Olson JW, Lory S, et al.The chaperone/usher pathways ofPseudomonas aeruginosa: Identification of fimbrial gene clusters(cup)and their involvement in biofilm formation.Proc Natl Acad Sci USA,2001,98(12):6911–6916.
[61]Giraud C, Bernard CS, Calderon V, et al.The PprA-PprB two-component system activates CupE,the first non-archetypalPseudomonas aeruginosachaperone-usher pathway system assembling fimbriae.Environ Microbiol,2011,13(3):666–683.
[62]Kulasekara HD, Ventre I, Kulasekara BR, et al.A novel two-component system controls the expression ofPseudomonas aeruginosafimbrial cup genes.Mol Microbiol,2005,55(2):368–380.
[63]de Kievit TR.Quorum sensing inPseudomonas aeruginosabiofilms.Environ Microbiol,2009,11(2):279–288.
[64]Parsek MR, Greenberg EP.Sociomicrobiology: the connections between quorum sensing and biofilms.Trends Microbiol,2005,13(1):27–33.
[65]Jimenez PN, Koch G, Thompson JA, et al.The multiple signaling systems regulating virulence inPseudomonas aeruginosa.Microbiol Mol Biol Rev,2012,76(1):46–65.
[66]Lee J, Zhang LH.The hierarchy quorum sensing network inPseudomonas aeruginosa.Protein Cell,2015,6(1):26–41.
[67]Gilbert KB, Kim TH, Gupta R, et al.Global position analysis of thePseudomonas aeruginosaquorum-sensing transcription factor LasR.Mol Microbiol,2009,73(6):1072–1085.
[68]Sakuragi Y, Kolter R.Quorum-sensing regulation of the biofilm matrix genes(pel)ofPseudomonas aeruginosa.J Bacteriol,2007,189(14):5383–5386.
[69]Yang L, Barken KB, Skindersoe ME, et al.Effects of iron on DNA release and biofilm development byPseudomonas aeruginosa.Microbiology,2007,153(5):1318–1328.
[70]Fraser GM, Hughes C.Swarming motility.Curr Opin Microbiol,1999,2(6):630–635.
[71]Harshey RM.Bees aren’t the only ones: swarming in gram-negative bacteria.Mol Microbiol,1994,13(3):389–394.
[72]Daniels R, Vanderleyden J, Michiels J.Quorum sensing and swarming migration in bacteria.FEMS Microbiol Rev,2004,28(3):261–289.
[73]Mattick JS.Type IV pili and twitching motility.Annu Rev Microbiol,2002,56:289–314.
[74]Patriquin GM, Banin E, Gilmour C, et al.Influence of quorum sensing and iron on twitching motility and biofilm formation inPseudomonas aeruginosa.J Bacteriol,2008,190(2):662–671.
[75]Van Gennip M, Christensen LD, Alhede M, et al.Inactivation of therhlAgene inPseudomonas aeruginosaprevents rhamnolipid production,disabling the protection against polymorphonuclear leukocytes.APMIS,2009,117(7):537–546.
[76]Dusane DH, Zinjarde SS, Venugopalan VP, et al.Quorum sensing: implications on Rhamnolipid biosurfactant production.Biotechnol Genet Eng Rev,2010,27(1):159–184.
[77]Pamp SJ, Tolker-Nielsen T.Multiple roles of biosurfactants in structural biofilm development byPseudomonas aeruginosa.J Bacteriol,2007,189(6):2531–2539.
[78]Davey ME, Caiazza NC, O'Toole GA.Rhamnolipid surfactant production affects biofilm architecture inPseudomonas aeruginosaPAO1.J Bacteriol,2003,185(3):1027–1036.
[79]Boles BR, Thoendel M, Singh PK.Rhamnolipids mediate detachment ofPseudomonas aeruginosafrom biofilms.Mol Microbiol,2005,57(5):1210–1223.
[80]Diggle SP, Stacey RE, Dodd C, et al.The galactophilic lectin, LecA, contributes to biofilm development inPseudomonas aeruginosa.Environ Microbiol,2006,8(6):1095–1104.
[81]Tielker D, Hacker S, Loris R, et al.Pseudomonasaeruginosalectin LecB is located in the outer membrane and is involved in biofilm formation.Microbiology,2005,151(5):1313–1323.
[82]Winzer K, Falconer C, Garber NC, et al.ThePseudomonas aeruginosalectins PA-IL and PA-IIL are controlled by quorum sensing and by RpoS.J Bacteriol,2000,182(22):6401–6411.
[83]Rodrigue A, Quentin Y, Lazdunski A, et al.Cell signalling by oligosaccharides.Two-component systems inPseudomonas aeruginosa: why so many? Trends Microbiol,2000,8(11):498–504.
[84]Parkins MD, Ceri H, Storey DG.Pseudomonas aeruginosaGacA, a factor in multihost virulence, is also essential for biofilm formation.Mol Microbiol,2001,40(5):1215–1226.
[85]Brencic A, McFarland KA, McManus HR, et al.The GacS/GacA signal transduction system ofPseudomonas aeruginosaacts exclusively through its control over the transcription of the RsmY and RsmZ regulatory small RNAs.Mol Microbiol,2009,73(3):434–445.
[86]Irie Y, Starkey M, Edwards AN, et al.Pseudomonasaeruginosabiofilm matrix polysaccharide Psl is regulated transcriptionally by RpoS and post-transcriptionally by RsmA.Mol Microbiol,2010,78(1):158–172.
[87]Kay E, Humair B, Dénervaud V, et al.Two GacA-dependent small RNAs modulate the quorum-sensing response inPseudomonas aeruginosa.J Bacteriol,2006,188(16):6026–6033.
[88]Reimmann C, Beyeler M, Latifi A, et al.The global activator GacA ofPseudomonas aeruginosaPAO positively controls the production of the autoinducerN-butyryl-homoserine lactone and the formation of the virulence factors pyocyanin,cyanide, and lipase.Mol Microbiol,1997,24(2):309–319.
[89]Jimenez PN, Koch G, Thompson JA, et al.The multiple signaling systems regulating virulence inPseudomonas aeruginosa.Microbiol Mol Biol Rev,2012,76(1):46–65.
[90]Goodman AL, Kulasekara B, Rietsch A, et al.A signaling network reciprocally regulates genes associated with acute infection and chronic persistence inPseudomonas aeruginosa.Dev Cell,2004,7(5):745–754.
[91]Kong W, Chen L, Zhao JQ, et al.Hybrid sensor kinase PA1611 inPseudomonas aeruginosaregulates transitions between acute and chronic infection through direct interaction with RetS.Mol Microbiol,2013,88(4):784–797.
[92]Ventre I, Goodman AL, Vallet-Gely I, et al.Multiple sensors control reciprocal expression ofPseudomonas aeruginosaregulatory RNA and virulence genes.Proc Natl Acad Sci USA,2006,103(1):171–176.
[93]Goodman AL, Merighi M, Hyodo M, et al.Direct interaction between sensor kinase proteins mediates acute and chronic disease phenotypes in a bacterial pathogen.Genes Dev,2009,23(2):249–259.
[94]Records AR, Gross DC.Sensor kinases RetS and LadS regulatePseudomonas syringaetype VI secretion and virulence factors.J Bacteriol,2010,192(14):3584–3596.
[95]Hickman JW, Tifrea DF, Harwood CS.A chemosensory system that regulates biofilm formation through modulation of cyclic diguanylate levels.Proc Natl Acad Sci USA,2005,102(40):14422–14427.
[96]Merighi M, Lee VT, Hyodo M, et al.The second messenger bis-(3′-5′)-cyclic-GMP and its PilZ domain-containing receptor Alg44 are required for alginate biosynthesis inPseudomonas aeruginosa.Mol Microbiol,2007,65(4):876–895.
[97]Hengge R.Principles ofc-di-GMP signalling in bacteria.Nat Rev Microbiol,2009,7(4):263–273.
[98]Lee VT, Matewish JM, Kessler JL, et al.A cyclic-di-GMP receptor required for bacterial exopolysaccharide production.Mol Microbiol,2007,65(6):1474–1484.
[99]Finkelstein RA, Sciortino CV, McIntosh MA.Role of iron in microbe-host interactions.Rev Infect Dis,1983,5(S4): S759–S777.
[100]Cornelissen CN, Sparling PF.Iron piracy:acquisition of transferrin-bound iron by bacterial pathogens.Mol Microbiol,1994,14(5):843–850.
[101]Braun V, Killmann H.Bacterial solutions to the iron-supply problem.Trends Biochem Sci,1999,24(3):104–109.
[102]Boukhalfa H, Crumbliss AL.Chemical aspects of siderophore mediated iron transport.Biometals,2002,15(4):325–339.
[103]Hider RC, Kong XL.Chemistry and biology of siderophores.Nat Prod Rep,2010,27(5):637–657.
[104]Schalk IJ, Mislin GLA, Brillet K.Structure,function and binding selectivity and stereoselectivity of siderophore-iron outer membrane transporters.Curr Top Membr,2012,69:37–66.
[105]Schalk IJ, Guillon L.Fate of ferrisiderophores after import across bacterial outer membranes: different iron release strategies are observed in the cytoplasm or periplasm depending on the siderophore pathways.Amino Acids,2013,44(5):1267–1277.
[106]Meyer JM.Pyoverdines: pigments, siderophores and potential taxonomic markers of fluorescentPseudomonasspecies.Arch Microbiol,2000,174(3):135–142.
[107]Ravel J, Cornelis P. Genomics of pyoverdine-mediated iron uptake inpseudomonads.Trends Microbiol,2003,11(5):195–200.
[108]Visca P, Imperi F, Lamont IL.Pyoverdine siderophores: from biogenesis to biosignificance.Trends Microbiol,2007,15(1):22–30.
[109]Albrecht-Gary AM, Blanc S, Rochel N, et al.Bacterial iron transport: coordination properties of pyoverdin PaA, a peptidic siderophore ofPseudomonas aeruginosa.Inorg Chem,1994,33(26):6391–6402.
[110]Meyer JM, Neely A, Stintzi A, et al.Pyoverdin is essential for virulence ofPseudomonas aeruginosa.Infect Immun,1996,64(2):518–523.
[111]Takase H, Nitanai H, Hoshino K, et al.Requirement of thePseudomonas aeruginosa tonBgene for high-affinity iron acquisition and infection.Infect Immun,2000,68(8):4498–4504.
[112]Takase H, Nitanai H, Hoshino K, et al.Impact of siderophore production onPseudomonas aeruginosainfections in immunosuppressed mice.Infect Immun,2000,68(4):1834–1839.
[113]Imperi F, Massai F, Facchini M, et al.Repurposing the antimycotic drug flucytosine for suppression ofPseudomonas aeruginosapathogenicity.Proc Natl Acad Sci USA,2013,110(18):7458–7463.
[114]Lamont IL, Beare PA, Ochsner U, et al.Siderophore-mediated signaling regulates virulence factor production inPseudomonas aeruginosa.Proc Natl Acad Sci USA,2002,99(10):7072–7077.
[115]Rédly GA, Poole K.Pyoverdine-mediated regulation of FpvA synthesis inPseudomonas aeruginosa: involvement of a probable extracytoplasmic-function sigma factor, FpvI.J Bacteriol,2003,185(4):1261–1265.
[116]Rédly GA, Poole K.FpvIR control offpvAferric pyoverdine receptor gene expression inPseudomonas aeruginosa: demonstration of an interaction between FpvI and FpvR and identification of mutations in each compromising this interaction.J Bacteriol,2005,187(16):5648–5657.
[117]Cornelis P.Iron uptake and metabolism inpseudomonads.Appl Microbiol Biotechnol,2010,86(6):1637–1645.
[118]Banin E, Vasil ML, Greenberg EP.Iron andPseudomonas aeruginosabiofilm formation.Proc Natl Acad Sci USA,2005,102(31):11076–11081.
[119]Glick R, Gilmour C, Tremblay J, et al.Increase in rhamnolipid synthesis under iron-limiting conditions influences surface motility and biofilm formation inPseudomonas aeruginosa.J Bacteriol,2010,192(12):2973–2980.
[120]Cornelis P, Dingemans J.Pseudomonas aeruginosaadapts its iron uptake strategies in function of the type of infections.Front Cell Infect Microbiol,2013,3:75.
[121]Cox CD, Rinehart KL Jr, Moore ML, et al.Pyochelin: novel structure of an iron-chelatinggrowth promoter forPseudomonas aeruginosa.Proc Natl Acad Sci USA,1981,78(7):4256–4260.
[122]Ankenbauer RG, Toyokuni T, Staley A, et al.Synthesis and biological activity of pyochelin, a siderophore ofPseudomonas aeruginosa.J Bacteriol,1988,170(11):5344–5351.
[123]Brandel J, Humbert N, Elhabiri M, et al.Pyochelin,a siderophore ofPseudomonas aeruginosa:physicochemical characterization of the iron(Ⅲ),copper(Ⅳ)and zinc(Ⅱ)complexes.Dalton Trans,2012,41(9):2820–2834.
[124]Serino L, Reimmann C, Visca P, et al.Biosynthesis of pyochelin and dihydroaeruginoic acid requires the iron-regulated pchDCBA operon inPseudomonas aeruginosa.J Bacteriol,1997,179(1):248–257.
[125]Dumas Z, Ross-Gillespie A, Kümmerli R.Switching between apparently redundant iron-uptake mechanisms benefits bacteria in changeable environments.Proc Roy Soc B Biol Sci,2013,280(1764):20131055.
[126]Coffman TJ, Cox CD, Edeker BL, et al.Possible role of bacterial siderophores in inflammation.Iron bound to thePseudomonassiderophore pyochelin can function as a hydroxyl radical catalyst.J Clin Invest,1990,86(4):1030–1037.
[127]Britigan BE, Roeder TL, Rasmussen GT, et al.Interaction of thePseudomonas aeruginosasecretory products pyocyanin and pyochelin generates hydroxyl radical and causes synergistic damage to endothelial cells.Implications forPseudomonas-associated tissue injury.J Clin Invest,1992,90(6):2187–2196.
[128]Britigan BE, Rasmussen GT, Cox CD.Augmentation of oxidant injury to human pulmonary epithelial cells by thePseudomonas aeruginosasiderophore pyochelin.Infect Immun,1997,65(3):1071–1076.
[129]Lyczak JB, Cannon CL, Pier GB.Lung infections associated with cystic fibrosis.Clin Microbiol Rev,2002,15(2):194–222.
[130]Hare NJ, Soe CZ, Rose B, et al.Proteomics ofPseudomonas aeruginosaAustralian epidemic strain1(AES-1)cultured under conditions mimicking the cystic fibrosis lung reveals increased iron acquisition via the siderophore pyochelin.J Proteome Res,2012,11(2):776–795.
[131]Ochsner UA, Johnson Z, Vasil ML.Genetics and regulation of two distinct haem-uptake systems,phuandhas, inPseudomonas aeruginosa.Microbiology,2000,146(1):185–198.
[132]Wyckoff EE, Lopreato GF, Tipton KA, et al.Shigella dysenteriaeShuS promotes utilization of heme as an iron source and protects against heme toxicity.J Bacteriol,2005,187(16):5658–5664.
[133]Létoffé S, Redeker V, Wandersman C.Isolation and characterization of an extracellular haem-binding protein fromPseudomonas aeruginosathat shares function and sequence similarities with theSerratia marcescensHasA haemophore. Mol Microbiol, 1998, 28(6):1223–1234.
[134]Wandersman C, Delepelaire P.Bacterial iron sources: from siderophores to hemophores.Annu Rev Microbiol,2004,58:611–647.
[135]Wandersman C, Delepelaire P.Haemophore functions revisited.Mol Microbiol,2012,85(4):618–631.
[136]Bhakta MN, Wilks A.The mechanism of heme transfer from the cytoplasmic heme binding protein PhuS to the δ-regioselective heme oxygenase ofPseudomonas aeruginosa.Biochemistry,2006,45(38):11642–11649.
[137]Barker KD, Barkovits K, Wilks A.Metabolic flux of extracellular heme uptake inPseudomonas aeruginosais driven by the iron-regulated heme oxygenase(HemO).J Biol Chem,2012,287(22):18342–18350.
[138]O’Neill MJ, Bhakta MN, Fleming KG, et al.Induced fit on heme binding to thePseudomonas aeruginosacytoplasmic protein(PhuS)drives interaction with heme oxygenase(HemO).Proc Natl Acad Sci USA,2012,109(15):5639–5644.
[139]Bodilis J, Ghysels B, Osayande J, et al.Distributionand evolution of ferripyoverdine receptors inPseudomonas aeruginosa.Environ Microbiol,2009,11(8):2123–2135.
[140]Cornelis P, Bodilis J.A survey of TonB-dependent receptors influorescent pseudomonads.Environ Microbiol Rep,2009,1(4):256–262.
[141]Cornelis P, Matthijs S, Van Oeffelen L.Iron uptake regulation inPseudomonas aeruginosa.Biometals,2009,22(1):15–22.
[142]Hartney SL, Mazurier S, Kidarsa TA, et al.TonB-dependent outer-membrane proteins and siderophore utilization inPseudomonas fluorescensPf-5.Biometals,2011,24(2):193–213.
[143]Mettrick KA, Lamont IL.Different roles for anti-sigma factors in siderophore signalling pathways ofPseudomonas aeruginosa.Mol Microbiol,2009,74(5):1257–1271.
[144]Ghysels B, Dieu BTM, Beatson SA, et al.FpvB, an alternative type Ⅰ ferripyoverdine receptor ofPseudomonas aeruginosa.Microbiology,2004,150(6):1671–1680.
[145]Traxler MF, Seyedsayamdost MR, Clardy J, et al.Interspecies modulation of bacterial development through iron competition and siderophore piracy.Mol Microbiol,2012,86(3):628–644.
[146]Andrews SC, Robinson AK, Rodríguez-Quiñones F.Bacterial iron homeostasis.FEMS Microbiol Rev,2003,27(2/3):215–237.
[147]Cartron ML, Maddocks S, Gillingham P, et al.Feo- transport of ferrous iron into bacteria.Biometals,2006,19(2):143–157.
[148]Worlitzsch D, Tarran R, Ulrich M, et al.Effects of reduced mucus oxygen concentration in airwayPseudomonasinfections of cystic fibrosis patients.J Clin Invest,2002,109(3):317–325.
[149]Yoon SS, Hennigan RF, Hilliard GM, et al.Pseudomonas aeruginosaanaerobic respiration in biofilms: relationships to cystic fibrosis pathogenesis.Dev Cell,2002,3(4):593–603.
[150]Wang Y, Newman DK.Redox reactions of phenazine antibiotics with ferric(hydr)oxides and molecular oxygen.Environ Sci Technol,2008,42(7):2380–2386.
[151]Wang Y, Wilks JC, Danhorn T, et al.Phenazine-1-carboxylic acid promotes bacterial biofilm development via ferrous iron acquisition.J Bacteriol,2011,193(14):3606–3617.
[152]Hunter RC, Klepac-Ceraj V, Lorenzi MM, et al.Phenazine content in the cystic fibrosis respiratory tract negatively correlates with lung function and microbial complexity.Am J Respir Cell Mol Biol,2012,47(6):738–745.
[153]Hunter RC, Asfour F, Dingemans J, et al.Ferrous iron is a significant component of bioavailable iron in cystic fibrosis airways.mBio,2013,4(4):e00557–13.
[154]Barton HA, Johnson Z, Cox CD, et al.Ferric uptake regulator mutants ofPseudomonas aeruginosawith distinct alterations in the iron-dependent repression of exotoxin A and siderophores in aerobic and microaerobic environments.Mol Microbiol,1996,21(5):1001–1017.
[155]Matthijs S, Baysse C, Koedam N, et al.ThePseudomonassiderophore quinolobactin is synthesized from xanthurenic acid, an intermediate of the kynurenine pathway.Mol Microbiol,2004,52(2):371–384.
[156]Wilderman PJ, Sowa NA, FitzGerald DJ, et al.Identification of tandem duplicate regulatory small RNAs inPseudomonas aeruginosainvolved in iron homeostasis.Proc Natl Acad Sci USA,2004,101(26):9792–9797.
[157]Vasil ML.How we learnt about iron acquisition inPseudomonas aeruginosa: a series of very fortunate events.Biometals,2007,20(3/4):587–601.
[158]Cornelis P.Iron uptake and metabolism inPseudomonas.Appl Microbiol Biotechnol,2010,86(6):1637–1645.
[159]Martin LW, Reid DW, Sharples KJ, et al.Pseudomonassiderophores in the sputum of patients with cystic fibrosis.Biometals,2011,24(6):1059–1067.
[160]De Vos D, De Chial M, Cochez C, et al.Study ofpyoverdine type and production byPseudomonas aeruginosaisolated from cystic fibrosis patients:prevalence of type Ⅱ pyoverdine isolates and accumulation of pyoverdine-negative mutations.Arch Microbiol,2001,175(5):384–388.
[161]Lamont IL, Konings AF, Reid DW.Iron acquisition byPseudomonas aeruginosain the lungs of patients with cystic fibrosis.Biometals,2009,22(1):53–60.
[162]Konings AF, Martin LW, Sharples KJ, et al.Pseudomonas aeruginosauses multiple pathways to acquire iron during chronic infection in cystic fibrosis lungs.Infect Immun,2013,81(8):2697–2704.
[163]Singh PK, Parsek MR, Greenberg EP, et al.A component of innate immunity prevents bacterial biofilm development.Nature,2002,417(6888):552–555.
[164]Rogan MP, Taggart CC, Greene CM, et al.Loss of microbicidal activity and increased formation of biofilm due to decreased lactoferrin activity in patients with cystic fibrosis.J Infect Dis,2004,190(7):1245–1253.
[165]Berlutti F, Morea C, Battistoni A, et al.Iron availability influences aggregation, biofilm,adhesion and invasion ofPseudomonas aeruginosaandBurkholderia cenocepacia.Int J Immunopathol Pharmacol,2005,18(4):661–670.
[166]Cornelis P, Wei Q, Andrews SC, et al.Iron homeostasis and management of oxidative stress response in bacteria.Metallomics,2011,3(6):540–549.
[167]Wiens JR, Vasil AI, Schurr MJ, et al.Iron-regulated expression of alginate production,mucoid phenotype, and biofilm formation byPseudomonas aeruginosa.mBio,2014,5(1):e01010–013.
[168]Yu S, Wei Q, Zhao TH, et al.A survival strategy forPseudomonasaeruginosathat uses exopolysaccharides to sequester and store iron to stimulate Psl-dependent biofilm formation.Appl Environ Microbiol,2016,82(21):6403–6413.
(本文责编 郝丽芳)
Iron uptake and biofilm formation inPseudomonas aeruginosa
Shan Yu1, and Luyan Ma2
1Institute of Biophysics,Chinese Academy of Sciences,Beijing100101,China
2Institute of Microbiology,Chinese Academy of Sciences,Beijing100101,China
Biofilms are surface-associated communities of microorganisms embedded within self-secreted extracellular polymeric substances, and a major cause of chronic and persistent infections.RespiratoryPseudomona aeruginosainfection is the leading reason for morbidity and mortality in cystic fibrosis patients.The formation of biofilms byP.aeruginosain the airway is thought to increase persistence and antibiotic resistance during infection.Biofilm formation ofP.aeruginosais regulated by complicated signaling systems including quorum sensing and two-component systems that control the synthesis of extracellular polymeric substances.Furthermore, iron is an essential and scarce nutrient for bacteria and an important signal factor.P.aeruginosahas developed multiple iron uptake systems to sequester enough iron for its survival,with important regulatory roles in both release of virulence factors and formation of biofilms.In this review, we summarize recent advances in biofilm formation and its regulation along with the iron-uptake strategies inP.aeruginosa, to provide new insights and understanding to fight bacterial biofilms.
Pseudomonas aeruginosa, biofilm, iron uptake, qurom sensing, regulation factor
April5,2017;Accepted:May8,2017
Luyan Ma.Tel: +86-10-64807437; Fax: +86-10-64806101; E-mail: luyanma27@im.ac.cn

于珊, 马旅雁.铜绿假单胞菌铁摄取与生物被膜形成研究进展.生物工程学报,2017,33(9):1489–1512.
Yu S, Ma LY.Iron uptake and biofilm formation inPseudomonas aeruginosa.Chin J Biotech,2017,33(9):1489–1512.
Supported by:National Basic Research Program of China(973 Program)(No.2014CB846002), National Natural Science Foundation of China(No.31570126).
国家重点基础研究发展计划(973计划)(No.2014CB846002),国家自然科学基金(No.31570126)资助。
马旅雁 中国科学院微生物研究所研究员,博士生导师。1996年于中国农业大学获理学博士学位;中国科学院“百人计划”入选者;中国农业生物技术学会微生物生物技术分会理事;Microbiology Open副主编,Frontiers in Microbiology编委。马旅雁研究员致力于细菌生物被膜(Biofilm,亦称生物膜)的研究,旨在了解细菌的群体行为以及微生物间的信息交流,解析细菌生物被膜形成与瓦解的分子机理,为环境治理中有效发挥生物被膜的功能奠定理论基础,为防治和控制生物被膜相关问题提供理论依据及可能的解决办法。

