利用EST-SSR标记构建中国老芒麦品种DNA指纹图谱及种质遗传多样性
张俊超,谢文刚,赵旭红,张宗瑜,赵永强,王彦荣
(草地农业生态系统国家重点实验室 农业部草牧业创新重点实验室 草业科学国家级实验教学示范中心,兰州大学草地农业科技学院,甘肃 兰州 730020)
利用EST-SSR标记构建中国老芒麦品种DNA指纹图谱及种质遗传多样性
张俊超,谢文刚,赵旭红,张宗瑜,赵永强,王彦荣
(草地农业生态系统国家重点实验室 农业部草牧业创新重点实验室 草业科学国家级实验教学示范中心,兰州大学草地农业科技学院,甘肃 兰州 730020)
植物生产层
本研究利用EST-SSR分子标记,对国内的52份老芒麦(Elymussibiricus)材料进行遗传多样性的研究,并构建了7个老芒麦品种及栽培材料的DNA指纹图谱。20对EST-SSR引物共产生204个条带,平均每对引物扩增出10.2条带,176(86.27%)条带为多态性条带,表明供试材料具有较高水平的多态性。同时,引物Elw404和Elw195构建的指纹图谱较为容易地将品种和栽培材料与其他材料区别开。分子方差分析(AMOVA)表明,老芒麦地理区域内(73.94%)的遗传变异远高于地理区域间(26.06%)。结构分析将52份材料分为5组,主成分分析(PCoA)结果与之相似。与品种及栽培材料相比,野生材料具有更高的遗传多样性[多态性条带数(NPB)为174,多态性百分比(PPB)为85.29%,香农多样性信息指数(I)为0.296 6,Nei’s基因多样性(H)为0.179 8,观察等位基因数(Na)为1.852 9],可作为重要的遗传资源用于后续的育种工作。本研究结果表明,EST-SSR分子标记可以有效地评价老芒麦种质遗传多样性及进行品种鉴定。研究结果为制定老芒麦种质原位和非原位保护策略提供参考。
老芒麦;DNA指纹图谱;EST-SSR;遗传变异;种质保护
准确地鉴别植物栽培材料、品种及野生材料,并明确它们的遗传关系,是育种研究、品种管控与登记,以及保护育种者权利的基础[1]。传统方法中,植物品种的鉴别常使用形态学与生育期特性相结合的方法,但这种方法具有比较困难、定义不明确、耗时且具主观性的缺点。同时,鉴定的准确性会受环境因素的影响而受限制。DNA指纹图谱技术则可有效地弥补传统方法的缺陷,这种新方法可以将植物任何生理时期的任何组织作为材料。很多分子标记,诸如RAPD、ISSR、SSR、SRAP、SCOT[2-6]等,已经成功地在许多作物和牧草物种中用于品种鉴别和遗传多样性的测定。其中,SSR标记具有高突变率、共显性和高重复性的优点,可作为理想的标记方法,用于牧草及一些农业和园艺植物的品种鉴定和遗传多样性的研究[7-11]。
披碱草属(Elymus)是小麦族(Triticeae)中最大的属,它包含遍及世界的150个物种[12]。老芒麦(E.sibiricus),又称西伯利亚野黑麦(Siberian wild rye),在世界范围分布广泛,如在东欧、西伯利亚、中亚、西亚、蒙古、朝鲜、及印度均有分布[13],在北美洲也有少量分布,主要集中在亚北极地区,即阿拉斯加和加拿大北部[14-15]。国内老芒麦的种质丰富且多样化,它的主要分布地区包括河北、内蒙古、山西、陕西、西藏、青海、四川、甘肃及新疆等省(区)[4-6,16]。老芒麦是一种多年生、自花授粉的异源四倍体牧草[14]。它具有对环境适应性强,耐寒、耐旱性良好以及出色的草品质,已广泛应用于天然草地和栽培草地。但老芒麦在品种选育和遗传改良方面的进展较为缓慢。到目前为止,国内仅登记了8个老芒麦品种,其中的一些品种有着相对狭窄的遗传背景。这可能与育种的原始材料在自然或人工条件下与已有推广品种发生杂交有关[17]。因此,老芒麦品种选育进展缓慢的原因之一是缺少有效的遗传资源和遗传变异。鄢家俊等[4]采用SRAP和SSR分子标记研究了青藏高原地区的野生老芒麦种质遗传多样性,结果表明该地区的老芒麦种质具有丰富的遗传多样性。Xie等[18]基于EST-SSR标记研究发现,来自青藏高原地区具有不同落粒性的老芒麦种质的遗传多样性水平较高。Xie等[6]采用ScoT分子标记,Ma等[19]运用醇溶蛋白标记,均发现世界来源的老芒麦材料具有高水平的遗传变异,且遗传分化与其生态地理环境有密切关系。然而,针对国内老芒麦主要分布地区的种质遗传多样性和遗传关系并未有较好的论述。构建老芒麦品种和栽培材料的DNA指纹图谱,在国内外报道较少[17,20]。因此,借助EST-SSR分子标记构建国内老芒麦品种指纹图谱,并对国内野生老芒麦材料和品种间进行深层次的遗传多样性比较分析,可以对老芒麦遗传多样性分布提供新的见解。
本研究利用EST-SSR分子标记构建7个老芒麦品种及栽培材料的DNA指纹图谱,评价与比较品种、栽培材料和野生材料间的遗传多样性和亲缘关系,以期促进老芒麦种质收集、保护和育种工作。
1 材料与方法
1.1样本收集
研究所用的52份老芒麦材料均来自国内,主要包括品种、栽培材料和野生材料(表1)。其中包括4个栽培品种(同德、青牧1号、川草2号、红原),18份引种材料由美国农业部提供,其他材料由兰州大学草地农业生态系统国家重点实验室,四川农业大学和四川省草原科学研究院提供。所有的材料按起源地和自然区划可归结为5个地理区域,分别为青海(QH)、新疆(XJ)、四川(SC)、内蒙古(NM)和甘肃(GS)。

表1 试验中老芒麦材料的信息Table 1 Elymus sibiricus accessions used in the study
1.2DNA提取
每份材料选取25个幼苗的叶片等量混合,经冻干处理后采用改进的CTAB法提取DNA[21]。通过使用Nanodrop分光光度计和琼脂糖凝胶电泳对DNA的浓度和质量进行测定。最后,DNA的浓度使用ddH2O调整为25 ng·μL-1用以进一步的分析。
1.3引物筛选及PCR扩增
本研究中,100对EST-SSR引物主要来源于披碱草属、拟鹅观草属(Pseudoroegneria)及赖草属(Leymus)[22-23]。最终,筛选出20对多态性高的EST-SSR引物用于52份老芒麦材料遗传多样性评价。PCR扩增反应在PTC-200热循环仪上进行,扩增程序:94 ℃变性15 s,65→60 ℃退火30 s,72 ℃延伸1 min,共5个循环;接下来是94 ℃变性30 s,60 ℃退火30 s,72 ℃延伸1min,共30个循环;最后72 ℃延伸7 min;4 ℃保存。扩增片段点样于6%变性聚丙烯酰胺凝胶,电泳后,凝胶用AgNO3溶液染色,显影后在凝胶成像系统拍照。
1.4数据分析
对清晰度高、分辨度好的扩增条带按照条带的有无分别记为1,0。参照以下公式[24]计算每一对EST-SSR引物的多态信息含量值(PIC):
PIC=1-p2-q2.
式中:p表示有带所占的频率,q表示无带所占的频率。条带的信息量(Ib)按照如下公式[25]计算:
Ib=1-(2×|0.5-p|).
式中:p表示有带的品种或基因型所占的比例。
条带的分辨率(Rp)通过如下公式来计算[24]:
Rp=∑Ib.
POPGENE 32 v1.31软件[26]用在假定Hardy-Weinberg平衡条件下计算这些材料的遗传一致度(GI)、遗传距离(GD)、多态性条带数(NPB)、多态性百分比(PPB)、香农多样性信息指数(I)、Nei’s基因多样性(H)及观察等位基因数(Na)。采用AMOVA v1.55软件分析类群内和类群间遗传变异[27]。STRUCTURE v2.3.4软件[28-29]进行遗传结构分析。由NTSYS v2.10软件进行主坐标分析,同时利用品种和栽培材料的数据,结合Winboot软件构建系统树图[30-31]。其中,POPGENE和AMOVA的输入文件由DCFA v1.1软件准备[32]。
1.5DNA指纹图谱的构建和计算机化
老芒麦DNA指纹图谱的构建主要参考鸭茅指纹图谱构建的方法[33]。DNA指纹模型通过数字0和1转换为计算机语言来表示,即1代表有带,0代表无带[34]。
2 结果
2.1基于EST-SSR标记构建DNA指纹图谱
本研究中,20对EST-SSR引物是由100对EST-SSR引物筛选而来,这些引物可扩增出多态性高且清晰的条带(表2)。每对引物扩增出的多态性条带数量从2(Ps3577)到21(Elw306)不等,平均值为8.8。20对EST-SSR引物的多态信息含量(PIC)变化范围为0.26(Ps3577)~0.50(Ps1475,Elw228)。条带分辨率(Rp)从0.27(Ltc0055)到7.42(Elw404)不等,平均值为3.01。基于以上结果,Elw306(总条带数和多态性条带数均为最大值)、Elw404(Rp为最大值)、Ps2283、Elw195和Elw187这5对引物在遗传多样性分析中具有更多的潜力,可以较少的引物研究更多的个体和取样点。
引物Elw404和Elw195的条带分辨率及多态信息含量值较大,被选作用于构建7份老芒麦品种和栽培材料的DNA指纹图谱(图1)。两对引物扩增的条带数分别为17和14,条带大小范围为109~335 bp,这些独一无二的指纹图谱很容易将7份供试材料相互区别开。
2.2老芒麦种质遗传多样性
20对引物共扩增出204条带,其中176条带(86.27%)为多态性条带(表1),表明52份老芒麦材料具有高水平的遗传多态性。此外,与品种和栽培材料(PPB=50.98%,I=0.234 7,H=0.151 0,Na=1.509 8)相比,野生老芒麦材料具有更大的变异性(PPB=85.29%,I=0.296 6,H=0.179 8,Na=1.852 9)(表3),这一结果可能与试验中栽培材料数量较少有关。同时表明野生老芒麦材料有着广泛的遗传背景,可作为重要的基因库用于品种改良。
2.3遗传关系和类群结构
AMOVA分析表明,老芒麦地理类群内部的遗传变异为73.94%,地理类群间的遗传变异为26.06%(表4)。5个地区材料的遗传一致度(GI)从0.941 0到0.895 2不等(表5)。青海和新疆材料间的遗传一致度最小,为0.895 2。四川与甘肃材料间的遗传一致度最大,为0.941 0,两个类群材料均采样于青藏高原的东北缘。
借助STRUCRURE V 2.3.4软件,基于哈迪温伯格(Hardy-Weinberg)平衡,对52份材料的遗传结构进行评估,根据最大似然值和ΔK值可得到类群数目的最佳值(图2)。K=5时,所有的材料聚集为5个类群,这与老芒麦地理分布几乎一致,因此5就是最合适7份野生材料;类群Ⅳ包含来自内蒙古的2个栽培材料和4份野生材料;最后一个类群包括来自甘肃省的22份野生材料。主坐标分析遗传关系的结果与结构分析一致:前3个主成分可解释69.44%的总变异,它们分别占39.61%、29.83%和13.77%(图3)。

表2 20对EST-SSR引物用于老芒麦品种、栽培材料和野生材料基因型的研究Table 2 20 EST-SSR primers used to genotype Elymus sibiricus cultivars and, cultivated and wild accessions
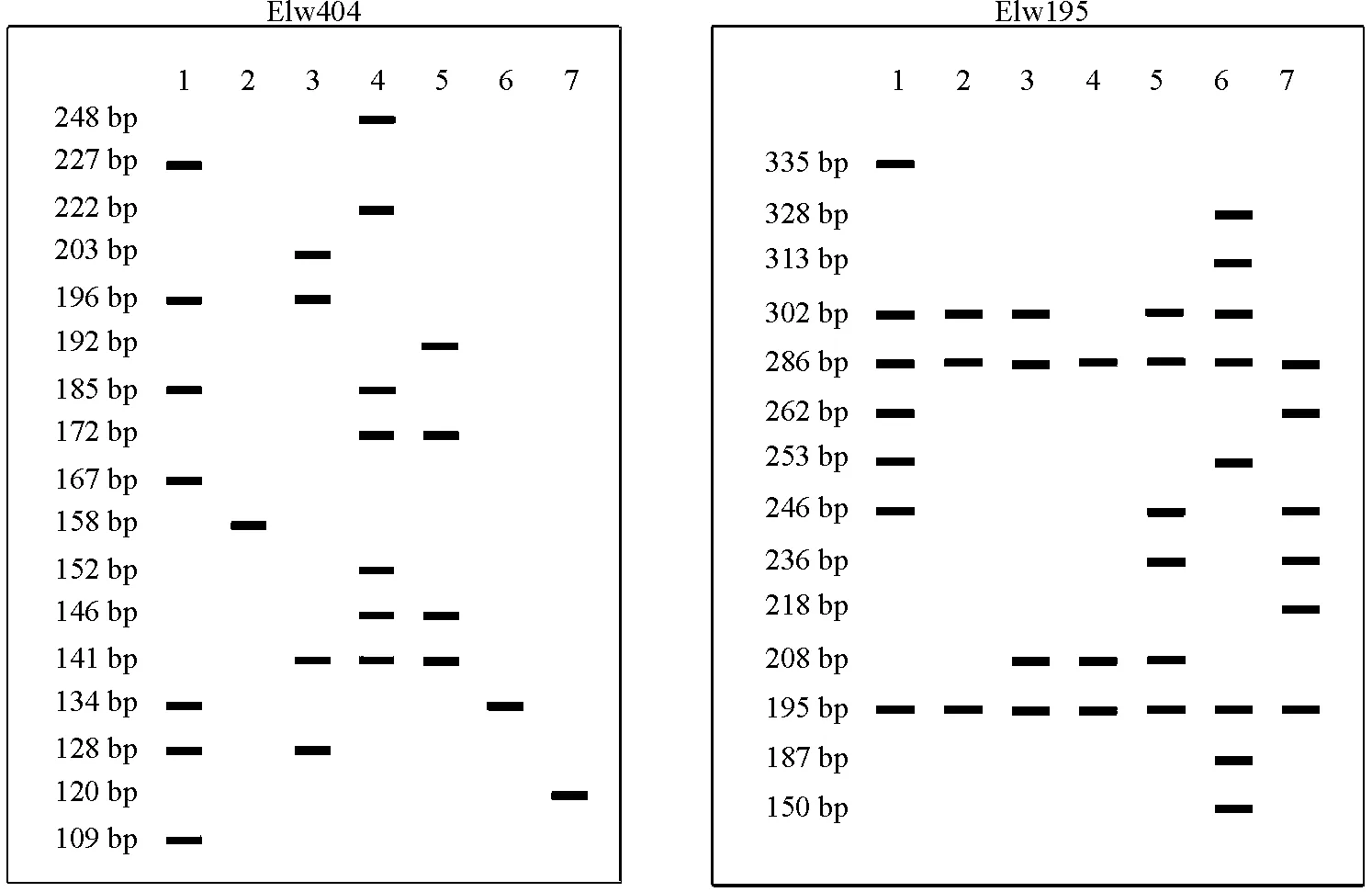
图1 通过Elw404和Elw195两对引物构建老芒麦品种(栽培种)指纹图谱Fig. 1 Molecular fingerprinting of Elymus sibiricus cultivars (cultivated accessions) using the primers Elw404 and Elw195
注:1号材料为同德,2号材料为青牧1号,3号材料为PI499468,4号材料为川草2号,5号材料为红原,6号材料为PI499457,7号材料为PI499456。
Note: 1, Tongde; 2, Qingmu No.1; 3, PI499468; 4, Chuancao No.2; 5, Hongyuan; 6, PI499457; 7, PI499456.

表3 EST-SSR标记用于老芒麦品种和栽培材料与野生材料的遗传变异性Table 3 Genetic variability between cultivar & cultivated accessions and wild groups of Elymus sibiricus detected by EST-SSR markers
Note: PB,Polymorphic bands;I,Shannon information index of diversity;H,Nei’s genetic diversity;Na,observed number of alleles values.的类群数目。类群Ⅰ包括来自青海省的2个品种和3份野生材料;类群Ⅱ包含来自新疆的1个栽培材料和9份野生材料;类群Ⅲ包括来自四川省的2个品种和
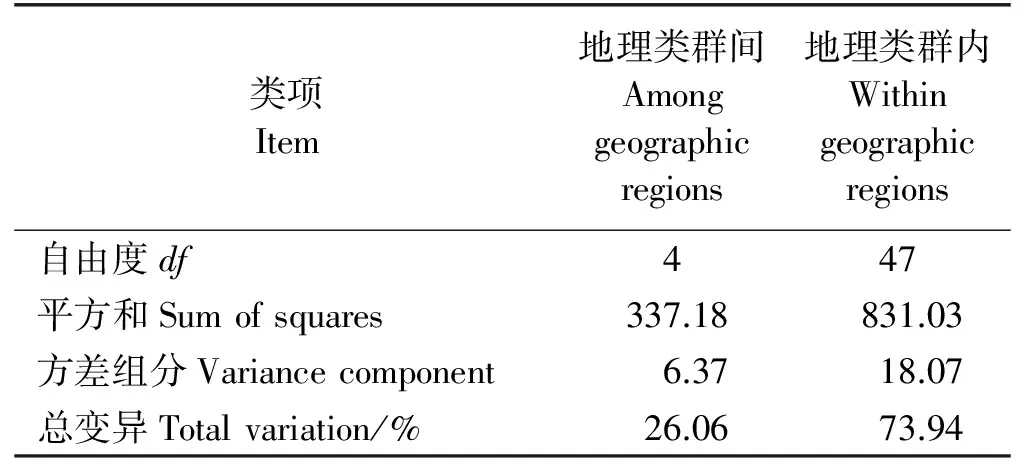
表4 基于EST-SSR标记对老芒麦的分子方差分析Table 4 Analysis of molecular variance of Elymus sibiricus based on EST-SSR markers

表5 老芒麦5个地理类群间Nei’s遗传一致度(对角线上部)和遗传距离(对角线下部) Table 5 Pairwise Nei’s genetic identity (above diagonal) and genetic distance (below diagonal) for five geographical populations of Elymus sibiricus

图2 中国52份老芒麦材料遗传结构分析Fig. 2 STRUCTURE analysis of 52 Elymus sibiricus accessions in China
注:类群编码同表1所示,不同的个体具有相同的颜色表示它们归属为同一类群。
Note: The population codes are indicated in Table 1. Same color in different individuals indicates that they belong to the same cluster.
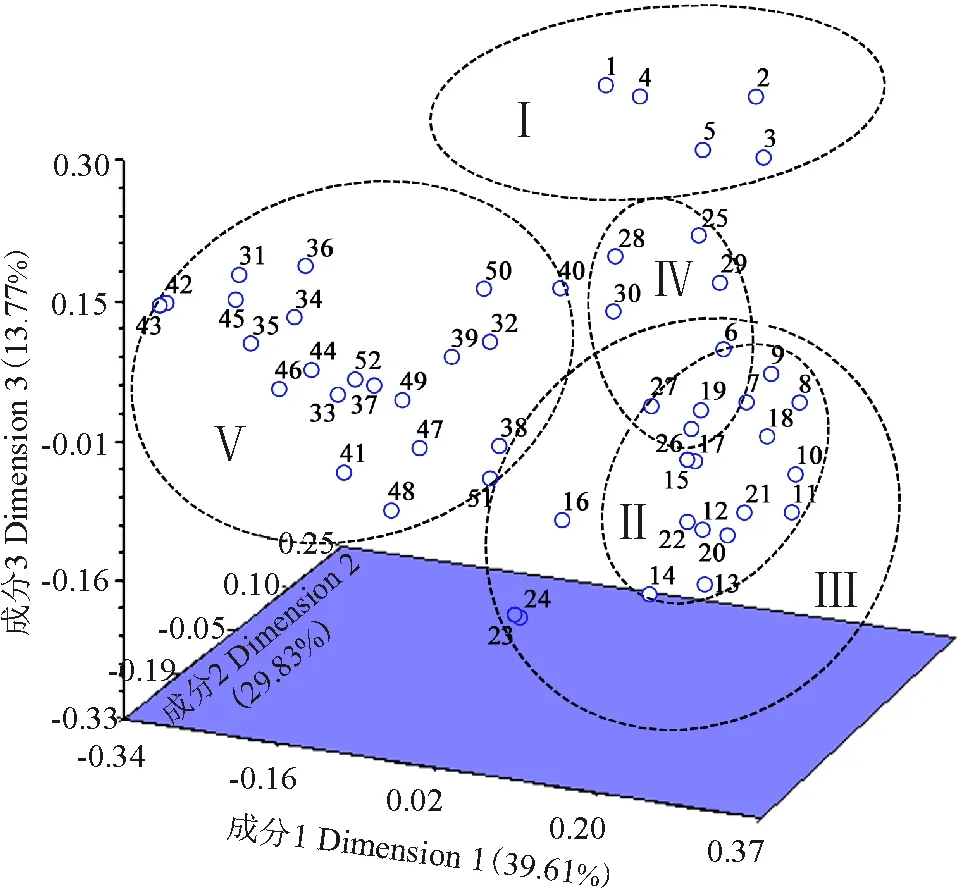
图3 基于EST-SSR标记使用Dice遗传相似矩阵对52份老芒麦进行主坐标分析的三维发散图Fig. 3 Principal coordinates analysis of three-dimensional scatter diagram for EST-SSR markers using the Dice genetic similarity matrix for 52 Elymus sibiricus accessions
利用统计所得数据,将3个品种和4份栽培材料单独进行聚类,由此得到的系统树图揭示了这几份材料间的遗传关系(图4)。这些材料聚为两大类(Ⅰ和Ⅱ),并且类群Ⅰ包括3个亚类群(A,B,C)。3个省(区)的6份材料聚在类群Ⅰ,同一地区的2份材料又形成单独的亚类群,而类群Ⅱ仅包括来自新疆的1个栽培材料。
3 讨论与结论
3.1老芒麦种质遗传多样性及遗传变异
老芒麦种质遗传多样性的研究,既可促进对该物种种质资源的管理、评价及利用,也是创制老芒麦优异新品种的基础工作[35]。本研究中,利用EST-ISSR引物扩增的多态性条带百分比(PPB)为86.27%,表明老芒麦具有高水平的遗传多样性。该结果与前人的研究结果相似,如SRAP(PPB=86.48%)[4],SSR(PPB=86.88%)[5],高于ISSR(PPB=77.20%)[3]和RAPD(PPB=78.65%)[2],但是低于SCoT(PPB=89.0%)[6]和醇溶蛋白(PPB=90.4%)[19]。老芒麦高水平的遗传多样性可能是由于其地理来源多样化和分子标记的高效性,在象草(Pennisetumpurpureum)[36]和大豆(Glycinemax)[37]的研究报道中已证明地理来源和遗传多样性之间有较强的相关性,严学兵等[38]认为地理生态环境与披碱草属植物的遗传变异有关,并指出青藏高原与蒙古高原多变的气候与环境因素会引起该属同种不同居群间遗传变异增大。本研究中,用于研究的52份老芒麦来自5个不同的地理区域,且它们具有不同的生态地理条件。有研究认为,引物平均多态信息含量值(PIC)与群体遗传变异程度正相关[39]。本研究所用20对EST-SSR引物PIC值范围为0.26~0.50,平均值为0.43,表明供试老芒麦材料间遗传变异程度较高。同时,这些引物扩增出204条带,其中176条具多态性。以上结果及前人的研究分析均表明EST-SSR标记用于评价老芒麦遗传多样性是一种有效的工具。

图4 基于Jaccard’s遗传相似性及非加权组平均法推导出老芒麦品种和栽培材料的系统树图Fig. 4 UPGMA-derived dendrogram of Elymus sibiricus cultivar and cultivated accessions based on Jaccard’s genetic similarity
注:图中只表示出bootstrap值大于50%的部分。
Note: Only bootstrap values higher than 50% are presented.
3.2品种和栽培材料与野生材料的遗传多样性比较
关于野生材料与品种/栽培材料间遗传多样性比较的研究未见报道。本研究结果表明,与品种/栽培材料(PPB=50.98%,I=0.234 7,H=0.151 0,Na=1.509 8)相比,野生材料(PPB=85.29%,I=0.296 6,H=0.179 8,Na=1.852 9)具有更高的遗传多样性。有研究表明,与野生材料相比,品种/栽培材料的遗传多样性较低,如荔枝(Litchichinensis)[40]、水稻(Oryzarufipogon)[41]、大豆[42]和珍珠粟(Pennisetumglaucum)[43]等。研究者曾指出与目的农艺性状相关的大多数位点(如生育期、叶面积和产量等)在育种进程中被选择并保留下来,但也有很多基因在此过程中丢失[44]。
单株选择法或系谱法,是自花授粉植物培育新品种的一种基本方法[45]。前人报道指出,单株选择法可以从混合系中分离出纯系,并可以在其子代保留最好的基因型[46]。因此,作为一种自花授粉植物,老芒麦在育种进程中可能因多次单株选择,导致杂合子的数目减少而纯合子增加[47]。以上因素降低了老芒麦品种/栽培材料的遗传多样性。相比之下,野生种质材料,因此它们具有更高的遗传多样性[48-49]。因此,具有丰富遗传多样性的野生材料在今后的育种工作中应作为重要的遗传资源。
本研究采用EST-SSR标记构建老芒麦指纹图谱,能够区别不同品种及栽培材料的遗传特异性,同时也为国内老芒麦品种知识产权和育种家的权益提供保障。
3.3老芒麦种质资源保护
其他的研究已证明国内的老芒麦种质资源丰富且具多样性,可以通过不同的标记方法揭示其丰富的表型性状和较高的遗传多样性[2-6,18-19]。本研究发现,与品种/栽培材料比较,野生材料具有更高的遗传多样性,且地理类群内具有更大的遗传变异。研究表明,近年来生态环境日益恶化加之自然灾害频发,造成大量野生老芒麦种质资源急剧下降[50]。同时,落后、单一的育种方法及种质收集保存不充分,使得国内只有少数的老芒麦品种[51-52]。因此,需要更加关注原位保护和非原位保护,以及特殊地理类群的老芒麦种质的收集与保护,从而为优异种质筛选和育种奠定基础。
综上,EST-SSR分子标记是研究老芒麦种质遗传多样性的有效方法,由此也证明我国老芒麦种质具有丰富的遗传变异,这在野生老芒麦材料中体现的尤为明显。为此,在开展老芒麦育种研究工作的同时,也更应注重老芒麦种质资源的收集、保护和利用等。
References:
[1] Rajora O P,Rahman M H.Microsatellite DNA and RAPD fingerprinting, identification and genetic relationships of hybrid poplar (Populusxcanadensis) cultivars.Theoretical and Applied Genetics,2003,106(3):470-477.
[2] Ma X,Chen S Y,Bai S Q,Zhang X Q,Li D X,Zhang C B,Yan J J.RAPD analysis of genetic diversity and population structure ofElymussibiricus(Poaceae) native to the southeastern Qinghai-Tibet Plateau,China.Genetics and Molecular Research,2012,11(3):2708-2718.
[3] Ma X,Zhang X Q,Zhou Y H,Bai S Q,Liu W.Assessing genetic diversity ofElymussibiricus(Poaceae:Triticeae) populations from Qinghai-Tibet Plateau by ISSR markers.Biochemical Systematics and Ecology,2008,36(7):514-522.
[4] 鄢家俊,白史且,张新全,游明鸿,张昌兵,李达旭,曾怡.青藏高原老芒麦种质基于SRAP标记的遗传多样性研究.草业学报,2010,19(1):173-183.
Yan J J,Bai S Q,Zhang X Q,You M H,Zhang C B,Li D X,Zeng Y.Genetic diversity of wildElymussibiricusgermplasm from the Qinghai-Tibetan Plateau in China detected by SRAP markers.Acta Prataculturae Sinica,2010,19(1):173-183.(in Chinese)
[5] 鄢家俊,白史且,张新全,常丹,游明鸿,张昌兵,李达旭.青藏高原东南缘老芒麦自然居群遗传多样性的SRAP和SSR分析.草业学报,2010,19(4):122-134.
Yan J J,Bai S Q,Zhang X Q,Chang D,You M H,Zhang C B,Li D X.Genetic diversity of nativeElymussibiricuspopulations in the Southeastern Margin of Qinghai-Tibetan Plateau as detected by SRAP and SSR markers.Acta Pratacultuae Sinica,2010,19(4):122-134.(in Chinese)
[6] Xie W G,Zhang J C,Zhao X H,Zhang J Q,Wang Y R.Siberian wild rye (ElymussibiricusL):Genetic diversity of germplasm determined using DNA fingerprinting and SCoT markers.Biochemical Systematics and Ecology,2015,60:186-192.
[7] Dias P M B,Julier B,Sampoux J P,Barre P,Dall’Agnol M.Genetic diversity in red clover (TrifoliumpratenseL.)revealed by morphological and microsatellite (SSR)markers.Euphytica,2008,160(2):189-205.
[8] Wang J P,Dobrowolski M P,Cogan N O I,Forster J W,Smith K F.Assignment of individual genotypes to specific forage cultivars of perennial ryegrass based on SSR markers.Crop Science,2009,49(1):49-58.
[9] He Q,Li X W,Liang G L,Ji K,Guo Q G,Yuan W M,Zhou G Z,Chen K S,Vande Eric W,Gao Z S.Genetic diversity and identity of Chinese loquat cultivars/accessions (Eriobotryajaponica) using apple SSR markers.Plant Molecular Biology Reporter,2011,29(1):197-208.
[10] Erfani J,Ebadi A,Abdollahi H,Fatahi R.Genetic diversity of some pear cultivars and genotypes using simple sequence repeat (SSR) markers.Plant Molecular Biology Reporter,2012,30(5):1065-1072.
[11] Ren X P,Jiang H F,Yan Z Y,Chen Y N,Zhou X J,Huang L,Lei Y,Huang J Q,Yan L Y,Qi Y.Genetic diversity and population structure of the major peanut (ArachishypogaeaL.)cultivars grown in China by SSR markers.PloS One,2014,9(2):e88091.
[12] Dewey D R.The Genomic System of Classification as a Guide to Intergeneric Hybridization with the Perennial Triticeae.US:Springer,1984:209-279.
[13] 董景实,张素贞.老芒麦的研究.中国草原,1980(2):29-33.
Dong J S,Zhang S Z.The study ofElymussibiricus.Chinese Journal of Grassland,1980(2):29-33.(in Chinese)
[14] Bowden W M.Cytotaxonomy of the species and interspecific hybrids of the genusElymusin Canada and neighboring areas.Canadian Journal of Botany,1964,42(5):547-601.
[15] Dewey D R.Cytogenetics ofElymussibiricusand its hybrids withAgropyrontauri,Elymuscanadensis,andAgropyroncaninum.Botanical Gazette,1974,135(1):80-87.
[16] 王比德.野生老芒麦的引种驯化.中国草原,1986(6):35-37.
Wang B D.Introduction and domestication in wildElymussibiricus.Chinese Journal of Grassland,1986(6):35-37.(in Chinese)
[17] 郭志慧,杨红宇,张成林,孙铭,符开欣,张新全,马啸.利用形态性状以及SSR标记鉴定4个川西北老芒麦品种(系).草业科学,2016,33(9):364-372.
Guo Z H,Yang H Y,Zhang C L,Sun M,Fu K X,Zhang X Q,Ma X.Identification of fourElymussibiricuscultivar (strains) from Northwest Plateau of Sichuan using morphological traits and SSR markers.Pratacultural Science,2016,33(9):364-372. (in Chinese)
[18] Xie W G,Zhao X H,Zhang J Q,Wang Y R,Liu W X.Assessment of genetic diversity of Siberian wild rye (ElymussibiricusL.)germplasms with variation of seed shattering and implication for future genetic improvement.Biochemical Systematics and Ecology,2015,58(58):211-218.
[19] Ma X,Chen S Y,Zhang X Q,Bai S Q,Zhang C B.Assessment of worldwide genetic diversity of Siberian wild rye (ElymussibiricusL.)germplasm based on gliadin analysis.Molecules,2012,17(4):4424-4434.
[20] 雷云霆,赵闫闫,喻凤,李媛,窦全文.利用基因组SSR分子标记对老芒麦品种(种质)鉴别和品种纯度鉴定.草地学报,2015,23(1):151-155.
Lei Y T,Zhao Y Y,Yu F,Li Y,Dou Q W.Cultivars (germplasms) discrimination and seed purity assessment of grassElymussibiricususing genomic SSR markers.Acta Agrestia Sinica,2015,23(1):151-155.(in Chinese)
[21] Doyle P J.DNA Protocols for Plants.Berlin:Springer-Verlag,1991:283-293.
[22] Larson S R,Kellogg E A.Genetic dissection of seed production traits and identification of a major-effect seed retention QTL in hybridLeymus(Triticeae) wildryes.Crop Science,2009,49(1):29-40.
[23] Mott I W,Larson S R,Jones T A,Robins J G,Jensen K B,Peel M D,Gustafson P.A molecular genetic linkage map identifying the St and H subgenomes ofElymus(Poaceae:Triticeae) wheatgrass.Genome,2011,54(10):819-828.
[24] Ghislain M,Zhang D P,Fajardo D,Huamán Z,Hijmans R J.Marker-assisted sampling of the cultivated Andean potatoSolanumphurejacollection using RAPD markers.Genetic Resources and Crop Evolution,1999,46(6):547-555.
[25] Prevost A,Wilkinson M J.A new system of comparing PCR primers applied to ISSR fingerprinting of potato cultivars.Theoretical and Applied Genetics,1999,98(1):107-112.
[26] Yeh F C,Yang R C,Boyle T.POPGENE Version 1.31:Microsoft Window Based Free Ware for Population Genetic Analysis.Edmonton:University of Alberta,1999:11-23.
[27] Mengoni A,Bazzicalupo M.The statistical treatment of data and the Analysis of MOlecular VAriance (AMOVA) in molecular microbial ecology.Annals of Microbiology,2002,52(2):95-102.
[28] Pritchard J K,Stephens M,Donnelly P.STRUCTURE, version 2-3-3,inference of population structure using multilocus genotype data.Genetics,2000,155(2):945-959.
[29] Evanno G,Regnaut S,Goudet J.Detecting the number of clusters of individuals using the software STRUCTURE:A simulation study.Molecular Ecology,2005,14(8):2611-2620.
[30] Rohlf F J.NTSYS-pc:Numerical Taxonomy and Multivariate Analysis System.New York:Exeter Software,1992:6-40.
[31] Yap I V,Nelson R J.Winboot:A Program for Performing Bootstrap Analysis of Binary Data to Determine the Confidence Limits of UPGMA-based Dendrograms.Manila:International Rice Research Institute,1996:14-14.
[32] 张富民,葛颂.群体遗传学研究中的数据处理方法IRAPD数据的AMOVA分析.生物多样性,2002,10(4):438-444.
Zhang F M,Ge S.Data analysis in population genetics Ⅰ analysis of RAPD data with AMOVA.Chinese Biodiversity,2002,10(4):438-444.(in Chinese)
[33] 蒋林峰,张新全,黄琳凯,马啸,罗登,张宗瑜,蒙芬.中国鸭茅主栽品种DNA指纹图谱构建.植物遗传资源学报,2014,15(3):604-614.
Jiang L F,Zhang X Q,Huang L K,Ma X,Luo D,Zhang Z Y,Meng F.Construction of DNA fingerprinting of dominant orchardgrass (Dactylisglomerata) varieties of China.Journal of Plant Genetic Resources,2014,15(3):604-614.(in Chinese)
[34] Weng M L,Liu B,Jin D M,Yang Q K,Zhao G,Ma J H,Xu P,Duan D L,Wang B.Identification of 27Porphyralines (Rhodophyta) by DNA fingerprinting and molecular markers.Journal of Applied Phycology,2005,23(1):251-262.
[35] 王应芬,牟琼,李娟,雷霞,吴佳海.芜菁甘蓝种质资源评价.草业科学,2016,33(6):1200-1205.
Wang Y F,Mou Q,Li J,Lei X,Wu J H.Evaluation ofBrassicanapobrassicagermplasm resources.Pratacultural Science,2016,33(6):1200-1205.(in Chinese)
[36] Babu C,Sundaramoorthi J,Vijayakumar G,Ram S G.Analysis of genetic diversity in Napier Grass (PennisetumpurpureumSchum) as detected by RAPD and ISSR markers.Journal of Plant Biochemistry and Biotechnology,2009,18(2):181-187.
[37] Chen Y,Nelson R L.Relationship between origin and genetic diversity in Chinese soybean germplasm.Crop Science,2005,45(4):1645-1652.
[38] 严学兵,郭玉霞,周禾,王堃.影响披碱草属植物遗传分化和亲缘关系的地理因素分析.植物资源与环境学报,2006,15(4):17-24.
Yan X B,Guo Y X,Zhou H,Wang K.Analysis of geographical conditions affected on genetic variation and relationship among population ofElymus.Journal of Plant Resources and Environment,2006,15(4):17-24.(in Chinese)
[39] 王瑞晶,李培英,张延辉.偃麦草EST-SSR标记开发及应用.草业科学,2016,33(8):1526-1535.
Wang R J,Li P Y,Zhang Y H.Development ofElytrigiarepensEST-SSR markers and its application.Pratacultural Science,2016,33(8):1526-1535.(in Chinese)
[40] Fan Q,Chen S F,Zhou R C,Xiang X,Liao W B,Shi S H.Genetic variation of wild litchi (LitchichinensisSonn subspchinensis) revealed by microsatellites.Conservation Genetics,2011,12(3):753-760.
[41] Sun C Q,Wang X K,Li Z C,Yoshimura A,Iwata N.Comparison of the genetic diversity of common wild rice (OryzarufipogonGriff) and cultivated rice (OsativaL.) using RFLP markers.Theoretical and Applied Genetics,2001,102(1):157-162.
[42] Lam H M,Xu X,Liu X,Chen W B,Yang G H,Wong F L,Li M W,He W M,Qin N,Wang B,Li J,Jian M,Wang J,Shao G H,Wang J,Samuel S M S,Zhang G Y.Resequencing of 31 wild and cultivated soybean genomes identifies patterns of genetic diversity and selection.Nature Genetics,2010,42(12):1053-1059.
[43] Mariac C,Luong V,Kapran I,Mamadou A,Sagnard F,Deu M,Chantereau J,Gerard B,Ndjeunga J,Bezançon G,Pham J L,Vigouroux Y.Diversity of wild and cultivated pearl millet accessions (PennisetumglaucumL R Br) in Niger assessed by microsatellite markers.Theoretical and Applied Genetics,2006,114(1):49-58.
[44] Medini M,Hamza S,Rebai A,Baum M.Analysis of genetic diversity inTunisiandurumwheat cultivars and related wild species by SSR and AFLP markers.Genetic Resources and Crop Evolution,2005,52(1):21-31.
[45] Tokatlidis I S,Xynias I N,Tsialtas J T,Papadopoulos I I.Single-plant selection at ultra-low density to improve stability of a bread wheat cultivar.Crop Science,2006,46(1):90-97.
[46] Dimitriosn V,Anastasioss L,Demetriosg R.Effectiveness of single-plant selection at low density under organic environment:A field study with lentil.Crop Science,2001,51(1):41-51.
[47] 盘朝邦,刘国藩.川西北高原栽培牧草育种研究Ⅲ:老芒麦单株选择系统的性状变异及其育种效果.草业与畜牧,1988(1):28-32.
Pan C B,Liu G F.The breeding study of forage inthe Northwestern Plateau of Sichuan Province Ⅲ:Trait variability and breeding effect of single-plant selection inElymussibiricus.Prataculture and Animal Husbandry,1988(1):28-32.(in Chinese)
[48] Hawkes J G.The importance of wild germplasm in plant breeding.Euphytica,1977,26(3):615-621.
[49] Acostagallegos J A,Kelly J D,Gepts P.Prebreeding in common bean and use of genetic diversity from wild germplasm.Crop Science,2007,47(S3):44-59.
[50] 鄢家俊,白史且,张昌兵,游明鸿.川西北高原野生老芒麦种质资源考察初报.草业与畜牧,2006(12):23-26.
Yan J J,Bai S Q,Zhang C B,You M H.Preliminary investigation report ofElymussibiricusgermplasms resources from the Northwest Plateau of Sichuan Province.Prataculture and Animal Husbandry,2006(12):23-26.(in Chinese)
[51] 周国栋,李志勇,李鸿雁,师文贵,李兴酉,刘磊,韩海波.老芒麦种质资源的研究进展.草业科学,2011,28(11):2026-2031.
Zhou G D,Li Z Y,Li H Y,Shi W G,Li X Y,Liu L,Han H B.Research advances in germplasm resource ofElymussibiricus.Pratacultural Science,2011,28(11):2026-2031.(in Chinese)
[52] 翟夏杰,张蕴薇,黄顶,王堃.中美牧草育种的现状与异同.草业科学,2016,33(6):1213-1221.
Zhai X J,Zhang Y W,Huang D,Wang K.Status,similarities and differences on forage breeding in USA and China.Pratacultural Science,2016,33(6):1213-1221.(in Chinese)
EST-SSRmarkerbasedDNAfingerprintingforcultivaridentificationandgeneticdiversityanalysisofElymussibiricusgermplasminChina
Zhang Jun-chao, Xie Wen-gang, Zhao Xu-hong, Zhang Zong-yu, Zhao Yong-qiang, Wang Yan-rong
(State Key Laboratory of Grassland Agro-ecosystems, Key Laboratory of Grassland Livestock Industry Innovation, Ministry of Agriculture, National Demonstration Center for Experimental Grassland Science Education (Lanzhou University), College of Pastoral Agriculture Science and Technology, Lanzhou University, Lanzhou 730020, Gansu, China)
In this study, EST-SSR markers were used to analyze the genetic diversity of 52Elymussibiricusaccessions, and to construct DNA fingerprinting profiles of sevenE.sibiricuscultivated accessions and cultivars in China. A total of 204 bands were generated from 20 EST-SSR markers with an average of 10.2 bands per marker. Of the total, 176 (86.27%) bands were polymorphic, indicating that the tested materials possessed a high level of polymorphism. The primers Elw404 and Elw195 could distinguish the seven cultivated accessions and cultivarsfrom other wild materials. Analysis of molecular variance showed that genetic variation was greater within geographical regions (73.94%) than between them (26.06%). Structure analysis suggested that the 52 accessions were clustered into five groups; a similar result was obtained using principal coordinate analysis. Compared with cultivars and cultivated accessions,wild accessions[number of polymorphic bands=174, percentage of polymorphic bands (PPB)=85.29%, Shannon’s information index (I)=0.296 6, Nei’s gene diversity (H)=0.179 8, observed number of alleles (Na)=1.852 9] possessed higher genetic diversity, suggesting that wild accessions could be used as important genetic resources for future breeding programs. Furthermore, ex situ and in situ conservation strategies were suggested forE.sibiricusgermplasm. The results of the present study showed that EST-SSR markers provided an efficient means of assessing genetic diversity and identifying cultivars inE.sibiricus.
Elymussibiricus; DNA fingerprinting; EST-SSR markers; genetic and variation; germplasm conservation
Xie Wen-gang E-mail:xiewg@lzu.edu.cn;
10.11829/j.issn.1001-0629.2016-0580
张俊超,谢文刚,赵旭红,张宗瑜,赵永强,王彦荣.利用EST-SSR标记构建中国老芒麦品种DNA指纹图谱及种质遗传多样性.草业科学,2017,34(10):2052-2062.
Zhang J C,Xie W G,Zhao X H,Zhang Z Y,Zhao Y Q,Wang Y R.EST-SSR marker based DNA fingerprinting for cultivar identification and genetic diversity analysis ofElymussibiricusgermplasm in China.Pratacultural Science,2017,34(10):2052-2062.
S543+.9;Q943
A
1001-0629(2017)10-2052-11
2016-11-17
2017-03-13
国家基础研究项目(973项目)(2014CB138704);长江学者与创新团队发展计划项目(IRT-17R50);国家自然科学基金(31302023);中央高校基础研究基金(LZUJBKY-2016-7);草地农业生态系统国家重点实验室开放课题
张俊超(1990-),男,河南驻马店人,在读博士生,主要从事牧草种质资源与育种研究。E-mail:zhangjch16@lzu.edu.cn
谢文刚(1982-),男,四川资中人,副教授,博士,主要从事牧草遗传育种研究。E-mail:xiewg@lzu.edu.cn
王彦荣(1956-),女,吉林大安人,教授,博士,主要从事草类种子及种质资源研究。E-mail:yrwang@lzu.edu.cn
Wang Yan-rong E-mail:yrwang@lzu.edu.cn
(责任编辑 王芳)

