大豆转录因子GmMYB52的克隆、表达及结合功能分析
许 玲王元琮何晓兰 黄益洪 徐照龙 邵宏波张大勇
江苏省农业科学院农业资源与环境研究所 / 盐土农业研究中心, 江苏南京 210014
大豆转录因子GmMYB52的克隆、表达及结合功能分析
许 玲**王元琮**何晓兰 黄益洪 徐照龙 邵宏波*张大勇*
江苏省农业科学院农业资源与环境研究所 / 盐土农业研究中心, 江苏南京 210014
MYB类转录因子在植物的生长发育和逆境响应中有重要的调控作用。依据课题组前期获得的盐胁迫相关的数字表达谱(DGEP)数据, 获得盐胁迫响应显著上调基因GmMYB52, 利用RT-PCR方法从栽培大豆(Williams 82)中克隆该基因片段, 并与已公布的Williams 82基因组数据库序列比对, 该基因与GmMYB52序列一致。生物信息学分析表明, GmMYB52编码区(CDS)全长1083 bp, 编码360个氨基酸。其编码的氨基酸序列具有MYB类转录因子的共同特征, 其距N端110~160氨基酸残基处有MYB结构域; 系统进化树分析表明, 该基因编码的蛋白与GmMYB62、拟南芥AtMYBSt1、苜蓿MtMYB52、水稻OsMYBS3及木豆CcMYB-like protein J的亲缘关系最近; 实时荧光定量PCR结果表明, 大豆根部GmMYB52的转录水平受外界非生物逆境的调控, 用脱落酸(ABA)和低温(4℃)处理后12 h明显上调, 用氯化钠和PEG处理0.5~24.0 h后检测到GmMYB52的转录水平在50%~600%区间内呈现出先上调后下调再上调的趋势。GmMYB52为组成型表达, 在大豆的幼苗期和开花期表达较多, 在成熟期表达相对较低。GmMYB52在茎叶与开花期的花中表达较强, 在根的表达较弱, 而在成熟期的豆荚中几乎不表达。亚细胞定位的结果表明, GmMYB52定位于细胞核, 符合典型转录因子的定位特征。酵母杂交系统检测表明, GmMYB52具有转录激活特征,并且能够与MYB相关顺式作用元件基序相结合。本研究结果表明, GmMYB52编码典型的MYB转录因子, 具有转录激活活性及DNA结合活性, 在大豆中的表达可能与大豆的非生物胁迫和ABA信号转导途径有关, 推测其可能参与了大豆对非生物胁迫的响应。
大豆; GmMYB52; 表达分析; 转录激活功能; 结合功能
盐碱、高温、低温、高盐、干旱是影响植物生长发育的重要环境胁迫因子, 在进化中, 植物产生了较为完善的分子机制, 感受外界环境的变化, 从而调整自身基因的表达, 以应对这些逆境。转录因子与植物的抗逆反应密切相关, 当植物感受到外界环境刺激时, 往往会激发转录因子的表达, 这些被激活的转录因子会结合相应的顺式元件, 进而调控下游基因的表达, 从而调控植物的生长发育和生理状态。对植物转录因子的研究有助于帮助揭示目标植物应对环境胁迫的生理和分子机制。MYB转录因子是一大类功能多样的转录因子, 在各种植物中均有分布[1-2]。第1个在植物中被克隆出来的MYB基因是玉米中的 MYBC1, 它参与花青素的合成[3]。很多研究表明 MYB类转录因子与植物响应非生物逆境胁迫密切相关, 例如, 拟南芥中的AtMYB15负调控拟南芥的抗寒性, 可以与 ICE1互作来调控下游CBF基因的表达[4], AtMYB96参与了由ABA和JA介导的应对干旱胁迫的响应[5]; AtMYB2可以在干旱胁迫下作为转录激活因子激活脱落酸(ABA)诱导基因的表达[6], AtMYB30可以通过与油菜素内酯(BR)途径中的关键基因BES1协同调控BR信号通路中的基因表达来响应非生物胁迫[7]。甘蔗(Saccharum officinarum)中的ScMYBAS1在干旱与高盐胁迫下会大幅度上调表达[8-9], 水稻(Oryza sativa)中的OsMYB2则参与了对高盐、低温和干旱胁迫的响应[10]。
近年来生态环境的不断恶化, 给大豆的生产造成了巨大的损失。大豆在其生长过程中, 会遇到来自环境中的各种非生物胁迫, 如盐碱、高盐、低温、干旱等。因此, 寻找合适的参与逆境调控的基因可以为选育优良大豆品种提供良好的遗传基础。鉴定和分析大豆中的 MYB类转录因子也是研究的热点之一。依据MYB结构域的重复数, 含有MYB结构域的蛋白主要被分为4大类, 分别命名为1R (R1/2, R3-MYB)、2R (R2R3-MYB)、3R (R1R2R3-MYB)和4R (含有4个R1/R2-like重复)。在大豆中, 目前已经分析预测出127个1R-MYB、244个2R-MYB和 6个3R-MYB[11]。很多MYB类转录因子都参与了逆境胁迫的响应, Liao等[12]在之前鉴定出的 156个编码MYB类转录因子的基因中, 发现43个的表达会受到ABA处理, 高盐, 干旱以及低温的影响。在拟南芥中过表达 GmMYB76、GmMYB92和GmMYB177可以让植株不同程度获得对这些逆境的抗性。杜海等[13]在大豆中克隆出了 GmMYBJ6和GmMYBJ7, 并发现这2个基因的表达都受到ABA和NAA的诱导。GmMYBJ7的表达在高盐、干旱和紫外照射的处理下会明显降低, 过表达 GmMYBJ7的烟草中, 类黄酮的含量明显减少, 推测可能该基因是通过调控类黄酮的量参与植物的生理代谢[14]。R2R3-MYB类转录因子GmMYB12B2受NaCl与紫外线(UV)的诱导, 在拟南芥中过表达 GmMYB12B2不但可以增强拟南芥对高盐和 UV的耐受能力, 而且增加了拟南芥的类黄酮含量, 类黄酮合成相关基因的表达与野生型对照相比均上调[15-16]。杨文杰等[17]在大豆中分离并鉴定了GmMYBZ1和GmMYBZ2, 并发现GmMYBZ2也可能参与类黄酮的调控。魏麦玲等[18]分离克隆出 GmMYB174, 并发现其表达量在ABA和高盐处理下下调, 在干旱处理下上调。许玲等[19]从栽培大豆(Williams)中克隆到R2R3类型的转录因子GmMYB111, 其表达量受到ABA、高盐、干旱和低温的诱导。除了参与大豆对非生物逆境的响应和大豆类黄酮的调控, MYB类转录因子还直接参与调控大豆的生长发育, 例如GmMYB-G20-1参与调控大豆的花瓣颜色, 在病毒诱导沉默GmMYB-G20-1的大豆植株中, 大豆的花瓣颜色由原来的紫色变为部分或全部灰蓝色[20]。MYB类转录因子在植物中参与了包括胚形态发生、次生代谢、开花结果、植物激素信号转导、生物逆境、非生物逆境响应等过程[2,21]。尽管对MYB类转录因子在植物的生长发育和逆境响应过程中的作用已有较多研究, 但是目前尚未有针对大豆GmMYB52的相关报道。本研究在挖掘之前发表的大豆表达谱数据信息时发现GmMYB52在盐处理前后表达差异明显, 因此克隆出了MYB类转录因子 GmMYB52, 对该基因进行了生物信息学和表达模式分析, 鉴定了该基因的亚细胞定位并验证了其转录激活活性和结合功能。这将为进一步研究该基因在大豆逆境胁迫中的调控作用奠定基础。
1 材料与方法
1.1 植物材料、菌株和试剂
本实验所用大豆为Williams 82。用于载体构建的菌株 DH5α为江苏省农业科学院盐土农业研究中心实验室保存, 所用酵母Y187购于Clonetech公司。试验所需的引物为南京金斯瑞公司合成。用于PCR扩增的酶和试剂购自东洋坊(Toyobo)公司, 用于载体构建的内切酶、连接酶, 实时荧光定量PCR的试剂, DNA Marker, 反转录酶等其他试剂购自宝生物(TaKaRa)公司。质粒提取, 胶回收试剂盒为捷瑞生物技术公司生产。
1.2 总RNA提取与cDNA合成
分别用100 μmol L–1ABA、200 mmol L–1NaCl、4℃低温和20% PEG-6000处理大豆的三叶期幼苗。在处理后0、0.5、1、3、6、12和24 h取样, 液氮冻存, 用于逆境处理不同时间段的表达分析。取大豆幼苗期的根、茎、叶, 开花期的根、茎、叶、花以及成熟期的根、茎、叶、豆荚, 液氮冻存, 用于GmMYB52的时空表达分析。采用 Promega公司的RNA提取试剂盒提取总RNA, 经DNA酶(DNase I) 37℃消化 20 min后用 1.2%琼脂糖凝胶电泳鉴定提取质量。按照购自TaKaRa公司的RNA反转录试剂盒说明完成模板cDNA的合成。
1.3 基因克隆
根据之前发表的盐处理数字表达谱(DGEP)数据寻找处理前后表达差异明显的基因, 挖掘到GmMYB52[22]。由此设计引物, 正向引物为5′-ATGACT CGGCGCTGCTCCCACTGCA-3′, 反向引物为5′-TCA GACAGCTTGAATTGCGTTGTCC-3′, PCR体系含, cDNA 2 μL、10×PCR缓冲液5 μL、2.5 mmol L–1dNTPs 4 μL、2 mmol L–1MgSO43 μL, 10 μmol L–1上下游引物各1.5 μL、KOD DNA聚合酶1 μL, ddH2O补足至50 μL。PCR程序为98℃ 3 min; 98℃ 5 s, 55℃ 10 s, 68℃ 1min, 35个循环; 暂停PCR程序, 加入1 μL KOD DNA聚合酶, 68℃ 10 min。电泳检测将PCR产物送交南京金斯瑞生物科技有限公司测序, 获得目的基因序列。
1.4 序列信息分析
利用http://www.soybean.org/网站提供的数据库分析核实基因序列, 利用 http://www.ncbi.nlm.nih. gov/网站的 BlastP程序分析目的蛋白的结构域和预测保守氨基酸。采用 Invitrogen公司的 Vector NTI软件进行多重比对和系统进化树的构建。
1.5 基因表达分析
根据 GmMYB52序列, 设计适用于实时荧光定量PCR的引物。F: 5′-GAAGCACAGGTACGTGAT AGAG-3′, R: 5′-GAACTACCAACCGGGACATT-3′,选取大豆看家基因(Glycine max actin-1-like, GmActin登录号为XM_003552652)为内参基因(F: 5′-CGG TGGTTCTATCTTGGCATC-3′, R: 5′-GTCTTTCGCT TCAATAACCCTA-3′)。采用从TaKaRa公司购买的SYBR Green Super Mix, 参照Roche 2.0 Real-time PCR Detection System设计反应程序, 所有操作均按说明书进行, 试验重复3次。
1.6 GmMYB52的亚细胞定位分析
根据GmMYB52最长的开放阅读框设计特异性引物, F: 5′-TGCTCTAGA ATGACTCGGCGCTGCTCC CACTGCA-3′和R: 5′-CGCGGATCC GACAGCTTGA ATTGCGTTGTCC-3′ (下画线处分别为Xba I和BamH I酶切位点), 采用东洋坊公司的KOD高保真酶扩增获得GmMYB52全长ORF (去除终止密码子TGA), 与pJIT166-35S-EGFP载体连接构建pJIT166-GmMYB52-GFP融合表达载体, 在 PEG 介导下将 pJIT166-GmMYB52-GFP质粒转入拟南芥原生质体, 25℃培养24 h, 采用激光共聚焦显微镜(ZEISS LSM-510META, Germany)观察GFP信号, 确定其亚细胞定位。
1.7 转录激活功能验证分析
根据 GmMYB52的编码区(CDS)设计特异性引物, F: 5′-TCCCCCGGG ATGACTCGGCGCTGCTC CCACTGCA-3′, R: 5′-CGCGGATCC TCAGACAGCT TGAATTGCGTTGTCC-3′ (下画线处分别为Sma I和BamH I酶切位点), 扩增GmMYB52的ORF连入酵母表达载体pGBKT7, 构建与BD (binding domain)翻译融合的效应重组质粒 pGBKT7-GmMYB52, 参考Clonetech公司的酵母转化体系说明书, 经测序验证后用乙酸锂沉淀法转入酿酒酵母Y187, 同时转入响应的空载体为对照。均匀涂于SD/–Trp单缺陷平板上, 结合菌落PCR 筛选阳性转化子; 将筛选出的阳性转化子接入 YPDA培养基摇菌培养 2~3 d后,均匀涂在 SD/–Trp/–His/–Ade三缺陷平板上划板培养, 观察转化子的生长情况, 再挑选转化子涂于SD/–Trp/–X-gal显色平板, 进行 β-半乳糖苷酶活性检测, 并照相记录。
1.8 DNA结合功能分析
将GmMYB52的编码区与酵母表达载体pGADT7连接, 构建重组质粒 pGADT7-GmMYB52。参照之前的报道设计若干MYB转录因子基序序列[12,17], 通过引物设计将序列连接到酵母载体 pHIS2.1, 构建含有目标基序的重组质粒。测序验证后用乙酸锂沉淀法将2个重组质粒同时转入酿酒酵母Y187, 以转入空载体为阴性对照, 转入带有 P53的重组质粒为阳性对照, 然后均匀涂于 SD/–Trp/–His双缺陷平板上, 结合菌落PCR筛选转化子, 以YPDA培养基摇菌培养 2~3 d后, 在加有 80 mmol L–1的 3-AT的SD/–Trp/–Leu/–His三缺陷平板上画线培养, 观察转化子的生长情况, 并照相记录。
1.9 统计学分析
采用 t测验(t-test)测定数据间的差异显著性, P值小于等于 0.05 (P ≤ 0.05)为差异显著, P值小于0.01 (P ≤ 0.01)为差异极显著。方差分析使用SPSS软件, 采用最小显著差数(LSD)法进行多重比对, P值小于等于 0.05 (P ≤ 0.05)为差异显著。试验重复至少3次。
2 结果与分析
2.1 GmMYB52的克隆和序列分析
以栽培大豆的cDNA为模板, 利用RT-PCR技术扩增出 GmMYB52的开放阅读框(Open Reading Frame, ORF)。将获得序列与公布的Williams 82数据库比对显示, 编码 GmMYB52的基因序列位于大豆的第15条染色体。其mRNA全长为1367 nt, 编码区(CDS)为1083 bp。编码区内有2个内含子, 分别长1426 bp和216 bp (图1)。

图1 GmMYB52结构图Fig. 1 Gene structure of GmMYB52
GmMYB52编码蛋白共有 360个氨基酸, 与NCBI的数据库比对显示, 该蛋白与大豆蛋白GmMYB62, 木豆蛋白 MYB-like Protein J, 苜蓿蛋白 MtMYB52, 拟南芥蛋白 AtMYBSt1, 水稻蛋白OsMYBS3相似度较高, 其相似性分别为91%、82%、75%、56%和50%。生物信息学分析显示, 它们的N端具有一个保守的锌指结构域(图2中标*号部分), 位于GmMYB52的R4-P20; 中间都有R1一个MYB结构域(图2中下画线部分), 位于GmMYB52的P116-Y159。GmMYB52属于第一类MYB蛋白(1R-MYB)。
利用软件对相关MYB类蛋白构建系统进化树,表明, GmMYB52与GmMYB62的亲缘关系最近, 与木豆中的 CcMYB-like Protein J、拟南芥中的AtMYBSt1亲缘关系也比较近(图3)。推测这类蛋白有类似的功能。
2.2 GmMYB52的表达分析
分别用高盐、PEG (模拟干旱)、低温(4℃)以及脱落酸(ABA)处理大豆, 并在不同时间段提取根部RNA, 用 Real-time PCR监测 GmMYB52的转录水平。结果显示, 100 μmol L–1ABA处理和4℃处理, 会使得GmMYB52转录水平在处理后0.5 h开始上调,在处理后12 h达到最高峰(图4)。用200 mmol L–1的NaCl和20% PEG处理时, GmMYB52的转录水平在处理0.5~1.0 h后呈现出先上调再下调的振荡模式,其中用NaCl处理后在0.5 h发生微弱下调, 在6 h上调约6倍, 12 h明显下调, 24 h又明显上调(图4);用20% PEG处理在0.5~1.0 h就出现了上调, 随后出现波动升降, 处理后24 h表达量最高, 约为0 h的6倍(图4)。
用 Real-time PCR检测大豆不同生长时期组织的GmMYB52的RNA, 显示转录水平在幼苗期和开花期相对较高, 在成熟期相对较低。GmMYB52在茎、叶、花中有较高的表达, 在根中表达相对较低,在豆荚和成熟期的根中几乎不表达(图5)。
2.3 GmMYB52定位于细胞核部位
构建 pJIT166-GmMYB52-GFP重组质粒, 通过PEG介导的方法转入拟南芥原生质体。在25℃条件下培养 24 h, 在激光共聚焦显微镜下观察。显示, pJIT166-GFP空载体可以在细胞核、细胞质以及细胞膜中检测到 GFP的荧光信号, 而 pJIT166-GmMYB52-GFP的信号几乎都集中在细胞核中(图6), 符合转录因子的典型特征。

图2 GmMYB52与其他MYB类同源蛋白序列多重比对Fig. 2 Multi-alignment of GmMYB52 with other MYB type proteins
2.4 GmMYB52具有转录激活功能
构建具有GAL4 DNA 结合结构域的pGBKT7-GmMYB52酵母表达重组质粒, 分别将 pGBKT7-GmMYB52、pGBKT7 (阴性对照)、pGBKT7-P53 (阳性对照)转入酵母菌株Y187中。三者在酵母单缺培养基 S D (–T r p)中均能够正常生长。转入pGBKT7-P53和pGBKT7-GmMYB52的酵母菌株可以在二缺培养基 SD (–Trp, –His)中生长, 阴性对照转入pGBKT7的酵母菌株无法在二缺培养基中生长,说明效应基因都能够正常表达, 激活了下游的 his3报告基因, 使其能够在His缺陷培养基中正常生长。将含有这3种转化子的菌株在SD (–Trp/–X-Gal)进行显色反应, 转入pGBKT7-P53和pGBKT7-GmMYB52的酵母菌株显蓝色, 转入空载体pGBKT7的酵母菌株不显色(图7)。以上结果表明, GmMYB52具有转录激活活性。

图3 GmMYB52和其他植物MYB蛋白的系统进化树Fig. 3 Phylogenetic tree of GmMYB52 and MYBs from other plants
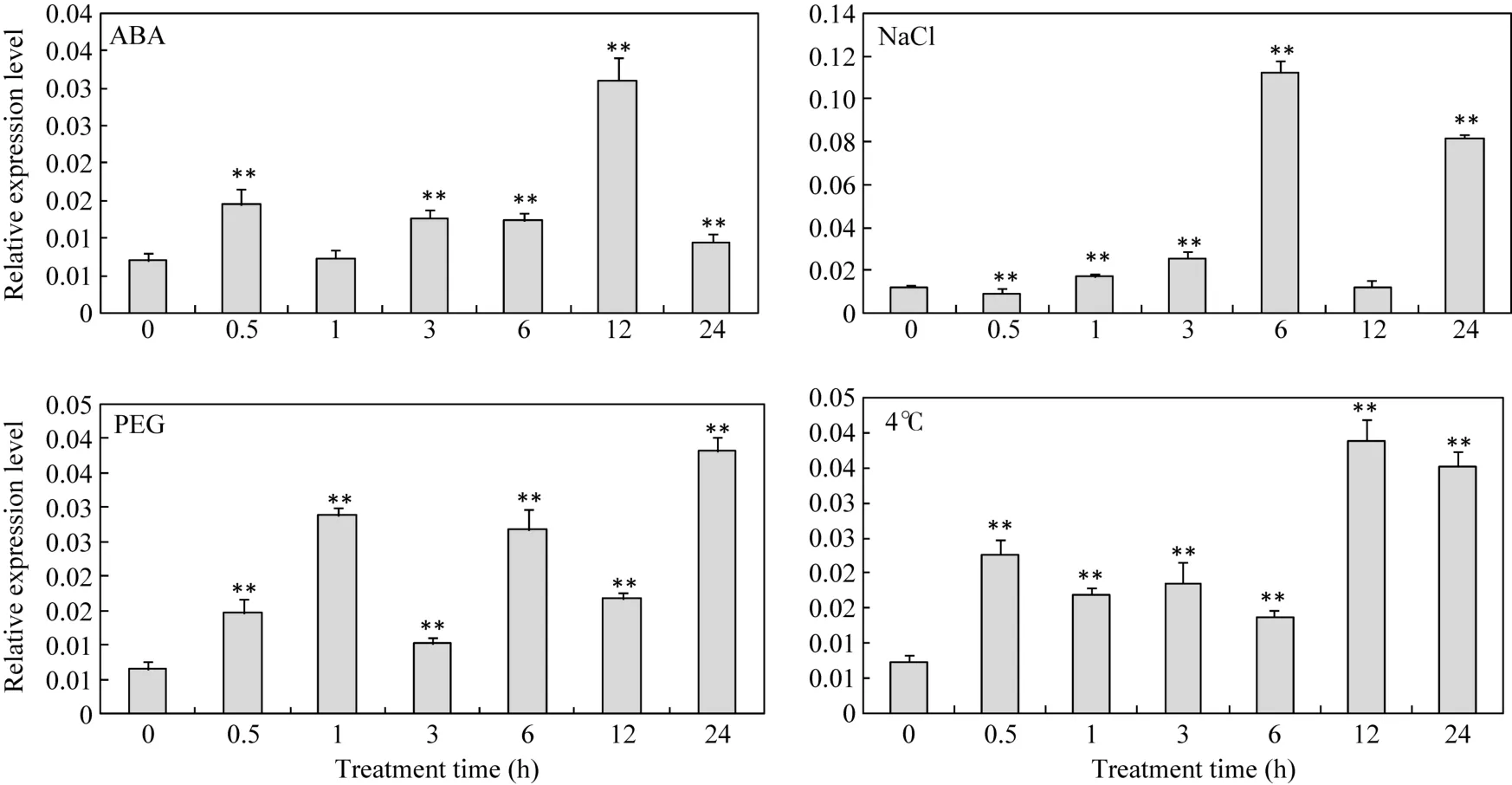
图4 GmMYB52在不同处理下的表达Fig. 4 Expression of GmMYB52 in soybean under different treatments

图5 GmMYB52的时空表达Fig. 5 Spatiotemporal expression of GmMYB52
2.5 GmMYB52可以结合MYB结合基序
为进一步分析GmMYB52可能的结合位点, 将GmMYB52连入 pGADT7载体, 同时选择一系列MYB结合系列序列[12], 连入了pHIS2.1, 将pGADT7-GmMYB52与连入相应序列的pHIS2.1共同转入酵母 Y187中, 检测 His报告基因的表达来分析GmMYB52的结合活性。有趣的是, 我们发现GmMYB52可以激活pHis2.1空载体的His报告基因的表达。因此, 我们将pHis2.1的多克隆位点序列在网站http://jaspar.genereg.net/进行结合位点分析, 发现其多克隆位点(MCS)上含有潜在的 MYB结合基序 AGTGGATCCA (图 8加粗斜体部分)。除了pHIS2.1空载体外, GmMYB52还与mMBSI、MRE4、MYB-motif[12,19]有较强的结合活性。与 MRE1、mMRE1、MRE3、mMRE4也有一定结合活性, 与MBS1、mMRE3没有结合活性(图 8)。以上结果表明, GmMYB52可以与特定的基序相结合, 并调控下游基因的表达。
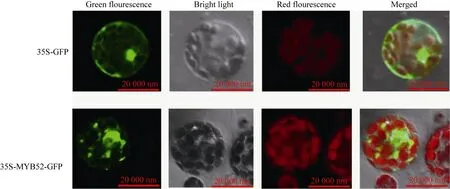
图6 GmMYB52的亚细胞定位Fig. 6 Sub-cellular localization of GmMYB52
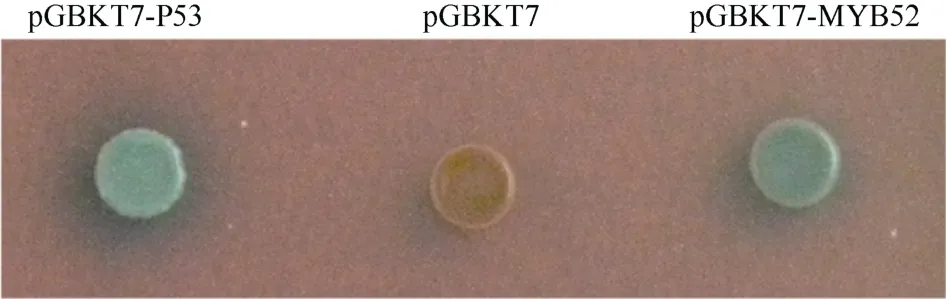
图7 GmMYB52蛋白在酵母单杂交系统中的转录激活活性分析Fig. 7 Assay of transcriptional activation ability of the GmMYB52 protein by yeast expression system
3 讨论
MYB类转录因子作为植物最大的转录因子家族之一, 在植物的生长发育, 激素信号通路, 逆境胁迫响应中都起重要的作用。本研究分离鉴定出的GmMYB52属于MYB转录因子家族当中的1R-MYB,该类 MYB蛋白与之前利用同一表达谱克隆出的2R(R2R3)类 GmMYB111分属于不同家族。一般认为, 1R类家族的基因一般参与胚形态发生、次生代谢途径、生物钟、磷饥饿以及花和果实的发育[21]。2R类MYB是植物MYB家族类最大的一个亚类, 主要参与调节初生和次生代谢, 生长发育以及响应生物和非生物胁迫[2,23-24]。我们的结果显示, 尽管GmMYB52属于1R类MYB转录因子, 仍然能够受到各种非生物逆境的诱导, 但 GmMYB52在逆境胁迫下的表达模式与GmMYB111不尽相同, GmMYB52在ABA的处理下, 并没有表现出GmMYB111的明显振荡模式, 而是先上调后下调。在高盐处理下, GmMYB52和GmMYB111都表现出一定的振荡模式。在干旱处理下, GmMYB52的转录表现出一定的振荡模式, GmMYB111则是先上调后下调, 在低温处理下, GmMYB111和GmMYB52的表达均上调。以上结果暗示了1R类MYB转录因子和2R类MYB转录因子在功能上既有重叠, 也有不同, 且GmMYB52也有可能参与非生物逆境的响应。时空表达模式的结果表明, GmMYB52在幼苗期和开花期的表达较高,在根茎叶花中的表达较高, 暗示 GmMYB52主要参与植株早期的生长发育调控。这与之前发表的GmMYB111表达模式相似[19]。
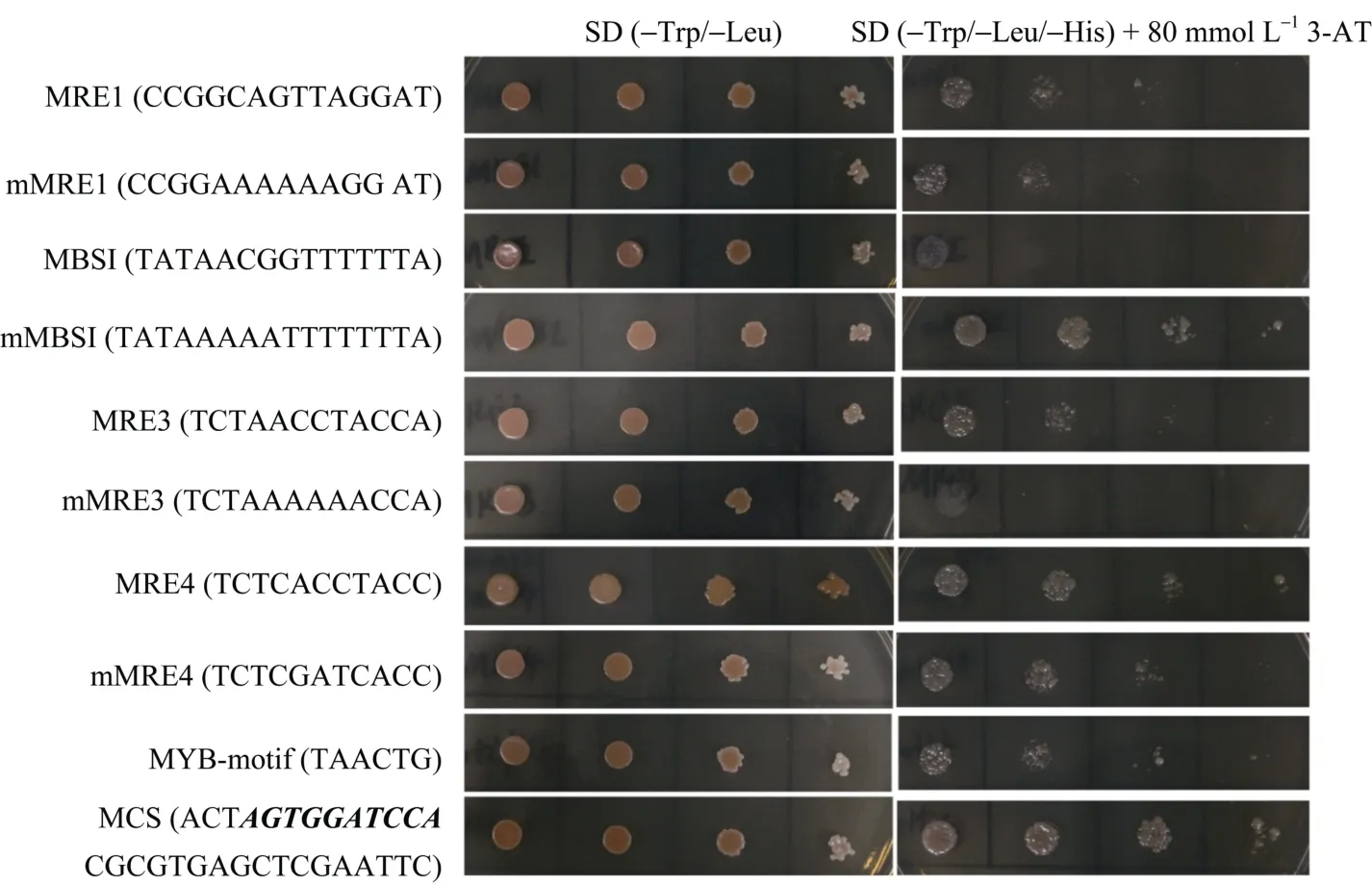
图8 GmMYB52蛋白在酵母单杂交系统中的结合基序分析Fig. 8 Motif sequence binding analysis of GmMYB52 by one-hybrid system
对植物中 MYB转录因子的特定结合基序已经有了较为广泛的研究。一般认为, MYB转录因子均可以结合一个或多个顺式 DNA序列。不同类型的MYB蛋白可以结合不同的基序。拟南芥中的AtMYB2和哺乳动物中的 MYB类转录因子(如c-MYB、A-MYB、B-MYB)都可以结合MBSI基序。大豆中的MYB类转录因子GmMYB76、GmMYB92、GmMYB177也可以与MBSI结合[12,25-26]。在本实验中, GmMYB52与MBSI序列没有结合, 但是与其相应的突变基序mMBSI却有较强的结合活性。这很可能意味着GmMYB52与之前报道的GmMYB76、GmMYB92和GmMYB177调控不同类型的下游基因。除了MBSI和mMRE3以外, GmMYB52与包括空载体多克隆位点在内的 DNA序列均有不同程度的结合, 说明GmMYB52参与调控的下游基因可能比较广泛。
大部分MYB类转录因子都具有转录激活活性,一般来说, 相对于DNA结合域, 转录激活区都在蛋白的中部或 C端, 富含酸性氨基酸[27]。但是相对于N端, C端的序列变化较大, 很可能是因为激活域的蛋白结构较为多变。并非所有的 MYB类转录因子都一定要具备转录激活功能, 也有研究表明, 有一些 MYB类转录因子可能具有转录抑制的功能[25]。本研究显示, GmMYB52具有转录激活功能。与GmMYB52同源性较高的MYB类转录因子C端保守区均富含谷氨酰胺(Q)和脯氨酸(P), 我们推测这个区域在激活下游基因方面起关键作用。
基因组分析显示, 大豆中共有206个MYB类蛋白, 进一步的研究确证了其中的156个[12,28]。这156个MYB蛋白中有55个与拟南芥中的很多MYB蛋白有较多的同源性。我们的结果显示, GmMYB52在拟南芥、水稻、苜蓿中都能找到相似度较高的同源蛋白, 这表明MYB类蛋白在进化中的功能相对保守。
植物在其生长过程中, 需要应对外界的各种非生物胁迫, 转录因子在其中起着非常重要的调控作用。研究显示, 编码156个MYB蛋白的基因中有大约27%受到ABA, 高盐、低温和干旱逆境的诱导[12],说明 MYB类转录因子对植物应答非生物胁迫非常关键。GmMYB52也同样受到这四种常见的非生物胁迫的诱导表达。这说明GmMYB52很可能也参与植物响应这些非生物胁迫的信号通路。对GmMYB52的详细生物学功能仍需要进一步探究。
4 结论
分离和鉴定了大豆中的 MYB类转录因子GmMYB52, 该基因属于 1R类 MYB转录因子。GmMYB52在植物中有组成型表达, 其在根部的表达受ABA、干旱、高盐、低温的诱导。GmMYB52定位于细胞核, 有转录激活活性, 并可以结合若干MYB相关顺式转录原件。推测 GmMYB52可能与ABA信号、非生物胁迫应答和苗期及开花期的生长发育密切相关。
[1] Ambawat S, Sharma P, Yadav N R, Yadav R C. MYB transcription factor genes as regulators for plant responses: an overview. Physiol Mol Biol Plants, 2013, 19: 307–321
[2] Dubos C, Stracke R, Grotewold E, Weisshaar B, Martin C, Lepiniec L. MYB transcription factors in Arabidopsis. Trends Plant Sci, 2010, 15: 573–581
[3] Paz-Ares J, Abe H, Urao T, Ito T, Seki M, Shinozaki K, Yamaguchi-Shinozaki K. The regulatory c1 locus of Zea mays encodes a protein with homology to myb proto-oncogene products and with structural similarities to transcriptional activators. EMBO J, 1987, 6: 3553–3558
[4] Agarwal M, Hao Y, Kapoor A, Dong C H, Fujii H, Zheng X, Zhu J K. A R2R3 type MYB transcription factor is involved in the cold regulation of CBF genes and in acquired freezing tolerance. J Biol Chem, 2006, 281: 37636–37645
[5] Seo P J, Park C M. Auxin homeostasis during lateral root development under drought condition. Plant Signal Behav, 2009. 4: 1002–1004
[6] Abe H, Urao T, Ito T, Seki M, Shinozaki K,Yamaguchi-Shinozaki K. Arabidopsis AtMYC2 (bHLH) and AtMYB2 (MYB) function as transcriptional activators in abscisic acid signaling. Plant Cell, 2003, 15: 63–78
[7] Li L X, Thompson A, Guo M, Yoshida S, Asami T, Chory J, Yin Y. Arabidopsis MYB30 is a direct target of BES1 and cooperates with BES1 to regulate brassinosteroid-induced gene expression. Plant J, 2009, 58: 275–286
[8] Prabu G. Structure of DNA binding MYB transcription factor protein (ScMYBAS1-3) from sugarcane-threading and Ab initio modelling. J Physiol, 2011, 3: 77–82
[9] Prabu G, Prasad D T. Functional characterization of sugarcane MYB transcription factor gene promoter (PScMYBAS1) in response to abiotic stresses and hormones. Plant Cell Rep, 2012, 31: 661–669
[10] Yang A, Dai X, Zhang W H. A R2R3-type MYB gene, OsMYB2, is involved in salt, cold, and dehydration tolerance in rice. J Exp Bot, 2012: 2541–2556
[11] Liu J, Osbourn A, Ma P. MYB transcription factors as regulators of phenylpropanoid metabolism in plants. Mol Plant, 2015, 8: 689–708
[12] Liao Y, Zou H F, Wang H W, Zhang W K, Ma B, Zhang J S, Chen S Y. Soybean GmMYB76, GmMYB92, and GmMYB177 genes confer stress tolerance in transgenic Arabidopsis plants. Cell Res, 2008, 18: 1047–1460
[13] 杜海, 杨文杰, 刘蕾, 唐晓凤, 吴燕民, 黄玉碧, 唐益雄. 大豆MYB转录因子基因GmMYBJ6和GmMYBJ7的克隆及表达分析. 作物学报, 2008, 34: 1179–1187 Du H, Yang W J, Liu L, Tang X F, Wu Y M, Huang Y B, Tang Y X. Cloning and functional identification of the two MYB transcription factors GmMYBJ6 and GmMYBJ7 in soybean. Acta Agron Sin, 2017, 43: 42–50 (in Chinese with English abstract)
[14] 杨文杰, 吴燕民, 唐益雄. 大豆转录因子基因GmMYBJ7的表达及功能分析. 华北农学报, 2012, 27(6): 24–29 Yang W J, Wu Y M, Tang Y X. Expressing and functional analysis of GmMYBJ7 from soybean. Acta Agric Boreali-Sin, 2012, 27(6): 24–29 (in Chinese with English abstract)
[15] Li X, Li J, Zhai Y, Zhao Y, Zhao X, Zhang H, Su L T, Wang Y, Wang Q. A R2R3-MYB transcription factor, GmMYB12B2, affects the expression levels of flavonoid biosynthesis genes encoding key enzymes in transgenic Arabidopsis plants. Gene, 2013, 532: 72–79
[16] Li X, Wang Y, Yan F, Li J, Zhao Y, Zhao X, Zhai Y, Wang Q. Overexpression of soybean R2R3-MYB transcription factor, GmMYB12B2, and tolerance to UV radiation and salt stress in transgenic Arabidopsis. Genet Mol Res, 2016, 15(2): 1–10
[17] 杨文杰, 杜海, 方芳, 杨婉身, 吴燕民, 唐益雄. 大豆两个MYB转录因子基因的克隆及表达分析. 中国农业科学, 2008, 41: 961–970 Yang W J, Du H, Fang F, Yamg W S, Wu Y M, Tang Y X. Cloning and characterization of two new MYB transcription factor genes from soybean. Sci Agric Sin, 2008, 41: 961–970 (in Chinese with English abstract)
[18] 魏麦玲, 裴丽丽, 刘佳明, 闵东红, 陈明, 李连城, 马有志, 徐兆师, 张小红. 大豆MYB转录因子基因GmMYB174的克隆及分子特性分析. 植物遗传资源学报, 2015, 16: 94–99 Wei M L, Pei L L, Liu J M, Min D H, Chen M, Li L C, Ma Y Z, Xu Z S, Zhang X H. Isolation and molecular characteristics analysis of soybean transcription factor gene GmMYB174. J Plant Genet Resour, 2015, 16: 94–99 (in Chinese with English abstract)
[19] 许玲, 卫培培, 张大勇, 徐照龙, 何晓兰, 黄益洪, 马鸿翔, 邵宏波. 大豆转录因子基因 GmMYB111的克隆及功能分析. 中国农业科学, 2015, 48: 3079–3089 Xu L, Wei P P, Zhang D Y, Xu Z L, He X L, Huang Y H, Ma H X, Shao H B. Expression and function analysis of the transcription factor GmMYB111 in soybean. Sci Agric Sin, 2015, 48: 3079–3089 (in Chinese with English abstract)
[20] Takahashi R, Yamagishi N, Yoshikawa N. A MYB transcription factor controls flower color in soybean. J Hered, 2013, 104, 149–153
[21] Feller A, Machemer K, Braun E L, Grotewold E. Evolutionary and comparative analysis of MYB and bHLH plant transcription factors. Plant J, 2011, 66: 94–116
[22] Ali Z, Zhang D Y, Xu Z L, Xu L, Yi J X, He X L, Huang Y H, Liu X Q, Khan A A, Trethowan R M, Ma H X. Uncovering the salt response of soybean by unraveling its wild and cultivated functional genomes using tag sequencing. PLoS One, 2012, 7: e48819
[23] Shelton D, Stranne M, Mikkelsen L, Pakseresht N, Welham T, Hiraka H, Tabata S, Sato S, Paquette S, Wang T L, Martin C, Bailey P. Transcription factors of lotus: regulation of isoflavonoid biosynthesis requires coordinated changes in transcription factor activity. Plant Physiol, 2012, 159: 531–547
[24] Stracke R, Stracke R, Ishihara H, Huep G, Barsch A, Mehrtens F, Niehaus K, Weisshaar B. Differential regulation of closely related R2R3-MYB transcription factors controls flavonol accumulation in different parts of the Arabidopsis thaliana seedling. Plant J, 2007, 50: 660–677
[25] Martin C, Paz-Ares J. MYB transcription factors in plants. Trends Genet, 1997, 13: 67–73
[26] Urao T, Yamaguchi-Shinozaki K, Urao S, Shinozaki K. An Arabidopsis MYB homolog is induced by dehydration stress and its gene product binds to the conserved MYB recognition sequence. Plant Cell, 1993, 5: 1529–1539
[27] Weston K. Myb proteins in life, death and differentiation. Cur Opin Genet Dev, 1998, 8: 76–81
[28] Tian A G, Wang J, Cui P, Han Y J, Xu H, Cong L J, Huang X G, Wang X L, Jiao Y Z, Wang B J, Wang Y J, Zhang J S, Chen S Y. Characterization of soybean genomic features by analysis of its expressed sequence tags. Theor Appl Genet, 2004, 108: 903–913
Isolation, Expression and Binding Function Analysis of the Transcription Factor GmMYB52 in Soybean
XU Ling**, WANG Yuan-Cong**, HE Xiao-Lan, HUANG Yi-Hong, XU Zhao-Long, SHAO Hong-Bo*, and ZHANG Da-Yong*
Institute of Agricultural Resources and Environment, Jiangsu Academy of Agricultural Sciences / Salt-soil Agricultural Research Center, Nanjing 210014, China
The MYB type transcription factors are involved in plant development and response to abiotic stress. GmMYB52 was significantly up-regulated after salt treatment. In order to gain more information about GmMYB52, GmMYB52 of Williams 82 was cloned by RT-PCR. Bioinformatic analysis showed the CDS of GmMYB52 was 1083 bp, encoding 360 amino acid residues. A MYB domain was found in the region of 110 to 160 amino acid residues from N-terminal. Blast results showed that GmMYB52 are highly homologous to GmMYB62, AtMYBSt1 from Arabidopsis, MtMYB52 from Medicago sativa, OsMYBS3 from Oryza sativa, and CcMYB-like protein J from Cajanus cajan. Quantitative PCR (qPCR) results indicated that the transcription level of GmMYB52 was upregulated under ABA and low temperature stresses, under salt, drought and cold stresses, show a bimodalpattern. GmMYB52 was nearly expressed in all detected tissues, except in pods at maturing stage, and its expression level was relatively higher at seedling or flowering stages than those at maturing stage. The transcription level of GmMYB52 was high in stem, leaf, and flower, during seedling and blooming stages, and low in root and pods during maturity stage. Subcellular localization results showed that GmMYB52 was located in the nucleus which is in agreement with the localization characteristics of typical transcription factors. Yeast hybrid assay indicated that GmMYB52 had transcriptional activation functions and could bind to several MYB cis-acting element motifs. In conclusion GmMYB52 is a typical 1R-MYB transcription factor, and able to bind MYB cis-acting element motifs. We speculate GmMYB52 is involved in response to the abiotic stress and ABA signal transduction pathway.
Soybean; GmMYB52; Expression analysis; MYB motif; Transcriptional activation activity
(
): 2017-02-08; Accepted(接受日期): 2017-04-20; Published online(网络出版日期): 2017-05-11.
10.3724/SP.J.1006.2017.01458
本研究由国家自然科学基金项目(31600211, 31101166), 江苏省自然科学基金项目(BK20151364)和江苏省农业科技自主创新项目(CX(15)1005)资助。
This study was supported by the National Natural Science Foundation of China (31600211, 31101166), the Natural Science Foundation of Jiangsu Province, and the Independent Innovation Project for Agricultural Science and Technology of Jiangsu Province [CX(15)1005].
*通讯作者(Corresponding authors): 张大勇, E-mail: cotton.z@126.com, Tel: 025-84391105; 邵宏波, E-mail: shaohongbochu@126.com
**同等贡献(Contributed equally to this work)
联系方式: E-mail: xuling@jaas.ac.cn, Tel: 025-84390351
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20170511.1152.006.html

