一种基于液相基因芯片技术对甲型和乙型流感病毒分型检测方法的建立
陈锦龙 吕刚,2 张优 符瑞佳 尹飞飞,2★
一种基于液相基因芯片技术对甲型和乙型流感病毒分型检测方法的建立
陈锦龙1吕刚1,2张优1符瑞佳1尹飞飞1,2★
目的建立能够对甲型流感病毒(influenza virus A,INFA)和乙型流感病毒(influenza virus B,INFB)进行区分的液相基因芯片技术,为甲型和乙型流感病毒的分型提供一种高通量、高灵敏度的检测和鉴定方法。方法从NCBI的GenBank下载具有代表性的流感病毒基因组序列,利用生物信息学软件BioEdit进行序列比对,分别设计针对INFA和INFB的简并引物和特异性探针,将设计好的引物和探针与GenBank中所有的基因序列进行比对,以验证其特异性。经探针合成、悬浮液相芯片制备和检测条件的优化获得用于甲型和乙型流感病毒分型的液相基因芯片。结果所检测INFA和INFB均可获得特异性杂交信号,非特异性杂交信号不明显,将2种病毒混合,模拟混合病毒感染也可以得到相应的特异性杂交信号。灵敏度评估结果表明INFA的检测灵敏度为1~10空斑形成单位(plaque forming unit,pfu),而INFB为10~100 pfu。将本方法用于检测11份临床样本,其结果与荧光定量分型逆转录聚合酶链反应(reverse transcriptase⁃polymerase chain reaction,RT⁃PCR)检测方法的结果一致。结论本研究初步建立了INFA和INFB的液相基因芯片检测技术,可用于流感病毒的初步筛查和鉴定,并且可以进行混合感染样本的检测,为流感病毒的分型和鉴定提供了一种新的手段。
甲型流感病毒;乙型流感病毒;液相基因芯片;病毒分型检测
流感病毒的感染是世界范围内的重大公共卫生问题。根据核衣壳蛋白和基质蛋白的同源性,流感病毒分为甲、乙和丙型[1⁃2]。甲型流感病毒(influenza virus A,INFA)抗原性易发生变异,可以进一步分为 H1N1、H3N2、H5N1、H7N9等亚型,多次引起世界范围的大流行。乙型流感病毒(influ⁃enza virus B,INFB)只在局部引起小范围的流行,丙型流感病毒只引起人类不明显的或轻微的上呼吸道感染,很少造成流行[3]。准确有效的流感病毒检测和分型方法对于临床采取及时有效的治疗方式和控制流感的传播具有重要的意义。但是目前对于流感病毒的检测临床上主要采用实时荧光定量PCR,其缺点为检测靶标单一,对多靶标检测易出现假阳性结果[4⁃5]。而液相基因芯片作为一种新兴检测技术,利用多标志物并行检测从而提高了检测通量[6]。本研究建立了一种能够对甲型和乙型流感病毒进行检测和分型的液相基因芯片技术,为流感病毒的筛查和流行病学本底调查提供重要的技术手段。
1 材料与方法
1.1 材料
1.1.1 细胞株与病毒株
本检测所用流感病毒株包括甲型流感病毒H1N1(A/PR/8/34,ATCC⁃VR95)和 H3N2(A/Victo⁃ria/210/2009型),乙型流感病毒(B/Lee/40,ATCC⁃VR101)。所有流感病毒在Vero⁃E6细胞系中进行增殖培养。病毒的增殖于中科院武汉病毒研究所BSL⁃2生物安全实验室进行。
1.1.2 所需试剂
M⁃MLV反转录酶购自Promega公司(美国),Taq酶购自宝生物工程(大连)有限公司,报告分子Streptavidin⁃phycoerythrin购于Molecular Probes公司(美国),杂交缓冲液TMAC和微球购自Luminex公司(美国)。病毒RNA提取试剂盒QIAamp viral RNAmini kit购自Qiagen公司(美国)。
1.2 方法
1.2.1 病毒的增殖培养和核酸提取
将病毒接种于Vero⁃E6细胞系,在含5%FBS的DMEM培养基中置于37℃、5%CO2培养箱中进行增殖培养,每日在显微镜下观察细胞病变效应(cytopathic effect,CPE)情况,当 90%以上细胞出现CPE时,收集病毒培养液,分装后-80℃保存备用。根据病毒RNA提取试剂盒的说明书提取病毒核酸。
1.2.2 简并引物和特异性探针的设计合成
在GenBank下载具有代表性的甲型流感病毒和乙型流感病毒全基因组序列各100条以上,利用BioEdit(版本7.0)软件进行序列同源性比对分析,在此基础上设计简并引物,并在简并引物的范围内设计特异性探针。初步设计的引物和探针经过Blast比对分析进行进一步的优化设计。正向引物进行生物素(Biotin)修饰,探针进行氨基化和C12修饰。所有引物和探针由上海英骏生物技术有限公司(中国)合成。
1.2.3 芯片制备
根据Luminex公司的说明书,将检测探针通过共价交联的方式偶联在微球上。4种检测探针分别偶联到不同编号的微球上,以利于多重检测。
1.2.4 病毒RT⁃PCR扩增及扩增产物的检测
按照M⁃MLV反转录酶试剂盒说明书进行反转录获得cDNA模板,然后进行PCR扩增,PCR反应体系组成如下:cDNA 模板 5μL,20 μmol/L 上下游引物各 1μL,10×PCR 缓冲液 5μL,10 mol/L dNTP 1μL,加水补至 50μL。PCR 反应条件:94℃预变性5min;94℃变性30 s,57℃复性30 s,72℃扩增30 s扩增,共30个循环;最后72℃延伸10min,降温至4℃。取5μL扩增产物至96孔PCR管,向管内加入用1.5×TMAC杂交液稀释的4种微球至终浓度150 个/μL,加入12μL TE,空白对照管加入 17μL TE,混匀后于 95℃变性 5min、55℃杂交15min,加入报告分子55℃继续孵育5min后用Luminex 200系统检测。
1.2.5 敏感性评估
将H1N1、H3N2和INFB梯度稀释,获得100000、10 000、1 000、100和10 pfu/mL的病毒浓度,各取0.5 mL提取病毒RNA,反转录后取1/5做模板进行液相基因芯片检测,即各检测模板量为10 000、1 000、100、10和1 pfu。
1.2.6 特异性和重复性检测
所有样本分别进行3次核酸提取、PCR扩增和液相芯片检测,以检测芯片的重复性。通过DNA琼脂糖凝胶电泳检测每对引物的PCR产物的条带是否为特异性。
1.2.7 模拟混合病毒感染的检测
将1 000 pfu的H1N1、H3N2和INFB病毒溶液两两组合,模拟H1N1和H3N2、H1N1和INFB、H3N2和INFB混合感染的样本,分别提取RNA并反转录,对模板进行液相基因芯片检测。
1.2.8 检测方法的初步应用
对本实验室收集和保存的11份临床样本,用本文建立的液相基因芯片方法进行检测,同时以H1N1、H3N2和INFB病毒培养液做为阳性对照,并与荧光定量分型RT⁃PCR方法(上海之江生物科技股份有限公司)进行比较,评价本方法的可靠性及临床实用性。
2 结果
2.1 引物的设计合成及特异性检测
针对流感病毒型特异性基因M基因分别设计针对流感病毒甲型和乙型的引物和探针[4⁃5],所设计合成的引物和探针序列如表1。为便于液相基因芯片的优化,针对甲型和乙型流感病毒分别设计2组引物和探针,进行后继系列评价比较。病毒RNA提取后,进行反转录获得cDNA模板,利用设计合成的引物进行PCR扩增,结果如图1所示,引物对INFA⁃F1/INFA⁃R1、INFA⁃F2/INFA⁃R2、INFB⁃F1/INFB⁃R1和 INFB⁃F2/INFB⁃R2分别扩增出304 bp、359 bp、453 bp和300 bp的特异性条带且无明显非特异性条带产生。

表1 用于甲型和乙型流感病毒分型的引物和探针Table 1 The primers and probes used for the typing of INFA and INFB
2.2 不同液相基因芯片检测效果的比较
分别利用各引物和探针进行PCR和液相基因芯片检测,比较不同的液相基因芯片的检测结果。结果如表2所示,利用引物对INFA⁃F1/INFA⁃R1进行PCR并用探针INFA⁃P1制备液相基因芯片检测,阳性样本H1N1和H3N2的平均荧光值分别为890和785,阴性样本乙型流感病毒平均荧光值为150,阴性对照结果为28;利用引物对INFA⁃F2/IN⁃FA⁃R2进行PCR并用探针INFA⁃P2制备液相基因芯片检测,阳性样本H1N1和H3N2的平均荧光值分别为1 800和1 980,阴性样本乙型流感病毒平均荧光值为98,阴性对照结果为43。2种液相基因芯片比较,引物对INFA⁃F2/INFA⁃R2和探针INFA⁃P2制备液相基因芯片效果较好。利用引物对INFB⁃F1/INFB⁃R1进行PCR 并用探针 INFB⁃P1制备液相基因芯片检测,阳性样本乙型流感病毒平均荧光值为1 540,阴性样本H1N1和H3N2的平均荧光值分别为91和87,阴性对照结果为59;利用引物对INFB⁃F2/INFB⁃R2进行PCR并用探针INFB⁃P2制备液相基因芯片检测,阳性样本INFB平均荧光值为687,阴性样本H1N1和H3N2的平均荧光值分别为149和205,阴性对照结果为60。2种液相基因芯片比较,引物对INFB⁃F1/INFB⁃R1和探针INFB⁃P1制备液相基因芯片效果较好。
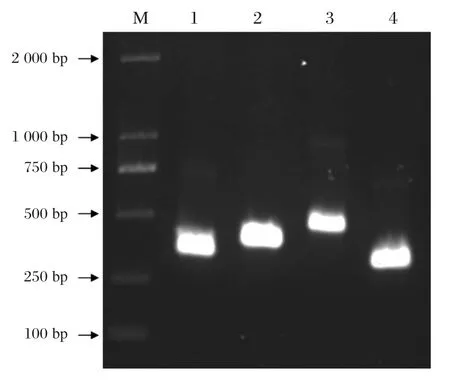
图1 引物特异性检测Figure 1 The specificity test of the primers
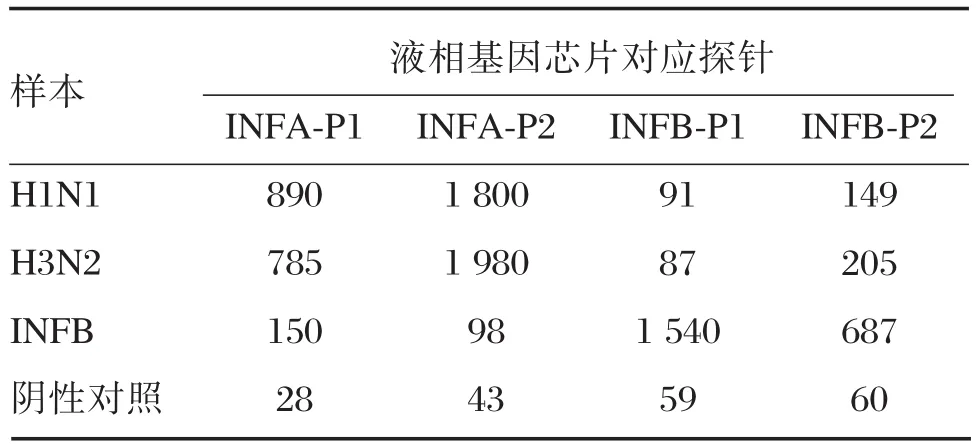
表2 不同液相基因芯片平均荧光值的比较Table 2 The comparison of the median fluorescence intensity of different microbead⁃based liquid assay
2.3 液相基因芯片检测的敏感性的评估
分别对引物对INFA⁃F2/INFA⁃R2和探针INFA⁃P2制备的液相基因芯片和引物对INFB⁃F1/INFB⁃R1和探针INFB⁃P1制备的液相基因芯片进行敏感性评估,结果如图2所示,INFA的液相基因芯片检测灵敏度为1~10空斑形成单位(plaque forming unit,pfu),而INFB的液相基因芯片检测灵敏度为10~100 pfu。

图2 液相基因芯片检测敏感性评估Figure 2 Evaluation of the sensitivity of the assay
2.4 液相基因芯片检测的特异性和重复性
所有样本分别进行3次核酸提取、PCR扩增和液相基因芯片检测,各样本所用病毒量均为100 pfu,结果如图3所示,3次检测实验的定性结果完全正确,而且探针间也没有交叉反应,表明该方法具有良好的特异性和可重复性。
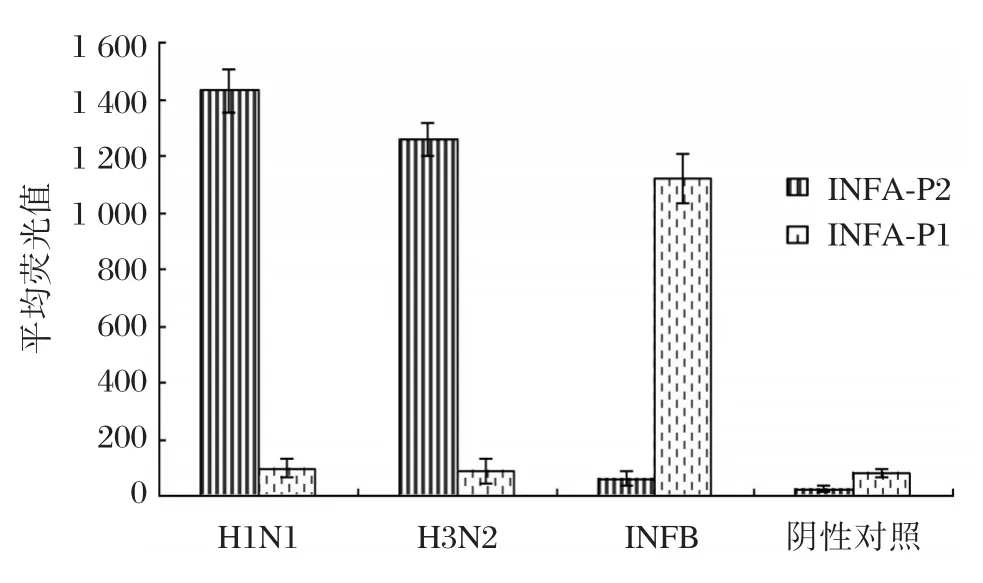
图3 液相基因芯片特异性和重复性检测结果Figure 3 Results of the specificity and reproducibility test of the microbead⁃based liquid assay
2.5 模拟混合病毒感染的检测评估
将H1N1、H3N2和INFB病毒溶液两两组合模拟混合病毒感染,对样本进行液相基因芯片检测,结果如图4所示,2种INFA病毒、H1N1和INFB、H3N2和INFB分别混合检测,都能特异性检测到相应样本,说明本检测方法能够进行混合种类病毒感染的检测。
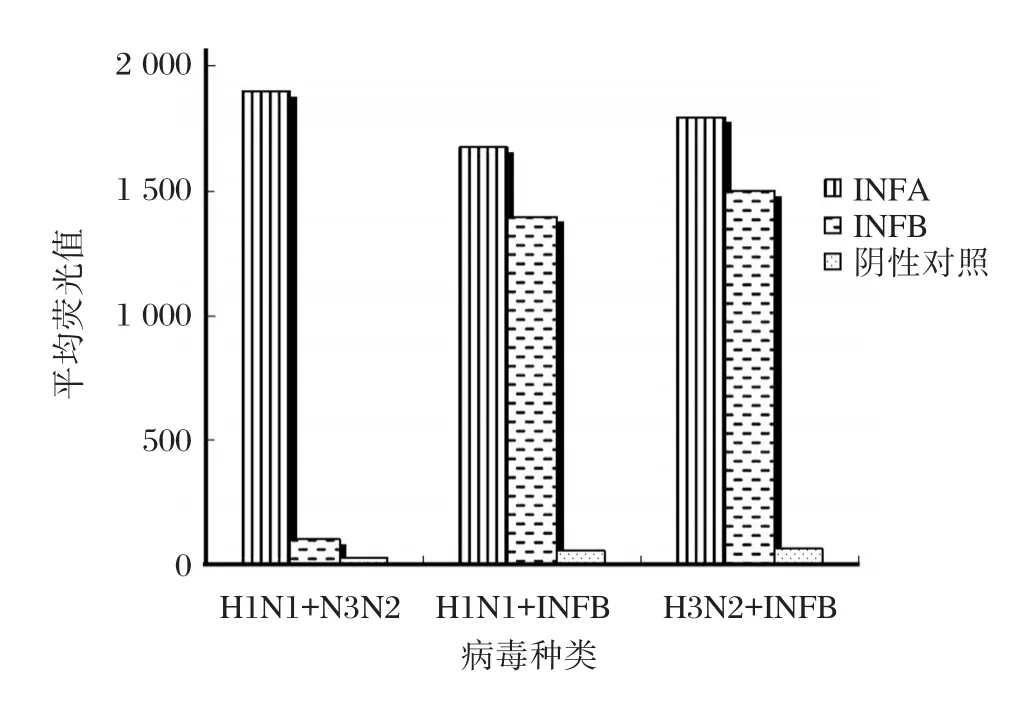
图4 模拟混合病毒检测结果Figure 4 The results of the detection of mixed viruses
2.6 临床样本的检测结果
检测的11份临床样本中,10份为INFA,1份为INFB,H1N1和H3N2病毒培养液检测结果为INFA,乙型流感病毒培养液检测结果为INFB,阴性对照检测结果为无病毒。其结果与荧光定量分型RT⁃PCR方法的结果一致,见表3。
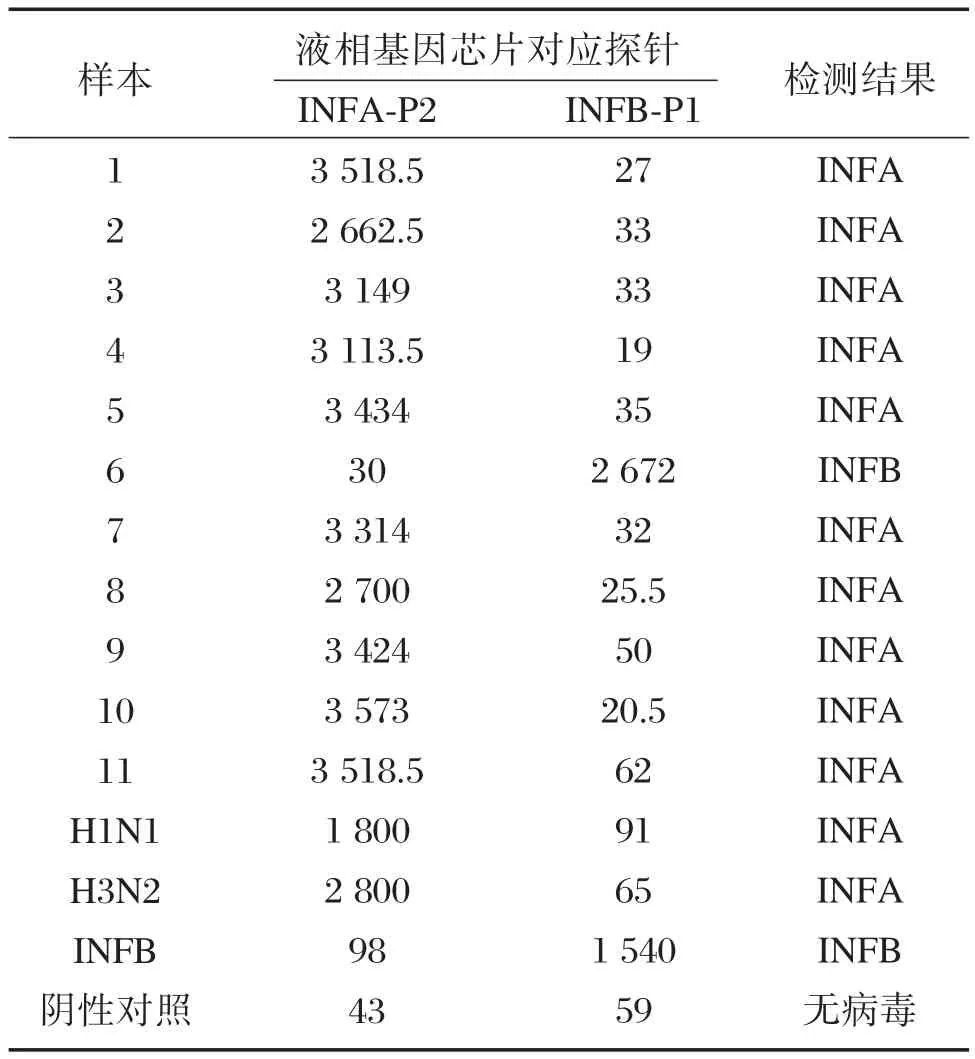
表3 临床样本检测结果Table 3 The results of clinical samples
3 讨论
由流感病毒引起的流感是一种世界范围内的传染性疾病,具有传染性强、传播快、潜伏期短、发病率高等特点,对公共卫生和人们身体健康带来重大威胁。流感的主要表现症状为高热、肌痛、咳嗽、流涕等,可伴有严重的肺炎,甚至可能引起心脏、肾脏等多种重要器官衰竭而导致死亡。对人类危害巨大的季节性流感主要是由甲型流感病毒引起,而乙型流感病毒只在局部引起小范围的流行,由于各型流感病毒所致疾病的治疗和防控策略不同,建立流感病毒分型的快速检测方法,对于流感的早期诊断和制定有效防治策略有重要意义[7]。本研究针对甲型和乙型流感病毒的保守基因M基因的核苷酸序列设计特异性引物和探针,用于甲型和乙型流感病毒的分型,经过优化选择所设计的液相基因芯片具有很好的重复性和特异性。通过将分型探针偶联在不同种类微球上,建立多重检测体系,可在一个反应中同时诊断甲型和乙型流感病毒,检测96个样本所需时间为5.5 h,从采样到分型检测获得结果可以在一天内完成,而传统的流感病毒分离培养需要3~5天的时间[8]。液相基因芯片体系由荧光染料编码的微球构成,每种微球上偶联有针对目的核苷酸区段的探针分子,与带有报告分子藻红蛋白的多重PCR反应产物进行杂交,当微球通过Luminex检测仪,检测结果通过荧光值直接判读,其特异性引物和针对目的核苷酸的探针设计双重保证检测特异性。所以本研究建立的基于液相基因芯片的流感病毒分型检测方法较基于抗原⁃抗体免疫反应的流感病毒酶联免疫吸附测定(enzyme⁃linked immuno⁃sorbent assay,ELISA)检测方法,有效避免了抗原抗体交叉反应的干扰[9]。同时由于液相基因芯片对流感病毒分型的基础是核酸分子的杂交,其检测的基础是PCR产物和探针之间的互补程度,而且每个反应结果的平均荧光值是100个微球上的荧光值的平均值,相当于100次反应的平均结果,检测结果更加准确[10]。近年来很多实验室建立了用于流感病毒分型的多重荧光RT⁃PCR方法,目前被广泛用于流感病毒的分型[11⁃14]。本研究采用多重PCR和液相基因芯片技术相结合的方法,克服了多重荧光检测方法设计难度高的弊端,在一次反应中能同时对甲型和乙型流感病毒进行分型检测,有利于流感病毒的快速鉴定和诊断。
本研究对检测引物、探针序列及多重PCR条件、杂交条件进行多次优化,选取阳性和阴性探针检测荧光值差异最明显、检测灵敏度相对较高的条件建立流感病毒分型的液相基因芯片,对标准品病毒和11份临床样本的检测结果,阳性探针对应的平均荧光值均大于1 000,而阴性探针和阴性对照对应的平均荧光值均小于100,较目前应用较多的将大于阴性对照平均荧光值的2倍作为阳性结果判断依据更为明确[15]。本方法检测灵敏度为 1~100 pfu,与Beck等[16]报道的利用荧光定量RT⁃PCR分型方法敏感性相当。
本项目建立的方法和流感病毒荧光定量RT⁃PCR分型方法相比,检测结果与其具有较高的一致性,但在高通量检测方面具有明显的优势。因实验条件的限制,本实验检测的临床样本数量较少,未来研究中要扩大样本量来对实验可靠性进行评价。该液相基因芯片检测方法的建立,对流感病毒的临床诊断分型和流感病毒的预防控制具有一定的意义。
[1]Barberis I,Myles P,Ault SK,et al.History and evo⁃lution of influenza control through vaccination:from the first monovalent vaccine to universal vaccines[J].J Prev Med Hyg,2016,57(3):E115⁃E120.
[2]Potter CW.A history of influenza[J].J Appl Microbi⁃ol,2001,91:572⁃579.
[3]Poeppl W,Hell M,Herkner H,et al.Clinical aspects of 2009 pandemic influenza A(H1N1)virus infection in Austria[J].Infection,2011,39(4):341⁃352.
[4]Wang C,Wang Q,Hu J,et al.A multiplex RT⁃PCR assay for detection and differentiation of avian⁃origin canine H3N2,equine⁃origin H3N8,human⁃origin H3N2,and H1N1/2009 canine influenza viruses[J].PLoS One,2017,2012(1):e0170374.
[5]Cui D,Zhao D,Xie G,et al.Simultaneous detection of influenza A subtypes of H3N2 virus,pandemic(H1N1)2009 virus and reassortant avian H7N9 virus in humans by multiplex one⁃step real⁃time RT⁃PCR as⁃say[J].Springerplus,2016,5(1):2054.
[6]Dunbar SA.Applications of Luminex xMAP technolo⁃gy for rapid,high throughput multiplexed nucleic acid detection[J].Clin Chim Acta,2006,363(1⁃2):71⁃82.
[7]Bedford T,Riley S,Barr IG,et al.Global circulation patterns of seasonal influenza viruses vary with antigen⁃ic drift[J].Nature,2015,523(7559):217⁃220.
[8]Abraham MK,Perkins J,Vilke GM,et al.Influenza in the emergency department:vaccination,diagnosis,and treatment:Clinical practice paper approved by American Academy of Emergency Medicine Clinical Guidelines Committee[J].J Emerg Med,2016,50(3):536⁃542.
[9]Woźniak⁃Kosek A,Kempińska⁃Mirosławska B,Hoser G.Detection of the influenza virus yesterday and now[J].Acta Biochim Pol,2014,61(3):465⁃470.
[10]Fraiture MA,Herman P,Taverniers,et al.Current and new approaches in GMO detection:challenges and solutions[J].Biomed Res Int,2015:392872.
[11]Cox NJ,Subbarao K.Global epidemiology of influen⁃za:past and present[J].Annu Rev Med,2000,51:407⁃421.
[12]Chen Y,Cui D,Zheng S,et al.Simultaneous detec⁃tion of influenza A,influenza B,and respiratory syn⁃cytial viruses and subtyping of influenza A H3N2 virus and H1N1(2009)virus by multiplex real⁃time PCR[J].J Clin Microbiol,2011,49(4):1653⁃1656.
[13]Kang X,Wu W,Zhang C,et al.Detection of avian influenza A/H7N9/2013 virus by real⁃time reverse tran⁃scription⁃polymerase chain reaction[J].J Virol Meth⁃ods,2014,206:140⁃143.
[14]Cui D,Zhao D,Xie G,et al.Simultaneous detection of influenza A subtypes of H3N2 virus,pandemic(H1N1)2009 virus and reassortant avian H7N9 virus in humans by multiplex one⁃step real⁃time RT⁃PCR as⁃say[J].Springerplus,2016,5(1):2054.
[15]史玲莉,闫冀焕,李云,等.登革病毒悬液芯片分型检测方法的建立[J].国际病毒学杂志,2016,23(3):173⁃176.
[16]Beck,ET,Jurgens LA,Kehl SC,et al.Development of a rapid automated influenza A,influenza B,and re⁃spiratory syncytial virus A/B multiplex real⁃time RT⁃PCR assay and its use during the 2009 H1N1 swine⁃ori⁃gin influenza virus epidemic in Milwaukee,Wisconsin[J].J Mol Diagn,2010,12(1):74⁃81.
Development of a method for detecting type influenza virus A and B based on liquid⁃phase gene chip technology
CHEN Jinlong1,LV Gang1,2,ZHANG You1,FU Ruijia1,YIN Feifei1,2★
(1.Department of Microbiology,Hainan Medical University,Haikou,Hainan,China,571199;2.Key Laboratory of Translation Medicine Tropical Diseases,Hainan Medical University,Haikou,Hainan,China,571199)
ObjectiveTo develop a microbead⁃based liquid assay for rapid and high throughput detection and the typing of influenza virus A and B(INFA,INFB).MethodsRepresentative sequences has been downloaded from the GenBank database and aligned with BioEdit software.Specific primers and probes have been designed based on conserved sequences.The specificity of the primers and probes has been tested by blast of them with all the gene sequences in the GenBank database.The microbead⁃based liquid assay was developed by covalent linkage of the probes and microbeads.ResultsSpecific signals of mean fluorescent intensity have been observed in all of the tested samples.Very low signals have been obtain for non⁃specific samples.Mixture of both influenza virus A and B can obtain their positive signal respectively.The sensitivityof the method for INFA is 1~10 pfu and 10~100 pfu for INFB.11 clinical samples were detected using this method and the results were consistent with the detection results of real time reverse transcriptase⁃polymerase chain reaction(RT⁃PCR).ConclusionThe microbead⁃based liquid assay developed in this study for the typing of influenza virus A and B can be used for the detection and typing of influenza viruses.The assay provides a new method for the detection of the viruses.
Influenza virus A;Influenza virus B;Microbead⁃based liquid assay;Virus typing
作者单位:1.海南医学院病原生物学教研室,海南,海口571199
2.海南医学院热带医学转化重点实验室,海南,海口571199
国家自然科学基金课题(31460017,81672072);海南省重点研发计划项目(ZDYF2017091);海南省自然科学基金项目(20158287);海南省高等学校科学研究项目(Hnkyzx2014⁃08,Hnky2017ZD⁃16)
★通讯作者:尹飞飞,E⁃mail:yinfeifeiff@163.com