PCR-DGGE技术监测自然发酵葛根中微生物菌群动态变化
冉淦侨张红艳任平
(1. 陕西省科学院酶工程研究所,西安 710600;2. 陕西省酶工程技术中心,西安 710600;3. 陕西省生物农业研究所,西安 710600)
PCR-DGGE技术监测自然发酵葛根中微生物菌群动态变化
冉淦侨1,3张红艳2任平1
(1. 陕西省科学院酶工程研究所,西安 710600;2. 陕西省酶工程技术中心,西安 710600;3. 陕西省生物农业研究所,西安 710600)
采用自然堆积发酵对葛根进行预处理,处理后葛根总异黄酮的提取量提高了5.7% 左右。随后,分别采用传统平板法和PCR-DGGE技术,对发酵过程中不同时期微生物菌群的变化进行分析,从而筛选出发酵中起主要作用的微生物。结果表明,自然发酵过程中的细菌呈升高-降低-再升高的变化趋势:升温期明显增多,高温期有所下降,降温期再次增多;真菌的数量在整个发酵过程中都呈持续升高的趋势。发酵过程中起主要作用的微生物也被初步确定,它们分别是地衣芽孢杆菌、桃色欧文氏菌、芽孢杆菌属、枯草芽孢杆菌、不可培养细菌、爪哇青霉、黑曲霉、溜曲霉、米根霉和不可培养曲霉。
自然发酵;微生物群落;总异黄酮;变性梯度凝胶电泳(DGGE)
葛根(Radix Puerariae),又名葛藤,甜葛藤等,为豆科植物葛的根,一般指野葛 Pueraria Lobata(Willd.)Ohwi 或甘葛藤[1]。葛根黄酮为葛根中起主要药理作用的黄酮类化合物,其主要成分有葛根素(Puerarin)、大豆苷元(daidzein)、大豆苷(daidzin)等[2-3],约占葛根干重的4%-12%[4],对严重威胁人类生命健康的心脑血管疾病具有重要的药理和治疗作用[5]。
近年来,微生物酶解法开始广泛应用于中草药工业,只要选用恰当的产酶菌株对原料进行预处理,不仅可以较温和地分解植物组织,加速释放有效成分,而且能够分解去除影响液体制剂的杂质,如淀粉、蛋白质、果胶等,促进某些极性低的脂溶性成分转化为易溶于水的糖苷类,而有利于植物活性成分的提取,这为中草药的提取提供一条新途径[6]。
本课题组前期研究发现,采集于陕西省洋县的葛根经自然堆积发酵预处理后,葛根黄酮的提取量能提高 5% 左右,这在国内尚属首次,在此之前国内未有关于自然发酵提高葛根黄酮提取量的相关报道。因此,有必要对葛根自然发酵过程中微生物种群多样性及其动态变化进行研究,从而筛选出发酵过程中起主要作用的微生物。传统的菌株筛选方法为平板培养法,该法需要先将样品中的微生物进行分离纯化,然后将纯化的菌株回接到基质中发酵,最后再根据发酵结果筛选出目标菌株。此法不仅费时费力,而且其适用范围也有待提高,尤其对于多个菌株共同作用的自然发酵而言,研究起来就更加困难。变性梯度凝胶电泳(Denaturing gradient gel electrophoresis,DGGE)技术目前已经成为研究微生物物种多样性和动态变化的分子检测工具之一[7]。该技术不依赖于微生物的分离培养,而是直接提取环境样品中总的DNA进行PCR-DGGE分析,由此包括了样品中可培养微生物和不可培养微生物的总遗传信息,从而真实地反映了微生物群落的原始组成。该技术目前已被广泛应用于各种环境微生物生态的研究,如湖泊[8]、海洋[9]、土壤[10]、根系[11]、和沙丘[12]等。本实验采用 PCR-DGGE 技术对自然发酵葛根中的微生物群落结构及其动态变化进行研究,并与传统的平板培养法进行对比,旨在明确发酵过程中微生物群落的组成及其在不同阶段的变化规律,确定发酵中起主要作用的微生物,旨为葛根粉的发酵由自然堆积转为人工控制下的发酵奠定基础,同时也为进一步提高葛根总黄酮的提取率奠定基础。
1 材料与方法
1.1 材料
葛根样品分别于 2014 年 12 月采集于陕西省洋县,经鉴定为野葛[Pueraria Lobata(Willd.)Ohwi]。葛根素标准品购买于陕西省食品药品检验所,其它试剂均为国药分析纯。
真菌培养用 PDA 培养基;真菌分离培养基是在孟加拉红培养基中加入过滤除菌的100 mg/L 链霉素;真菌纯化培养基采用孟加拉红培养基;细菌培养和纯化用营养琼脂培养基;细菌分离培养基是在营养琼脂培养基中加入过滤除菌的 100 mg/L 钠他霉素;放线菌培养和纯化用高氏一号培养基;放线菌分离培养基是高氏一号培养基加入过滤除菌的 0.075 g/L重铬酸钾和 0.002 g/L 青霉素。所有的培养基均参见冉淦侨[13]的方法配制。
主要溶液 100 mg/L 链霉素溶液,100 mg/L 纳他霉素溶液,参见冉淦侨[13]的方法配制。100 mg/L重铬酸钾溶液,100 mg/mL 青霉素溶液,参见冉淦侨[14]的方法配制。
葛根粉的制备:将采集的葛根洗净,去除表面泥沙,晾干、切成小块,于60℃烘干、粉碎,过80目筛后放于干燥器中保存备用。
50×电泳缓冲液(TAE):准确称取Tris 242 g,Na2EDTA·2H2O 37.2 g,加入800 mL去离子水,充分搅拌溶解,再加入57.1 mL的醋酸,充分搅拌,加去离子水定容至1 000 mL,室温保存。
1.2 方法
1.2.1 葛根的自然发酵 将经粉碎处理的葛根粉与自来水按1:1(W/V)充分混合后分装在呢绒袋中(每袋50 kg,共10袋),再将呢绒袋置于25℃环境中进行自然堆积发酵。每天定时记录发酵堆的温度,分别于0 d、1 d、5 d、12 d、15 d进行样品采集。采样点为发酵堆的上层、中间层和下层,每个点采样3份。将每次采集的样品混匀,一部分保存于4℃冰箱用于微生物分离培养以及葛根总黄酮的提取和检测;一部分保存于-20℃冰箱中用于细菌和真菌的总DNA提取。1.2.2 葛根黄酮的提取 提取方法参见[15],做了适当调整。将1.2.1中定时采集的葛根粉烘干至恒重(先用50℃烘8 h,去除大部分的水分,再用80℃烘至恒重)。精密称取烘干后的葛根粉2.5 000 g装于滤纸筒中,将开口端折叠封住,放入索氏提取器的提取筒中。将100 mL 80%乙醇作为提取液加入到圆底烧瓶中,安装好索氏提取装置,于85℃水浴中回流提取3 h。收集烧瓶中的提取液用于葛根黄酮的检测。
1.2.3 葛根黄酮含量的测定 取1.2.2中收集的提取液0.1 mL,用水定容至10 mL。以0.l mL 80%乙醇加水至10 mL为空白对照,在250 nm处测定吸光度。
标准曲线绘制:精密称取干燥至恒重的葛根素标准品5 mg,于25 mL量瓶中,加95%乙醇配制成0.2 mg/mL 的溶液,作为标准品溶液。精密吸取0.1、0.2、0.3、0.4、0.5、0.6、0.8和1.0 mL 置于10 mL量瓶中,用水调至刻度;以 l mL 乙醇加水至10 mL为空白对照,在 250 nm 处测定吸光度,得回归方程为:y=0.068 6x+0.009(R2=0.999 6)。
1.2.4 微生物纯培养法分析葛根自然发酵过程微生物种群的变化 对自然发酵过程中采集的葛根样品进行微生物种群变化分析。采用传统微生物纯培养法确定葛根自然堆积发酵过程中微生物的种类和数量的动态变化情况。
1.2.4.1 微生物的分离与计数 称取其中25 g发酵后的葛根粉放入含有250 mL 无菌水的三角瓶中(在瓶中加入玻璃珠),150 r/min摇床振荡30 min后制成10-1的菌悬液,再用无菌水将菌悬液按10-2、10-3、10-4、10-5、10-6做梯度稀释,取不同稀释梯度的菌悬液0.1 mL分别涂于真菌分离培养基、细菌分离培养基和放线菌分离培养基表面,每个稀释度设3次重复,30℃下培养2 d后开始细菌和放线菌计数,3 d后开始酵母菌和霉菌计数。根据菌落大小、形态和颜色等表型特征将真菌、细菌和放线菌进行初步划分,平板划线分离法进行纯化,得到纯种菌株,4℃贮藏备用。
1.2.4.2 微生物的鉴定 微生物的形态鉴定及分子鉴定均参考冉淦侨[13]的方法进行。
1.2.5 PCR-DGGE技术分析葛根自然发酵过程微生物种群的变化 对自然发酵过程中采集的葛根样品,分别进行细菌和真菌的总DNA提取,并用PCRDGGE技术分析葛根自然堆积发酵过程中微生物的种类和数量的动态变化情况。
1.2.5.1 葛根中微生物总DNA提取 将发酵后的葛根粉置于-70℃冰箱中冷冻3 h后,称取其中4 g样品经液氮充分研磨;接下来按照试剂盒操作说明(PowerSoil DNA Isolation Kit Sample,Catalog No.12888-S,MO BIO)和(PowerFecal DNA Isolation Kit Sample,Catalog No.12830-S,MO BIO)分别提取葛根样品中微生物基因组总DNA。
1.2.5.2 PCR扩增 细菌16S rDNA V3区PCR:采用的引物为338F-GC/518R(引物序列见表1,其中,cclamp序列:GC clamp:CGCCCGCCGCGCGC GGCGGGCGGGGCGGGGGCACGGGGGG),扩增目的片段为200 bp。50 μL的PCR反应体系组成如下:Template DNA 0.5 μL,Forward Primer(10 μmol/L)1 μL,Reverse Primer(10 μmol/L)1 μL,2×PCR Mix(Vazyme)25 μL,ddH20 to final volume 50 μL。PCR反应程序见表2(细菌第一轮PCR)。
真菌ITS区PCR:采用的引物为NS3F-GC/ Fun15R(引物序列见表1),扩增目的片段为200 bp。PCR反应体系与细菌16S rDNA PCR相同。PCR反应程序见表2(真菌第一轮PCR)。
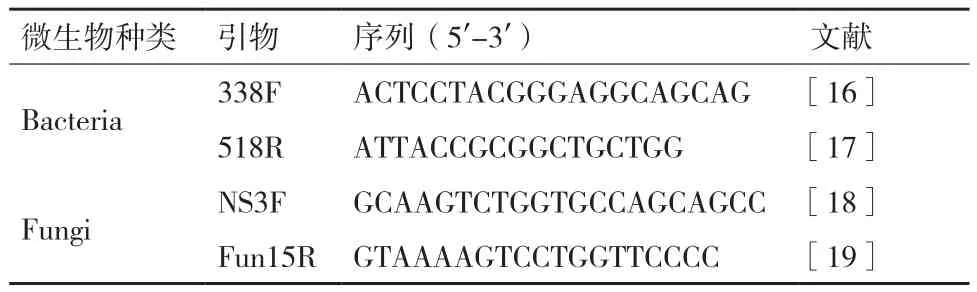
表1 PCR-DGGE扩增细菌和真菌所需引物的名称及序列
1.2.5.3 变性梯度凝胶电泳DGGE DGGE电泳分析使用德国Biorad仪器(DCode,Biorad,Germany),变性胶的范围为30%-65%(100%的变性剂为7 mol/L的尿素和40%的去离子甲酰胺的混合物),变性胶的浓度从上至下递增。待变性梯度胶完全凝固后,将胶板放入装有1×TAE电泳缓冲液的电泳槽中,取PCR产物30 μL加入加样孔中,200 V电压在60℃下电泳450 min。显色采用EB染色,应用Quantity One软件分析结果。
1.2.5.4 DGGE特征条带的回收、测序 将DGGE分析胶上的特征条带用无菌手术刀片切下,用30 μL 的TE溶液浸泡过夜,取浸泡液4 μL作为PCR模板进行扩增,引物不带GC夹子,扩增体系为:Template DNA 4 μL,Forward Primer(10 μmol/L)2 μL,Reverse Primer(10 μmol/L)2 μL,2×PCR Mix(Vazyme)25 μL,ddH20 to final volume 50 μL。PCR反应程序见表2(第二轮PCR)。扩增所得产物送至GENEWIZ公司测序,测序结果在GenBank中进行序列同源性比对。
2 结果
2.1 葛根自然发酵过程中总黄酮的变化
发酵地点为安康中科麦迪森天然药业公司的仓库中,葛根原料产地为陕西洋县,结果见图1。由图1可知,随着发酵时间的推移,葛根黄酮的得率也逐渐提高,在发酵第10天时达到最高点,与未经发酵处理的葛根相比,提取出的总黄酮量提高了约5.7%,发酵15 d时,葛根黄酮的得率略有下降。

表2 细菌(16S rDNA V3区)和真菌(ITS区)PCR 扩增条件
2.2 微生物纯培养法分析葛根自然发酵过程微生物种群的变化
以发酵过程中采集的葛根样品作为研究对象,通过对葛根样品中的微生物进行分离鉴定,研究葛根发酵过程中微生物的种类及数量的变化。
历代鸿儒对“权”之阐释表明,“权”是在具体的情景下,采取针对性的措施或者方法,需因地制宜,因时制宜。亦即权是对经的反叛。但行权有度。如董仲舒《春秋繁露·玉英》中说:“夫权虽反经,亦必在可以然之域。”权虽然是反经的,但是权是在允许的、正确的范围之内,行权的结果需要对事物本身有益。在这一点上,余治平(2008)曾指出,“权”的实施,不能是没有边际的,而应该以一定的“经”为前提、为背景。岳天雷(2013)也曾说,“权”作为权衡轻重利弊的智慧和灵活变通的方法,是儒家政治哲学、道德哲学和历史哲学的重要内容。
如图2所示,在整个自然发酵过程中,就微生物的菌群数量而言,随着堆体温度的升高,细菌和真菌的数量也迅速从102CFU/g升高至105CFU/g,并随着发酵时间的延长,真菌数量呈逐渐上升的趋势,而细菌除了在发酵第5天时数量略有下降之外,到第10天时又迅速上升至108CFU/g。分析原因,可能是由于从第3天开始到第5天,堆体温度一直维持在40℃以上,持续的高温会导致部分不耐热的细菌死亡,而其余细菌生长缓慢,所以导致第5天时细菌数量略有下降;而真菌由于细胞壁较厚,加之曲霉属的真菌还能产生抗逆性较强的孢子,所以真菌的数量没有受到高温的影响。
就微生物的菌群多样性而言,从采集的葛根样品中总共分离到6株不同的细菌,11株不同的真菌。经过菌落形态初步鉴定及16S rDNA和18S rDNA的分子鉴定,鉴定结果见表3和表4。整个发酵过程中,细菌和真菌的种群多样性都是呈先升高,后降低而后再升高的趋势。
如图3-A所示,在最初采集的样品中,分离出的4种细菌,分别是B1地衣芽孢杆菌(Bacillus licheniformis)、B2桃色欧文氏菌(Erwinia persicina)、B3污水杆菌(Lysinibacillus contaminans)和 B4大肠杆菌属(Escherichia sp),到第3天的时候,又多了B5枯草芽孢杆菌(Bacillus subtilis)。第5天,受持续高温的影响,样品中的革兰氏阴性细菌B3污水杆菌和B4大肠杆菌属几乎消失,而B5和B6由于是芽孢杆菌属能产生抗逆性较强的芽孢,逐渐成为优势菌,直至发酵结束。综上所述,确定发酵过程中起主要作用的细菌为:B1地衣芽孢杆菌、B2桃色欧文氏菌、B5芽孢杆菌属和B6枯草芽孢杆菌。
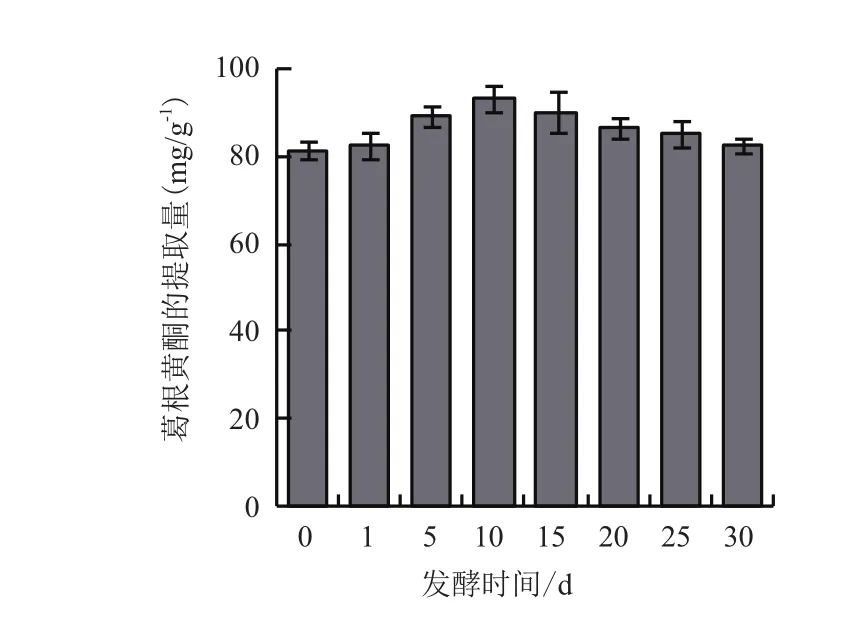
图1 葛根自然发酵过程中葛根总黄酮的动态变化
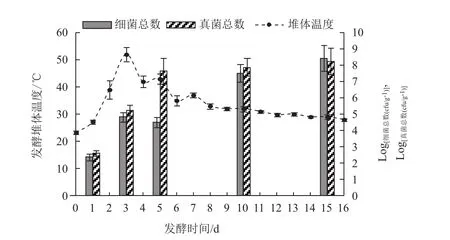
图2 葛根发酵过程中堆体温度及微生物菌群数量的变化

图3 葛根发酵过程中细菌(A)和真菌(B)种群多样性的变化

表3 葛根发酵样品中分离出的6株细菌的16S序列比对结果
真菌种群多样性的变化趋势与细菌类似,如图3-B所示,在最初采集的样品中,分离出6种真菌,且每种真菌所占比例都差不多,没有形成优势菌。随着发酵时间的推移,到第3天的时候,样品中分离出的真菌已达到8种,且F1爪哇青霉(Penicillium javanicum)逐渐成为优势菌。到第5天的时候,由于受持续高温的影响,真菌种类又下降至6种,此时F1爪哇青霉(Penicillium javanicum)在样品所占比例高达91%,成为绝对的优势菌。发酵第10天,随着堆体温度的降低,其余种类的真菌也开始生长,真菌种类又升高到8种,且F2/F3黑曲霉(Aspergillus niger)、F4溜曲霉(Aspergillus tamarii)和F7米根霉(Rhizopus oryzae)也开始快速生长,逐渐成为优势菌。综上所述,确定发酵过程中起主要作用的真菌为:F1爪哇青霉,F2/F3黑曲霉,F4溜曲霉,F7米根霉。2.3 PCR-DGGE技术分析葛根自然发酵过程微生物种群的变化
2.3.1 葛根样品中微生物基因组DNA的提取 采用PCR-DGGE技术进行微生态研究的第一步是提取环境样品的总 DNA。所提取的总DNA必须既能实际代表原始体系中生物多样性,又能满足后续PCR 等分子生物学反应的质量要求。考虑到葛根发酵样品的固态原料比例很高,且含有丰富的代谢产物,在查阅相关文献和咨询相关专业人员的基础上,选取了MO BIO公司的两种DNA提取试剂盒,分别是Catalog No.12888-S的 PowerSoil DNA Isolation Kit Sample和Catalog No.12830-S,PowerFecal DNA Isolation Kit Sample。
结果如表5所示,就DNA浓度而言,货号为12830-S试剂盒提取的DNA浓度普遍高于货号为12888-S试剂盒。就A260/A280值而言,货号为12888-S试剂盒的A260/A280值均在2.0以上,而货号为12830-S试剂盒的A260/A280值均在1.8-2.0之间,纯度较高,满足后续 PCR 反应要求。因此不管是DNA浓度还是纯度上,货号为12830-S的试剂盒提取葛根样品中的微生物总DNA效果较好。

表4 葛根发酵样品中分离出的11株苹真菌的18S序列比对结果

表5 发酵葛根样品中微生物总DNA的提取结果
2.3.2 PCR-DGGE分析葛根发酵过程中微生物的种群变化 以提取的基因组DNA为模板,用引物对338F/518R扩增细菌16S rDNA V3区,用引物对NS3F/Fun15R扩增真菌的ITS区,PCR产物在合适的变性剂浓度梯度和电泳条件下经DGGE分析,结果见图4。
图4中,0 d、1 d和5 d各泳道中优势条带的电泳迁移位置基本一致,表明这个阶段的优势微生物是相同的,且每个泳道中都仅有2-3条亮度较高的DNA 条带,说明这几个样品中的微生物多样性较低。10 d泳道亮度较高的 DNA 条带增多,说明样品中微生物多样性增多,优势菌群增多。
为了确定DGGE图谱中各条带对应的微生物种属信息,对胶中主要条带进行切胶、回收、克隆、验证、测序和序列比对。
细菌条带的鉴定结果(表6)表明,与条带1和条带3序列同源性最高的是芽孢杆菌属和不可培养细菌属,与条带10、11和12序列同源性最高的也分别是不可培养细菌属,芽孢杆菌属和不可培养细菌属,这说明在葛根的自然发酵过程中,不可培养细菌和芽孢杆菌属于优势菌,这与微生物纯培养的分析结果略有不同。虽然两种分析结果都表明芽孢杆菌属在整个发酵过程中占据优势,但由于不可培养细菌无法通过传统的微生物纯培养法进行分离培养,因此微生物纯培养法并不能将它检测出来,这是该方法的局限性。此外,与条带6序列同源性最高的是大肠杆菌属,该条带在5 d泳道中消失,这一结果与微生物纯培养的分析结果一致。由此可见,PCR-DGGE技术确定了发酵过程中起主要作用的细菌为:芽孢杆菌(条带1、5、7、11),不可培养细菌(条带3、9、10、12、13)和欧文氏菌(条带2、8)。综上所述,PCR-DGGE技术不仅能检测到传统微生物分离中获得的全部细菌,还能检测出那些含量低、较难培养的细菌。与传统的微生物纯培养技术相比,PCR-DGGE技术不仅涵盖的细菌种类更为全面,检测结果可信度高,还能更直观、快速地观察到细菌的演变过程,确定样品中的优势细菌。
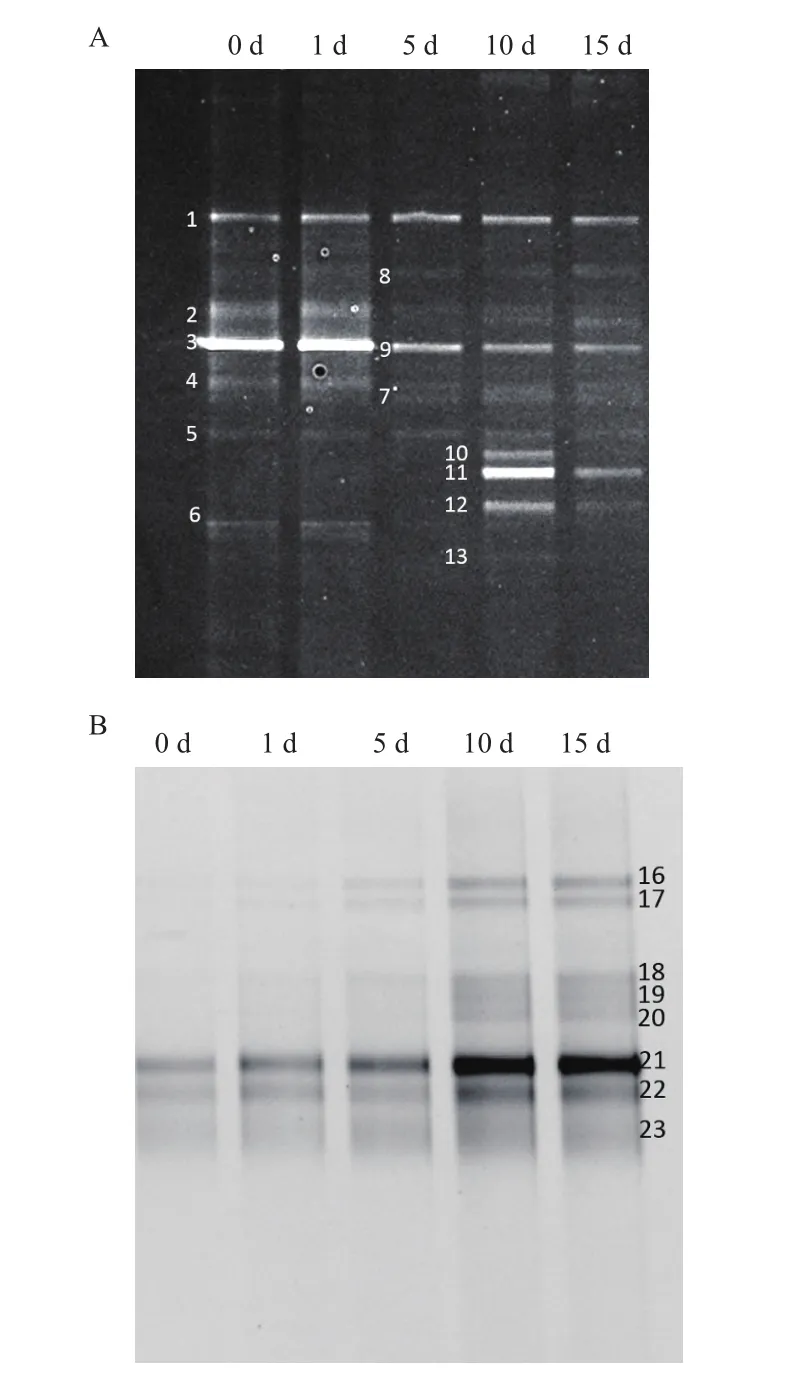
图4 不同葛根样品细菌16S rDNA V3区(A)和真菌ITS区(B)扩增片段的DGGE电泳分析
真菌条带的鉴定结果(表6)表明,与条带21序列同源性最高的是青霉属(Penicillium sp.),而微生物纯培养方法检测的结果也表明爪哇青霉为整个发酵过程中的优势菌,两种方法的检测结果一致。此外,与条带22和23序列同源性最高的都是不可培养曲霉属(Uncultured Aspergillus sp.)。与传统的微生物纯培养方法相比,PCR-DGGE技术的优点是能分析鉴定出不可培养真菌。但由于DGGE片段只有 bp,只含有18S rDNA 序列的一部分,因此序列比对结果的可靠性大大降低,只能鉴定到属,这给真菌的多样性分析带来了困难。同时,表6的鉴定结果可以看出,PCR-DGGE技术分析出的真菌多样性较低,只包含曲霉属和青霉属两类。而微生物纯培养方法分析结果表明,虽然样品中青霉属和曲霉属为发酵过程中的优势菌属,但样品中还存在大量的毛霉、根霉等其它菌属。由此可见,DGGE技术对真菌的多样性检测结果存在偏差。主要有以下两点原因:其一,总DNA提取的方法不够优化,涵盖面较窄导致部分DNA的遗漏;其二,PCR扩增时对模板有偏嗜性,导致有的DNA被大量扩增,而有的DNA被扩增的几率很小。因此,要提高DGGE检测结果的准确性,还需要进一步的实验探索。综上所述,PCR-DGGE技术确定了发酵过程中起主要作用的真菌为:曲霉属(条带16、17、18、19),青霉属(条带20、21),不可培养曲霉属(条带22、23)。
3 讨论
自然界中某些微生物可以分解植物组织(如果胶、木质素、纤维素等),加速植物有效成分的释放,有利于植物活性成分的提取。汤海鸥等[6]采用黑曲霉对葛根进行酶解处理,处理后葛根黄酮的提取量提高 21.1%。宋艳秋等[20]采用红曲酶发酵转化葛根,使葛根主要有效成分含量有所提高。邬建国等[21]从10株食药用真菌中筛选获得1株高效菌株 DS1,该菌株在发酵葛根渣后使葛根渣中异黄酮的含量提升了30.3%。这些都说明了利用产酶微生物发酵处理葛根,可以提高葛根中总异黄酮的提取量。本研究采用自然堆积发酵处理葛根,处理10 d后葛根黄酮的提取量提高了 5.7% 左右,这说明自然发酵处理也能提高葛根黄酮的提取量。然而,并不是所有葛根在任何地方经自然发酵后,其葛根黄酮的提取量都能得到提高,它取决于葛根的产地来源及发酵地点等诸多因素。在前期实验中,本课题组将产地不同的8种葛根分别在两个不同的地方进行自然发酵,仅有产自陕西洋县的葛根在安康某公司的仓库中进行发酵后,总黄酮的提取量才会提高(数据未显示)。这说明洋县的葛根中或安康某公司的仓库环境中肯定存在着某些特有的微生物,正是通过这些特有微生物的发酵处理,才使得葛根总黄酮的提取量得以提高。
为了确定发酵过程中起主要作用的微生物,本研究分别采用传统培养法和PCR-DGGE技术对葛根自然发酵过程的微生物菌群变化进行分析。结果表明,就微生物的数量而言,传统培养法与PCRDGGE技术得到了相似的变化规律;发酵过程中的细菌呈升高-降低-再升高的变化趋势:升温期明显增多,高温期有所下降,降温期再次增多;真菌的数量在整个发酵过程中都呈持续升高的趋势。就微生物的种类而言,传统培养法和PCR-DGGE方法得到的结果大致相同,但也存在差异。传统培养法确定起主要作用的细菌为:地衣芽孢杆菌、桃色欧文氏菌、芽孢杆菌属和枯草芽孢杆菌;PCR-DGGE技术分析表明,除了芽孢杆菌属和欧文氏菌属之外,还有大量的不可培养细菌在发酵中发挥主要作用。这说明PCR-DGGE技术不仅能直观、快速地观察到细菌的演变过程,确定样品中的优势细菌,且涵盖的细菌种类也较为全面,结果可信度较高。此外,传统培养法确定起主要作用的真菌为:爪哇青霉、黑曲霉、溜曲霉和米根霉;而PCR-DGGE技术虽然能检测出青霉属、曲霉属和不可培养的曲霉属,但多种其它真菌(如米根霉、粗糙链孢霉菌等)该技术未能检出,与纯培养技术相比,并不能完全反映出真菌菌群的原始组成,且由于扩增的片段较短,比对结果准确性也不够高。因此,还需进一步的研究,以提高PCR-DGGE技术在真菌检测上的准确性。

表6 PCR-DGGE条带的序列比对结果
4 结论
综合上述两种方法的结果,初步确定发酵过程中起主要作用的微生物有地衣芽孢杆菌、桃色欧文氏菌、芽孢杆菌属、枯草芽孢杆菌、不可培养细菌、爪哇青霉、黑曲霉、溜曲霉、米根霉及不可培养曲霉。后续我们将分别对这些微生物的作用机理进行研究,最终确定具体是其中哪一种或哪几种微生物通过何机制提高了葛根黄酮的提取率。
[1] 王慕邹. 常用中草药高效液相色谱分析[M]. 北京:科学出版社, 1999:354-355.
[2] Kazohiro H, Makoto M, Kaoru N. Phenolic glucosides from the root of Pueraria lobate[J]. Phytochemistry, 1997, 46(5):92-93.
[3] 汪洋. 葛根中葛根素的分离与纯化[D]. 武汉:武汉工程大学, 2011:2-3.
[4] 李悦, 李艳菊. 国内外葛根功能食品研究进展[J]. 食品研究与开发, 2007, 28(12):174-176.
[5] 叶淑静. 中药葛根研究进展[J]. 浙江中医学院学报, 2003, 27(6):94-96.
[6] 汤海鸥, 程茂基, 石秀侠, 等. 黑曲霉酶解提取葛根黄酮的研究[J]. 饲料工业, 2006, 27(24):14-17.
[7] 马俊孝, 季明杰, 孔健. PCR-DGGE技术在微生物物种多样性研究中的局限性及其解决措施[J]. 食品科学, 2008, 29(5):493-497.
[8] Bukowska A, Bielczyńska A, Karnkowska A, et al. Molecular(PCRDGGE)versus morphological approach:analysis of taxonomic composition of potentially toxic cyanobacteria in freshwater lakes[J]. Aquat Biosyst, 2014, 10(1):2.
[9] Ling J, Zhang YY, Dong JD, et al. Spatial variations of bacterial community and its relationship with water chemistry in Sanya Bay, South China Sea as determined by DGGE fingerprinting and multivariate analysis[J]. Ecotoxicology, 2015, 24(7-8):1486-1497.
[10] Dong XL, Reddy G B. Soil bacterial communities in constructed wetlands treated with swine wastewater using PCR-DGGE technique[J]. Bioresource Technology, 2010, 101:1175-1182.
[11] Yang CH, Crowley DE. Rhizosphere microbial community structure in relation to root location and plant iron nutritional status[J]. Appl Environ Microbiol, 2000, 66(1):345-351.
[12] 颜菲, 葛琴雅, 李强, 等. 云冈石窟石质文物表面及周边岩石样品中微生物群落分析[J]. 微生物学报, 2012, 52(5):629-636.
[13] 冉淦侨, 戴佳锟, 秦涛, 等. 芽孢杆菌Q13、Q14 在猕猴桃叶面的定殖及其对叶面菌群的影响[J]. 生物技术通报, 2012(1):162-167.
[14] 冉淦侨, 王楠, 戴佳锟, 等. 枯草芽孢杆菌BS24 在苹果叶面的定殖及其对叶面菌群的影响[J]. 生物技术通报, 2013(10):131-136.
[15] 崔九成, 宋小妹, 张培芳, 等. 葛根总黄酮提取工艺研究[J].西北药学杂志, 1999, 14(5):202.
[16] Lane DJ. 16S/23S rRNA sequencing[M]//Stackebrandt E, eds. Nucleic acid techniques in bacterial systematics. New York:John wiley and sons, 1991:115-175.
[17] Muyzer G, de Waal EC, Uitterlinden AG. Profiling of complex microbial population by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J]. Applied and Environmental Microbiology, 1993, 59(3):695-700.
[18] White TJ, Bruns TD, Lee SB, et al. Amplification and direct sequencing of fungal ribosomal RNA genes for phylogenetics[J]. PCR-Protocols:A guide to methods and applications, 1990, 18:315-322.
[19] Smit E, Leeflang P, Glandorf B, et al. Analysis of fungal diversity in the wheat rhizosphere by sequencing of cloned PCR-amplified genes encoding 18S rRNA and temperature gradient gel electrophoresis[J]. Applied and Environmental Microbiology, 1999, 65:2614-2621.
[20] 宋艳秋, 陈有为. 红曲霉转化中药葛根固体发酵条件研究[J].安徽农业科学, 2010, 38(4):1707-1708, 1745.
[21] 邬建国, 朱之光, 贾伟彦, 等. 葛根渣功能产品固态发酵菌株的筛选[J]. 食品与发酵工业, 2009, 35(1):27-30.
(责任编辑 狄艳红)
Dynamic Changes of Microbial Community in the Process of Natural Fermentation of Puerariae lotaba Monitored by PCR-DGGE
RAN Gan-qiao1,3ZHANG Hong-yan2REN Ping1
(1. Institute of Enzyme Engineering,Shaanxi Academy of Science,Xi’an 710600;2. Enzyme Engineering Technology Center of Shaanxi Province,Xi’an 710600;3. Shaanxi Bio-Agriculture Institute,Xi’an 710600)
In this study,the extraction yield of total isoflavones from Puerariae lotaba increased by 5.7% after pretreated with natural compost fermentation. After that,in order to determine the microorganisms playing a major role in this fermentation,the dynamic changes of the microbes in the process of natural fermentation of P. lotaba powder were analyzed using traditional flat culture method and PCR-DGGE technology respectively. The results showed that the number of bacteria showed an “increase-decrease-increase” trend in the whole fermentation process:increased significantly in the temperature-rising stage,then decreased slightly in the thermophilic stage,and finally rose again in the temperature-cooling stage,but the number of fungi presented a continuous increase trend. Besides,several microorganisms isolated from P. lotaba were initially identified to have positive effects on improving the extraction yield of total isoflavones,and these microbes included:Bacillus licheniformis,Rhizoctonia solani,Bacillus,Bacillus subtilis,non-cultivable bacteria,Penicillium japonicum,Aspergillus niger,Aspergillus,Asparagus,and non-cultivable Aspergillus.
natural fermentation;microbial community;total isoflavones;denatured gradient gel electrophoresis(DGGE)
10.13560/j.cnki.biotech.bull.1985.2017-0251
2017-03-30
陕西省科学院青年人才培养专项(2012k-31)
冉淦侨,女,硕士,助理研究员,研究方向:植物保护;E-mail:ranganqiao@163.com

