生物法合成5-氨基乙酰丙酸的研究进展
李智祥,赵 磊,梁云龙,张成林,2,3
(1. 天津科技大学 生物工程学院,天津 300457;2. 代谢控制发酵技术国家地方联合工程实验室,天津 300457; 3. 天津市氨基酸高效绿色制造工程实验室,天津 300457)
生物法合成5-氨基乙酰丙酸的研究进展
李智祥1,赵 磊1,梁云龙1,张成林1,2,3
(1. 天津科技大学 生物工程学院,天津 300457;2. 代谢控制发酵技术国家地方联合工程实验室,天津 300457; 3. 天津市氨基酸高效绿色制造工程实验室,天津 300457)
5-氨基乙酰丙酸(5-aminolevulinic acid,ALA)是血红素、叶绿素等吡咯化合物生物合成的重要前体,在农业和医药上得到广泛应用.5-氨基乙酰丙酸的生物合成途径主要包括C4和C5途径.对5-氨基乙酰丙酸的合成途径和调控机制以及利用大肠杆菌和谷氨酸棒杆菌通过C4途径和C5途径合成5-氨基乙酰丙酸的代谢工程研究进展进行了综述.
5-氨基乙酰丙酸;C4途径;C5途径;代谢工程
5-氨基乙酰丙酸(5-aminolevulinic acid,ALA)是生物体合成叶绿素、血红素和维生素B12等四吡咯类化合物的前体物质,是一种广泛存在于微生物、植物和动物细胞中的非蛋白类五碳氨基酸.近年来,ALA作为一种安全、选择性和渗透性好的光动力学药物在医学领域引起广泛关注.目前,ALA已经应用于诸多癌症(如皮肤癌、消化道癌及肺癌等)的诊断与光动力学治疗[1].作为第二代光动力学药物的显著优点是副作用小、渗透性好和疗效确切和对疾病的适用范围广等.同时,作为一种环境相容性及选择性很高的新型光活化农药,对农作物和人畜无害,可在农业上广泛用作光动力学杀虫剂、落叶剂和除草剂等[1-2].
目前主要采用化学合成法进行ALA的工业化生产[3],由于化学合成反应步骤多、副产物多、分离提纯难、ALA的得率低以及环境污染严重等问题,生物法合成ALA已成为未来研究发展的趋势[1].笔者介绍了5-氨基乙酰丙酸的合成途径和调控机制以及通过C4途径和C5途径生物合成5-氨基乙酰丙酸的代谢工程研究进展.
1 ALA的生物合成途径及代谢调控
自然界中,ALA的合成主要存在两条途径[1]:一条是以琥珀酸为前体物称为C4途径[4],另一条是以谷氨酸为前体物称为C5途径[5].C4途径是由5-氨基乙酰丙酸合酶(5-aminolevulic acid synthase, ALAS)催化琥珀酰-CoA和甘氨酸生成ALA,该途径主要存在于紫细菌属等光合细菌、真菌以及动物体中.C5途径是以谷氨酸为前体物经过三步酶促反应生成ALA[1],谷氨酸由谷氨酰-tRNA合成酶(由gltX基因编码)催化生成谷氨酰-tRNA;后者在谷氨酰-tRNA还原酶(由hemA基因编码)催化下生成谷氨酸-1-半醛;再经谷氨酸-1-半醛-氨基转移酶(由hemL基因编码)催化生成ALA,该途径广泛存在于植物、藻类以及细菌中.ALA可进一步用于合成吡咯类化合物,因此ALA的合成是吡咯类化合物合成的限速步骤,ALA合成的关键酶ALAS受其下游代谢物血红素的反馈抑制[4-5].ALA生物合成途径为
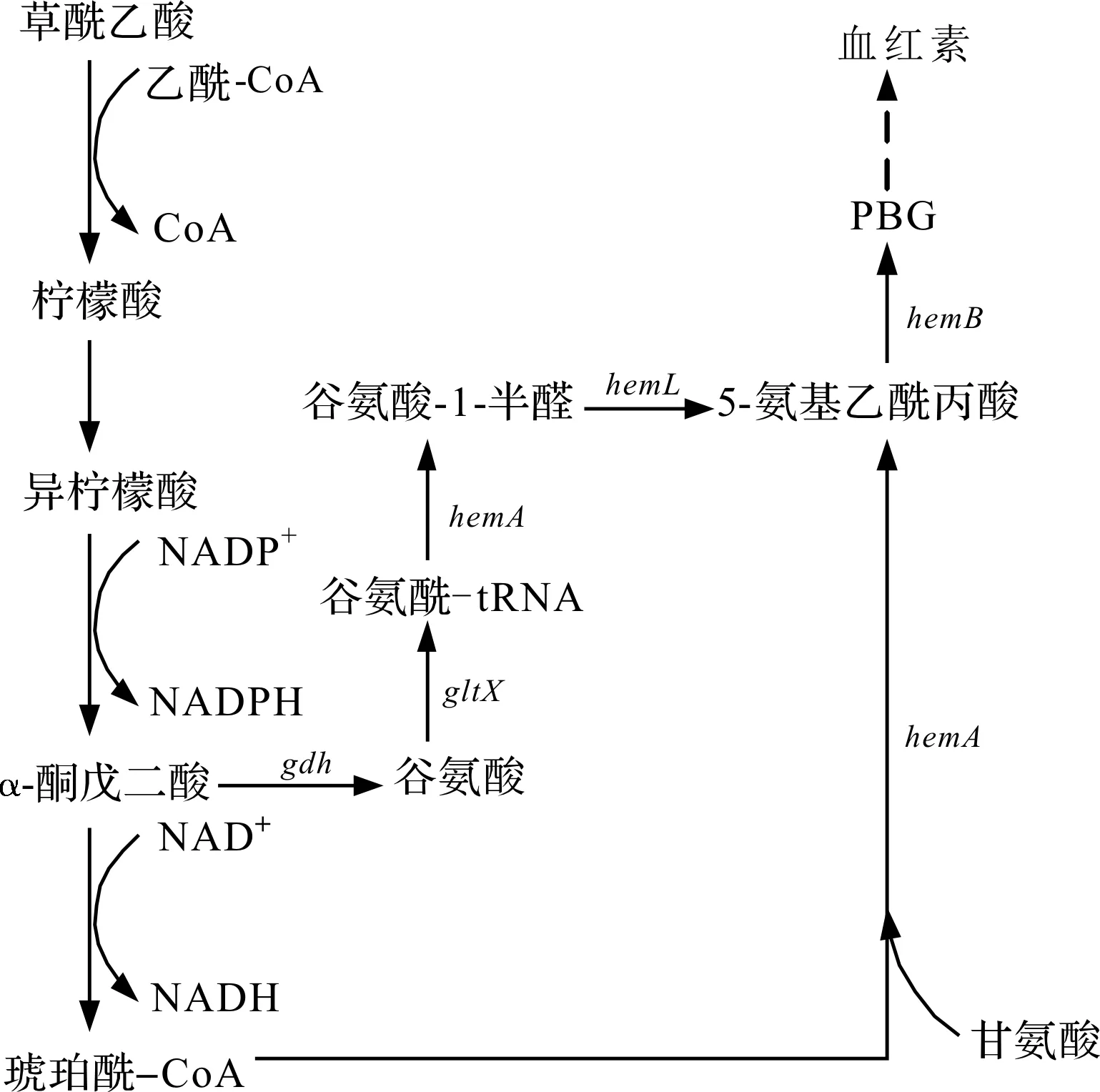
近年来,随着多种微生物基因组测序的完成以及基因重组技术的不断完善,使得通过基因工程等手段获得性状优良的ALA生产菌株已成为现实.目前,对生物法合成ALA的研究主要集中在大肠杆菌和谷氨酸棒杆菌,大多集中在关键酶谷氨酰-tRNA合成酶酶活的增强,也不乏通过发酵过程控制和优化发酵条件增加ALA的合成.
2 ALA产生菌选育的早期研究
早期主要是从自然界中筛选得到合成ALA的微生物.1970年,Beale等[6]筛选获得一株能够利用CO2积累ALA的小球藻(Chlorellavulgaris),随后相继发现其他小球藻也具有合成积累ALA的能力[7-8].但由于藻类自身的特性决定了其积累ALA的浓度较低,此后对藻类合成ALA的研究逐渐减少.1987年,Sasaki等[9]筛选到一株类球红细菌(Rhodobactersphaeroides),通过人为添加ALA合成前体物琥珀酸和甘氨酸,ALA积累量达到0.26 g/L,随后进一步添加乙酸和丙酸,ALA质量浓度可提高至0.55 g/L[10].
ALA在ALA脱水酶(HemB,由hemB基因编码)的催化作用下转化为胆色素原.研究发现乙酰丙酸是ALA脱水酶的竞争性抑制剂,因此,添加适量的乙酰丙酸能够抑制ALA脱水酶的活性从而积累ALA,这一手段成为发酵优化中的一个有效策略[11].另一个策略是通过对培养基和培养条件的优化,菌株R.sphaeroidesCR 720的ALA积累量达到了3.61 g/L[12].但是发酵优化的前提是获得具有高效合成ALA潜力的菌株,又因从自然界筛选合成ALA的工业菌种工作量大且具有盲目性,致使ALA的生物合成研究进展缓慢.随着基因工程、代谢工程及组学技术的迅猛发展,研究重点集中于ALA生产菌株的代谢工程改造.
3 利用C4途径合成ALA
C4途径是利用ALA合成酶通过一步酶促反应催化中心代谢产物琥珀酰-CoA和甘氨酸缩合生成ALA,方法简单.大肠杆菌(Escherichiacoli)和谷氨酸棒杆菌(Corynebacteriumglutamicum)作为模式菌株,遗传背景清晰,分子操作手段和工具完善齐备,因此目前利用C4途径合成ALA主要集中于这两种微生物.
3.1 大肠杆菌C4途径合成ALA
随着生物技术的发展,模式微生物表达异源蛋白的策略被广泛采用.而大肠杆菌作为一种具有诸多优点(如遗传背景清晰、遗传操作工具完善、操作简单、营养要求简单等)的模式微生物常被研究者用作异源蛋白的表达载体.野生型的大肠杆菌利用C5途径合成ALA,已有文献报道C4途径的重组大肠杆菌ALA最高产量已经达到了9.40 g/L[13].
Vander等[14]成功将来源于R.sphaeroides菌株中的hemA基因克隆至E.coli中,实现了hemA基因的异源活性表达以及ALA的异源生物转化(表1).基于提高ALAS(5-氨基乙酰丙酸合酶)的表达量及活力,Fu等[15]将来源于R.sphaeroides的hemA基因克隆至依赖于T7RNA聚合酶的表达系统中,并分析了其在E.coliBL21 (DE3)和E.coliRosetta (DE3)中的表达差异.研究发现,hemA基因在E.coliRosetta (DE3)菌株中得到了更好的表达,ALA 产量提高至3.80 g/L(表1).与此同时,多个研究团队尝试了来源于不同菌株的hemA基因的表达,他们分别从大豆根瘤菌(Bradyrhzobiumjaponicum)[16-17]、放射形土壤杆菌(Agrobacteriumradiobacter)[18-20]和沼泽红假单胞菌(Rhodopseudomonaspalustris)[21]菌株中克隆hemA基因并在E.coli中进行表达分析.研究表明,不同来源的ALAS在E.coli中的活性差异较大(表1).
2013年,Zhang等[22]克隆了来源于R.palustris的ALAS同工酶基因hemA和hemO,最终通过过表达hemO使ALA产量达到了6.31 g/L(表1).
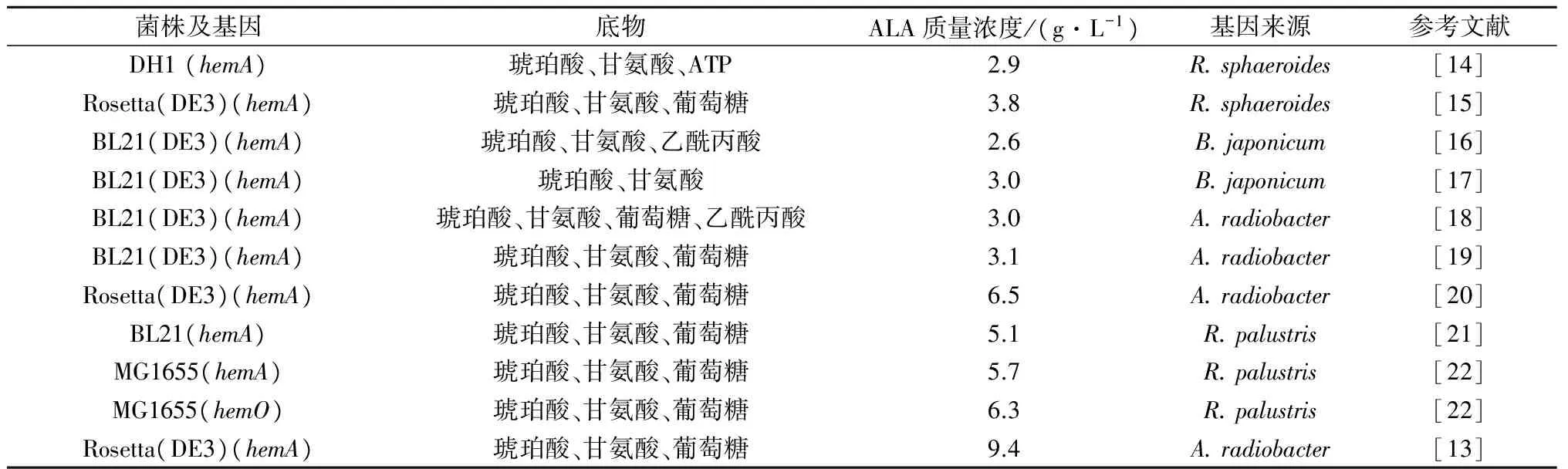
表1 大肠杆菌C4途径合成ALA
3.2 谷氨酸棒杆菌C4途径合成ALA
谷氨酸棒杆菌是一种革兰氏阳性菌,抗逆能力较强,发酵性能稳定.与大肠杆菌相比,谷氨酸棒杆菌不具有内毒素,被认为是具有安全性(Generally recognized as safe, GRAS)的微生物,因此被广泛用于谷氨酸、赖氨酸等食用和药用氨基酸的生产.近年来人们陆续开始利用谷氨酸棒杆菌进行ALA的合成,C4途径因其代谢路径简单而成为研究热点.
Feng等[23]以C.glutamicumATCC 13032作为出发菌株进行代谢改造,为提高关键酶的表达量,异源表达来自R.palustris的hemA基因,并对其进行密码子优化,使其能够在宿主菌株内高效表达.添加7.50 g/L的前体物甘氨酸经转化获得1.44 g/L ALA.随后为加强中心代谢,增加丙酮酸的代谢通量并减少丙酮酸转化为乳酸和乙酸,敲除乳酸合成基因ldhA和乙酸合成基因pqo.此外,为减少乙酰-CoA转化为乙酸,敲除乙酸合成基因pta,ackA及cat,并过表达编码乙酸合成乙酰-CoA的acsA基因,以增加乙酰-CoA的供应.结果显示,乙酸含量减少了58%,而ALA仅有0.65 g/L,暗示过量的乙酰-CoA可能会对ALA的合成具有反作用.此外,为增加ALA的外排,减弱底物抑制和毒害作用,分别敲除与细胞壁合成有关的高分子量青霉素结合蛋白编码基因pbp1a,pbp1b,pbp2a和pbp2b.结果表明敲除pbp1b后,目的产物ALA的积累量达到2.68 g/L,提高了29.5%.在此基础上过表达来源于E.coli的ALA运输载体编码基因rhtA.利用5 L发酵罐进行验证,流加4.60 g/L甘氨酸发酵33 h后,ALA的积累量为7.53 g/L.
Yang等[24]以C.glutamicumCgS1为出发菌株,过表达来源于不同菌株的ALAS,结果表明来源于R.capsulatus32 SB1003的ALAS具有较高的合成ALA的潜能.CgS1/pEC-SB菌株在5 L发酵罐中采用流加培养的方式,ALA积累量达到7.60 g/L.
虽然利用C4途径合成ALA的方法简单且较为有效,但微生物细胞在生长过程中额外消耗底物甘氨酸,造成ALA合成效率低,更重要的是甘氨酸对细胞具有毒害作用[14],进一步影响了其工业化生产的前景.
4 利用C5途径合成ALA
C4途径合成ALA需要在培养过程中添加前体物琥珀酸和甘氨酸,与C5途径以葡萄糖为碳源合成ALA相比,不仅极大地增加了成本,而且甘氨酸对于细胞生长具有抑制作用,阻碍了C4途径合成ALA的产业化发展.因此,对于利用C5途径合成ALA的研究日益增多.
4.1 大肠杆菌C5途径合成ALA
Kang等[25]首次通过改造E.coliALA合成C5途径,实现了在无机盐培养基中以葡萄糖为碳源一步发酵生产ALA的设想.研究发现由hemA基因编码的谷氨酰-tRNA还原酶以及由hemL基因编码的谷氨酸-1-半醛氨基转移酶是C5途径的限速酶,两者存在协同作用.通过单独表达hemA,以及共表达hemA和hemL使得ALA产量分别提高5.7倍和66倍.
此外,许多用于转运氨基酸的潜在转运蛋白已经被预测[26],且在ALA代谢工程改造中被加以应用[27].为寻找高效的ALA转运蛋白,研究者对蛋白运输载体YeaS[28](亮氨酸胞外转运蛋白,由yeaS基因编码)以及RhtA[29](苏氨酸和高丝氨酸胞外转运蛋白,由rhtA基因编码)进行了分析.结果表明,RhtA转运蛋白对ALA具有较强的运输能力,通过共表达rhtA基因,ALA的产量提高了约46%.
张良程等[30]以ALA高产菌株大肠杆菌E.coliZDEcA8为出发菌株,以活性丧失但能正常表达的ALA合成酶突变体大肠杆菌作为对照,通过不同基因转录组的测定,对潜在的ALA转运蛋白进行研究.已有文献报道rhtA过表达可以提高ALA的产量,促进胞内ALA的外排.EamA与RhtA属于同一个转运蛋白家族,并且属于同源蛋白,推测EamA也参与ALA的向外转运.转录组数据分析表明,这两个基因的表达都没有发生显著差异,推测这两个基因不受ALA的诱导.该研究鉴定出RhtA,EamA和EmrD等为ALA转运蛋白,可对其进行深入研究,探索其转运机制、转运活性和底物亲和性等.
4.2 谷氨酸棒杆菌C5途径合成ALA
Yu等[31]首次报道谷氨酸棒杆菌具有ALA C5合成途径,并证明谷氨酸棒杆菌能够合成ALA.为增加关键酶的酶活,共表达了来源于Salmonellaarizona的hemA基因和来源于大肠杆菌的hemL基因.为抑制ALA代谢为血红素,在发酵的时候添加抑制剂乙酰丙酸、马来酸和硝基苯甲酸.此外,还对培养基中Fe2+的浓度和转速进行了优化.最后,在5 L生物反应器中积累了1.79 g/L的ALA.该研究首次证明了谷氨酸棒状杆菌能够通过C5途径合成ALA,为后续代谢工程育种提供了依据.
Ramzi等[32]以C.glutamicumATCC 13032为出发菌株,比较了过表达不同来源的hemA对ALA合成的影响,结果表明源于S.typhimurium的hemA表达效果最好(204 mg/L),随后又共表达了hemA和hemL基因,ALA积累至457 mg/L,两者分别为出发菌株(17 mg/L)的11.6倍和25.9倍.随后在添加谷氨酸的条件下,摇瓶优化培养产量达到2.20 g/L.
Yu等[33]在谷氨酸棒杆菌中过表达异源hemA和hemL,测定了相关基因的转录水平.转录组分析表明糖酵解途径受到抑制,三羧酸循环、磷酸戊糖途径和氧化呼吸链增强.转录组数据分析表明二元信号转导系统HrrSA参与血红素合成的调控.而ALA作为血红素合成的前体物,其积累有助于血红素合成和呼吸代谢.当血红素存在的时候,HrrSA能够促进血红素的降解.HrrSA激活了hmuO的表达(hmuO编码血红素氧化酶),同时,HrrSA能够激活含有血红素并且与呼吸链有关的组件,抑制与血红素合成有关基因的转录,hrrA和hrrS全部上调,抵消了heme的积累.
5 结 论
近年来,通过C4和C5途径生物合成ALA均取得了较为长足的进展,为后续研究提供了必要的依据,奠定了坚实的基础,并且加快了其工业化的进程.但是,C5途径合成ALA相较C4途径而言合成途径更长,并且关键酶HemA的催化反应需要还原力NADPH.因此,如何增加反应体系的还原力也将是后续研究重点.随着转录组学技术研究的迅猛发展,对谷氨酸棒杆菌合成ALA转录组学和代谢组学的研究也将成为热门并可为其代谢工程改造提供依据.此外,随着CRISPR interference技术在谷氨酸棒杆菌中的应用[34],利用该技术对ALA脱水酶编码基因hemB的转录进行干扰以减少ALA的分解也将成为研究的热点.
[1] YANG X, LI W, PALASUBERNIAM P, et al. Effects of silencing heme biosynthesis enzymes on 5-aminolevulinic acid-mediated protoporphyrin IX fluorescence and photodynamic therapy[J]. Photochemistry & photobiology, 2015, 91(4): 923-930.
[2] ZHANG J, ZHEN K, JIAN C, et al. Optimization of the heme biosynthesis pathway for the production of 5-aminolevulinic acid inEscherichiacoli[J]. Scientific peports, 2015, 5(17): 8584-8592.
[3] ERIKSSON E S E, ERDTMAN E, ERIKSSON L A. Permeability of 5-aminolevulinic acid oxime derivatives in lipid membranes[J]. Theoretical chemistry accounts, 2016, 135(1): 1-9.
[4] LIU S, ZHANG G, LI X, et al. Microbial production and applications of 5-aminolevulinic acid[J]. Applied microbiology and biotechnology, 2014, 98(17): 7349-7357.
[5] 康振, 张俊丽, 杨森, 等. 微生物发酵生产5-氨基乙酰丙酸研究进展[J]. 生物工程学报, 2013, 29(9): 1214-1222.
[6] BEALE S I. The biosynthesis of δ-aminolevulinic acid inChlorella[J]. Plant physiology, 1970, 45(4): 504-506.
[7] SASAKI K, WATANABE K, TANAKA T, et al. 5-Aminolevulinic acid production byChlorellasp.during heterotrophic cultivation in the dark[J]. World journal of microbiology biotechnology, 1995, 11(3): 361-362.
[8] ANO A, FUNAHASHI H, NAKAO K, et al. Effect of glycine on 5-aminolevulinic acid biosynthesis in heterotrophic culture ofChlorellaregularisYA-603[J]. Journal of bioscience and bioengineering, 1999, 88(1): 57-60.
[9] SASAKI K, IKEDA S, NISHIZAWA Y, et al. Production of 5-aminolevulinic acid by photosynthetic bacteria[J]. Journal of fermentation technology, 1987, 65(5): 511-515.
[10] SASAKI K, TANAKA T, NISHIZAWA Y, et al. Production of a herbicide, 5-aminolevulinic acid, byRhodobactersphaeroidesusing the effluent of swine waste from an anaerobic digestor[J]. Applied microbiology biotechnology, 1990, 32(6): 727-731.
[11] NISHIKAWA S, WATANABE K, TANAKA T, et al.Rhodobactersphaeroidesmutants which accumulate 5-aminolevulinic acid under aerobic and dark conditions[J]. Journal of bioscience and bioengineering, 1999, 87(6): 798-804.
[12] KAMIYAMA H, HOTTA Y, TANAKA T, et al. Production of 5-aminolevulinic acid by a mutant strain of a Photosynthetic bacterium[J]. Journal of bioscience and bioengineering, 2000, 78(2): 48-55.
[13] LIN J P, FU W Q, CEN P L. Characterization of 5-aminolevulinate synthase fromAgrobacteriumradiobacter, screening new inhibitors for 5-aminolevulinate dehydratase fromEscherichiacoliand their potential use for high 5-aminolevulinate production[J]. Bioresource technology, 2009, 100(7): 2293-2297.
[14] VANDER W, M J ZEILUS J G. 5-Aminolevulinate production byEscherichiacolicontaining theRhodobactersphaeroideshemAgene[J]. Applied & environmental microbiology, 1996, 62(10): 3560-3566.
[15] FU W Q, LIN J P, CEN P L. 5-Aminolevulinate production with recombinantEscherichiacoliusing a rare codon optimizer host strain[J]. Applied microbiology biotechnology, 2007, 75(4): 777-782.
[16] CHOI C, HONG B S, SUNG H C, et al. Optimization of extracellular 5-aminolevulinic acid production fromEscherichiacolitransformed with ALA synthase gene ofBradyrhizobiumjaponicum[J]. Biotechnology letters, 1999, 21(6): 551-554.
[17] LEE D H, JUN W J, SHIN D H, et al. Effect of culture conditions on production of 5-aminolevulinic acidby recombinantEscherichiacoli[J]. Bioscience, biotechnology and biochemistry, 2005, 69(3): 470-476.
[18] QIN G, LIN J, LIU X, et al. Effects of medium composition on production of 5-aminolevulinic acid by recombinantEscherichiacoli[J]. Journal of bioscience and bioengineering, 2006, 102(4): 316-322.
[19] LIU X X, WANG L, WANG Y J, et al. D-glucose enhanced 5-aminolevulinic acid production in recombinantEscherichiacoliculture[J]. Applied biochemistry and biotechnology, 2010, 160(3): 822-830.
[20] FU W Q, LIN J P, CEN P L. Expression of ahemAgene fromAgrobacteriumradiobacterin a rare codon optimizingEscherichiacolifor improving 5-aminolevulinate production[J]. Applied biochemistry and biotechnology, 2010, 160(2): 456-466.
[21] CHOI H P, LEE Y M, YUN C W, et al. Extracellular 5-aminolevulinic acid production byEscherichiacolicontaining theRhodopseudomonaspalustrisKUGB306hemAgene[J]. Journal of microbiology biotechnology, 2008, 18(6): 1136-1140.
[22] ZHANG L, CHEN J, CHEN N, et al. Cloning of two 5-aminolevulinic acid synthase isozymes HemA and HemO fromRhodopseudomonaspalustriswith favorable characteristics for 5-aminolevulinic acid production[J]. Biotechnology letters, 2013, 35(5): 763-768.
[23] FENG L, ZHANG Y, FU J, et al. Metabolic engineering ofCorynebacteriumglutamicumfor efficient production of 5-aminolevulinic acid[J]. Biotechnology & bioengineering, 2016, 113(6): 1284-1293.
[24] YANG P, LIU W, CHENG X, et al. A new strategy for the production of 5-aminolevulinic acid in recombinantCorynebacteriumglutamicumwith high yield[J]. Applied & environmental microbiology, 2016, 82(9): 2709-2717.
[25] KANG Z, WANG Y, GU P, et al. EngineeringEscherichiacolifor efficient production of 5-aminolevulinic acid from glucose[J]. Metabolic engineering, 2011, 13(5): 492-498.
[26] BURKOVSKI A, KRAMER R. Bacterial amino acid transport proteins: occurrence, functions, and significance for biotechnological applications[J].Applied microbiology biotechnology, 2002, 58(3): 265-274.
[27] PARK J H, LEE S Y. Fermentative production of branched chain amino acids: a focus on metabolic engineering[J]. Applied microbiology biotechnology, 2010, 85(3): 491-506.
[28] KUTUKOVA E A, LIVSHITS V A, ALTMAN I P, et al. TheyeaS(leuE) gene ofEscherichiacoliencodes an exporter of leucine, and the Lrp protein regulatesits expression[J]. FEBS letters, 2005, 579(21): 4629-4634.
[29] LIVSHITS V A, ZAKATAEVA N P, ALESHIN V V, et al. Identification and characterization of the new generhtAinvolved in threonine and homoserine efflux inEscherichiacoli[J]. Research in microbiology, 2003, 154(2): 123-135.
[30] 张良程. 大肠杆菌中5-氨基乙酰丙酸转运蛋白的研究[D]. 天津: 天津科技大学,2016.
[31] YU X, JIN H, LIU W, et al. EngineeringCorynebacteriumglutamicumto produce 5-aminolevulinic acid from glucose[J]. Microbial cell factories, 2015, 14(1): 1-10.
[32] RAMZI A B, HYEON J E, KIM S W, et al. 5-Aminolevulinic acid production in engineeredCorynebacteriumglutamicumvia C5 biosynthesis pathway[J]. Enzyme & microbial technology, 2015, 81: 1-7.
[33] YU X, JIN H, CHENG X, et al. Transcriptomic analysis for elucidating the physiological effects of 5-aminolevulinic acid accumulation onCorynebacteriumglutamicum[J]. Microbiological research, 2016, 192: 292-299.
[34] CLETO S, JENSEN J V, WENDISCH V F, et al.Corynebacteriumglutamicummetabolic engineering with CRISPR interference (CRISPRi)[J]. Acssynthetic biology, 2016, 5(5): 375-380.
(责任编辑:朱小惠)
Advance on biosynthesis of 5-aminolevulinic acid
LI Zhixiang1, ZHAO Lei1, LIANG Yunlong1, ZHANG Chenglin1,2,3
(1.College of Biotechnology, Tianjin University of Science and Technology, Tianjin 300457, China; 2.National and Local United Engineering Lab of Metabolic Control Fermentation Technology, Tianjin 300457, China; 3.Tianjin Engineering Lab of Efficient and Green Amino Acid Manufacture, Tianjin 300457, China)
As the key intermediate for biosynthesis of tetrapyrrole compounds, such as heme and chlorophyll, 5-aminolevulinic acid (ALA) is widely used in agriculture and pharmaceutical field. There are mainly two pathways for ALA biosynthesis found in microorganisms, C4 and C5 pathway. In this review, metabolic engineering and fermentation engineering research on ALA biosynthesis byEscherichiacoliandCorynebacteriumglutamicumvia C4 and C5 pathway was summarized.
ALA; C4 pathway; C5 pathway; metabolic engineering
2017-04-24
国家自然科学基金资助项目(31300069);天津科技大学青年教师创新基金(2016LG07);天津市科委科技特派员项目(15JCTPJC62800);天津市大学生创新创业训练计划项目(201710057039)
李智祥(1991—),男,山东日照人,硕士研究生,研究方向为5-氨基乙酰丙酸生产菌株代谢工程育种,E-mail: 415323662@qq.com.通信作者:张成林副教授,E-mail: zcl@tust.edu.cn.
TQ922
A
1674-2214(2017)03-0178-05

