ACE基因I/D多态性与心房颤动相关性的Meta分析
皮阳,钱成,魏宝柱,曹建雷,冯鸿,王扬淦
ACE基因I/D多态性与心房颤动相关性的Meta分析
皮阳1,钱成2,魏宝柱2,曹建雷2,冯鸿2,王扬淦2
目的 评价ACE I/D基因多态性对心房颤动(房颤)发病风险的影响。方法 检索PubMed、Web of Science、谷歌学术、中国知网、万方数据库从建库到2016年3月发表的关于ACE I/D基因多态性与房颤发病风险相关性的研究。对纳入的相关文献使用STATA 12.0进行统计学分析,使用固定效应模型或随机效应模型合并的比值比(OR)和95%置信区间(CI)来评价ACE I/D基因多态性和房颤发病风险,同时检测发表偏倚。对各纳入研究按种族,房颤类型,年龄进行亚组分析,并通过敏感性分析寻找可能的异质性来源。结果 本文纳入了20篇有关ACE I/D基因多态性与房颤发病风险的研究,其中病例组3006例,对照组4861例,Meta分析结果显示,显性基因模型合并后的OR值为1.22(95%CI:0.97~1.52),等位基因模型合并后的OR值为1.22(95%CI:1.04~1.43),隐性基因模型合并后的OR值为1.41(95%CI:1.13~1.76),加性模型合并后的OR值为1.48(95%CI:1.11~1.98),共显性基因模型(DD vs. DI:OR=1.36,95%CI:1.09~1.70;DI vs. II:OR=1.12(95%CI:0.89~1.39)。在以民族为亚组的亚组分析中,高加索人可以在隐性基因模型中发现ACE I/D基因多态性与房颤有很强的联系(OR=1.53,95%CI:1.09~2.95);在以患者疾病背景为亚组的亚组分析中,高血压患者可在所有五个模型中发现较强的联系;在以年龄为亚组的亚组分析中,年龄≥65岁的患者可以在隐性基因模型和共显性基因模型中发现较强联系。研究中未发现有明显发表偏倚。结论 ACE I/D基因多态性是房颤发病的一个重要危险因素,DD基因型总体上可增加患者房颤发病风险,特别是在高加索人和高血压患者中,同时DD基因型可明显增加65岁以上病例房颤的发病风险,但在65岁以下病例中并未增加其房颤发病风险。
ACE;插入/缺失;基因多态性;心房颤动
目前心房颤动是临床上最常见的心律失常,房颤患者由于心房节律紊乱,血流动力学发生改变,左心房内易形成附壁血栓,随着血流的不断冲击,左心耳血栓易发生脱落,可造成脑动脉栓塞、周围动脉栓塞、肺动脉栓塞、急性心肌梗死、心力衰竭、心源性猝死等严重后果。房颤的发病机制复杂,大多数器质性心脏病,如高血压、风湿性瓣膜病、急性心肌梗死[1]、扩张性心肌病、心肌肥厚[2]等都可以导致心房电重构和电生理紊乱,进而触发房颤。
肾素-血管紧张素-醛固酮系统(RAAS)是心血管疾病的发生发展的重要影响因素。血浆中血管紧张素转化酶(ACE)催化血管紧张素Ⅰ(AngⅠ)转化为AngⅡ。AngⅡ可以与血管紧张素Ⅱ受体-1(AT1)结合,发挥收缩血管和兴奋交感神经系统作用,进而导致血压升高和心室重构。研究辨明,ACE基因I/D基因多态性与心血管系统疾病有密切关系[3]。血浆中ACE水平与ACE基因16号基因内区插入(I)和缺失(D)一个长度为287个碱基对的DNA序列密切相关[4],形成DD,DI,II型三种基因型,ACE基因DD型携带者血浆中ACE的含量比II型基因型携带者大约高出两倍,而DI基因型携带者血浆中ACE的含量介于DD和II之间,提示ACE基因I/D基因多态性是D等位基因和I等位基因的共显性表达[5]。
进来研究证实,房颤的诱发是与RAAS激活紧密相连的,尤其是ACE基因I/D基因多态性。在很多房颤患者中可以观察到心房内和心房组织中ACE高表达。ACE表达增加可以间接增加血浆AngⅡ的含量,而AngⅡ可以通过激活丝裂原活化蛋白激酶(MAPK)信号通路[6,7],引起成纤维细胞增殖和心肌肥大[8]。研究发现,有不同ACE基因型的患者进行房颤的相关治疗时对ACEI和ARB类药物的反应性有明显差异[9,10],所以对ACE基因多态性的研究将会给临床治疗带来更多指导。
但由于研究的病例数,病例的种族,人群基本情况不同会造成结果的不统一,一些已发表文章的结果更是存在一定的矛盾,极大影响了结论的准确性,因此需通过Meta分析来增加样本含量来寻找到ACE基因I/D基因多态性和房颤发病风险的准确关系。我们进行了这篇包括3006个房颤病例和4861个对照病例的20项临床研究的Meta分析,为临床房颤的诊治提供更多循证医学证据。
1 资料和方法
1.1 文献纳入标准 将本文所纳入的研究限定为病例对照研究,并满足如下纳入标准:①研究中评估了ACE基因I/D基因多态性与房颤发病风险之间的关系;②病例组房颤都是根据欧洲心脏病学会房颤管理指南诊断的。所有房颤的诊断都基于患者的症状,体征,病史和12导联持续性或临时心电图的检查结果;③病例组病例均有房颤病史,但对照组病例无房颤病史,且病例组和对照组其他病史和治疗方案无显著性差异;④病例组和对照组都提供了ACE基因的基因型频率和等位基因频率;⑤研究为公开发表的文章,可搜索到全文并可以提取出完整的基因型频率和等位基因频率等数据;⑥研究中对照组病例基因型分布符合哈迪-温伯格平衡(HWE)定律。
1.2 文献排除标准 ①动物实验;②搜索到的文章为综述或Meta分析;③无法获取全文或文章实验数据不全,联系作者却未收到回复的文章;④相同机构发表的2项研究,研究过程雷同,研究结果相似的;⑤非病例对照研究或实验组与对照组病例间存在较大差异的。
1.3 文献检索方法 检索PubMed、Web of Science、谷歌学术、中国知网、万方数据库,从建库到2016年3月公开发表的关于ACE基因I/D基因多态性与房颤发病风险相关性的研究。人工检索每一篇所纳入的文献的参考文献,通过已查找到的参考文献以及在数据库搜索时每次搜索到的相关文献进行再次搜索。如果搜索到的相关文献只有摘要或相关数据不完整,则通过电子邮件与文章作者进行联系,以求得到完整的文章及实验数据。在中文数据库中检索时,检索词为“血管紧张素转化酶”、“插入/缺失”、“基因多态性”、“心房颤动”;在英文数据库中进行检索时,检索词为“ACE”、“angiotensin converting enzyme”、“I/D polymorphisms”、“atrial fibrillation”。所有纳入的文献所用语言限定为中文和英文。
1.4 资料提取 两名独立研究者分别独立分析所有符合纳入标准的研究,并提取各研究有关病例和研究特点的相关数据。在数据提取过程和分析过程中的分歧由第三位研究者通过讨论或咨询专家等方式进行调节。从各符合纳入标准的研究中提取以下的相关信息:①文章第一作者,文章发表年份和实验设计方案;②实验组和对照组病例的相关信息:国籍、病例种族、病例数目、平均年龄、病例组房颤的类型及其他基本情况;③病例组和对照组的基因型分布和等位基因频率,对照组的HWE情况。
1.5 统计学分析 用STATA软件(12.0版本)合并各纳入研究的相关数据,并用比值比(OR)和95%置信区间(CI)来评估ACE基因I/D基因多态性和房颤发病风险之间的关系。ACE基因I/D基因多态性分别使用五种基因模型来评价,分别是显性基因模型(DD+DI vs. II),隐性基因模型(DI+II vs. DD),共显性基因模型(DI vs. DD,II vs. DI),加性基因模型(DD vs. II)和等位基因模型(D vs. I)。用χ2检验评估对照组的基因频率是否符合HWE[11],采用I2统计量行异质性分析,当存在显著异质性(P<0.1,I2>50%)的时候,采用随机效应模型,否则采用固定效应模型。通过敏感性分析(每次排除一项研究)来评估每个功效指标的稳定性。根据纳入病例的种族,房颤类型及病例的平均年龄,将研究分为若干亚组,进行亚组分析。使用Begg’s漏斗图和Egger检验来评价纳入的研究的发表偏倚,当P<0.05是认为研究存在发表偏倚。
2 结果
2.1 检索结果及纳入研究特征 通过数据库检索共检索到78篇相关研究,其中英文30篇,中文48篇。通过查看摘要或全文,最终我们在此篇meta分析中共纳入20项研究[12-31],其中13篇英文文献,7篇中文文献。文献筛选过程如图1所示。
20项纳入研究的特征如表1所示。我们共纳入了7867组病例,其中包括实验组病例3006例,对照组病例4861例。13项研究探究了亚洲人中ACE基因I/D基因多态性与房颤发病风险的关系,7项研究探究了高加索人中ACE基因I/D基因多态性与房颤发病风险间的关系。所有20项研究均为全文发表。各项研究对照组都进行了哈迪-温伯格平衡,各项研究具体的基因型频率,等位基因频率如表2所示。
2.2 各基因模型汇总分析结果 各模型中I2>50%,所以我们采用随机效应模型来合并各项研究的OR值和95%CI。当使用显性基因模型(OR=1.22,95%CI=0.97~1.52,P>0.05)时ACE基因I/D基因多态性与房颤发病风险无显著性差异。而采用隐性基因模型(OR=1.41,95%CI:1.13~1.76,P<0.05),加性基因模型(OR=1.48,95%CI:1.11~1.98,P<0.05)和等位基因模型(OR=1.22,95%CI:1.04~1.43,P<0.05)时,ACE基因I/D基因多态性与房颤发病风险有显著性差异。对于共显性基因模型,当DD vs. DI时有显著性差异(OR=1.36,95%CI:1.09~1.70,P<0.05),当DI vs. II时无显著性差异(OR=1.12,95%CI:0.89~1.39,P<0.05)。
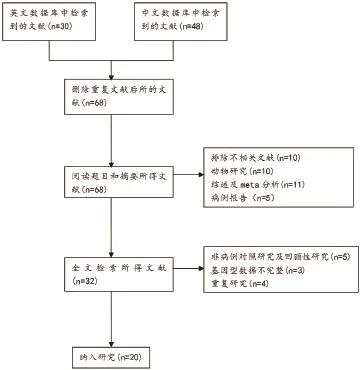
图1 文献筛选及检索流程图
2.3 发表偏倚评估 所有纳入研究的发表偏倚使用Begg’s漏斗图和Egger检验进行评价。Begg’s 漏斗图和Egger检验结果如图2、图3。Begg检验和Egger检验的结果均提示无发表偏倚。
2.4 敏感性分析和亚组分析 为了寻找研究中造成异质性的原因,我们对纳入研究进行了敏感性分析和亚组分析。敏感性分析的结果如图4所示,可以看出剔除所纳入的研究中的某一项研究,其余研究合并后的结果并未见显著差异。
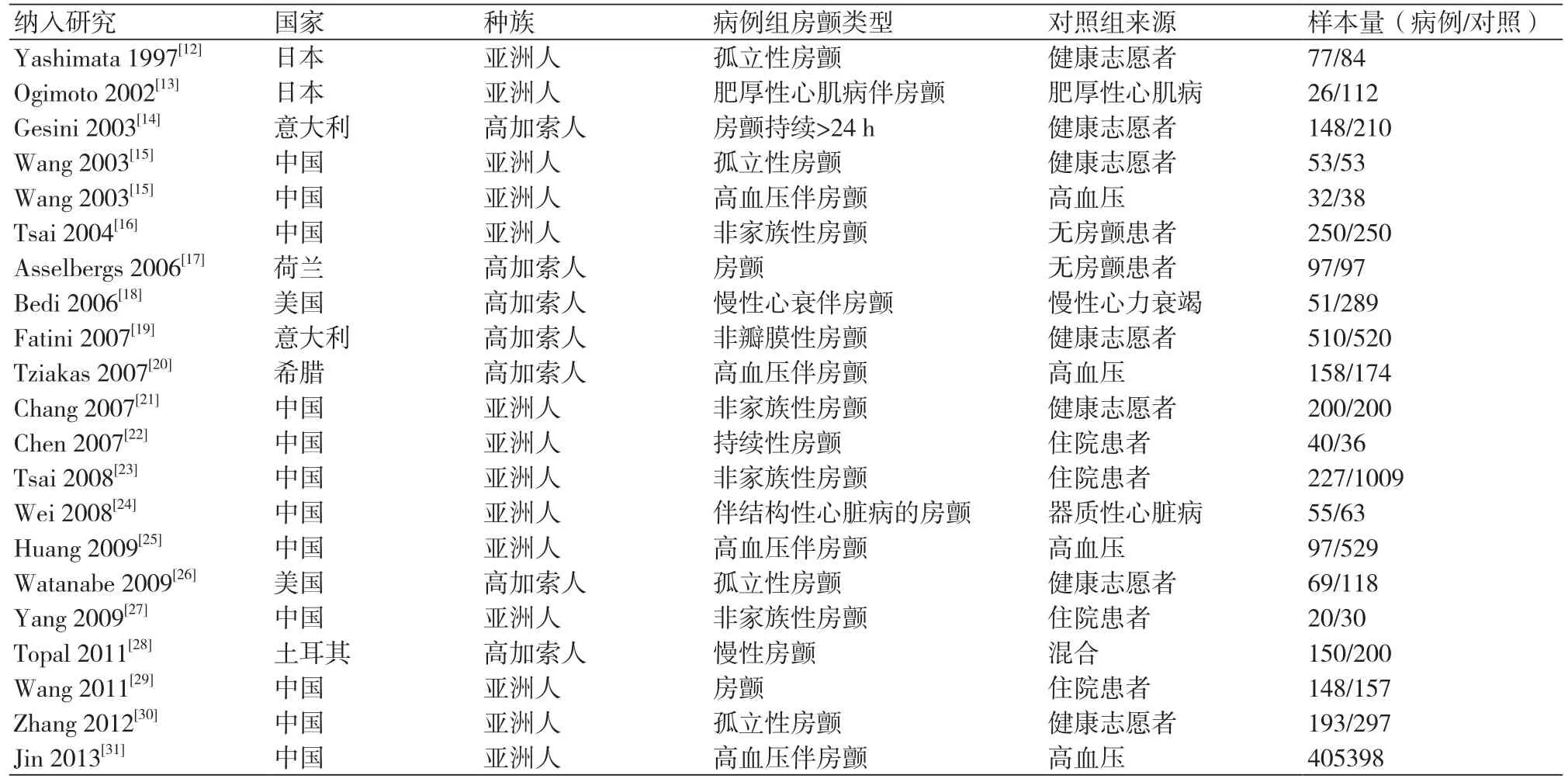
表1 纳入研究的基本特征
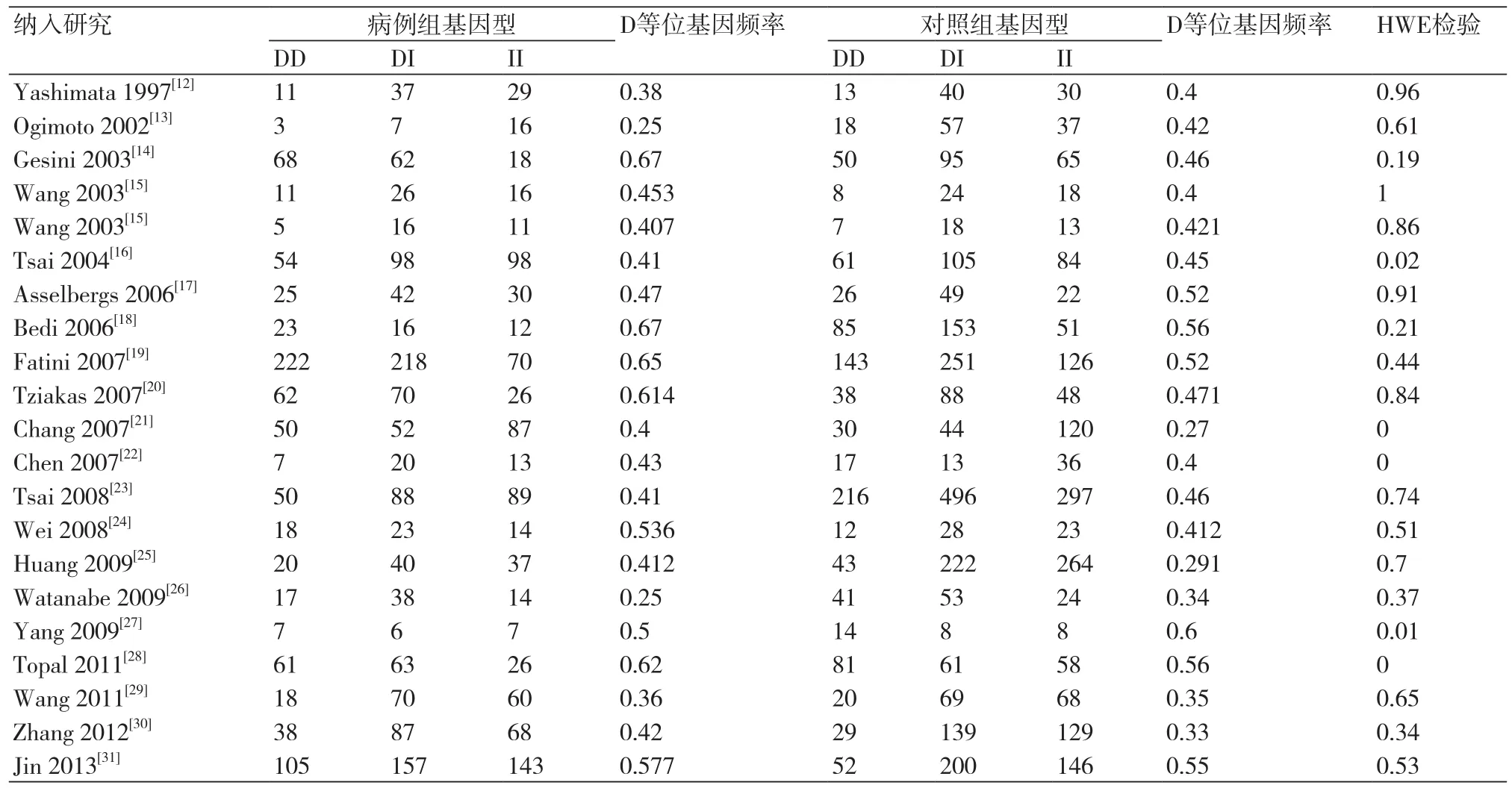
表2 纳入研究各组基因型频率和D等位基因频率
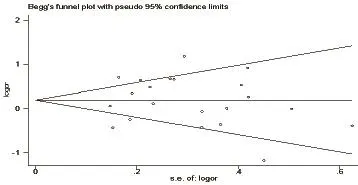
图2 Begg’s 漏斗图
在有关种族的亚组分析中,将纳入研究分为高加索人组和亚洲人组,在高加索人组中,ACE基因I/D基因多态性在隐性基因模型(OR=1.53,95%CI:1.06~2.21,P<0.05),加性基因模型(OR=1.79,95%CI:1.09~2.95,P<0.05)和等位基因模型(OR=1.37,95%CI:1.05~1.79,P<0.05)中可见组间存在显著性差异,但在显性基因模型,共显性基因模型各组间则无显著性差异。结果如图5~10所示。提示ACE基因DD基因型可显著增加高加索人的发病风险,而DI、II基因型并未增加房颤发病风险。在亚洲人中,所有基因模型中并未观察到显著性差异,提示ACE基因I/D基因多态性可能对亚洲人房颤发病风险并没有显著影响。
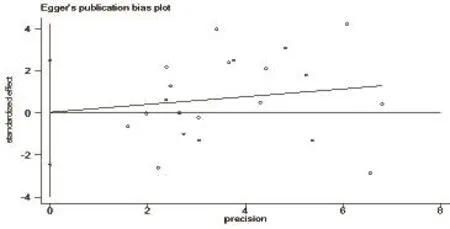
图3 Egger检验
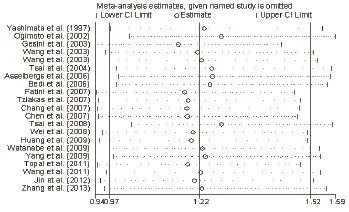
图4 敏感性分析
在房颤类型的亚组分析中,我们根据各研究中实验组病例的房颤类型将纳入研究分为高血压伴房颤组,孤立性房颤组,混合性房颤(房颤伴其他心脏疾病)组。通过亚组分析可以发现,高血压伴房颤组ACE基因I/D基因多态性在隐性基因模型(OR=2.32,95%CI:1.77~3.05,P<0.05),共显性基因模型DD vs. II(OR=2.30,95%CI:1.74~3.02,P<0.05),加性基因模型(OR=2.41,95%CI:1.66~3.51,P<0.05),等位基因模型(OR=1.49,95%CI:1.22~1.83,P<0.05)中均有显著性差异。在孤立性房颤和混合性房颤患者中,所有基因模型中均未发现显著性差异,提示ACE基因I/D基因多态性只影响部分类型的房颤的发病风险,ACE基因I/D基因多态性对不同心血管背景的患者房颤的发病风险的影响截然不同。

图5 ACE基因I/D基因多态性与房颤发病风险亚组分析(DD+DI vs. II)
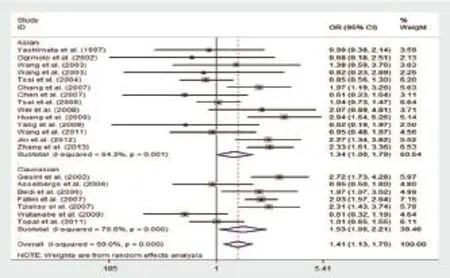
图6 ACE基因I/D基因多态性与房颤发病风险亚组分析(DD vs. DI+II)
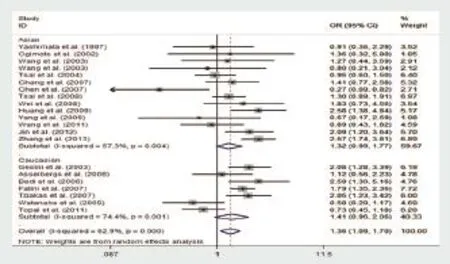
图7 ACE基因I/D基因多态性与房颤发病风险的亚组分析(DD vs. DI)
此外,我们还按照年龄进行亚组分析,将平均年龄为65岁以上的研究归为一个亚组,将65岁以下的归为另一个亚组。在65岁以上病例的亚组分析中,在隐性基因模型(OR=1.54,95%CI:1.15~2.06,P<0.05),共显性基因模型DD vs. DI (OR=1.52,95%CI:1.17~1.97,P<0.05),加性基因模型(OR=1.64,95%CI:1.08~2.48,P<0.05)中可观察到显著性差异,而在65岁以下病例的亚组分析中均未观察到显著性差异。提示ACE基因I/D基因多态性对房颤发病风险的影响对于不同年龄段的病例是不同的。所有相关数据都
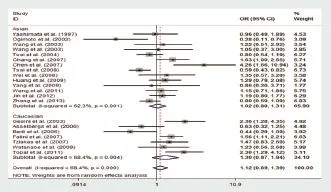
图8 ACE基因I/D基因多态性与房颤发病风险的亚组分析(DI vs. II)

图9 ACE基因I/D基因多态性与房颤发病风险的亚组分析(DD vs. II)
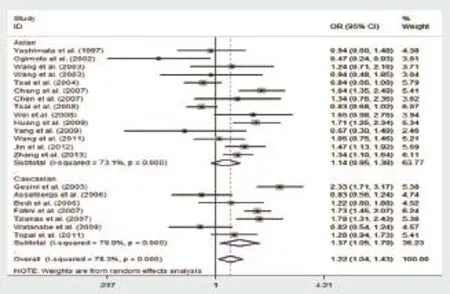
图10 ACE基因I/D基因多态性与房颤发病风险的亚组分析(D vs. I)
如表3中所示。
3 讨论
本文搜索到了所有在中英文数据库中可查找到的有关ACE基因I/D基因多态性与房颤发病风险之间联系的文章,共包括7867组病例,其中包括实验组病例3006例,对照组病例4861例。我们发现ACE基因DD基因型可显著增加高加索人的房颤发病风险,但在亚洲人中却并未显著增加房颤发病风险,提示相同基因型的病例由于种族差异和生存环境的不同,房颤发病风险也有所不同。另一方面,ACE基因I/D基因多态性对不同心血管背景的病例的发病风险有不同的影响。ACE基因DD基因型可以显著增加高血压患者的房颤发病风险,但对于伴有其他心脏病的患者并未观察到明显增加的房颤风险。同时ACE基因DD基因型也并未增加无任何心血管疾病的病例的房颤的发病风险。除此之外,ACE基因DD基因型可明显增加65岁以上患者房颤的发病风险,但并不增加65岁以下患者房颤的发病风险,提示ACE基因I/D基因多态性对房颤发病风险的影响在不同年龄段的患者中是不同的,通常是对患者房颤发病风险造成慢性影响,随着年龄增加,DD基因型携带者患房颤的风险会显著增高,而DI和II基因型携带者房颤发病风险并无明显变化。
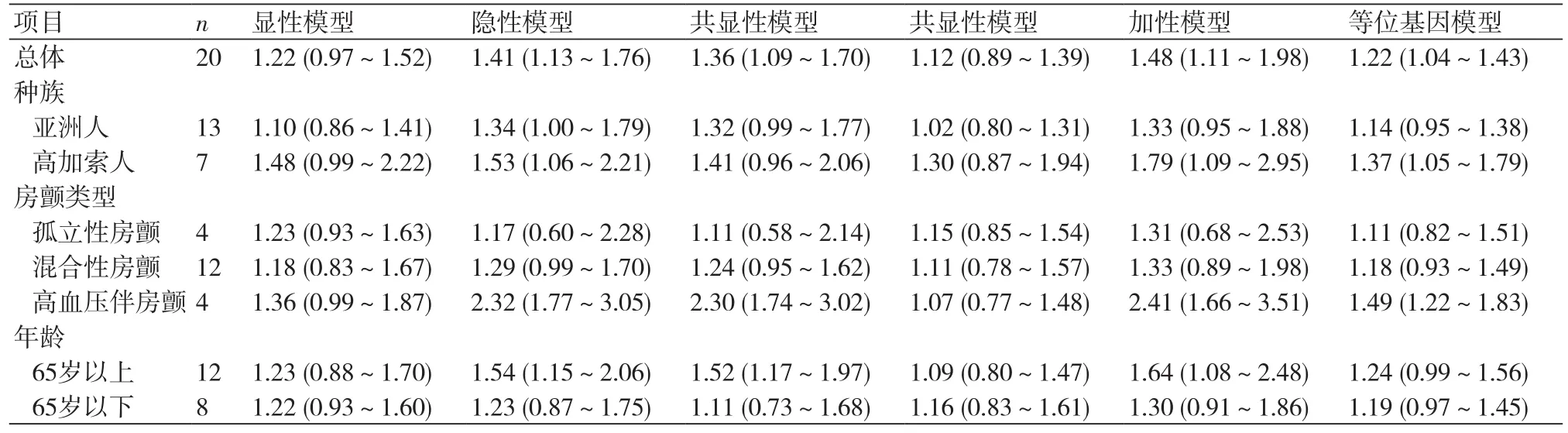
表3 ACE基因I/D基因多态性与房颤发病风险关系亚组分析数据
在房颤发生发展过程中,RAAS系统扮演了重要角色。RAAS系统已被证实可促进炎症发生,氧化应激和纤维化[32]。在人体心脏,AngⅠ通过血浆或组织内ACE转化是生成AngⅡ的主要途径,心脏中AngⅡ含量增加可以促进心脏重构,特别是心肌间质纤维化。而ACE基因可调控血浆和组织中ACE的表达,ACE基因DD基因型相比于Ⅱ基因型可显著增加血浆中ACE的含量,而心房中ACE含量的增加可以引起心房中AngⅡ的含量增加。AngⅡ通过增加心房内压力,并激活MAPK信号通路,导致成纤维细胞增殖和心肌肥大[8],引起心房纤维化和心房电重构,引起电传导紊乱,促进房颤的发生和发展。
Xiao等[33]2010年进行了一项纳入12项有关ACE基因I/D基因多态性与房颤发病风险临床病例对照研究的Meta分析;Liu等[34]之后也进行了一项关于ACE基因I/D基因多态性与房颤发病风险的研究,共纳入了18项病例对照研究,但他们没有足够的证据说明ACE基因I/D基因多态性与房颤发病风险的明确关系,因为纳入研究的异质性较大。我们在其研究的基础上进行了这篇Meta分析,并增加了2012~2015年发表的有关这一主题的最新研究,纳入了更多来自不同地区,不同种族,不同心血管背景,不同年龄的病例,并从多角度对ACE不同基因型间对房颤发病影响的联系进行评价以增加统计学效能,使结果更加准确和真实。
然而,Meta分析中部分亚组之间仍存在较明显的异质性,因为各纳入研究之间存在着差异,而且不能用偶然因素来进行解释。所以我们进行了亚组分析和敏感性分析去寻找异质性的来源。敏感性分析结果显示,去除20项研究中的任何一项研究并不会对合并后的结果造成较大影响,提示所纳入研究具有较好的稳定性。进行亚组分析后,我们发现,种族不同和年龄的不同并不会导致Meta分析明显的异质性,而且不同对照组病例选择方式也并不会引起明显的异质性。异质性最可能的来源是不同研究中患者不同的心血管背景,不同患者选择的纳入标准和每项研究不同的样本量,及不同研究之间不同的实验设计方案。
本文有一定的局限性。首先,并没用提到基因-基因和基因-环境之间的相互关系,只进行了单核苷酸基因多态性的分析,而ACE基因其他的基因多态性或其他基因的多态性是否与ACE基因I/D基因多态性有连锁反应尚不清楚。同时,不同生存环境对同一基因型的表型是否有明确影响,本文也并没有进行相关分析,因为对这些领域研究的文章较少,相关机制较为复杂,相关实验数据也较为缺乏。第二,我们在数据库中搜索到的一些研究中并没有得到完整的数据或只能获得文章摘要而无法获得文章全文,我们努力去联系文章作者,但没有得到回应,所以不得不将这些研究排除在此Meta分析之外,这可能会造成一些有代表性的数据的缺失和一定程度的偏倚。除此之外,我们只能在数据库中搜索到公开发表的文章,而阴性结果可能并不会发表,可能会造成一定程度的发表偏倚,虽然我们从Begg’s漏斗图和Egger检验并没有观察到明显的发表偏倚。
总之,此Meta分析表明,ACE基因DD基因型总体上可增加房颤的发病风险,而DI和II基因型并未显著增加房颤发病风险。但此种联系在高加索人中比较明显,而在亚洲人当中此种联系并不明显。在以年龄为分组标准的亚组分析中,只有65岁以上年龄的病例中ACE基因DD基因型可以明显增加其房颤发病风险,而65岁以上年龄的病例中并未明显增加房颤发病风险。对于高血压患者,含有一个或两个D等位基因均可显著增加高血压患者房颤的发病风险。对于有其他心血管疾病的患者,ACE基因多态性并未被证实可以明显增加房颤的发病风险。由于纳入研究数量有限,且多为回顾性研究,未来需要更多的多中心大样本前瞻性研究与回顾性研究共同验证,共同寻找更加准确全面的ACE基因I/D基因多态性对房颤发病风险的影响。
[1] Kannel WB,Abbott RD,Savage DD,et al. Epidemiologic features of chronic atrial fibrillation: the Framingham study[J]. N Engl J Med, 1982,306(17):1018-22.
[2] Benjamin EJ,Levy D,Vaziri SM,et al. Independent risk factors for atrial fibrillation in a population-based cohort. The Framingham Heart Study[J]. JAMA,1994,271(11):840-4.
[3] Sayed-Tabatabaei FA,Oostra BA,Isaacs A,et al. ACE polymorphisms[J]. Circ Res,2006,98(9):1123-33.
[4] Rigat B,Hubert C,Corvol P,et al. PCR detection of the insertion/ deletion polymorphism of the human angiotensin converting enzyme gene (DCP1) (dipeptidyl carboxypeptidase 1)[J]. Nucleic Acids Res,1992,20(6):1433.
[5] Rigat B,Hubert C,Alhenc-Gelas F,et al. An Insertion/Deletion Polymorphism in the Angiotensin I-converting Enzyme Gene Accounting for Half the Variance of Serum Enzyme Levels[J]. J Clin Invest,1990,86(4):1343-6.
[6] Goette A,Staack T,Rocken C,et al. Increased Expression of Extracellular Signal-Regulated Kinase and Angiotensin-Converting Enzyme in Human Atria During Atrial Fibrillation[J]. J Am Coll Cardiol,2000,35(6):1669-77.
[7] Li D,Shinagawa K,Pang L,et al. Effects of Angiotensin-Converting Enzyme Inhibition on the Development of the Atrial Fibrillation Substrate in Dogs With Ventricular Tachypacing-Induced Congestive Heart Failure[J]. Circulation,2001,104(21):2608-14.
[8] KomuroS I,Kaida T,Shibazaki Y,et al. Stretching Cardiac Myocytes Stimulates Protooncogene Expression[J]. J Biol Chem,1989,265(7):3595-8.
[9] Healey JS,Baranchuk A,Crystal E,et al. Prevention of atrial fibrillation with angiotensin-converting enzyme inhibitors and angiotensin receptor blockers: a meta-analysis[J]. J Am Coll Cardiol, 2005,45(11):1832-9.
[10] Darbar D,Motsinger AA,Ritchie MD,et al. Polymorphism modulates symptomatic response to antiarrhythmic drug therapy in patients with lone atrial fibrillation[J]. Heart Rhythm,2007,4(6):743-9.
[11] Schaid DJ,Jacobsen SJ. Biased tests of association: comparisons of allele frequencies when departing from Hardy-Weinberg proportions[J]. Am J Epidemiol,1999,149(8):706-11.
[12] Yamashita T,Hayami N,Ajiki K,et al. Is ACE gene polymorphism associated with lone atrial fibrillation?[J]. Jpn Heart J,1997,38(5):637-41.
[13] Ogimoto A,Hamada M,Nakura J,et al. Relation between angiotensin-converting enzyme II genotype and atrial fibrillation in Japanese patients with hypertrophic cardiomyopathy[J]. J Hum Genet,2002,47(4):184-9.
[14] Gensini F,Padeletti L,Fatini C,et al. Angiotensin-converting enzyme and endothelial nitric oxide synthase polymorphisms in patients with atrial fibrillation[J]. Pacing Clin Electrophysiol,2003,26(1 Pt 2):295-8.
[15] 王淑敏,萍张,陈凤荣,等. 心房颤动与血管紧张素-1转换酶基因多态性的相关性研究[J]. 中国实用内科杂志,2003,23(9):537-8.
[16] Tsai CT,Lai LP,Lin JL,et al. Renin-angiotensin system gene polymorphisms and atrial fibrillation[J]. Circulation,2004,109(13):1640-6.
[17] Asselbergs FW,Moore JH,van den Berg MP,et al. A role for CETP TaqIB polymorphism in determining susceptibility to atrial fibrillation: a nested case control study[J]. BMC Med Genet,2006,7:39.
[18] Bedi M,McNamara D,London B,et al. Genetic susceptibility to atrial fibrillation in patients with congestive heart failure[J]. Heart Rhythm,2006,3(7):808-12.
[19] Fatini C,Sticchi E,Gensini F,et al. Lone and secondary nonvalvular atrial fibrillation: role of a genetic susceptibility[J]. Int J Cardiol,2007, 120(1):59-65.
[20] Tziakas DN,Chalikias GK,Stakos DA,et al. Effect of angiotensinconverting enzyme insertion/deletion genotype on collagen type I synthesis and degradation in patients with atrial fibrillation and arterial hypertension[J]. Expert Opin Pharmacother,2007,8(14):2225-34.
[21] 常慧颖,杨树森,韩薇. 非家族性心房颤动与血管紧张素基因I/D多态性关系的研究[J]. 黑龙江医学,2007,31(10):731-2.
[22] 陈旭娇,凌锋,沈法荣. 血管紧张素转换酶基因多态性与房颤的相关性研究[J]. 浙江医学,2007,29(9):908-10.
[23] Tsai CT,Hwang JJ,Chiang FT,et al. Renin-angiotensin system gene polymorphisms and atrial fibrillation: a regression approach for the detection of gene-gene interactions in a large hospitalized population[J]. Cardiology,2008,111(1):1-7.
[24] 魏玲,魏文婷,卢国良,等. ACE基因插入/缺失及β2肾上腺素受体基因多态性与房颤的相关性研究[J]. 西南国防医药,2008,18(4):495-8.
[25] Huang M,Gai X,Yang X,et al. Functional polymorphisms in ACE and CYP11B2 genes and atrial fibrillation in patients with hypertensive heart disease[J]. Clin Chem Lab Med,2009,47(1):32-7.
[26] Watanabe H,Kaiser DW,Makino S,et al. ACE I/D polymorphism associated with abnormal atrial and atrioventricular conduction in lone atrial fibrillation and structural heart disease: implications for electrical remodeling[J]. Heart Rhythm,2009,6(9):1327-32.
[27] 杨纯玉,梁旭,黄璐璐,等. ACE及AGT基因多态性与非家族性房颤的相关性卑[J]. 广东医学,2009,30(1):86-7.
[28] Topal NP,Ozben B,Hancer VS,et al. Polymorphisms of the angiotensin-converting enzyme and angiotensinogen gene in patients with atrial fibrillation[J]. J Renin Angiotensin Aldosterone Syst,2011,12(4):549-56.
[29] 王亚珠,李瑛,范晋奇,等. ACE基因I/D和ACE2基因A9570G多态性与心房颤动的相关性研究[J]. 解放军医学杂志,2011,36(9):904-8.
[30] Zhang XL,Wu LQ,Liu X,et al. Association of angiotensin-converting enzyme gene I/D and CYP11B2 gene -344T/C polymorphisms with lone atrial fibrillation and its recurrence after catheter ablation[J]. Exp Ther Med,2012,4(4):741-7.
[31] 靳钰,邱春光,侯允天,等. 血管紧张素转换酶基因插入缺失多态性与原发性高血压患者合并心房颤动的相关性研究[J]. 中华老年心脑血管病杂志,2013,15(9):906-8.
[32] Rahman ST,Lauten WB,Khan QA,et al. Effects of eprosartan versus hydrochlorothiazide on markers of vascular oxidation and inflammation and blood pressure (renin-angiotensin system antagonists, oxidation, and inflammation)[J]. Am J Cardiol,2002,89(6):686-90.
[33] Xiao P,Ling Z,Woo K,et al. Renin-angiotensin system-related gene polymorphisms are associated with risk of atrial fibrillation[J]. Am Heart J,2010,160(3):496-505.
[34] Liu T,Korantzopoulos P,Xu G,et al. Association between angiotensinconverting enzyme insertion/deletion gene polymorphism and atrial fibrillation: a meta-analysis[J]. Europace,2011,13(3):346-54.
本文编辑:翁鸿,田国祥
The impact of polymorphism of angiotensin converting enzyme gene I/D on atrial fibrillation: A Meta- analysis
Pi Yang*, Qian Cheng, Wei Baozhu, Cao Jianlei, Feng Hong, Wang Yanggan.
*Intensive Care Unit, Nanjing Drum Tower Hospital, The Affiliated Hospital of Nanjing University, Nanjing 210008, China.
Corresponding author: Wang Yanggan,E-mail:ygwang2013@163.com
Objective To evaluate the impact of angiotensin converting enzyme (ACE) I/D polymorphism on the risk of atrial fibrillation (AF). Methods Eligible studies about the impact of ACE I/D polymorphism on the risk of atrial fibrillation were searched in PubMed, Web of Science, Google Scholar, CNKI and Wanfang from when the databases established to 2016.3. STATA 12.0 is used to statistically analyze the relevant articles. The associations between ACE I/D polymorphism and AF risk were estimated by pooled odds ratio (OR) and 95% confidence interval (CI) using a fixed or random-effects model. Publication bias was also calculated. Subgroup analysis and sensitivity analysis are used to find the possible source of heterogeneity. And subgroup analyses are conducted with respect to ethnicity, AF type and age. Results A total of 20 studies which evaluated the association between ACE I/D polymorphisms and AF were included, with 3006 cases and 4861 controls. The pooled OR was 1.22 (95%CI:0.97~1.52) for dominant model, 1.22 (95%CI: 1.04~1.43) for allele model, 1.41 (95%CI: 1.11~1.98) for recessive model, 1.48 (95%CI: 1.11~1.98) for additive model, 1.36 (DD vs. DI, 95% CI: 1.09~1.70) and 1.12 (DI vs. II, 95% CI: 0.89 to 1.39) for codominant gene model, respectively. In subgroup analysis, a strong association was found in Caucasians in recessive model (OR=1.53, 95%CI: 1.09~2.95). Strong associations were also found in hypertensive patients of all five models and subjects ≥ 65-year-old of recessive model and co-dominant model. No publication bias was found in this study. Conclusion ACE I / D polymorphism is an important risk factor for the onset of atrial fibrillation. DD genotype significantly increases the risk of atrial fibrillation, especially in the Caucasus and hypertensive patients. Meanwhile, DD genotype significantly increase AF risk in patients over 65 years old, but no association was observed in patients below 65 years old.
ACE; Insertion/Deletion; Polymorphism; Atrial fibrillation
R541.75
A
1674-4055(2017)08-0900-07
1210008 南京,南京大学医学院附属鼓楼医院重症医学科;2430071 武汉,武汉大学中南医院心血管内科
王扬淦,E-mail:ygwang2013@163.com
10.3969/j.issn.1674-4055.2017.08.02

