碱胁迫相关基因GsWRKY15的克隆及其转基因苜蓿的耐碱性分析
朱娉慧陈冉冉于 洋 宋雪薇 李慧卿 杜建英 李 强丁晓东 朱延明
东北农业大学农业生物功能基因重点实验室, 黑龙江哈尔滨 150030
碱胁迫相关基因GsWRKY15的克隆及其转基因苜蓿的耐碱性分析
朱娉慧**陈冉冉**于 洋 宋雪薇 李慧卿 杜建英 李 强丁晓东 朱延明*
东北农业大学农业生物功能基因重点实验室, 黑龙江哈尔滨 150030
WRKY蛋白属于锌指型转录调控因子, 能够参与植物多种逆境响应。本研究利用前期野生大豆盐碱胁迫RNA-seq测序数据, 从构建的碱胁迫基因调控网络中筛选并克隆到GsWRKY15基因。分析GsWRKY15在碱胁迫下野生大豆根中的表达模式, 发现该基因受碱胁迫诱导显著上调表达, 且在胁迫后1 h表达量最高。分析GsWRKY15基因在野生大豆各组织中的表达特异性, 发现该基因在各组织中均有表达, 花中表达量最高。采用根癌农杆菌侵染苜蓿子叶节方法, 将GsWRKY15转化肇东苜蓿, 获得39株抗性植株。通过PCR、Southern blot和RT-PCR方法分析抗性植株, 获得了超量表达GsWRKY15基因的转基因株系并对其进行了耐碱性分析。在150 mmol L–1NaHCO3处理2周后转基因苜蓿生长状态良好, 而非转基因苜蓿出现萎蔫、变黄, 甚至死亡; 非转基因苜蓿的相对质膜透性和丙二醛含量显著高于转基因苜蓿, 而叶绿素含量显著低于转基因苜蓿; 同时分析碱胁迫下转基因植株中胁迫相关基因的表达模式, 发现H+-Ppase、NADP-ME、KIN1、RD29A基因的表达量高于非转基因苜蓿。结果表明GsWRKY15基因的超量表达能够显著增强苜蓿的耐碱能力。
野生大豆; GsWRKY15; 肇东苜蓿; 农杆菌; 耐碱性
非生物胁迫严重制约着农业的发展, 其中土地盐碱化是限制粮食产量, 影响植物生长和分布的主要非生物环境因素。盐碱胁迫影响植物生长、发育、繁殖等过程进而导致作物减产或绝收。随着全球人口的不断增加, 粮食供需矛盾日益尖锐, 开发和利用盐碱地, 增加耕地面积, 提高盐碱地作物产量,成为解决粮食问题的有效手段之一[1]。紫花苜蓿(Medicago sativa)素有“牧草之王”的美称, 是我国重要的豆科植物, 具有产量高, 适应性广的特性, 是我国重要的饲料作物, 同时也是改良和利用盐碱化土地的重要作物之一[2]。因此培育具有耐盐碱特性的苜蓿新品种, 具有重要的现实意义[3]。
已有大量文献报道 WRKY类转录因子在植物响应生物胁迫和非生物胁迫中扮演着重要角色[4]。例如, Li等[5]发现WRKY70作为茉莉酸、水杨酸介导的信号通路中的关键节点基因, 其表达被水杨酸激活并被茉莉酸抑制, 同时WRKY70是茉莉酸诱导基因的激活剂和水杨酸响应基因的阻遏物, 通过整合来自这些相互拮抗途径的信号能够提高植株的抗病性。Zhou等[6]通过超量表达 GmWRKY13、GmWRKY21和 GmWRKY54基因, 分析转基因拟南芥与非转基因拟南芥在非生物胁迫耐受性方面的不同; 发现 GmWRKY21提高了拟南芥的耐冷性; GmWRKY54可能通过调节DREB2A和STZ/Zat10来响应盐和干旱胁迫; 而超表达GmWRKY21基因降低了拟南芥对盐和甘露醇的耐性, 同时抑制了拟南芥侧根的发生。因此, WRKY类转录因子在植物抵御各种逆境的过程中具十分重要的作用[7]。然而, WRKY类转录因子在植株耐碱方面的研究仍鲜有报道。
野生大豆(Glycine soja)具有很强的抗逆性和适应能力, 是我国宝贵的野生种质资源, 也是克隆耐逆基因、研究耐逆分子机制的理想材料[8]。目前通过生物技术的手段, 将野生大豆优良抗逆基因导入其他物种, 来创造优秀的新种质资源, 尤其是大豆和苜蓿, 它们与野生大豆的亲缘关系更近, 遗传物质更容易交流。已有研究结果表明采用生物技术手段, 利用野生大豆的优良抗逆性状培育优质高抗苜蓿新品种是可行的[9-14]。因此, 实验室前期利用RNA-seq测序数据, 构建了野生大豆盐碱胁迫基因调控网络[15], 从中筛选出了碱胁迫应答关键基因GsWRKY15。本研究通过同源克隆的方法获得了GsWRKY15基因的 CDS区全长序列, 并构建了由35S启动子调控的植物表达载体, 用以转化肇东苜蓿, 以期获得耐碱能力显著提高的转基因苜蓿新株系。本研究对于充分利用我国盐碱地资源, 提高苜蓿种植面积, 具有重要的研究价值和广泛的开发应用前景。
1 材料与方法
1.1 试验材料
耐盐碱东北野生大豆(Glycine soja) G07256、肇东苜蓿(Medicago sativa)种子由东北农业大学农业生物功能基因重点实验室保存; 大肠杆菌DH5α、酵母菌株AH109、农杆菌EHA105、植物超量表达载体pCAMBIA330035Su由东北农业大学农业生物功能基因重点实验室构建保存; 胶回收和质粒小提试剂盒购自Transgene公司, 由哈尔滨英俊生物公司完成引物合成及测序。
1.2 GsWRKY15基因的克隆
以TRIzol法[16]从野生大豆的根中提取总RNA,并反转录成cDNA。通过同源克隆方法, 以目的基因在大豆中的同源基因Glyma05g20710序列为模板设计基因特异引物(5′-GGCTTAAUATGGCCGTGGAC CTC-3′和5′-GGTTTAAUTCAAGACGATTCTAGAA TGAGATT-3′)。以稀释5倍的总cDNA为模板, 利用pfu DNA聚合酶进行PCR扩增[17]。根据PCR产物对照Marker的位置回收目的条带。
1.3 GsWRKY15基因的表达特异性分析
对21 d龄的野生大豆幼苗进行碱胁迫(50 mmol L–1NaHCO3)处理, 分别在处理 0、1、3、6、12和24 h后迅速剪取根尖部分; 此外, 选取2个月左右,正处于生殖生长阶段的野生大豆的根、茎、胚、下胚轴、老叶、新叶、花和豆荚。提取总RNA, 并反转录成cDNA, 利用荧光定量PCR检测基因表达量。采用比较 CT 法(2–ΔΔCT)定量分析, 以 GAPDH (GenBank登录号为 DQ355800)为内参基因, 设置 3次独立生物学重复和3次技术重复, 数据取3次重复的平均值。参照文献[18]原始数据标准化处理的方法。
1.4 GsWRKY15基因对苜蓿的遗传转化
利用 USER 技术[19-20]将目的片段与pCAMBIA330035Su载体快速融合, 以Bar基因作为筛选标记基因, 采用冻融法将构建成功的植物表达载体转化到根癌农杆菌EHA105中。
参照盛慧等[21]方法, 稍作优化。对苜蓿子叶节进行2~3 d的预培养后, 用含有GsWRKY15基因的根癌农杆菌 EHA105对子叶节进行侵染, 将侵染后的子叶节转移到共培养基中培养3~4 d, 再转移到含有100 mg L–1阿莫西林-克拉维酸钾的液体植物培养基中冲洗3~4次, 随后转移到具有草甘膦(0.5 mg L–1)筛选压力的除菌培养基上, 进行 2周的除菌筛选培养, 待外植体分化出抗性不定芽。将其转入具有相同筛选压力的伸长除菌培养基上继代培养 2周, 获得大量抗性不定芽, 当抗性芽伸长至 3~4 cm高时,将其从外植体基部剪下, 接种到生根培养基上培养。当植株根系生长至2~3 cm长时, 进行2~3 d的驯化。然后将抗性苗移栽到花盆中培养。
1.5 再生植株的分子生物学检测
提取再生植株叶片DNA作为模板, 非转基因植株叶片DNA作为阴性对照的模板。利用Bar基因特异引物(5′-TGCACCATCGTCAACCACTACATCG-3′和 5′-CCAGCTGCCAGAAACCCACGTCATG-3′)对抗性植株进行PCR检测, PCR产物长度为450 bp。
以CTAB法提取PCR阳性植株的总DNA, 参照丁晓东等[22]方法。利用GsWRKY15基因特异性引物,从植物表达载体上克隆探针。方法参照Mishra等[23]总DNA经过酶切后纯化、电泳、转移印记、固定、预杂交、杂交等步骤, 对PCR阳性植株进行Southern blot检测。
提取PCR阳性植株的总RNA, 以其为模板, 用内参基因GAPDH的特异性引物(5′-GACTGGTATG GCATTCCGTGT-3′和5′-GCCCTCTGATTCCTCCTT G-3′)进行PCR扩增, 验证基因组DNA, 确定无污染后反转录成cDNA。以稀释5倍的苜蓿cDNA为模板, 用基因特异引物进行半定量RT-PCR检测。
1.6 碱胁迫下转基因苜蓿的表型分析
方法参照王臻昱等[10], 稍作改进。通过扦插繁殖的方式获得转基因苜蓿扦插苗, 对扦插苗进行耐碱性分析。在苜蓿生长茂盛的季节, 以每 2~3个节点剪为一段, 在稀释1∶5000的生根粉溶液中浸泡2 h后, 插入蛭石、草炭土和珍珠岩(1∶1∶1)混合的基质中。白天用遮阳网覆盖降温、保湿, 早晚各喷灌一次, 每次0.5 h, 并保持湿度80%和温度26℃, 在温室中培养约 2周至生根, 移栽到蛭石和珍珠岩(1∶1)的混合基质中, 每2 d用1/8 Hoagland营养液浇灌一次。培养 4周左右, 挑选生长健壮且状态一致的扦插苗进行碱胁迫处理, 用含有0、100和150 mmol L–1NaHCO3营养液浇灌, 2周后观察照相并记录表型。
1.7 碱胁迫下转基因植株相关生理指标检测
采用硫代巴比妥酸(TBA)比色法[24]测定丙二醛(MDA)含量; 利用电导率仪检测叶片的相对电导率[25],测定相对质膜透性; 采用 80%丙酮抽提比色法[26]测定叶片中叶绿素含量。所有生理指标均设置 3次重复, 用SPSS13.0检验显著性, Microsoft Excel 2007制图。
1.8 转 GsWRKY15苜蓿胁迫相关基因的表达模式分析
将非转基因和转基因苜蓿分别置于 100 mmol L–1NaHCO3条件下处理0、1、3、6、12和24 h后, 剪取苜蓿叶片。参照1.2方法, 提取苜蓿叶片总 RNA并反转录成cDNA。参照1.4方法, 选取胁迫相关基因H+-Ppase、NADP-ME、KIN1和RD29A, 分析其在非转基因和转基因苜蓿中的表达模式。
2 结果与分析
2.1 GsWRKY15基因的筛选与克隆
2.1.1 基于RNA-seq测序数据的GsWRKY15筛选
利用前期野生大豆盐碱胁迫RNA-seq测序数据,从构建的碱胁迫基因调控网络中筛选得到碱胁迫下显著上调表达的GsWRKY15基因。由RNA-seq测序数据结果(图1)可以看出, 野生大豆根中GsWRKY15基因受碱胁迫诱导表达, 在碱胁迫下处理 1 h时表达量最高。因此推测GsWRKY15可能在植物响应碱胁迫的过程中具关键作用。
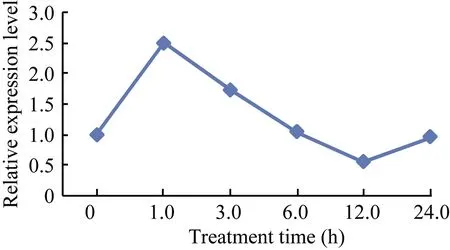
图1 GsWRKY15基因在碱胁迫下RNA-seq测序数据分析Fig. 1 RNA-seq data analysis of GsWRKY15 gene expression patterns under alkaline stress
2.1.2 GsWRKY15基因的克隆 根据与GsWRKY15同源的大豆基因 Glyma05g20710序列,设计基因特异性引物, 以野生大豆 cDNA为模板,使用高保真酶Pfu DNA聚合酶进行PCR扩增。通过电泳结果(图2)及NCBI序列分析, 发现GsWRKY15基因大小为1005 bp, 编码335个氨基酸。

图2 GsWRKY15的克隆Fig. 2 Cloning GsWRKY15 geneM: DL2000 marker.
2.2 GsWRKY15基因的表达模式分析
2.2.1 碱胁迫下GsWRKY15基因的表达模式分析
以荧光定量 PCR验证 GsWRKY15基因在 50 mmol L–1NaHCO3胁迫下野生大豆根中的表达特性表明, GsWRKY15基因受碱胁迫明显上调表达, 且在1 h时表达量最高(图3), 虽然表达量与RNA-seq数据有所不同, 但趋势是基本一致的, 说明该基因受碱胁迫诱导表达。
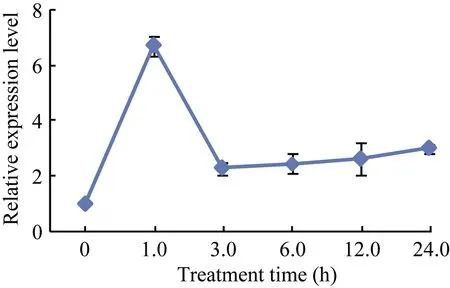
图3 GsWRKY15基因在碱胁迫下表达模式Fig. 3 GsWRKY15 gene expression pattern under alkaline stress
2.2.2 GsWRKY15基因的组织定位分析 荧光定量PCR分析表明, 该基因在野生大豆根、下胚轴、茎、老叶、幼叶、果实和花中均有表达, 且不同组织之间的表达量存在差异, 该基因在花中的表达量最高, 其次是幼叶, 在胚中的表达量最低(图 4)。说明 GsWRKY15基因在野生大豆各组织中的分布具有特异性, 其行使功能的主要部位可能在花和幼叶中。
2.3 GsWRKY15基因对苜蓿的遗传转化
2.3.1 植物表达载体构建 将克隆得到的GsWRKY15基因插入由35S启动子调控的植物表达载体 pCAMBIA330035Su, 成功构建植物超量表达载体(图5-A)。将构建的植物超量表达载体转入大肠杆菌DH5α, 挑取阳性克隆摇菌并提取质粒(图5-B),送交公司测序。以测序结果正确的质粒, 利用冻融法转化根癌农杆菌EHA105。
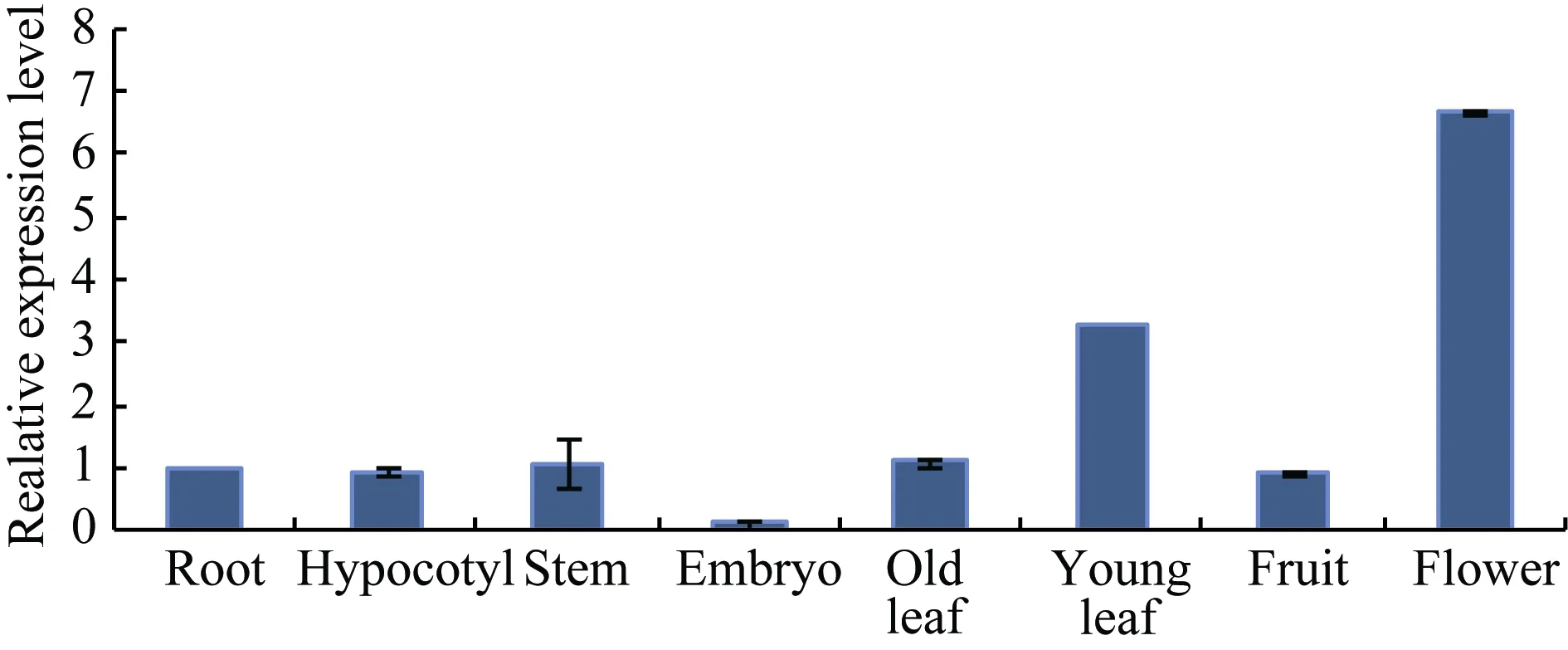
图4 GsWRKY15基因的组织定位分析Fig. 4 Locating analysis of GsWRKY15 gene in different tissues

图5 植物表达载体构建与转化农杆菌后菌落PCR鉴定Fig. 5 Construction of plant expression vector and PCR identification of transformed agrobacteriumM: DL2000 maker; +: 阳性对照; –: 阴性对照; 1~3: 菌落PCR产物。M: DL2000 maker; +: positive plasmid; –: negative control; 1–3: PCR products of bacterial colony.
2.3.2 转基因苜蓿抗性植株的获得 采用农杆菌介导法将植物表达载体 pCAMBIA330035Su-GsWRKY15转入苜蓿中。经过种子萌发、形成无菌苗、子叶节农杆菌共培养、抗性不定芽诱导及筛选、诱导生根等过程, 共获得39株抗性再生植株。
2.4 再生植株的分子生物学检测
2.4.1 再生植株的PCR、Southern blot鉴定 对获得的转 GsWRKY15基因的 39株再生植株进行PCR检测。其中, 含有GsWRKY15基因的重组质粒作为阳性对照, 水和非转基因植株的DNA作为阴性对照(图6-A)。经PCR检测, 最终获得转GsWRKY15基因 PCR阳性植株 16株, 转化率为 41.03%。Southern blot (图6-B)结果显示, #5、#17、#23株系为Southern blot阳性株系, 且均为单拷贝株系。
2.4.2 Southern blot阳性植株的RT-PCR鉴定
半定量RT-PCR方法对Southern blot阳性株系检测(图6-C)显示, GsWRKY15基因在#5、#17、#23转基因苜蓿中能够转录表达, 而非转基因株系中未检测到阳性信号。最终确定3株转基因株系。
2.5 转基因苜蓿的耐碱性生物学分析
2.5.1 碱胁迫下转基因苜蓿的表型分析 选取RT-PCR阳性植株#5、#17转基因株系进行表型分析。对非转基因和转基因株系分别进行 0、100和 150 mmol L–1NaHCO3处理, 2周后发现, 未处理组中,非转基因和转基因株系生长状况良好且相对一致(图7-A)。在100 mmol L–1NaHCO3胁迫处理下, 转基因植株生长状况良好, 而非转基因株系苜蓿叶片逐渐萎蔫失绿(图7-B)。在150 mmol L–1NaHCO3处理下, 转基因苜蓿受到影响较轻, 仍能正常生长,而非转基因植株碱害较重, 生长发育受到抑制, 叶片萎蔫、失绿(图7-C), 说明转基因株系耐碱性明显好于非转基因株系。

图6 再生植株的分子生物学鉴定Fig. 6 Molecular biological identification of regeneration plantsA: 再生植株的PCR检测; B: PCR阳性植株的Southern blot鉴定; C: PCR阳性植株的RT-PCR检测。M: DL2000 marker; +: 阳性对照;–: 阴性对照; WT: 非转基因株系; 1~19: 部分转基因株系; #5, #17, #23: 转基因株系。A: PCR identification of regeneration plants; B: Southern identification of PCR positive plants; C: RT-PCR identification of Southern blot positive plants. M: DL2000 marker; –: negative control; WT: non-transgenic plants; +: positive control; 1–19: partial transgenic lines; #5, #17, #23: transgenic lines.
在0、100和150 mmol L–1NaHCO3胁迫处理下,所有株系的株高和根长随 NaHCO3处理浓度的增高而下降, 但转基因株系#5、#17的下降幅度均小于非转基因株系(图8)。由此表明, GsWRKY15基因的超量表达, 提高了转基因苜蓿的耐碱性。
2.5.2 碱胁迫下转基因苜蓿的生理指标检测 图9-A显示, 转基因和非转基因株系的MDA含量随碱胁迫浓度的升高而增加, 但2个转GsWRKY15基因苜蓿株系的 MDA含量增加幅度显著低于非转基因对照株系(P<0.05), 从而说明超量表达 GsWRKY15基因可降低碱胁迫对转基因苜蓿细胞膜的过氧化损伤, 转基因苜蓿耐碱性更高。
图 9-B显示, 随着碱胁迫浓度的增加, 2个转GsWRKY15基因苜蓿株系和非转基因对照株系的叶绿素含量均随之降低, 但相同的碱胁迫浓度处理条件下, 转基因株系的叶绿素含量均高于非转基因株系, 在150 mmol L–1NaHCO3处理条件下最为明显, 2个转基因株系的叶绿素含量均显著高于非转基因对照株系(P<0.05), 显然在碱胁迫条件下转GsWRKY15基因苜蓿株系的叶绿体受伤害的程度小,具有更强的光合能力, 具有更高的耐碱性。

图7 转基因苜蓿碱胁迫下的表型Fig. 7 Phenotype of transgenic alfalfa with GsWRKY15 under alkaline stressA、B和C分别为0、100和150 mmol L–1NaHCO3处理2周后的表型; WT: 非转基因对照; #5和#17: 转GsWRKY15基因株系。A, B, and C are phenotype of alfalfa treated with 0, 50, and 100 mmol L–1NaHCO3for 14 days; WT: non-transgenic alfalfa; #5 and #17: transgenic alfalfa plants with GsWRKY15.
图 9-C表明, 转基因和非转基因苜蓿的相对质膜透性均随着碱胁迫浓度的增加而增加, 但转基因植株相对质膜透性的增加幅度显著低于非转基因对照植株(P<0.05), 说明在碱胁迫条件下, 转GsWRKY15基因株系细胞膜所受的损害较轻, 具有较高的耐碱能力。
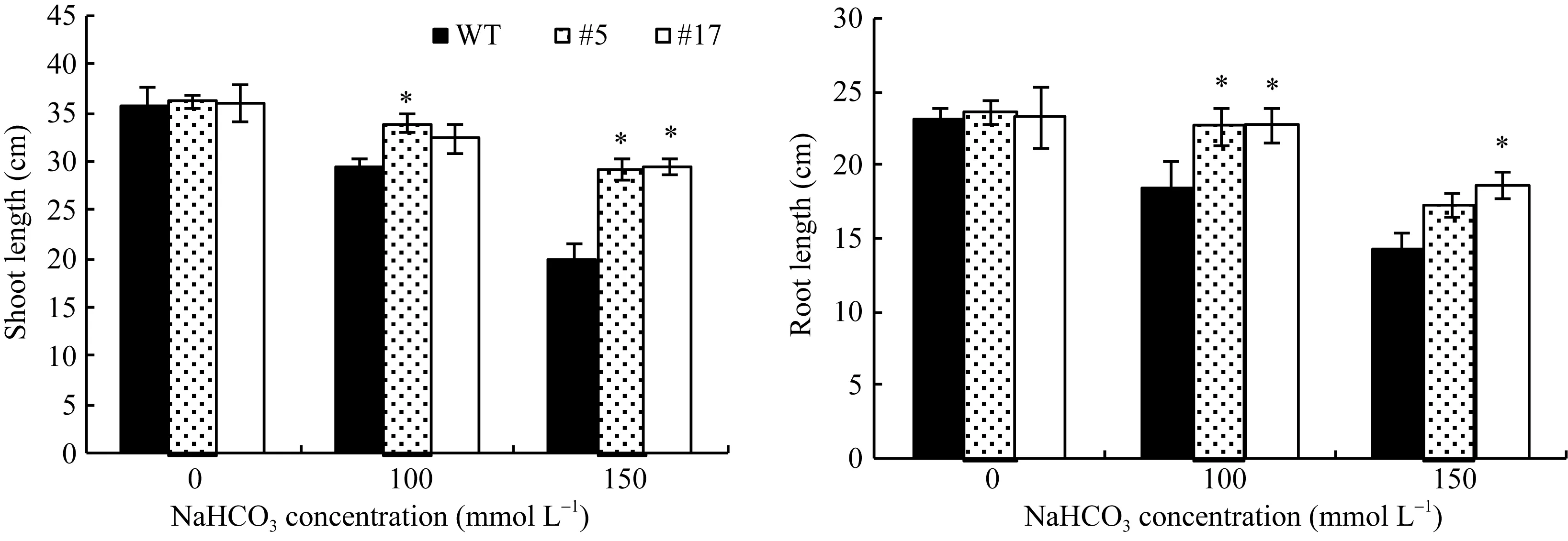
图8 碱胁迫对转GsWRKY15基因苜蓿株高和根长的影响Fig. 8 Changes of plant height and root length of transgenic alfalfa with GsWRKY15 under alkine stress *表示在P<0.05水平差异显著。* indicates significantly different at P<0.05.
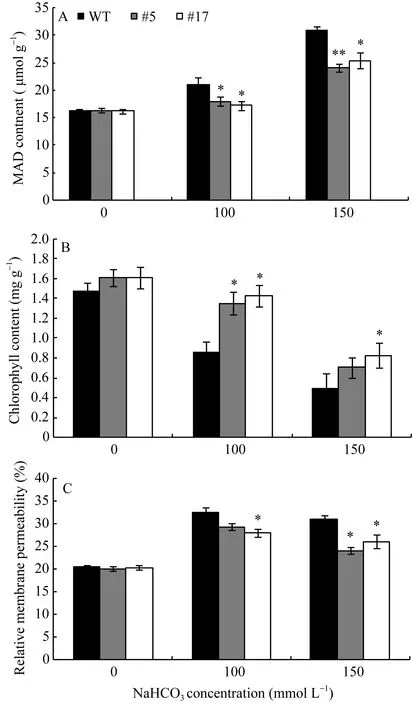
图9 碱胁迫下转基因植株的生理指标Fig. 9 Physiological indexes of transgenic plants under alkali stress**和*分别表示在P<0.01和P<0.05水平差异显著。A: 丙二醛含量; B: 叶绿素含量; C: 相对质膜透性。** and * indicate significantly different at P<0.01 and P<0.05, respectively. A: MAD content; B: chlorophyll content; C: relative membrane permeability.
2.6 转基因苜蓿中胁迫相关基因的表达模式分析
为了检验GsWRKY15基因在苜蓿中的超量表达是否影响了胁迫相关基因的表达, 从而使转基因苜蓿在生理水平上表现出耐碱特性, 对胁迫相关基因在转基因和非转基因苜蓿中的表达量进行了分析,包括H+-Ppase、NADP-ME、KIN1、RD29A。通过荧光定量PCR分析发现, 转基因和非转基因植株中这4个基因都被碱胁迫诱导表达, 且具有相似的表达模式, 但转基因株系的表达量均高于非转基因植株(图10)。由此表明, GsWRKY15基因作为转录因子可能通过调节这些基因的转录来提高植株耐碱性。
3 讨论
在以往的研究中得知 WRKY类转录因子是一个庞大的基因家族, 不同的家族成员在功能上存在特异性, 可以响应不同的生物或非生物胁迫。目前, WRKY类转录因子的研究主要集中在拟南芥、水稻、小麦等植物中进行[27-30], 而苜蓿中的研究较少。在与苜蓿近缘的野生大豆中已克隆到多个 WRKY家族基因, Luo等[31]通过超量表达GsWRKY20, 降低了拟南芥叶片气孔密度, 及其离体叶片失水速率, 从而提高了转基因植株的干旱胁迫耐性。以上研究为我们探究该基因的功能提供了一定的理论基础。
土壤盐碱化是限制农业产量的重要因素之一,而野生大豆具有较强耐盐碱能力, 是分析耐碱分子机制和确定胁迫相关基因的理想适材。实验室前期,以野生大豆的根为样本, 通过RNA-seq数据确定了3380个基因能够响应碱胁迫应答[15]。我们从中筛选出GsWRKY15基因作为研究对象, 从RNA-seq数据(图 1)和荧光定量 PCR结果显示(图 3), GsWRKY15基因在野生大豆根中的表达量能够迅速受碱胁迫诱导表达; 同时, 组织定位分析发现(图 4), 在野生大豆中的表达具有组织特异性且在花中的表达量最高,其次为幼叶。据此推测该基因可能在碱胁迫中发挥一定的作用, 其作用的部位可能是花和幼叶。
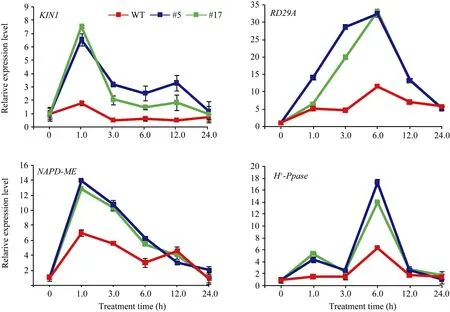
图10 胁迫相关基因在转基因和非转基因苜蓿中的表达模式Fig. 10 Expression patterns of stress-responsive genes in transgenic and non-transgenic alfalfa
紫花苜蓿是我国最重要的豆科牧草之一, 对我国畜牧业有着重要的意义。然而, 土壤盐渍化正严重制约着紫花苜蓿的生长。因此, 培育出具有强耐逆性的苜蓿新品种具有重要的生产价值。针对上述研究结果, 本研究利用GsWRKY15基因转化肇东苜蓿, 期望能够提高苜蓿的耐碱性。本研究构建了植物表达载体 pCAMBIA330035Su-GsWRKY15, 其筛选标记基因为Bar, Bar基因不仅可以作为筛选标记基因使用, 还具有农艺价值且是国家公认的安全性基因[32]; 通过PCR, Southern blot对再生植株进行分子生物学检测, 确定GsWRKY15基因已整合到苜蓿的基因组中(图6-A, B)。我们发现16株PCR阳性植株中, 仅有3株为Southern blot阳性植株; 这可能与外源基因的整合机制相关, 如染色体的重组, 异位等现象的发生致使目的基因片段丢失。通过RT-PCR检测, 最终确定GsWRKY15基因在转录水平上已成功表达(图6-C)。
当植物受到碱胁迫时, 最直接的伤害是由碱胁迫造成的生理干旱; 细胞内产生活性氧诱发氧化胁迫, 破坏质膜完整性、改变蛋白活性, 引起代谢失调;随着时间的增长也会出现胞内离子稳态失衡, 发生离子毒害; 最终造成生理紊乱, 使植物的形态发育发生变化[33]。因此, 针对以上几点, 选择与其相关的生理指标进行评价, 更能说明转基因材料是否真正耐碱。本研究选取的评价指标包括反映植物生长抑制情况的株高、根长等形态指标; 反映植物光合作用及受伤害程度的叶绿素含量; 反映碱胁迫下质膜氧化损伤程度的丙二醛含量; 反映细胞膜完整性的相对质膜透性。结果显示, 超量表达GsWRKY15基因的苜蓿, 在碱胁迫条件下, 与非转基因株系相比其株高和根长受影响较小, 基本可以正常生长(图8)。由此可见, GsWRKY15在碱胁迫下能够减缓高浓度碱对苜蓿生长的损害。通过分析转GsWRKY15基因苜蓿相关的生理指标, 发现GsWRKY15能够减缓碱胁迫对苜蓿的生长抑制, 从而使转基因苜蓿在碱胁迫条件下仍能保持良好的生长状态。
此外, 为了探究超量表达GsWRKY15基因对提高苜蓿耐碱能力的分子机制, 我们对胁迫相关基因在转基因和非转基因苜蓿中的表达量分析发现, 转基因苜蓿中 H+-Ppase、NADP-ME、KIN1、RD29A的表达量明显高于非转基因植株(图 10)。KIN1、RD29A 基因在调节植物胞内生理过程中起到关键作用[37], 碱胁迫下KIN1、RD29A的诱导表达可能有助于减缓碱对植株的伤害。H+-Ppase、NADP-ME基因同样受碱胁迫诱导表达, 其功能在于调节胞内的pH值[34-35]。H+-Ppase、NADP-ME基因的表达有助于植物细胞内的pH稳态, 提高植株的耐碱能力。因此, GsWRKY15基因可能通过调控 H+-Ppase、NADP-ME、KIN1、RD29A的表达来降低碱胁迫对植株的伤害。
综上所述, 我们推断GsWRKY15基因的超量表达能够提高苜蓿的耐碱性。而GsWRKY15作为转录因子, 对其在异源植株体内的表达及行使其功能的机制, 还需通过进一步的实验来深入探究。
4 结论
筛选出了一个在碱胁迫下明显上调表达的基因GsWRKY15, 该基因在野生大豆中的表达具有组织特异性。获得了超量表达的转GsWRKY15基因苜蓿, GsWRKY15提高了转基因苜蓿耐碱性。
[1] 李彬, 王志春, 孙志高, 陈渊, 杨福. 中国盐碱地资源与可持续利用研究. 干旱地区农业研究, 2005, 23(2): 154–158
Li B, Wang Z C, Sun Z G, Chen Y, Yang F. Resources and sustainable resource exploitation of salinized land in China. Agric Res Arid Res, 2005, 23(2): 154–158 (in Chinese with English abstract)
[2] 王鑫马, 马永祥, 李娟. 紫花苜蓿营养成分及主要生物学特性.草业科学, 2003, 20(10): 39–41
Wang X M, Ma Y X, Li J. Alfalfa nutrient composition and main biological characteristics. Acta Pratac Sin, 2003, 20(10): 39–41 (in Chinese with English abstract)
[3] 乔建江, 堃王 , 杨青川. 苜蓿转基因的研究现状和前景. 中国草地学报, 2006, 28(5): 98–103
Qiao J J, Wang K, Yang Q C. Research situation and future of transgenic alfalfa. Chin J Grassland, 2006, 28(5): 98–103 (in Chinese with English abstract)
[4] Ülker B, Somssich I E. WRKY transcription factors: from DNA binding towards biological function. Curr Opin Plant Biol, 2004, 7: 491–498
[5] Li J, Brader G, Palva E T. The WRKY70 transcription factor: a node of convergence for jasmonate-mediated and salicylate- mediated signals in plant defense. Plant Cell, 2004, 16: 319–331
[6] Zhou Q Y, Tian A G, Zou H F, Xie Z M, Lei G, Huang J, Wang C M, Wang H W, Zhang J S, Chen S Y. Soybean WRKY-type transcription factor genes, GmWRKY13, GmWRKY21, and GmWRKY54, confer differential tolerance to abiotic stresses in transgenic Arabidopsis plants. Plant Biotechnol J, 2008, 6: 486–503
[7] Eulgem T, Rushton P J, Robatzek S, Somssich I E. The WRKY superfamily of plant transcription factors. Trends Plant Sci, 2000, 5: 199–206
[8] 李福山. 中国野生大豆资源的地理分布及生态分化研究. 中国农业科学, 1993, 26(2): 47–55
Li F S. Studies on the ecological and geographical distribution of the chinese resources of wild soybean (G. soja). Sci Agric Sin, 1993, 26(2): 47–55 (in Chinese with English abstract)
[9] Bai X, Liu J, Tang L, Cai H, Chen M, Ji W, Liu Y, Zhu Y. Overexpression of GsCBRLK from Glycine soja enhances tolerance to salt stress in transgenic alfalfa (Medicago sativa). Funct Plant Biol, 2013, 40: 1048–1056
[10] 王臻昱, 才华, 柏锡, 纪巍, 李勇, 魏正巍, 朱延明. 野生大豆GsGST19基因的克隆及其转基因苜蓿的耐盐碱性分析. 作物学报, 2013, 38: 971–979
Wang Z Y, Cai H, Bai X, Ji W, Li Y, Wei Z W, Zhu Y M. Isolation of GsGST19 from Glycine soja and analysis of saline-alkaline tolerance for transgenic Medicago sativa. Acta Agron Sin, 2013, 38: 971–979 (in Chinese with English abstract)
[11] 魏正巍, 朱延明, 化烨, 才华, 纪巍, 柏锡, 王臻昱, 文益东.转GsPPCK1基因苜蓿植株的获得及其耐碱性分析. 作物学报, 2013, 39: 68–75
Wei Z W, Zhu Y M, Hua Y, Cai H, Ji W, Bai X, Wang Z Y, Wen Y D. Transgenic alfalfa with GsPPCK1 and its alkaline tolerance analysis. Acta Agron Sin, 2013, 39: 68–75 (in Chinese with English abstract)
[12] 赵阳, 朱延明, 柏锡, 纪巍, 吴婧, 唐立郦, 才华. 转GsCBRLK/SCMRP双价基因苜蓿耐碱性及氨基酸含量分析.作物学报, 2014, 40: 431–438
Zhao Y, Zhu Y M, Bai X, Ji W, Wu J, Tang L L, Cai H. Over-expressing GsCBRLK/SCMRP enhances alkaline tolerance and methionine content in transgenic Medicago sativa. Acta Agron Sin, 2014, 40: 431–438 (in Chinese with English abstract)
[13] Sun M Z, Jia B W, Cui N, Wen Y D, Duanmu H Z, Yu Q Y, Xiao J L, Zhu Y M. Functional characterization of a Glycine soja Ca2+ATPase in salt–alkaline stress responses. Plant Mol Biol, 2016, 90: 419–434
[14] Sun M Z, Sun X L, Zhao Y, Zhao C Y, Duanmu H Z, Yu Y, Ji W, Zhu Y M. Ectopic expression of GsPPCK3 and SCMRP in Medicago sativa enhances plant alkaline stress tolerance and methionine content. PLoS One, 2013, 9: e89578
[15] Duanmu H Z, Wang Y, Bai X, Cheng S F, Deyholos M K, Wong G K, Li D, Zhu D, Li R, Yu Y, Cao L, Chen C, Zhu Y M. Wild soybean roots depend on specific transcription factors and oxidation reduction related genesin response to alkaline stress. Funct Integr Genomics, 2015, 15: 651–660
[16] Rio D C, Ares M, Hannon G J, Nilsen T W. Purification of RNA using TRIzol (TRI Reagent). Cold Spring Harbor Protocols, 2010. pdb.prot5439
[17] Cline J, Braman J C, Hogrefe H H. PCR fidelity of pfu DNA polymerase and other thermostable DNA polymerases. Nucl Acids Res, 1996, 24: 3546–3551
[18] Willems E, Leyns L, Vandesompele J. Standardization of real-time PCR gene expression data from independent biological replicates. Anal Biochem, 2008, 379: 127–129
[19] Geu-Flores F, Nour-Eldin H H, Nielsen M T, Halkier B A. USER fusion: a rapid and efficient method for simultaneous fusion and cloning of multiple PCR products. Nucl Acids Res, 2007, 35: e55
[20] Nour-Eldin H H, Hansen B G, Nørholm M H, Jensen J K, Halkier B A. Advancing uracil-excision based cloning towards an ideal technique for cloning PCR fragments. Nucl Acids Res, 2006, 34: e122
[21] 盛慧, 朱延明, 李杰, 柏锡, 才华. DREB2A基因对苜蓿遗传转化的研究. 草业科学, 2007, 24(3): 40–45
Sheng H, Zhu Y M, Li J, Bai X, Cai H. Genetic transformation of DREB2A gene into alfalfa. Acta Pratac Sin, 2007, 24(3): 40–45
[22] 丁晓东, 吕柳新. 从顽拗植物荔枝中提取基因组DNA技术的研究. 应用与环境生物学报, 2000, 6: 142–145
Ding X D, Lyu L X. Study on genomic DNA extraction from recalcitrant litchi. Chin J Appl Environ Biol, 2000, 6: 142–145 (in Chinese with English abstract)
[23] Mishra S, Bansal S, Sangwan R S, Sangwan N S. Genotype independent and efficient Agrobacterium-mediated genetic transformation of the medicinal plant Withania somnifera. J Plant Biochem Biot, 2016, 25: 191–198
[24] Hodges D M, Delong J M, Forney C F, Prange R K. Improving the thiobarbituric acid-reactive-substances assay for estimating lipid peroxidation in plant tissues containing anthocyanin and other interfering compounds. Planta, 1999, 207: 604–611
[25] Gibon Y, Larher F. Cycling assay for nicotinamide adenine dinucleotides: NaCl precipitation and ethanol solubilization of the reduced tetrazolium. Anal Biochem, 1997, 251: 153–157
[26] Wellburn A R, Lichtenthaler H. Formulae and program to determinetotal carotenoids and chlorophylls a and b of leaf extracts in different solvents. Adv Photosynth Res, 1984: 9–12
[27] Shimono M, Sugano S J, Nakayama A, Jiang C J, Ono K, Toki S, Takatsuji H. Rice WRKY45 plays a crucial role in benzothiadiazole inducible blast resistance. Plant Cell, 2007, 19: 2064–2076
[28] Marè C, Mazzucotelli E, Crosatti C, Francia E, Stanca A M, Cattivelli L. Hv-WRKY38: a new transcription factor involved in cold and drought response in barley. Plant Mol Biol, 2004, 55: 399–416
[29] 秦伟, 赵光耀, 曲志才, 张立超, 段佳磊, 李爱丽, 贾继增, 孔秀英. 小麦白粉病菌诱导的TaWRKY34基因的鉴定与分析. 作物学报, 2010, 36: 249–255
Qin W, Zhao G Y, Qu Z C, Zhang L C, Duan J L, Li A L, Jia J Z, Kong X Y. Identification and analysis of TaWRKY34 gene induced by wheat powdery mildew (Blumeria graminis f. sp. tritici). Acta Agron Sin, 2010, 36: 249–255 (in Chinese with English abstract)
[30] 王瑞, 吴华玲, 王会芳, 黄珂, 霍春艳, 倪中福, 孙其信. 小麦TaWRKY44基因的克隆、表达分析及功能鉴定. 作物学报, 2013, 39: 1944–1951
Wang R, Wu H L, Wang H F, Huang K, Huo C Y, Ni Z F, Sun Q X. Cloning, characterization, and functional analysis of TaWRKY44 gene from wheat. Acta Agron Sin, 2013, 39: 1944–1951 (in Chinese with English abstract)
[31] 田云, 卢向阳, 彭丽莎, 方俊. 植物WRKY转录因子结构特点及其生物学功能. 遗传, 2006, 28: 1607–1612
Tian Y, Lu X Y, Peng L S, Fang J. The structure and function of plant WRKY transcription factors. Hereditas (Beijing), 2006, 28: 1607–1612 (in Chinese with English abstract)
[32] 江淑琼, 周守标, 刘坤, 程龙玲. 干旱胁迫对中国石蒜叶片形态和部分生理指标的影响. 北方园艺, 2010, (7): 16–19
Jiang S Q, Zhou S B, Liu K, Cheng L L. Effects of drought stress on morphology and partial physiological indexes of leaves in lycoris chinensis. Northern Hort, 2010, (7): 16–19 (in Chinese)
[33] Tardieu F, Davies W J. Stomatal response to abscisic acid is a function of current plant water status. Plant Physiol, 1992, 98: 540–545
[34] Sze H, Li X, Palmgren M G. Energization of plant cell membranes by H+-pumping ATPases regulation and biosynthesis. Plant Cell, 1999, 11: 677–690
[35] Davies D D. The fine control of cytosolic pH. Physiol Plant, 1986, 67: 702–706
Cloning of Gene GsWRKY15 Related to Alkaline Stress and Alkaline Tolerance of Transgenic Plants
ZHU Ping-Hui**, CHEN Ran-Ran**, YU Yang, SONG Xue-Wei, LI Hui-Qing, DU Jian-Ying, LI Qiang, DING Xiao-Dong, and ZHU Yan-Ming*
Key Laboratory of Agricultural Biological Functional Genes, Northeast Agricultural University, Harbin 150030, China
WRKY proteins are members of a transcription factor family with zinc-finger structure in higher plant, which participate in various responses to multiple stresses. In this study, we constructed a gene expression profile under alkaline treatment using RNA-seq data, from which we cloned a gene GsWRKY15. We analyzed the expression pattern of GsWRKY15 in roost of Glycine soja under alkaline stress, and found that this gene was up-regulated by alkaline stress, wirh the highest expression at one hour after alkaline treatment. We analyzed the expression pattern of GsWRKY15 in different tissues of Glycine soja, and found that this gene could express in all tissues, with the highest expression level in flowers. GsWRKY15 was transformed into Zhaodong alfalfa by Agrobacterium tumefaciens-mediated infection of alfalfa cotyledonary nodes, and 39 resistant plants were obtained. The results of PCR, Southern blot and RT-PCR showed that GsWRKY15 was integrated into the genome of Zhaodong alfalfa and expressed in transgenic plants. Alkaline tolerance analysis showed that the growth of transgenic alfalfa after two weeks of treatment with 150 mmol L–1NaHCO3was better than those of non-transgenic alfalfa. MDA content and relative membrane permeability were significantly higher while chlorophyll content was significantly lower in non-transgenic alfalfa than in transgenic alfalfa.And by analyzing t some stress response marker genes, we found that the expression levels of H+-Ppase, NADP-ME, KIN1, and RD29A were higher in transgenic alfalfa than in non-transgenic alfalfa. Taken together, we suggest that the expression of GsWRKY15 gene can enhance the alkaline-resistant ability of alfalfa.
Glycine soja; GsWRKY15; Zhaodong alfalfa; Agrobacterium tumefaciens; Alkaline tolerance
(
): 2016-12-03; Accepted(接受日期): 2017-05-10; Published online(网络出版日期): 2017-05-22.
10.3724/SP.J.1006.2017.01319
本研究由国家自然科学基金项目(31171578), 黑龙江省高校科技创新团队建设计划项目(2011TD005)和东北农业大学学科团队建设项目(团队1)资助。
This study was supported by the National Natural Science Foundation of China (31171578), Heilongjiang Provincial Science and Technology Innovation Team Building Program for Universities (2011TD005), and the Northeast Agricultural University Discipline Team Construction Project (Group 1).
*通讯作者(Corresponding author): 朱延明, E-mail: ymzhu2001@neau.edu.cn, Tel: 18645035310
**同等贡献(Contributed equally to this work)
联系方式: E-mail: zhupinghui@outlook.com, Tel: 15604601520
URL: http://kns.cnki.net/kcms/detail/11.1809.S.20170522.0915.002.html

