三维肝脏模型个性化UV映射方法研究
高 聪,陈国栋,王 娜
(1.福州大学 物理与信息工程学院,福建 福州 350116; 2.福州大学 计算机图像图形研究所,福建 福州 350116;3.福建师范大学 福清分校,福建 福州 350300)
三维肝脏模型个性化UV映射方法研究
高 聪1,2,陈国栋1,2,王 娜3
(1.福州大学 物理与信息工程学院,福建 福州 350116; 2.福州大学 计算机图像图形研究所,福建 福州 350116;3.福建师范大学 福清分校,福建 福州 350300)
虚拟手术是计算机图形学里的一个重要应用。三维器官模型UV映射(UV贴图)方法一直是虚拟手术领域里的一个研究热点。UV映射就是将一个2D纹理映射到一个3D模型上,肝脏模型传统的UV映射方法就是将二维纹理的坐标位置映射到三维肝脏模型的网格顶点上。提出了一种全新的三维肝脏模型UV映射方法,该方法使UV贴图独立于网格的表面,能够有效减少GPU运算量,缩短渲染时间,并且同一张纹理图像能够应用于多个不同分辨率的相似的三维肝脏模型,充分体现出肝脏的个性化,对虚拟手术中个体的研究具有重要意义。
虚拟手术;计算机图形学;肝脏;UV映射;个性化
0 引言
随着计算机图形学技术的飞速发展,如今计算机图形学与现代医学紧密结合,虚拟手术仿真系统应运而生,目前虚拟手术仿真系统已经是医学教育和术前规划的重要工具[1]。本文所作的研究便是虚拟手术仿真系统中的一部分。虚拟的三维肝脏模型在虚拟手术上的应用具有独特的优势,一个3D肝脏模型上良好的UV贴图能够更好地提升虚拟手术中的真实体验[2]。另外,由于每个个体的肝脏形状都存在着微小的差异,因此,突出三维肝脏模型的个性化的UV映射方法成为了当前虚拟手术中针对每个个体的研究热点,对于虚拟手术及现代医学的发展也有着重要的意义。
目前,国内外对于3D肝脏模型的UV映射方法一般采用计算机图形学里传统的2D纹理映射方法。传统的UV贴图方法的本质就是将3D模型的每个顶点与2D的纹理图像绑定在一起,然后利用插值函数让2D纹理图像作缩放等操作,从而能够产生3D的效果[3]。但是,传统的UV映射方法需要对3D模型的每个顶点进行运算,存在着GPU运算量大、资源占用率高等缺点,而且这种方法应用在肝脏模型中也无法反映出不同个体中肝脏存在的微小差异,无法突出其个性化。本文提出的这种UV映射方法是一种基于立体的UV贴图方法,该方法消除了传统对于3D模型每个顶点上的运算,能够有效减少GPU运算量与资源占用率,同时能够应用于多个相似的肝脏模型,高分辨率和低分辨率的肝脏模型也能共享相同的几何纹理[4],能够有效突出3D肝脏模型UV映射的个性化。
1 肝脏表面体素化
体素化就是将一个物体连续的几何形式表示转换成最接近该物体离散的体素表示形式[5],产生体数据集,其包含了模型的表面信息和一些内部属性。对于肝脏模型表面的体素化,根据三维肝脏模型分别在x、y、z三个方向上的定义域得到肝脏模型的包围盒B,定义包围盒B为一个逻辑空间L:
L=[0,Sx-1]×[0,Sy-1]×[0,Sz-1]
(1)
三个自然数S[x,y,z]是体素化的空间分辨率。在L的每个整数位置存储UV坐标位置(UV空间Ω=[0,1]×[0,1]),三线性插值也存储在体素的角落中。在L里的一个单位大小的立方体,其在整数位置的八个角落称为一个体素。对于三个自然数S[x,y,z]的选取,一般介于4~64之间,选取的数值越小越能减少内存的占用,数值越大则表示UV贴图上更大的自由度以及更高的质量,本研究中将这三个值都取为16。
定义映射函数f:
f(p)=τ(σ(p))
(2)
(3)
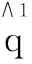
在肝脏表面进行体素化运算可得到图1。

图1 肝脏表面体素化
其中颜色的深浅变化表示相邻的不同体素。
在计算机进行渲染时,L空间中整数位置的UV坐标值被存储为一个与二通道纹理相同分辨率的三维立体纹理,片段处理器只需进行一个简单的q=σ(p)运算便可以解出对每个顶点的计算,然后在q中进行三线性插值计算[6],并返回τ(q),返回的值直接作为最终纹理的地址。这样便可以用这一操作代替传统上对于每个顶点进行线性插值的UV贴图模式,有效节省了纹理的内存带宽,降低了GPU的运算量。
2 连续UV贴图的构建
首先需要对肝脏模型表面S进行采样,每一个表面样品用si表示,并带有一个3D位置坐标pi,一个法向量ni和一个区域面积ai。定义模型表面积与纹理面积之间的比值ri,ri与si相关联,标量r随着S的变化而变化。
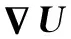
(4)
(5)

(6)
(7)
(8)
另外,定义E来衡量上述向量等式,并对所有表面的样本进行面积加权:
(9)
其中:
(10)
(11)
(12)
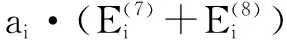


(13)
(14)
同一组变量在这里将产生一个新的二次最小二乘系统的下一次迭代。计算机执行除了最后一个的所有迭代。另一个优化则在于合并了样本共享的相同体素。
最后阶段,为了进一步提高贴图的几何独立性,非空体素的角落所产生的UV值将传到所有的其他位置的整数位置。首先分配一个平均的法线向量到L中的所有体素,作为该体素内所有样品面积加权的平均法向量;然后,用一个简单的扩散算法将这个法向量扩展到空体素,将空体素的法向量归零,再将所有近邻的平均法向量迭代分配到每个空体素;最后,将存储在L空间中的整数位置传到未分配的位置。
3 完成贴图
切线空间法线贴图是目前应用较为广泛的一种法线贴图技术[9],其与正常空间的法线贴图相比具有很大的优势:法线贴图被独立地定义在它被应用到的表面的方向上。这意味着相同的法线贴图可以让不同的模型进行共享,或者是同一模型的不同部分也可进行共享。
本文所述的贴图方式与正常的切线空间的法线贴图相似,因为在三维肝脏模型表面上处处都有切线方向的定义。在传统的贴图方式中,切线和双切线方向都是预先计算好的并作为顶点的属性进行存储,然后在各面片上进行插值。在本课题中,先计算三维纹理,然后将其存储在三维空间中(比如L空间),然后在渲染时对其进行插值。
构造过程可以归结为一个简单的算法:一个纹理元素中的t和b在x方向上的坐标值已经预先计算为u值(v的值)的一半差值,与在x方向上的3D纹理的之前之后都相关,同理,在y和z方向上也一样。之后每个纹理元素上的t和b向量将会进行重新计算。
最终,选取其中一种肝脏模型为例,使用该方法进行UV贴图,结果如图2所示。

图2 肝脏表面UV贴图
4 结果
本课题使用的操作系统为64位Windows 7,编程环境为Visual Studio 2013,编程语言采用C++,并搭载Qt创建可视化界面。选用的三维肝脏模型为OBJ格式,纹理图片的为PNG格式,另外还有OpenGL图形库及其扩展库GLUT和GLEW,使用着色语言GLSL对顶点着色器和片段着色器进行编写。
由于肝脏表面是一个盘状表面,表面上并无缺口,因此其表面的映射函数f是一个不间断的连续函数。这里选取以下四个三维的肝脏模型,这四个肝脏模型在形态上、顶点数和面片数上都不相同,具体见表1。

表1 四个三维肝脏模型对比
最后,选取同一张2D纹理图像对这四个不同的三维肝脏模型进行UV贴图,结果如图3和图4所示。
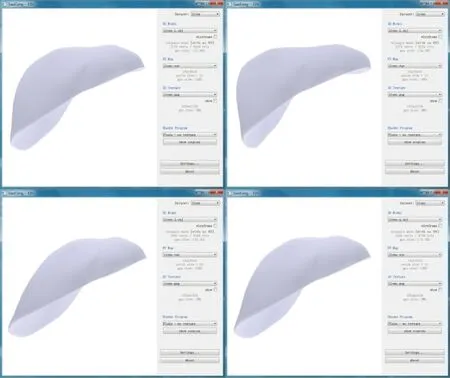
图3 贴图前肝脏模型

图4 贴图后肝脏模型
5 结论
本文针对三维肝脏模型提出了一种个性化的UV贴图方法,该方法先将肝脏模型作体素化处理,然后将UV坐标存储在一个三维的立体空间中,形成一个三维立体纹理,之后与切线空间的法线贴图类似,在渲染的时候对其进行三线性插值。该方法与传统上对于每个顶点进行线性插值的UV贴图模式相比,有效节省了纹理的内存带宽,降低了GPU的运算量,同时能使同一张二维纹理图像应用于多个不同分辨率的相似的三维肝脏模型,充分体现出三维肝脏模型的个性化,在虚拟手术中对于每个个体的肝脏的研究具有重要的意义。
由于肝脏表面纹理比较单一,因此该贴图方法能够发挥极大的优势,实现个性化。但是对于虚拟手术中更复杂的三维器官模型、更复杂的纹理图像的UV贴图以及如何在实现肝脏模型个性化的同时进一步提升肝脏模型的真实感等问题,还有待进一步研究。
[1] 赖颢升. 虚拟手术中流血效果模拟研究[D]. 济南:山东大学, 2014.
[2] 刘景丰, 林科灿. 虚拟肝技术在肝脏外科中的应用[J]. 外科理论与实践, 2010, 15(3): 214-216.
[3] 王波, 孙蔚. 基于OpenGL的新式OBJ文件纹理贴图方法研究[J]. 计算机与数字工程, 2015(8):1497-1500.
[4] TARINI M. Volume-encoded UV-maps[J]. ACM Transactions on Graphics, 2016, 35(4):107.
[5] 朱晓涛. 面向正则体积显示的三维模型体素化研究[D]. 杭州:浙江大学, 2013.
[6] 诸葛斌, 冯焕清, 周荷琴. 医学图像体绘制中的快速三线性插值算法[J]. 航天医学与医学工程, 2003, 16(3):206-209.
[7] LÉVY B, PETITJEAN S, RAY N, et al. Least squares conformal maps for automatic texture atlas generation[J]. ACM Transactions on Graphics, 2002, 21(3):362-371.
[8] DESBRUN M, MEYER M, ALLIEZ P. Intrinsic parameterizations of surface meshes[J]. Computer Graphics Forum, 2010, 21(21):209-218.
[9] 姚超. 浅谈切线空间法线贴图在三维建模中的应用[J]. 大众文艺, 2016(23): 96-97.
Research on personalized UV mapping method for 3D liver model
Gao Cong1,2, Chen Guodong1,2, Wang Na3
(1. College of Physics and Information Engineering, Fuzhou University, Fuzhou 350116, China;2. Institute of Computer Image and Graphics, Fuzhou University, Fuzhou 350116, China;3. Fuqing Branch of Fujian Normal University, Fuzhou 350300, China)
Virtual surgery is an important application in computer graphics. The UV mapping method of 3D organic model is a hot topic in the field of virtual surgery. UV mapping is to map a 2D texture to a 3D model. The traditional UV mapping method of liver model is to map the coordinates of 2D texture to the grid vertices of 3D liver model. In this paper, a new UV mapping method for 3D liver model is proposed. This method makes the UV map be independent of the surface of the mesh, which can effectively reduce the amount of GPU computation and reduce the rendering time, and the same texture image can be applied to several similar 3D liver models with different resolutions, fully reflecting the personalized liver. It is of great significance to study the individual in virtual surgery.
virtual surgery; computer graphics; liver;UV mapping; individualization
TP391.41
A
10.19358/j.issn.1674- 7720.2017.16.006
高聪,陈国栋,王娜.三维肝脏模型个性化UV映射方法研究[J].微型机与应用,2017,36(16):19-22.
2017-02-22)
高聪(1992-),男,硕士研究生,主要研究方向:计算机图形学、虚拟现实技术。
陈国栋(1979-),通信作者,男,博士,副研究员,主要研究方向:计算机图形学、虚拟现实技术。E-mail: fzucgd@163.com。
王娜(1978-),女,硕士,副教授,主要研究方向:计算机图形学、虚拟现实技术。

