DREB/CBF转录因子在植物非生物胁迫中的作用及研究进展
柏星轩, 闫 雪, 要宇晨, 宁 蕾, 王曙光, 孙黛珍
(1. 山西农业大学 农学院, 太谷 030801; 2. 山西农业大学 经济管理学院, 太谷 030801)
DREB/CBF转录因子在植物非生物胁迫中的作用及研究进展
柏星轩1, 闫 雪1, 要宇晨2, 宁 蕾1, 王曙光1, 孙黛珍1
(1. 山西农业大学 农学院, 太谷 030801; 2. 山西农业大学 经济管理学院, 太谷 030801)
DREB/CBF转录因子即干旱应答元件结合蛋白质/C-重复序列结合子,能特异地结合顺式激活元件DRE/CRT,从而激活植物细胞中与非生物胁迫抗性相关基因的表达,提高植物的抗逆性。系统地介绍了DREB/CBF转录因子的结构特点、功能、物种分布以及信号转导途径,阐述了DREB/CBF转录因子在植物抗逆基因工程育种中的作用、存在的问题,为DREB/CBF转录因子的深入研究和应用提供参考。
DREB/CBF;转录因子;转基因植物;抗逆育种
由于极端环境条件的影响,在过去的几十年中作物损失不断上升[1-2]。气候模型预测洪水、干旱和极端温度等恶劣气候条件的发生概率也都有所增加[1,3];综合气候变化和作物生产的模型预测,21世纪主要粮食作物水稻、小麦和玉米的产量会下降[1]。这将对全球粮食生产产生严重的影响,因而对于优良种质的选育显得尤为重要。研究表明,20世纪60年代以来,主要粮食作物产量的提升是通过对种质资源的开发和提高农艺措施实现的[1-3]。在较高的播种密度下,作物增加了对水分、光照和养分的竞争,这种模式对气候变异的敏感性也在上升,所以迅速开发能适应气候变化的种质对于确保粮食安全是必要的。一个有效的新模式是鉴定出在自然进化过程中对胁迫适应性有重要作用的基因,并且精确地渗入到优良品种中。这些基因位点常常与调控作用相联系,能够明显地改良植物对各种不良环境的适应性[1]。
非生物胁迫,一般是指能够通过影响植物细胞的稳定性最终使其生长和健康状况出现波动的生长条件,多是指未达最佳标准的气体和土壤条件。对于陆生植物而言,胁迫条件包括水分过饱或亏损,离子毒害(如Al3+、Cl-、Cd2+、Fe2+和Na+),离子短缺(如Fe3+、N、P、S和Zn2+),极端温度和对流层臭氧(O3)等。胁迫按照持续时间可以分为瞬时胁迫(如午时高温)或慢性胁迫(如高含量Na+的长期作用会碱化土壤)[4]。在作物早期的营养生长阶段,胁迫虽然会延缓作物的增长(阻碍细胞分裂和增大),但可能不会对高产农作物的产量有太多影响;在作物生殖生长期,胁迫会大大减少作物的产量。通常情况下,非生物胁迫常常组合出现(如高温和干旱,高温和臭氧)或连续出现(如洪涝接着干旱),因而在胁迫条件多变的环境中想要保持产量稳定增长可能需要多重防御机制的增强。结合帕累托最优原理可知,当非生物胁迫引致的减产值最小时,农业的综合生产力提升[1,4]。
转录因子是与启动子区域DNA相互作用的蛋白质,能够抑制或激活其他基因转录表达。近年来,已经相继从很多高等植物中分离出了一系列调控低温、高盐、干旱、激素等相关基因表达的转录因子,包括NAC、MYB和AP2/EREBP等[5];其中,AP2/EREBP转录因子在调节植物生长发育和对外界环境响应方面有着十分重要的作用,DREB/CBF是其一个亚族[6]。DREB转录因子能与DRE/CRT顺式作用元件或具有DRE元件的核心序列(CCGAC)特异结合,参与调控逆境相关基因的表达,介导非生物胁迫信号的传递。
1 DREB/CBF转录因子的结构特点和功能
DREB/CBF反式作用因子与DRE/CRT顺式元件能够专一性识别并结合。DRE/CRT顺式作用元件的核心序列是TACCGACAT,其中CCGAC序列是两者能够特异性结合的最小序列单位,而排在第4、第5和第7位的C碱基是两者能够结合的关键因素[7]。在干旱、高盐和低温等逆境条件下,DREB/CBF转录因子对胁迫响应基因的表达起着非常重要的作用。目前在一些受低温、高盐或干旱胁迫响应基因的启动子区域也发现了DRE核心序列或DRE元件[7],说明DRE/CRT作用元件普遍存在于逆境响应基因的启动子区域,所以,DREB/CBF转录因子对于调控逆境胁迫响应基因的表达具有十分重要的作用。
Sakuma等[7]采用凝胶移位分析,对拟南芥的DREB转录因子DREB1A和DREB2A[8]与DRE元件的特异性结合做了非常细致的研究与分析,结果如图1。
可以看出,如果DRE元件的第1、第8和第9位碱基被其他任意碱基所替换或者第2位的A被G替换,不会影响到DREB1A和DREB2A与DRE元件的结合;如果第2位的A被T或C所替换,DREB2A完全不能与之结合而DREB1A与DRE的结合能力也下降很多;如果第3位至第7位的碱基被全部替换,DREB1A和DREB2A与DRE元件的结合能力均大大降低甚至完全丧失结合的能力。这些结果显然证实了DREB1A和DREB2A转录因子的核心序列为A/GCCGAC。
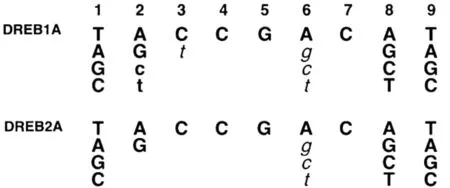
图1 DREB转录因子特异性结合序列(大写黑体表示DREB与DRE元件之间的结合力很强;小写黑体表示DREB与DRE元件之间的结合力适中;斜体表示DREB与DRE元件之间的结合力很弱)[7]
Fig 1 Binding sequence of DREB transcription factor(Capital boldface indicates that DREB and DRE components have strong adhesion; Lower-case boldface indicates that DREB and DRE components have moderate adhesion; Italic boldface indicates that DREB and DRE components have weak adhesion)[7]
迄今为止报道的DREB/CBF转录因子中,基因内均没有内含子,并且二级结构具有典型转录调控因子的特点,即C端含有大量的酸性氨基酸残基,仅有少量的碱性氨基酸残基,隶属于转录激活区;N端则含有大量的碱性氨基酸残基,隶属于核定位区。例如山菠菜的DREB1转录因子,其C端含有14个酸性氨基酸,而仅含有2个碱性氨基酸;小麦的DREB1转录因子,其C端含有15个酸性氨基酸,而碱性氨基酸仅有3个[9-10]。Qin等[11]将玉米DREB转录因子的氨基酸进行了点突变试验,更加验证了这一结果,当十四位的缬氨酸变成丙氨酸后,DREB不能与DRE序列实现特异性结合,DREB转录因子也丧失了转录激活的能力;当十九位的谷氨酸变成天冬氨酸时,DREB转录因子可以与DRE序列实现结合,但其转录激活能力大大降低。
2 DREB/CBF转录因子的克隆与鉴定
Stockinger等[12]首次从拟南芥中克隆并得到了AtCBF1,之后在许多高等植物中克隆到了DREB/CBF同源基因,DREB/CBF的克隆也因此得到了很大的发展。表1汇总了近20年来已报道的从不同植物中分离出的DREB/CBF基因,同时罗列了各基因对多种逆境胁迫的抗性。
3 DREB/CBF转录因子的信号转导途径
植物在逆境胁迫条件下能够产生大量的ABA,大多数胁迫应答基因被ABA诱导表达,对于提高植物的胁迫耐受性具有重要的作用[14]。然而在aba(ABA缺陷突变体)或abi(ABA不敏感突变体)拟南芥突变体中,同样存在一些基因,在高盐、低温和干旱等诱导胁迫条件下,不需要ABA诱导也能转录表达[15-16]。这些基因包括rd29a/lti78/cor78、cor47/rdl17、cor6.6/kin2和kin1[9]。DREB/CBF反式作用因子家族与DRE/CRT顺式元件能够专一性识别并结合,从而达到调控lti78、kin1、kin2等多种基因表达的目的[16]。受DREB/CBF调控的逆境响应基因,其启动子区域含有DRE/CRT顺式作用元件,所以该转录因子在调控抗逆基因时存在不依赖ABA的信号途径,进而增强了植物对胁迫的耐受性。

表1 DREB/CBF基因从不同植物中的克隆及对各种逆境胁迫的抗性[13]
续表1 (Continued table 1)

物种DREB/CBF类别逆境胁迫的抗性参考文献SpeciesDREB/CBFtype干旱Drought盐Salt低温ColdABAReferences黑麦草LoliumperenneLpCBF3低温[46]辣椒CapsicumannuumCaDREBLP1干旱、盐[47]狼尾草PennisetumglaucumPgDREB2A低温、干旱、盐[48]番薯IpomoeabatatasSwDREB1低温、干旱、盐[49]SwDREB1B低温[50]蓝桉EucalyptusglobulusEgCBF1低温[51]狗牙根CynodondactylonBeDREB1低温[52]BeDREB2低温芦荟Aloeveravar.chinensisAvDREB2低温、干旱、盐、ABA[53]银新杨Populusalba×P.albavar.pyramidalisPaDREB2低温、干旱、盐[54]巴西橡胶树HeveabrasiliensisHbCBF1*[55]盐芥ThellungiellaalsugineaTsCBF1*[56]榆钱菠菜AtriplexhortensisAhDREB1*[57]
*表示未知
截至目前的研究成果显示,从植物中克隆分离得到的DREB/CBF转录因子均能与DRE/CRT顺式作用元件进行交互[17]。其中,A-1与A-2组的转录因子DREB2和DREB1A/B/C,分别参与了不依赖于ABA的逆境胁迫应答途径中;其他受逆境诱导的DREB/CBF转录因子,如DREB1D/CBF4、GmDREBa、GmDRE-B2、ZmDBP2、ZmDBF1、TaDREBA6、GhDBP2和GhDBP3等参与了依赖ABA的脱水应答途径。这表明DRE/CRT顺式作用元件在参与胁迫应答的过程中,存在着依赖ABA和非依赖ABA两种信号途径,这两种途径通过一些具有相同性质的组分紧密地联系在一起,共同构成了一个纷繁复杂的信号转导网,最终使得植物的逆境胁迫耐受性增强,这一系列变化过程如图2所示。
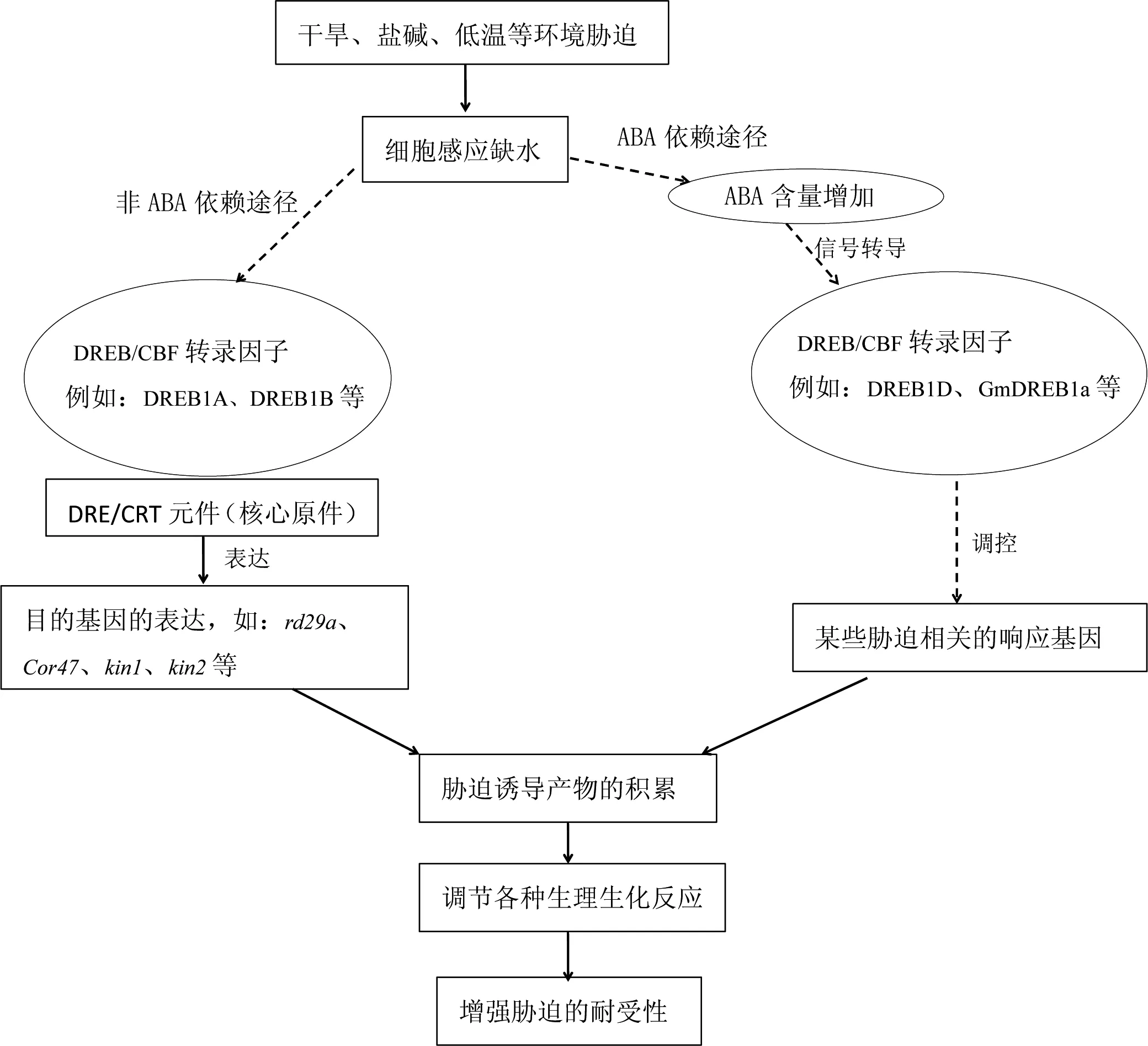
图2 逆境胁迫条件下DREB/CBF转录因子的两种信号途径
4 DREB/CBF转录因子在植物抗逆基因工程育种中的作用与应用
目前,在许多植物中已经检测到DREB/CBF的同源基因,例如小麦、大麦、水稻、玉米和大豆等[3,11]。从玉米中分离得到了一个DREB/CBF同源基因ZmDREB1A,该基因表达蛋白具有调控低温响应基因转录表达的能力,在拟南芥中过表达这一基因能够明显增加其对于低温或干旱胁迫的耐受性[11]。在超表达大豆GmDREBL的转基因拟南芥中,能够通过调控脂肪酸合成途径中的基因增加种子中的脂肪酸含量,促进转基因植株种子的增大,从而显著增加植物中的油含量[18]。目前,已从大豆中分离出6个DREB同源基因GmDREBa、GmDREBb、GmDREBc、GmDREB1、GmDREB和GmDREB3[15-16,18],其中GmDREB1、GmDREB2和GmDREB3同属于DREB家族的A-5亚家族,在拟南芥中超表达GmDREB1、GmDREB2或GmDREB3后,转基因植株对于盐碱、低温或干旱胁迫的抗性明显增强,这表明DREB/CBF的A-5亚族对提高植物抗逆性的效果更为明显[15-16,19]。济麦22、石麦15以及石麦366等常规育种得到的优良小麦品系转大豆GmDREB3后明显提高了小麦的干旱、高盐等逆境胁迫的耐受性并且提高了约10%至20%的产量(如图3)。
以上一系列的研究和试验结果表明,DREB/CBF类转录因子在促进作物抗逆性改良、提升作物产量等领域都具有十分重要的应用价值。

图3 常规育种材料济麦22株系转大豆GmDREB3后,孕穗期表型
注:该图片于2016年5月拍摄于石家庄农林科学研究院
5 DREB/CBF转录因子在现阶段研究中存在的问题及展望
目前对于DREB/CBF因子在胁迫中的转录表达方式和调控机制的研究有了一定进展,但依旧存在很大的空白区域有待深入研究。
首先,DREB/CBF转录因子的作用机理较为复杂。在转OsDREB1A和AtDREB1A的拟南芥植株中,两种基因功能相同但在植株中的作用方式又有所不同,AtDREB1A能特异地结合GCCGAC与ACCGAC两个DRE核心序列,而OsDREB1A只能结合GCCGAC序列,不能与ACCGAC序列特异结合。AtDREB1A和AtDREB1D具有特别高的同源性,在转基因植株中,AtDREB1A能被低温胁迫诱导表达,但AtDREB1D不能被低温胁迫诱导表达。此外,DREB/CBF转录因子在转基因植物中的表达也较为复杂,在转35S∶AtDREB1A和35S∶OsDREB1A拟南芥中,35S∶AtDREB1A植株与35S∶OsDREB1A植株在正常生长条件下,前者表现为严重的生长矮化,后者则表现出严重的生长延迟,这极有可能是DREB/CBF因子引起抗逆基因过表达的结果,相关胁迫应答蛋白的过量累加,使得转基因植物在正常的生长条件下表现出较高的胁迫耐受性。
其次,已有的关于DREB/CBF转录因子的功能验证主要集中在拟南芥,对于其他植物的研究少之又少。一方面,缺乏能够应用于田间种植推广的转DREB/CBF基因的抗逆转基因植物;另一方面,对于其他一些亲缘关系较远的植物能否产生相同的作用,还有待于进一步研究。同时,在调控外源的DREB/CBF基因的有效转录表达方面,除了已知的rd29a的启动子区域调控DREB/CBF基因外,还需要更多的探究。
最后,在DREB/CBF转录因子研究对象的选取方面,主流的研究多集中于草本植物,对于木本植物中的研究极为罕见。基于植株属性的巨大差异,普遍的草本植物研究成果并不能完全适用于木本植物。木本植物体形高大、结构复杂、生命周期长,大部分属于根、茎多年生,而叶一年生的类型,还有少部分木本植物的根、茎、叶均为多年生,这就注定了木本植物与草本植物存在着不同的抗逆分子机制,而且木本植物的进化程度更高、分子机制也更为复杂。
长远看来,从最开始的植物感应逆境信号,到拥有抗逆基因转录表达的整个信号传递途径中,DREB/CBF类转录因子起到至关重要的作用。同时,在样本植株选取方面,木本植物将会是一类颇具潜力的研究对象,也势必开辟一片植物非生物胁迫研究的新天地。
[1]MICHAEL M V, HASEGWAWA P M, BAILEYSERRES J. Genetic mechanisms of abiotic stress tolerance that translate to crop yield stability [J]. Nat Rev Genet, 2015, 16(4): 237-251.
[2]KIDOKORO S, WATANABE K, OHORI T, et al. Soybean DREB1/CBF-type transcription factors function in heat and drought as well as cold stress-responsive gene expression [J]. Plant J, 2015, 81(3): 505-518.
[3]杜洪伟,陈 芬,肖国樱.提高作物耐旱性的DREB转录因子研究进展[J]. 生物技术通报, 2008, 23(6): 1-6.
[4]张 梅,刘 炜,毕玉平.植物中DREBs类转录因子及其在非生物胁迫中的作用[J]. 遗传, 2009, 31(3): 236-244.
[5]倪志勇,徐兆师,李连城,等.DREB转录因子在植物抗逆胁迫中的作用机理及应用研究进展[J]. 麦类作物学报, 2008, 28(6): 1100-1106.
[6]陈金焕,夏新莉,尹伟伦.植物DREB转录因子及其转基因研究进展[J]. 分子植物育种, 2007, 5(6s): 29-35.
[7]SAKUMA Y, LIU Q, DUBOUZET J G, et al. DNA-binding specificity of the ERF/AP2 domain of Arabidopsis DREBs, transcription factors involved indehydration and cold-inducible gene expression [J]. Biochem Biophys Res Commun, 2002, 290(3): 998-1009.
[8]LIU Q, KASUGA M, SAKUMA Y, et al. Two transcription factors, DREB1 and DREB2, with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in drought-and low-temperature-responsive gene expression, respectively, in Arabidopsis [J]. Plant Cell, 1998, 10(8): 1391-1406.
[9]SHEN Y G, ZHANG W K, YAN D Q, et al. Characterization of DRE-binding transcription factor from a halophyte atriplex hortensis [J]. Theor Appl Genet, 2003, 107(1): 155-161.
[10]SHEN Y G, ZHANG W K, HE S J, et al. An EREBP/AP2 type protein inTriticcumaestivumwas a DRE-binding transcription factor induced by cold, dehydration and ABA stress[J]. Theor Appl Genet, 2003, 106(5): 923-930.
[11]QIN F, SAKUMA Y, LI J, et al. Cloning and functional analysis of a novel DREB1/CBF transcription factor involved in cold-responsive gene expression inZeamaysL.[J]. Plant Cell Physiol, 2004, 45(8): 1042-1052.
[12]STOCKINGER E, GILMOUR S, THOMASHOW M.ArabidopsisthalianaCBF1 encodes an AP2 domain-containing transcriptional activator that binds to the C-repeat /DRE, acis-acting DNA regulatory element that stimulates transcription in response to low temperature and water deficit [J]. Proc Natl Acad Sci USA, 1997, 94(3): 1035-1040.
[13]李科友,朱海兰.植物非生物逆境胁迫DREB/CBF转录因子的研究进展[J]. 林业科学, 2011, 47(1): 124-134.
[14]YAMAGUCHI S K, SHINOZAKI K. Transcriptional regulatory networks in cellular responses and tolerance to dehydration and cold stresses [J]. Annu Rev Plant Biol, 2006, 57(1):781-803.
[15]CHEN M, WANG Q, CHENG X, et al. GmDREB2, a soybean DRE-binding transcription factor, conferred drought and high-salt tolerance in transgenic plants [J]. Biochem Biophys Res Commun, 2007, 353(2): 299-305.
[16]CHEN M, XU Z, XIA L, et al. Cold-induced modulation and functional analyses of the DRE-binding transcription factor gene, GmDREB3, in soybean (GlycinemaxL.) [J]. J Exp Bot, 2009, 60(1): 121-135.
[17]高世庆,徐惠君,程宪国,等.转大豆GmDREB基因增强小麦的耐旱及耐盐性[J]. 科学通报, 2005, 50(23): 2617-2625.
[18]ZHANG Y Q, LU X, ZHAO F Y, et al. Soybean GmDREBL increases lipid content in seeds of transgenic arabidopsis[J]. Sci Rep, 2016, 6:1-13.
[19]AGARWAL P K, AGARWAL P, REDDY M K, et al. Role of DREB transcription factors in abiotic and biotic stress tolerance in plants [J]. Plant Cell, 2006, 25(12): 1263-1274.
[20]HAAKE V, COOK D, RIECHMANN J, et al. Transcription factor CBF4 is a regulator of drought adaptation in Arabidopsis[J]. Plant Physiol, 2002, 130(2): 639-648.
[21]MAGOME H, YAMAGUCHI S, HANADA A, et al. Dwarf and delayed-flowering1, a novel Arabidopsis mutant deficient in gibberellin biosynthesis because of overexpression of a putative AP2 transcription factor[J]. Plant J, 2004, 37(5): 720-729.
[22]SHI H, QIAN Y, TAN D, et al. Melatonin induces the transcripts of CBF/DREB1s and their involvement in both abiotic and biotic stresses in Arabidopsis[J]. J Pineal Res, 2015, 59(3): 334-342.
[23]CHEN J, DONG Y, WANG Y, et al. An AP2/EREBP-type transcription-factor gene from rice is cold-inducible and encodes a nuclear-localized protein[J]. Theor Appl Genet, 2003, 7(6): 972-979.
[24]DUBOUZET J, SAKUMA Y, ITO Y, et al. OsDREB genes in rice,OryzasativaL, encode transcription activators that function in drought-high-salt- and cold-responsive gene expression[J]. Plant J, 2003, 33(4): 751-763.
[25]WANG Q, GUAN Y, WU Y, et al. Overexpression of a rice OsDREB1F gene increases salt, drought, and low temperature tolerance in both Arabidopsis and rice[J]. Plant Mol Biol, 2008, 67(6): 589-602.
[26]HERATH V. Small family, big impact: in silico analysis of DREB2 transcription factor family in rice[J]. Comput Biol Chem, 2016, 65(6):128-139.
[27]李 月,代培红,刘 超,等.海岛棉5个CBF/DREB基因的克隆与表达分析[J]. 棉花学报, 2016, 28(1): 42-51.
[28]李 月,孔丽颖,李才运,等.海岛棉GbCBF6基因克隆及其在逆境胁迫下的表达分析[J]. 西北植物学报, 2015, 35(10): 1949-1955.
[29]GAO M, AIIARD G, BYASS L, et al. Regulation and characterization of four CBF transcription factors fromBrassicanapus[J]. Plant Mol Biol, 2002, 49(5): 459-471.
[30]LI X, TIAN A, LUO G, et al. Soybean DRE-binding transcription factors that are responsive to abiotic stresses[J]. Theor Appl Genet, 2005, 110(8): 1355-1362.
[31]EGAWA C, KOBAYASHI F, ISHIBASHI M, et al. Differential regulation of transcript accumulation and alternative splicing of a DREB2 homolog under abiotic stress conditions in common wheat[J]. Genes Genet Syst, 2006, 81(2): 77-91.
[32]SHAVRUKOV Y, BAHO M, LOPATO S, et al. The TaDREB3 transgene transferred by conventional crossings to different genetic backgrounds of bread wheat improves drought tolerance[J]. Plant Biotechnol J, 2016, 14(1): 313-322.
[33]CHOI D, RODRIGUEZ E, CIOSE T, et al. Barley CBF3 gene identification, expression pattern, and map location[J]. Plant Physiol, 2002, 129(4): 1781-1787.
[34]XUE G, LOVERIDGE C. HvDRF1 is involved in abscisic acid-mediated gene regulation in barley and produces two forms of AP2 transcriptional activators, interacting preferably with a CT-rich element[J]. Plant J, 2004, 37(3): 326-339.
[35]KIZIS D, PAGES M. Maize DRE-binding proteins DBF1 and DBF2 are involved in rab17 regulation through the drought-responsive element in an ABA-dependent pathway[J]. Plant J, 2002, 30(6): 679-689.
[36]QIN F, KAKIMOTO M, SAKUMA Y, et al. Regulation and functional analysis of ZmDREB2A in response to drought and heat stresses inZeamaysL.[J]. Plant J, 2007, 50(1): 54-69.
[37]WANG Q, MAILLARD M, SCHIBLER U, et al. Cardiac hypertrophy, low blood pressure, and low aldosterone levels in mice devoid of the three circadian PAR bZip transcription factors DBP, HLF, and TEF[J]. Am J Physiol Regul Integr Comp Physiol, 2010, 299(4): 1013-1019.
[38]LIU S, WANG X, WANG H, et al. Genome-wide analysis ofZmDREBgenes and their association with natural variation in drought tolerance at seedling stage ofZeamaysL.[J]. PLoS Genet, 2013, 9(9): 1003-1790.
[39]GU L, ZHANG Y, ZHANG M, et al. ZmGOLS2, a target of transcription factor ZmDREB2A, offers similar protection against abiotic stress as ZmDREB2A[J]. Plant Mol Biol, 2016, 90(1-2): 157-170.
[40]何 姣.冰岛虞美人(Papavernudicaule)不同品种耐寒性评价及其冷诱导基因PnDREB1的克隆[D]. 雅安:四川农业大学,2015.
[41]WU J, FOLTA K, XIE Y, et al. Overexpression ofMuscadiniarotundifoliaCBF2 gene enhances biotic and abiotic stress tolerance in Arabidopsis[J]. Protoplasma, 2017, 254(1): 239-251.
[42]HUANG B, JIN L, LIU J. Identification and characterization of the novel gene GhDBP2 encoding a DRE-binding protein from cotton (Gossypiumhirsutum) [J]. J Plant Physiol, 2008, 165(2): 214-223.
[43]HUANG B, LIU J. Cloning and functional analysis of the novel gene GhDBP3 encoding a DRE-binding transcription factor fromGossypiumhirsutum[J]. Biochim Biophys Acta, 2006, 1759(6): 263-269.
[44]HUANG B, LIU J. A cotton dehydration responsive element binding protein functions as a transcriptional repressor of DRE-mediated gene expression[J]. Biochem Biophys Res Commun, 2006, 343(4): 1023-1031.
[45]阳文龙,刘敬梅,刘 强,等.高羊茅DREB类转录因子基因的分离及鉴定分析[J]. 核农学报, 2006, 20(3): 187-192.
[46]XIONG Y, FEI S. Functional and phylogenetic analysis of a DREB/CBF-like gene in perennial ryegrass (LoliumperenneL.) [J]. Planta, 2006, 224(4): 878-888.
[47]HONG J, KIM W. Isolation and functional characterization of theCa-DREBLP1 gene encoding a dehydration-responsive element binding-factor-like protein 1 in hot pepper[J]. Planta, 2005, 220(6): 875-888.
[48]AGARWAL P,NAIR S, SOPORY S K, et al. Stress-inducible DREB2A transcription factor fromPennisetumglaucumis a phosphoprotein and its phosphorylation negatively regulates its DNA-binding activity [J]. Mol Genet Genomics, 2007, 277(2): 189-198.
[49]KIM Y, YANG K, RYU S, et al. Molecular characterization of a cDNA encoding DRE-binding transcription factor from dehydration-treated fibrous roots of sweetpotato [J]. Plant Physiol Biochem, 2008, 46(2): 196-204.
[50]赵晓飞.甘薯冷诱导转录组测序及转录因子SwDREB1B在抗冷调节中的作用[D]. 杭州:浙江农林大学, 2015.
[51]GAMBOA M, RASMUSSEN P, VALENZUELA P, et al. Isolation and characterization of a cDNA encoding a CBF transcription factor fromE.globulus[J]. Plant Physiol Biochem, 2007, 45(1): 1-5.
[52]谢永丽,王自章,刘 强,等.草坪草狗牙根中抗逆基因BeDREB的克隆及功能鉴定[J]. 中国生物化学与分子生物学报, 2005, 21(4): 521-527.
[53]张 倩.中华芦荟抗逆基因AlDREB2和AlTINY的克隆与功能的初步分析[D]. 重庆:西南大学, 2009.
[54]秦红霞,贾志平,张海超,等.银新杨中与DRE元件结合的转录因子的克隆及鉴定分析[J]. 生物工程学报, 2005, 21(6) :906-910.
[55]程 汉,安泽伟,黄华孙.巴西橡胶树CBF1基因的克隆和序列分析[J]. 热带作物学报, 2005, 26(3): 50-54.
[56]高 峰,高 强,岳桂东,等.小盐芥(Thellungiellasalsuginea)CBF1基因的克隆[J]. 山东大学学报, 2005, 40(5): 113-117.
[57]SHEN Y, ZHANG W, YAN D, et al. Characterization of a DRE-binding transcription factor from a halophyte Atriplex hortensis [J]. Theor Appl Genet, 2003, 107(1): 155-161.
The role and research progress of DREB/CBF transcription factors in plant abiotic stress
BAI Xing-xuan1, YAN Xue1, YAO Yu-chen2, NING Lei1, WANG Shu-guang1, SUN Dai-zhen1
(1. College of Agriculture; 2. College of Economics and Management, Shanxi Agricultural University, Taigu 030801, China)
DREB/CBF (Dehydration responsive element binding protein/C-repeat binding factor)transcription factor, the drought responsive element-binding protein/C-repeat sequence,can specifically interact with the DRE/CRT (Dehydration-responsive element/Cis-acting elements). It activates the expressions of genes associated with abiotic stress resistance in plant cells and thus enhances the plant resistance to various stresses. In this paper, the structure, function, species distribution and signal transduction of DREB/CBF transcription factors were systematically introduced. The roles and existing problems of DREB/CBF transcription factors in plant genetic engineering breeding were discussed.All these above are aimed at providing references for the further research and application of DREB/CBF transcription factor.
DREB/CBF; transcription factor; transgenic plant; stress tolerance breeding
2016-11-18;
2016-11-30
国家自然科学基金(31671607);国家转基因生物新品种培育科技重大专项(2014ZX0800203B-003);山西省自然科学基金(2014011004-3)
柏星轩,硕士,研究方向为小麦分子育种,E-mail:bxx1015@126.com
孙黛珍,博士,教授,研究方向为小麦分子育种,E-mail:sdz64@126.com
Q945.78
A
2095-1736(2017)04-0088-06
doi∶10.3969/j.issn.2095-1736.2017.04.088

