猪繁殖与呼吸系统综合征病毒华南株GDgz的分离鉴定及遗传进化分析
于林洋,董建国,2,张乐宜,刘燕玲,梁鹏帅,王 磊,宋长绪
(1.华南农业大学动物科学学院,广东 广州 510642;2.信阳农林学院牧医工程学院,河南 信阳 464000)
猪繁殖与呼吸系统综合征病毒华南株GDgz的分离鉴定及遗传进化分析
于林洋1,董建国1,2,张乐宜1,刘燕玲1,梁鹏帅1,王 磊1,宋长绪1
(1.华南农业大学动物科学学院,广东 广州 510642;2.信阳农林学院牧医工程学院,河南 信阳 464000)
为了解华南地区猪繁殖与呼吸综合征病毒(PRRSV)流行株的变异特性,用Marc-145细胞从广东省疑似猪繁殖与呼吸综合征发病猪场采集的肺组织中分离得到1株PRRSV(命名为GDgz),该毒株能产生明显的细胞病变。全基因组进化分析和同源性比对分析结果显示,GDgz与中国高致病性毒株位于同一分支,与欧洲型毒株Lelystad-virus同源性最低、仅为60.3%,与美洲型经典毒株VR-2332的同源性为88.8%,与中国高致病性毒株JXA1和HuN4同源性为98.8% 和98.9%,与JXA1的疫苗株同源性为91.1%,与NADC30和NADC30-like毒株 CHsx1401和JL580同源性分别为84.5%、87.2%和83.6%。NSP2序列比对结果显示,GDgz在高变区存在30个不连续氨基酸缺失。GP5序列比对结果显示,GDgz在GP5抗原表位上有氨基酸突变,表明GDgz属于美洲型高致病性毒株传代致弱的疫苗株,且在抗原表位上有一定程度的突变。
猪;繁殖与呼吸综合征病毒;分离鉴定;遗传进化
猪繁殖与呼吸系统综合征(P o r c i n e reproductive and respiratory syndrome,PRRS)是造成当今世界养猪业重大经济损失的传染性疾病之一,它主要造成母猪的繁殖系统障碍和各年龄猪群的呼吸系统疾病[1-2]。该病的病原是猪繁殖与呼吸系统综合征病毒(PRRSV)。PRRSV为有囊膜的单股正链RNA病毒,属于套式病毒目、动脉炎病毒科、动脉炎病毒属[3]。
PRRSV基因组全长约15 kb,含有10个开放阅读框(ORFs)、5′端UTR、3′端UTR和poly(A)尾巴。开放阅读框分别为ORF1a、ORF1b、ORF2a、ORF2b、ORF3~7和ORF5a[4-5]。其中ORF1a和ORF1b进行12个非结构蛋白Nsp1~Nsp12的编码,在病毒复制、转录和拮抗干扰素等方面起着重要作用[6-7]。ORF2~7进行结构蛋白GP2、GP3、GP4、GP5、M和N的编码[8]。病毒中和抗体生成有关的抗原表位主要位于结构蛋白上[9]。5′端UTR用于调节病毒RNA功能及结构蛋白的表达,3′端UTR位于ORF7下游,主要进行病毒复制酶识别并结合以启动负链RNA的合成[10-11]。根据PRRSV变异以及流行情况,PRRSV可分为以Lelystad-virus为代表的欧洲型(Ⅰ型)和以VR2332为代表的北美型(Ⅱ型)[12]。我国PRRSV主要以基因Ⅱ型为主。PRRS对我国养猪业造成巨大的经济损失,尤其是1996年以低致病性毒株CH-1a为代表和2006年以高致病性毒株JXA1为代表的两次PRRS暴发对我国养猪业造成了重大经济损失[13]。2014年,有研究报道NADC30-like毒株在我国广泛流行,进一步打击了我国养猪业,也给PRRS的防控带来了新挑战[14-17]。
为了进一步了解我国华南地区PRRSV的流行及变异情况,我们于2016年10月对广东省广州市某种猪场暴发疾病死亡的30日龄保育小猪进行检测,结果呈PRRSV阳性,最后通过在Marc-145细胞上将病毒成功分离并对其全基因组进行测定和分析,将测序结果与其他代表PRRSV毒株进行序列比对和进化树分析,进而分析所分离的毒株与目前国内PRRSV流行毒株的相似性,从而了解广东省目前PRRSV毒株的流行情况。
1 材料与方法
1.1 试验材料
1.1.1 病料 广东省广州市疑似暴发PRRS的自然发病猪,采集发病猪肺脏组织于-80℃冻存。
1.1.2 细胞、菌体及载体 本试验所使用的Marc-145细胞、克隆宿主菌E. coliDH5α感受态细胞以及阳性对照病毒株GDzj株均由华南农业大学动物科学学院猪病防控研究室保存。pMD-19T克隆载体购自宝生物工程(大连)有限公司。
1.1.3 主要试剂 病毒DNA/RNA提取试剂盒购自Magen公司;反转录试剂盒、胶回收试剂盒购于Promega公司;RT-PCR相关试剂盒购自TOYOBO生物科技有限公司和Vazyme公司;DNA Marker DL2000等购自TaKaRa公司; EB替代染料购自广州华奇盛生物科技有限公司;pEASY-Blunt Simple Cloning Vector和Trans-5α感受态细胞均购自北京全式金生物技术有限公司。
1.2 试验方法
1.2.1 引物设计与合成 根据GenBank中发布的VR-2332等PRRSV毒株全基因组序列进行生物信息学分析,选取保守区域设计13对特异性引物(表1)用于扩增全基因组序列。引物由苏州金维智生物公司合成。

表1 PRRSV全基因组扩增引物
1.2.2 病料处理及PCR检测 将采集的发病猪肺脏组织剪碎,加入1 mL PBS研磨后收集研磨液于1.5 mL EP管中,12 000 r/min离心5 min,取上清用0.22 μm微孔滤器过滤除菌,收集滤液并取200 μL进行病毒DNA/RNA核酸提取及PRRSV NSP2、CSFV、PPV的 RT-PCR检测与PRV、PCV2的常规PCR检测,剩余滤液于-80℃保存。
1.2.3 病毒分离 使用12孔细胞培养板培养Marc-145细胞,待Marc-145细胞长满单层后弃去上清,用PBS洗3遍,接种50 μL过滤过的含有病毒的病料上清,加入100 μL不含血清、含有2%双抗的1 640培养液。37℃下孵育1 h后,弃去上清,加入含2%血清的1 640维持液1 mL,置于细胞培养箱中培养3~5 d,约80%细胞出现细胞病变效应CPE后即可收取样品。将毒液置于-80℃冰箱保存。
1.2.4 病毒接毒 使用25 mL培养瓶培养Marc-145细胞,待Marc-145细胞长满单层后弃去上清,用PBS洗3遍,接种200 μL过滤的含有病毒的病料上清,加入1 mL不含血清的含有2%双抗的1 640培养液。37℃下孵育1 h,弃去上清,加入含2%血清的1 640维持液5 mL,置于细胞培养箱中培养3~5 d,约80%细胞出现CPE后即可收取样品。将毒液置于-80℃冰箱保存。
1.2.5 核酸提取及反转录 取在Marc-145细胞上分离的第3代GDgz毒株,根据Magen公司产品试剂盒进行病毒核酸提取,然后根据Promega反转录试剂盒对所提取的病毒核酸进行反转录。
1.2.6 PRRSV GDgz毒株目的片段分段扩增 使用TOYOBO生物科技有限公司高保真酶对目的片段进行扩增。扩增体系为50 μL,KOD-Plus-Neo 1 μL,10×PCR Buffer for KODPlus-Neo 5 μL,2 mmol/L dNTP 5 μL,25 mmol/L MgSO43 μL,cDNA模板4 μL,上游引物2 μL,下游引物2 μL,RNase Free H2O 28 μL。PCR扩增程序:94℃预变性2 min;98℃变性10 s,62℃退火30 s,68℃延伸30 s/kb,35个循环;72℃延伸10 min。取5 μL的PCR产物进行1%琼脂糖凝胶电泳。
1.2.7 序列测定 PCR扩增产物通过1%琼脂糖凝胶电泳鉴定后,使用胶回收试剂盒对目的片段进行胶回收。然后连接到北京全式金生物有限公司的pEASY-Blunt Simple Cloning Vector上。经过转化挑菌鉴定后送往测序公司进行测序。
1.2.8 序列比对 利用Lasergene及Mega 5.0软件将分离的毒株全基因组与国内外分离毒株进行序列比对并构建进化树,并分别将NSP2基因序列和GP5氨基酸序列与其他国内外代表毒株的NSP2基因序列和GP5氨基酸序列进行比对分析。PRRSV代表毒株的基因序列均来源于GenBank。
2 结果与分析
2.1 病料的RT-PCR检测
用NSP2基因检测引物对处理好的病料样品进行检测,扩增出NSP2基因片段,琼脂糖凝胶电泳分析结果表明,与阳性对照片段大小一致(图1),而样品中RNA病毒CSFV、PPV与DNA病毒PRV、PCV2等检测均呈阴性(图略)。
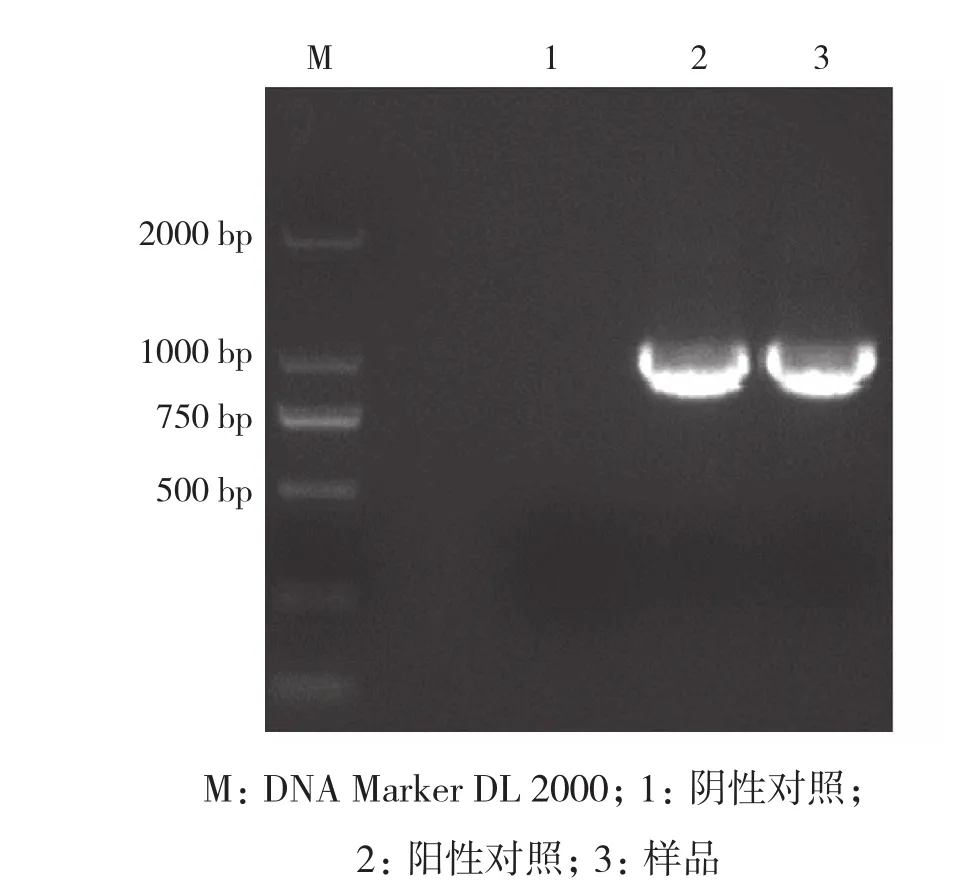
图1 PRRSV NSP2基因片段扩增电泳结果
2.2 病毒分离
将经处理的病料接种到Marc-145细胞,培养48~96 h,每天观察细胞情况,待细胞聚集、皱缩并脱落等有明显CPE时,收取病毒液(图2,封三)。
2.3 PRRSV全基因组测定
运用13对相互重叠的特异性引物,通过RT-PCR法扩增病毒的13个cDNA片段。琼脂糖凝胶电泳分析显示,扩增的13 个RT-PCR 产物大小与预期长度一致(图3)。将扩增的片段胶回收后连接pEASY-Blunt Simple Cloning Vector上送公司测序,最后对13个片段测序结果进行分析,SeqMan软件拼接序列,获得全长的PRRSV基因组序列,全长为15 353 nt,命名为GDgz。
2.4 PRRSV全基因进化树的构建

图3 PRRSV基因片段的RT-PCR扩增结果
运用分子生物学软件对GDgz毒株和其他国内外具有代表性的毒株进行进化树构建,结果(图4)显示,整个遗传进化树可分为以Lelystad virus为代表的欧洲型和以VR2332为代表的美洲型两大分支,以VR-2332为代表的美洲型分支又可进一步分为以美国NADC30株为代表的分支、以VR2332和我国低致病性毒株CH-1a为代表的分支以及以高致病性毒株JXA1、HUN4为代表的分支。结果表明,GDgz与国内的高致病性毒株JXA1、HUN4、JXwn06属于同一分支,且与高致病性毒株的疫苗株JXA1-P80同源性最高。

图4 基于PRRSV全基因组的进化树分析
2.5 PRRSV全基因同源性分析

图5 PRRSV全基因序列比对结果
用DNAStar软件进行PRRSV全基因的进化分析,结果(图5)显示,GDgz与JXA1-P80同源性最高、为99.1%,与HP-PRRSV毒株JXA1、HUN4和JXwn06同源性分别为98.8%、 98.9%和98.9%,与NADC30和NADC30-like毒株 JL580、CHsx1401同源性分别为84.5%、87.2%和83.6%,与美洲型代表株VR-2332同源性为88.8%,与欧洲型代表株Lelystad virus同源性仅为60.2%。
2.6 PRRSV NSP2氨基酸序列比对
使用DANStar对GDgz和国内外代表毒株进行NSP2氨基酸的序列比对,结果(图6)显示,GDgz与高致病性毒株HUN4、JXA1、JXwn06和高致病疫苗株JXA1-P80一样,NSP2在511、534~562位存在30个不连续氨基酸的缺失,在其他位点没有氨基酸的缺失或插入。

图6 PRRSV NSP2氨基酸序列比对结果
2.7 PRRSV GP5氨基酸序列比对
运用DNAStar软件对GDgz和国内外参考毒株进行GP5蛋白的氨基酸序列比对,已有研究表明美洲型毒株GP5蛋白的表位有3个,2个(aa27~aa30 和 aa180~aa197)为非中和表位,1个(aa37~aa45)为中和表位(图7),与美洲型代表毒株VR-2332相比,GDgz在非中和表位中有1个A29V的变异,与其他代表毒株相似;在185位有V185A突变,该突变与高致病性毒株及其疫苗株相似;在189位有I189L突变,该突变与国内经典毒株和高致病性毒株及其疫苗株相似。中和表位中,在39位有1个L39I突变,该突变与高致病性毒株及其疫苗株相似。

图7 PRRSV GP5 氨基酸序列比对结果
3 讨论
自从美国分离了第1株PRRSV毒株VR-2332以及1996年中国分离了第1株PRRSV CH-1a以来,PRRS已经成为严重影响养猪业发展的疾病之一,每年导致巨大的经济损失。我国分离的毒株以北美型PRRSV毒株为主[18-19],目前猪场主要通过疫苗免疫该病,国内用于PRRSV防控的疫苗主要有经典毒株和高致病性毒株减毒活疫苗以及灭活苗。由于我国PRRSV毒株种类众多,研发生产疫苗的厂家众多,致使疫苗使用混乱,一个猪场可能存在免疫多种疫苗的情况,导致不同毒株间发生遗传重组的可能。加上RNA病毒复制酶低保真性和免疫系统压力的影响,进而加速PRRSV病毒不断发生变异[20-21]。2006年出现的高致病性猪繁殖与呼吸综合征病毒和2014年出现的NADC30-like毒株说明我国PRRSV变异情况越来越严重,致使现有疫苗免疫效果差,不能很好地提供免疫保护,给疾病防控造成了巨大困难。本研究通过细胞培养分离病毒和RT-PCR方法对GDgz毒株全基因组进行分段扩增,并克隆、测序,通过相关软件对序列进行拼接后得到GDgz毒株的全基因序列,并对其与其他PRRSV毒株同源性进行比较和构建进化树。结果显示GDgz毒株与中国高致病性毒株JXA1疫苗株JXA1-P80同源性最高,表明广东猪场由于广泛使用减毒活疫苗,导致疫苗株在猪场广泛流行,给PRRSV的变异增加了风险。
NSP2基因是PRRSV基因组上变异最大的基因之一,遗传进化分析表明,NSP2区域存在广泛的基因缺失、插入和突变。有研究证实HPPRRSV毒株与经典毒株相比在NSP2区域存在30个不连续氨基酸的缺失[13]。对比国内外NSP2氨基酸序列可知,GDgz的NSP2存在30个不连续氨基酸的缺失,符合高致病性PRRSV毒株的特征。
抗原表位是决定病毒免疫特性的重要因素之一。目前,已有研究者证实美洲型 PRRSV GP5 蛋白抗原表位有1个中和表位和2个非中和表位[22]。GP5 蛋白由PRRSV ORF5 基因编码,是病毒主要结构蛋白之一,为囊膜糖蛋白,具有较高的免疫原性和中和活性[23]。与美洲型代表毒株VR-2332相比,GDgz在非中和表位中有1个A29V的变异,在185位有V185A突变,在189位有I189L突变;中和表位中,在39位有1个L39I突变。这些突变很可能影响蛋白的免疫原性,进而影响疫苗的免疫效果,导致猪场免疫失败,PRRS暴发和流行。
最近国内新出现了NADC30-like毒株,有研究证实该类毒株具有高致病性,是由中国高致病性毒株和美国NADC30毒株发生重组所致,商品化疫苗对该类型毒株缺乏免疫保护作用。我们分离的毒株与商品化疫苗JXA1-P80高度同源,这些结果表明弱毒苗的广泛使用导致中国 PRRSV 毒株变异情况越来越严重,有必要开发新型疫苗预防疾病发生,早日解决PRRS感染与流行的问题。
[1] Dekkers J,Rowland R R,Lunney J K,et al. Host genetics of response to porcine reproductive and respiratory syndrome in nursery pigs[J/OL]. Vet Microbiol,2017,http://www.sciencedirect. com/science/article/pii/S0378113516306782,2017-03-22.
[2] Ke H,Yoo D. The viral innate immune antagonism and an alternative vaccine design for PRRS virus[J]. Vet Res,1997,28:305-352.
[3] Snijder E J,Meulenberg J M. The molecular biology of arteriviruses[J]. Vet Microbiol,2017,81(Pt 4):853–879.
[4] Han J,Zhou L,Ge X,et al. Pathogenesis and control of the Chinese highly pathogenic porcine reproductive and respiratory syndrome virus[J/ OL]. Vet Microbiol,http://www.sciencedirect. com/science/article/pii/S0378113516305120,2017-03-02.
[5] Aeksiri N,Jantafong T. Structural insights into typeⅠ and typeⅡ of nsp4 porcine reproductive and respiratory syndrome virus (nsp4 PRRSV)by molecular dynamics simulations[J]. J Mol Graph Model,2017,74:125-134.
[6] 刘准,王凤雪,武华,等. 猪繁殖与呼吸综合征病毒非结构蛋白研究进展[J]. 动物医学进展,2010,31(12):120-124.
[7] Fang Y,Snijder E L. The PRRSV replicase:Exploring the multifunctionality of an intriguing set of nonstructural proteins[J]. Virus Res,2010,154(1-2):61-76.
[8] Wang Q,Li Y,Dong H,et al. Identification of host cellular proteins that interact with the M protein of a highly pathogenic porcine reproductive and respiratory syndrome virus vaccine strain [J]. Virol J,2017,14(1):39.
[9] 李国娟,田永强,李建强,等. 猪繁殖与呼吸综合征病毒分子生物学研究进展[J]. 生物技术通报,2009(12):37-41.
[10] 刘光清,薛 强,仇华吉,等. 猪繁殖与呼吸综合征病毒CH-1a株非结构基因的分子克隆及其基因特征的研究[J]. 中国预防兽医学报,2002,24(2):81-87.
[11] Leng C L,Tian Z J,Zhang W C,et al. Characterization of two newly emerged isolates of porcine reproductive and respiratory syndrome virus frome Northeast China in 2013[J]. Vet Microbiol,2014,171(1-2):41-52.
[12] Won I K,Jac J K,Sang H C,et al. Different biological characteristics of wild-type porcine respiratory syndrome viruses and vaccine viruses and identification of the corresponding gentic determinants[J]. J Clin Microbiol,2008,46(5):1758-1768.
[13] Bi C,Shao Z,Zhang Y,et al. Identification of a linear B-cell epitope on non-structural protein 12 of porcine reproductive and respiratory syndrome virus,using a monoclonal antibody[J/OL]. Arch Virol,http://www.doc88.com/p-1611301780273. html,2017-04-01.
[14] Li X,Bao H,Wang Y,Tian K Widespread of NADC30-like PRRSV in China:Another Pandora's box for Chinese pig industry as the outbreak of highly pathogenic PRRSV in 2006[J]? Infect Genet Evol. 2017,49:12-13.
[15] Zhang Q,Jiang P,Song Z,et al. Pathogenicity and antigenicity of a novel NADC30-like strain of porcine reproductive and respiratory syndrome virus emerged in China[J]. Vet Microbiol,2016,197:93-101.
[16] Li X,Wu J,Tan F,et al. Genome characterization of two NADC30-like porcine reproductive and respiratory syndrome viruses in China[J].Springerplus,2016,5(1):1677.
[17] Sun Z,Wang J,Bai X,et al. Pathogenicity comparison between highly pathogenic and NADC30-like porcine reproductive and respiratory syndrome virus[J]. Arch Virol,2016,161(8):2257-2261.
[18] Zhou Y J,Yu H,Tian Z J,et al. Genetic diversity of the ORF5 gene of porcine reproductive and respiratory syndrome virus isolates in China from 2006 to 2008[J]. Virus Res,2009,144(1-2):136-144.
[19] Zhou L,Yang H. Porcine reproductive and respiratory syndrome in China[J]. Virus Res,2010,154(1-2):31-37.
[20] Yuan S,Nelsen C J,Murtaugh M P,et al. Recombination between North American strains of porcine reproductive and respiratory syndrome virus[J]. Virus Res,1999,61:87-98.
[21] Leng X L,Li Z,Xia M,et al. Mutations in the genome of the highly pathogenic porcine reproductive and respiratory syndrome virus potentially related to attenuation[J]. Vet Microbiol,2012,157(1-2):50-60.
[22] Kijona F K,Gholamreza H,Denis K G,et al. Genetic variationand phylogentic analyses of the ORF5 gene of acute porcine respiratory and reproductive syndrome viruse isolate[J]. Vet Microbiol,2001,83(3):249-263.
[23] Zhang Q Y,Xu X J,You S M,et al. Emerging of two new subgenotypes of porcine reproductive and respiratory syndrome viruses in Southeast China[J]. Microb Pathog,2016,97:27-33.
[24] Bai X,Wang Y,Xu X,et al. Commercial vaccines provide limited protection to NADC30-like PRRSV infection[J]. Vaccine,2016,34(46):5540-5545.
[25] Zhao K,Ye C,Chang XB,et al. Importation and recombination are responsible for the latest emergence of highly pathogenic porcine reproductive and respiratory syndrome virus in China[J]. J Virol,2015,89(20):10712-10716.
(责任编辑 崔建勋)
Isolation,identification and genetic variation analysis of South China strain GDgz of porcine reproductive and respiratory syndrome virus
YU Lin-yang1,DONG Jian-guo1,2,ZHANG Le-yi1,LIU Yan-ling1,LIANG Peng-shuai1,WANG Lei1,SONG Chang-xu1
(1. College of Animal Science,South China Agricultural University,Guangzhou 510642,China;2. College of Animal Husbandry and Veterinary,Xinyang College of Agriculture and Forestry,Xinyang 464000,China)
To understand the evolution of porcine reproductive and respiratory syndrome virus(PRRSV)in South China,we isolated a PRRSV strain in Marc-145 cell from lung of suspected PRRSV-positive pig in South China and named GDgz. GDgz strain could cause typical cenotaphic effect. The results of phylogenetic tree and homology alignment analysis showed that GDgz belonged to the same cluster with high pathogenetic PRRSV(HPPRRSV) strains in China;GDgz had a far distance with European strain Lelystad virus,the nucleotide homology was only 60.3%,with American classic strain VR-2332 was 88.8%,with Chinese HP-PRRSV strains JXA1 and HuN4 were 98.8% and 98.9%,with the vaccine strain JXA1-P80 of JXA1 was 99.1%,with NADC30 and NADC30-like strains CHsx1401 and JL580 were 84.5%,87.2 and 83.6%,respectively. The result of sequence alignment showedthat there were 30 discontinuous amino acids deletion in the high variable region of NSP2. These results showed that GDgz belonged to vaccine strain of high pathogenetic North American type PRRSV and had some mutations in the epitope region.
porcine;reproductive and respiratory syndrome virus;isolation and identification;genetic variation

S855.3
A
1004-874X(2017)04-0138-08
于林洋,董建国,张乐宜,等. 猪繁殖与呼吸系统综合征病毒华南株GDgz的分离鉴定及遗传进化分析[J].广东农业科学,2017,44(4):138-145.
2017-02-19
国家科技支撑计划项目(2015BAD12B02-5)
于林洋(1992-),男,在读硕士生,E-mail: 396329755@qq.com
宋长绪(1965-),男,博士,研究员,E-mail: cxsong2004@163.com

