河北省平菇主栽品种分子身份证的构建*
李 慧,王朝江,高春燕
(河北省农林科学院遗传生理研究所,河北 石家庄 050051)
〈生理生化〉
河北省平菇主栽品种分子身份证的构建*
李 慧,王朝江,高春燕**
(河北省农林科学院遗传生理研究所,河北 石家庄 050051)
以河北省主栽的17个平菇品种为材料,对构建平菇种质分子身份证的方法进行探讨研究。首先应用CO1对平菇物种进行鉴定,然后利用SSR Locator软件从Pleurotus ostreatus PC15 v2.0全基因组序列上查找SSRs位点并设计引物,采用筛选后的SSR引物对供试菌株进行扩增,然后根据引物对不同菌株扩增产生的带型进行编码、组合,构建SSR分子身份证。结果显示,17个平菇品种包括4株糙皮侧耳(P.ostreatus)、8株白黄侧耳(P. cornucopiae)和5株肺形侧耳(P.pulmonarius)。从48对SSR引物中筛选出9对引物,在17个平菇品种间共检测到53种带型,将不同带型赋值编码后组合成17个平菇品种的特异分子身份证。该研究表明CO1结合SSR标记技术可有效地用于平菇种质分子身份证的构建。
平菇;分子身份证;CO1;SSR
平菇(Oyster mushroom)是一种栽培历史悠久,栽培范围广泛的食用菌[1-2]。平菇子实体肉质鲜美,营养丰富且具有一定的保健功效[3]。据中国食用菌协会统计,2015年我国平菇产量约占食用菌总产量的17%,位居第3位。
平菇原专指糙皮侧耳(Pleurotus ostreatus),现常将侧耳属几个可以栽培的近缘种泛称为平菇[4]。平菇近缘种的鉴定方法,传统上是依靠形态学[5]或结合拮抗试验和同工酶分析等多项分类方法[6]。随着现代分子生物学技术的迅速发展,多种快捷、稳定、准确的DNA分子标记技术应用到真菌鉴定中来。ITS(internal transcribed spacer) 序列分析是侧耳属鉴定的常用方法[7-8],但是由于没有界定种的明确界限并且ITS序列存在近缘种种内差异比种间大的问题[9],以及数据库序列信息的不准确性[10-11],造成了该方法的不准确。黄晨阳[12]根据侧耳属不同物种的CO1(细胞色素c氧化酶亚基Ⅰ基因)开发出特异引物可以对平菇的糙皮侧耳(P.ostreatus)、白黄侧耳(P. cornucopiae)以及肺形侧耳(P.pulmonarius)进行准确鉴别。平菇栽培品种繁多,据报道有些省份栽培用平菇品种多达十几至几十个,这些品种存在严重的同物异名、同名异物现象[7,13]。如何准确有效地进行品种鉴别,对于平菇品种繁育、引种育种具有重要意义,也是品种知识产权保护的需要。
近年来,分子生物学手段因具有不受环境影响且变异丰富等优点,被广泛应用到食用菌菌株鉴别中来[14-15]。尤其是分子身份证概念的提出,不仅给品种本身赋予了一个识别品种的标准,而且数字化的DNA指纹,使品种鉴别更加直观简洁[16]。SSR(simple sequence repeats)分子标记常用于分子身份证的构建,具有多态性高、稳定性和重复性好、易于检测而又识别率高等优点[17]。王慧玲等[18]利用国际通用的9对SSR引物构建了13个中国葡萄优新品种的分子身份证;陈昌文等[16]从16对SSR引物中筛选出8对核心引物构建了176份桃种质的可辩分子身份证;张肖雅等[19]利用5对SSR引物将11株灵芝菌种完全区分开,并构建其分子身份证。王新新等[20]根据8对SSR引物产生的不同扩增谱型,构建了29份双孢蘑菇菌株的分子身份证。平菇品种繁多,种质较为混乱,然而关于平菇SSR分子身份证的研究未见报道。本研究选取河北省常栽的17个平菇品种,在对平菇近缘种进行CO1物种鉴定的基础上,通过SSR分子标记技术,构建平菇品种的分子身份证,为当地平菇品种鉴定提供理论依据,也对平菇种质分子身份证的构建提供一定的方法参考。
1 材料与方法
1.1 材料
供试菌株为河北省主栽平菇品种,来源于科研单位或菌种公司。经多年的栽培试验测试,17个平菇品种均表现为稳定而特异的表型性状,并且拮抗试验显示17个菌株两两之间均有拮抗。供试菌株的品种名称和来源见表1。

表1 供试菌株名称和来源Tab.1 Name and source of tested strains
1.2 菌丝培养和DNA提取
将活化好的平菇菌种块转接至铺有玻璃纸的PDA平板上,25℃暗光培养5 d,刮取新鲜菌丝体100 mg左右,加入液氮研磨,按照植物基因组DNA提取试剂盒(购于天根生化科技有限公司) 说明,提取基因组DNA。采用1%琼脂糖凝胶电泳和紫外分光光度计检测提取的DNA浓度和纯度。将DNA浓度稀释至20 ng·μL-1~30 ng·μL-1,于-20℃下保存备用。
1.3 CO1扩增和分析
参考黄晨阳[12]方法,对17个菌株进行CO1基因扩增,根据扩增条带的大小进行物种鉴定,片段大小约为1 700 bp的为P.ostreatus,约为2 300 bp的为P.cornucopiae,约为3 600 bp的为P.pulmonarius。
1.4 SSR引物设计
从JGI数据库中搜索并下载Pleurotus ostreatus PC15 v2.0全基因组序列 (http://genome.jgi.doe.gov/ PleosPC15_2/PleosPC15_2.download.html)。利用SSR Locator软件对其全基因组序列进行单至六核苷酸重复SSR序列的查找[21],并随机选取4条位于不同scaffold上的SSR序列进行引物设计。共设计48条SSR引物用于PCR扩增。SSR引物由生工生物工程股份有限公司合成。
1.5 SSR引物筛选
选取4个平菇菌株的基因组DNA为模板进行SSR引物初筛。SSR-PCR反应体系:总体积20 μL,包括10×PCR buffer 2 μL,25 mmol·L-1MgCl22 μL,
10 mmol·L-1dNTP 0.4 μL,5 U·μL-1Taq DNA酶0.2 μL,Forward Primer(10 μmol·L-1) 1.5 μL,Reverse Primer(10 μmol·L-1)1.5 μL,Template DNA 2 μL,ddH2O 10.4 μL。PCR反应条件为:94℃预变性5 min;94℃变性30 s,55℃~57℃退火30 s,72℃延伸30 s,30个循环;72℃后延伸7 min。扩增产物用6%浓度的聚丙烯酰胺凝胶进行分离,银染技术检测。
1.6 SSR分析和分子身份证构建
根据4个菌株扩增结果筛选出扩增条带清晰、稳定、多态性丰富且区分度高的SSR引物。分别对17个供试菌株进行扩增试验。
根据供试菌株的扩增图谱,参考每个引物扩增目的片段的分子量大小,将强带或可分辨/重复性好的弱带记为有效的等位基因谱带,重复性不好的弱带不考虑,由小到大依次统计等位基因谱带,分析每对引物对17个菌株扩增产生的带型,并对不同带型进行赋值编码,标记为1~9。为了保证每位编码只有1位数字,第10种带型标记为0,将菌株带型转成零一矩阵,采用NTSYS-2.10e软件计算同一物种菌株间相似度。根据菌株间相似度分析结果,如果存在相似度为100%的菌株,则需设计新的SSR引物进行扩增,直至17个菌株的相似度小于100%,即将17个菌株全部区分开。根据带型编码构建17个平菇品种的SSR分子标记身份证。
2 结果与分析
2.1 CO1扩增结果及分析
CO1扩增结果见图1。
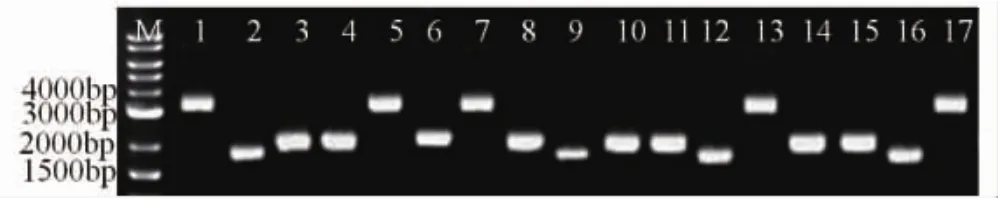
图1 供试菌株的CO1扩增结果Fig.1 PCR results of tested strains amplified by CO1 primers
17个菌株均能扩增得到单一条带,根据条带大小,供试菌株分为3组。第2号、第9号、第12号和第16号菌株为P.ostreatus,共4株;第3号、第4号、第6号、第8号、第10号、第11号、第14号和第15号菌株为P.cornucopiae,共8株;第1号、第5号、第7号、第13号、第17号菌株为P. pulmonarius,共5株。
2.2 SSR引物筛选及扩增多态性分析
根据4个菌株对48对SSR引物的筛选结果,经过多次扩增试验验证,最终选取9对扩增条带清晰、稳定、多态性丰富且区分度高的SSR分子标记,见表2。这九对SSR分子标记所对应的SSR位点分别分布在Pleurotus ostreatus PC15 v2.0全基因组12条scaffold的第3条~第8条上。9对SSR标记在17个平菇菌株中共检测到52个等位基因。不同SSR标记检测到等位基因数目范围为3个~8个,平均检测效率为5.8个/标记,扩增的等位基因谱带片段大小见表3。各位点的多态信息含量(PIC=1-∑Pi2,Pi为第i个等位基因的频率)范围为0.410~0.828,平均值为0.704,说明供试菌株具有较高的杂合度,可能是由于长期人工选择具有杂种优势品种的结果。其中引物P25的多态信息含量最高为0.828,说明该引物最能反映不同品种之间的差异。

表2 9对SSR分子标记序列及扩增多样性信息Tab.2 Sequence and genetic diversity of 9 SSR primers for oyster mushroom
进一步根据9对引物对17个菌株扩增的分子指纹图谱,将带型转成零一矩阵,计算同一物种内菌株间的相似度。4株P.ostreatus菌株间的遗传相似性系数为0.44~0.89,8株P.cornucopiae菌株间的遗传相似性系数为0.48~0.85,5株P.pulmonarius菌株间的遗传相似性系数为0.33~0.93,说明这九对SSR引物可以将17个平菇品种完全区分开。

表3 每对引物扩增产生的等位基因片段大小Tab.3 Size range of alleles amplified by SSR primer

图2 引物P4对17个供试菌株扩增产生的标准带型图及赋值Fig.2 Electrophoresis band pattern standards amplified by primer P4 for 17 tested strains
2.3 编码和分子身份证构建
9对引物在17个菌株中共产生53种带型,其中在P.ostreatus组中检测到18种、P.cornucopiae组27种、P.pulmonarius组27种,不同物种的菌株间可以产生相同的带型。9个引物分别扩增的带型数为4种~10种。引物P25扩增的带型数最多为10种。图2为引物P4对17个菌株扩增产生的全部带型和赋值情况,共有6种带型。每对引物产生的不同标准带型和赋值情况见表3、表4。
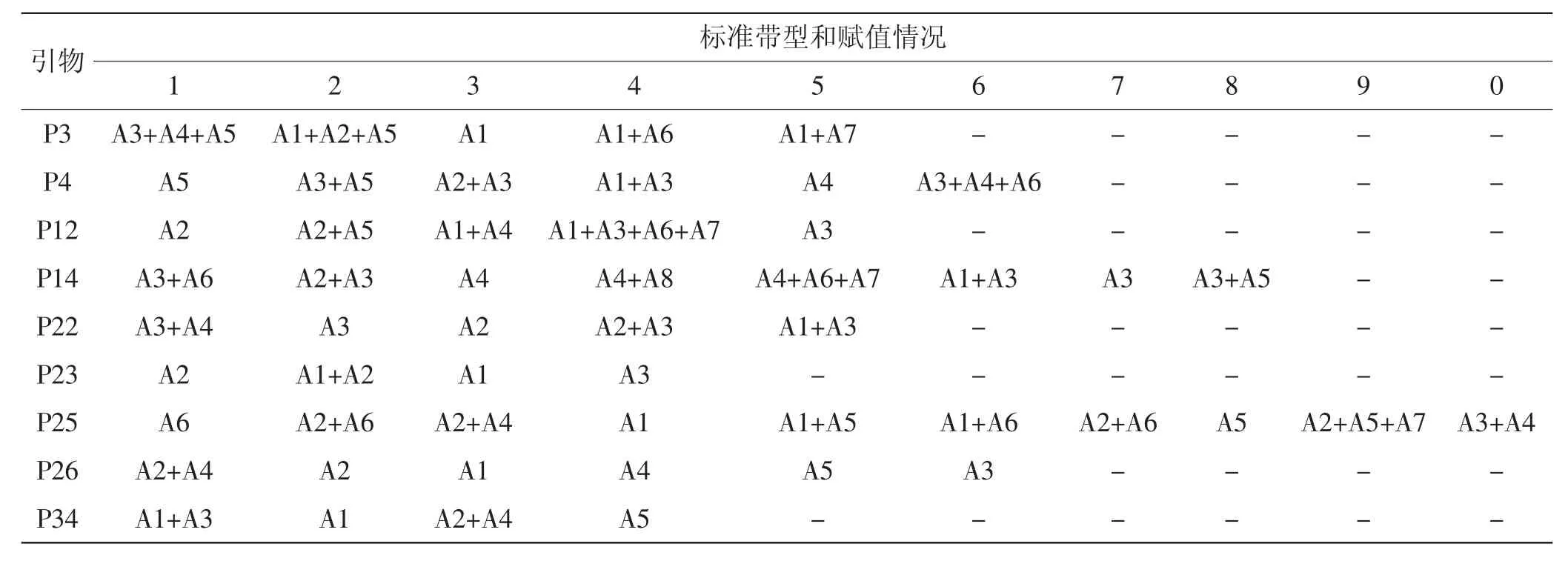
表4 每对SSR引物扩增产生的标准带型和赋值情况Tab.4 Band pattern and encoded standard amplified by each SSR primer
对17个供试菌株的分子身份证进行编码,每个菌株的分子身份证分别由9个字符串组成,从左到右依次代表引物P3、P4、P12、P14、P22、P23、P25、P26、P34产生的第几种带型,结果见表5。
从表5可以看出,17个平菇品种分别具有特异的分子身份证编码。
3 讨论
3.1 SSR分子身份证的编码方法
对于SSR分子身份证的构建方法,不同的研究者有不同的编码原则。于潇等[22]构建的野生黑木耳菌株的分子ID是由1、0字符串组合而成,代表某对引物某个基因位点有无扩增条带,1代表有扩增条带,0代表无扩增条带,该分子ID是依赖于资源特征分析软件(ID Analysis 1.0)分析构建而成,简单快捷,但具有一定的使用局限性。杜晶晶等[23]构建的葡萄种质分子身份证与陈昌文等[16]的方法类似,即分子身份证字符串的每位上的字符对应同一引物扩增的较小等位基因编码值,但对于杂合带型的菌株,如果较小谱带相同,那么就需要通过增加引物来区分种质,这样会增加所需引物数量和分子身份证位数。
本研究选取9对SSR引物对17个菌株扩增结果显示,大部分菌株都是杂合带型,即包括两个以上的等位基因,这可能是由于平菇栽培过程中容易产生孢子而发生自然杂交[24],再经不断的人工选择的结果。所以,本研究构建的平菇品种分子身份证是由不同带型的编码组合而成,每个标准带型包括所有的有效等位基因,这与王新新等[20]对双孢蘑菇分子身份证的构建方法类似。即分子身份证每一位数字代表一个引物产生的第几种带型,对于引物P25扩增的7个等基因片段共组成10种带型,为了保证每一位点只有1位数字,第10种带型赋值为0,以达到书写简洁的目的。本研究认为SSR分子身份证的编码是一种人为制定的标准,以简洁直观且不影响区分效率为目的即可。
3.2 SSR分子标记在平菇研究中的应用
SSR分子标记多用于平菇种质资源遗传多样性分析及菌株鉴定等研究[14,25]。对于SSR标记的开发主要有3种策略,包括基因文库筛选法,例如Barroso等[26]通过建立基因组文库,从双孢蘑菇(Agaricus bisporus)中分离出四核苷酸(TATG)4,并扩增出预期的产物;富集文库筛选法,例如Ma等[14]采用生物素-磁珠富集法首次开发出糙皮侧耳(P.ostreatus)微卫星序列引物,成功区分37个侧耳属栽培菌株;数据库筛选法,例如刘春滟等[27]利用香菇(Lentinus edodes) 的EST数据库设计了40对SSR引物,22对具有多态性,多态率为55%。随着糙皮侧耳菌株全基因组序列的测序成功公布[28-29],基于糙皮侧耳全基因组的SSR信息分析越来越深入[30],也为糙皮侧耳(P.ostreatus)SSR位点的获取提供了便捷有效的途径。本研究利用P.ostreatus PC15 v2.0全基因组序列成功设计9对SSR引物用于17个平菇菌株的区分,但为了更全面地反映种质间的基因信息,需要开发更多的SSR位点,覆盖更多的基因组序列。本研究选取的9对SSR分子标记,只分布在糙皮侧耳基因组12条scaffold中的其中6条,虽然达到了区分全部菌株的目的,但对于更多新菌株尤其是亲缘关系近的菌株,还需要不断开发新的SSR标记进行区分。
3.3 平菇种质分子身份证构建过程中的物种鉴定
在我国,不同地区称谓的平菇所指的生物学物种不完全相同,但是总体上说,俗称为平菇的侧耳属物种包括糙皮侧耳(P.ostreatus)、白黄侧耳(P. cornucopiae)、佛罗里达侧耳(P.florida) 和肺形侧耳(P.pulmonarius)4个种[31]。在对平菇种质进行菌种鉴别或分子身份证构建之前应首先对平菇物种进行准确鉴定。对于平菇近缘种的鉴定,至今没有一个准确的方法,本研究采用的CO1标记方法简便快速,虽然能够克服ITS测序方法中的数据库信息不准确、种内变异大于种间变异等不足,但是CO1引物是黄晨阳[12]仅对侧耳属9个种、20个菌株的CO1序列进行测序而设计的通用引物,由于试验材料有限,所以不能断定CO1基因种内保守,再者侧耳属CO1基因中频繁出现内含子,密码子的摆动效用可能使设计的通用引物失效或扩增出假基因而影响结果[32]。所以CO1鉴定平菇近缘种的方法适应性还有待于进一步验证。
[1]Falck R.Uber die Waldkultur des Austernpilzes(Agaricus ostreatus)[J].Laubholzstubben.Z.Forst-u.Jagdween,1917,49 (1917):159-165.
[2]Larraya LM,Perez G,Ritter E,et al.Genetic linkage map of the edible basidiomycete Pleurotus ostreatus[J].Applied& Environmental Microbiology,2000,66 (12):5290-5300.
[3]黄毅.食用菌栽培学[M].北京:高等教育出版社,2008.
[4]吕作舟.食用菌栽培学[M].北京:高等教育出版社,2006.
[5]卯晓岚.中国大型真菌[M].郑州:河南科学技术出版社,2000.
[6]郑素月,张金霞,王贺祥,等.我国栽培平菇近缘种的多相分类[J].中国食用菌,2003,22(3):3-6.
[7]申进文,赵旭,李燕,等.河南省栽培平菇品种的种质资源评价[J].河南农业大学学报,2011,45(3):297-301.
[8]郑和斌,马志刚,吕作舟,等.基于ITS序列分析对我国主要栽培的侧耳品种的鉴定及评价[J].菌物学报,2006,25(3):398-407.
[9]黄晨阳,陈强,张金霞,等.侧耳属主要种类ITS序列分析[J].菌物学报,2010,29(3):365-372.
[10]Nilsson RH,Ryberg M,Kristiansson E,et al.Taxonomic reliability of DNA sequences in public sequence databases:a fungal perspective[J].PLoS One,2006,1(1):59.
[11]Brock PM,Döring H,Bidartondo MI.How to know unknown fungi:the role of a herbarium[J].New Phytologist,2009,181 (3):719-724
[12]黄晨阳.ITS和CO1在侧耳属物种鉴定中应用[D].北京:中国农业大学,2011.
[13]郑素月,张庆桥.生化标记在河北省栽培平菇种质资源鉴别中的应用[J].北方园艺,2011(14):162-164.
[14]Ma KH,Lee GA,Lee SY,et al.Development and characterization of new microsatellite markers for the oyster mushroom (Pleurotus ostreatus)[J].Journal of Microbiology Biotechnology,2009,19(9):851-857.
[15]叶翔,黄晨阳,陈强,等.中国主栽香菇品种SSR指纹图谱的构建[J].植物遗传资源学报,2012,13(6):1067 -1072.
[16]陈昌文,曹珂,王力荣,等.中国桃主要品种资源及其野生近缘种的分子身份证构建[J].中国农业科学,2011,44(10):2081-2093.
[17]Morgante M,Olivivieri A.PCR-amplified microsatellites as markers in plant genetics[J].The Plant Journal,1993,3 (1): 175-182.
[18]王慧玲,闫爱玲,孙磊,等.13个中国葡萄优新品种的分子身份证构建[J].生物技术通报,2016,32(4):137 -142.
[19]张肖雅,许修宏,刘华晶.11个灵芝菌株的分子ID构建[J].微生物学通报,2013,40(2):249-255.
[20]王新新,李丹,宋冰,等.双孢蘑菇种质SSR分子身份证的构建[J].食用菌学报,2016,23(2):6-11.
[21]da Maia LC,Palmieri DA,de Souza VQ,et al.SSR locator: tool for simple sequence repeat discovery integrated with primer design and PCR simulation[J].International Journal of Plant Genomics,2008,2008:412696.
[22]于潇,许修宏,刘华晶.黑龙江部分野生黑木耳菌株的分子ID构建[J].中国农学通报,2012,28(13):171-175.
[23]杜晶晶,刘国银,魏军亚,等.基于SSR标记构建葡萄种质资源分子身份证[J].植物研究,2013,33(2):232 -237.
[24]李红,肖千明,刘娜,等.食用菌菌种退化原因分析及复壮方法的探讨[J].辽宁农业科学,2010(4):53-55.
[25]李慧,陈强,黄晨阳,等.基于SSR标记构建平菇栽培品种核心样本方法的探讨[J].园艺学报,2012,39(10):2023-2032.
[26]Barroso G,Sonnenberg AS,Van Griensven LJ,et al.Molecular cloning of a widely distributed microsatellite core sequence from the cultivated mushroom Agaricus bisporus[J]. Fungal Genetics and Biology,2000,31(2):115-123.
[27]刘春滟,李南羿,张玉琼.香菇EST-SSR标记的开发及应用[J].食用菌学报,2010,17(2):1-6.
[28]Grigoriev IV,Nordberg H,Shabalov I,et al.The genome portal of the department of energy joint genome institute[J]. Nucleic Acids Research,2012,40(Database issue):26-32.
[29]Riley R,Salamov AA,Brown DW,et al.Extensive sampling of basidiomycete genomes demonstrates inadequacy of the white-rot/brown-rot paradigm for wood decay fungi[J].Proceedings of the National Academy of Sciences,2014,111 (27):9923-9928.
[30]Qu J,Huang C,Zhang J.Genome-wide functional analysis of SSR for an edible mushroom Pleurotus ostreatus[J].Gene, 2014,575(2):524-530.
[31]张金霞,黄晨阳,郑素月.平菇新品种-秀珍菇的特征特性[J].中国食用菌,2005,24(4):25-26.
[32]Song H,Buhay JE,Whiting MF,et al.Many species in one: DNA barcoding overstimates the number of species when nuclear mitochondrial pseudogenes are coamplified[J].Proceedings of the National Academy of Sciences of the United States of America,2008,105(36):13486-13491.
Molecular ID Establishment of 17 Oyster Mushroom Cultivated in Hebei Province
LI Hui,WANG Chao-jiang,GAO Chun-yan
(Institute of Genetics and Physiology of Hebei Academy of Agriculture and Forestry Sciences,Shijiazhuang 050051,China)
The method of establishing the oyster mushroom germplasm molecular ID was studied by using 17 strains of oyster mushroom cultivated in Hebei province.Cytochrome oxidase subunit 1 gene (CO1)was used to identify the species of 17 oyster mushroom strains.Using SSR Locator software to screen SSRs(simple sequence repeats)in complete genome sequence of P.ostreatus PC15 v2.0 and to design primers for the selected SSR locus,suitable primers were selected to amplify genomic DNA.The assemblage of standard electrophoresis band patterns that amplified by each maker were coded and combined for the establishment of oyster mushroom germplasm molecular ID.The results showed that 17 oyster mushroom strains were identified as 4 strains of P.ostreatus,8 strains of P.cornucopiae,5 strains of P.pulmonarius.After screening of 48 primers,53 electrophoresis band patterns were detected using 9 selected SSR makers.Coding the standard electrophoresis band patterns,the distinctive molecule ID for 17 strains of oyster mushroom were established.The 17 strains were completely distinguished by 4 SSR primers.The results suggested CO1 combied with SSR maker techniques can be used for establishing molecular ID of oyster mushroom germplasm.
oyster mushroom;molecular ID;CO1;SSR
S646.9
A
1003-8310(2017)04-0035-07
10.13629/j.cnki.53-1054.2017.04.009
河北省农林科学院科技创新基本科研业务费项目(A2015110103);国家现代农业产业技术体系建设专项资金(CARS-24)。
李慧(1986-),女,硕士,助理研究员,主要从事食用菌栽培育种。E-mail:lihuiviphappy@163.com
**通信作者:高春燕(1964-),女,本科,副研究员,主要从事食用菌栽培研究。E-mail:ycsgcy@163.com
2017-05-15

