基于表型和SSR分子标记构建芝麻核心种质
刘艳阳,梅鸿献,杜振伟,武轲,郑永战,崔向华,郑磊
基于表型和SSR分子标记构建芝麻核心种质
刘艳阳1,梅鸿献1,杜振伟1,武轲1,郑永战1,崔向华2,郑磊3
(1河南省农业科学院芝麻研究中心,郑州450002;2河南省驻马店市农业科学院,河南驻马店 463000;3河南省漯河市农业科学院,河南漯河 462300)
【目的】便于管理、研究和利用芝麻种质资源,为芝麻育种提供优异基因资源。【方法】利用新收集和种质库保存的5 020份芝麻种质资源为基础,首先基于标准化的表型数据按地理来源分组后采用组内比例法聚类抽样构建初级核心种质,然后基于SSR分子标记应用位点优先取样策略逐步聚类,使用检验检测每次聚类形成的核心种质与初级核心种质的Nei’s基因多样度()和Shannon-Wiener指数(),直到核心种质的遗传多样性与初级核心种质开始有显著差异时,终止多次聚类取样,选择上一个与初级核心资源没有显著差异的核心种质作为最佳核心种质。利用Nei’s 多样性指数、Shannon-Wiener多样性指数、多态条带百分率(PB,%)、多态条带保留率(PBR,%)、变异系数符合率(VR)、极差符合率(CR)、方差差异百分率(VD,%)、均值差异百分率(MD,%)等参数进行核心种质代表性检验和评价。【结果】构建了含有816份资源的初级核心种质和含有501份资源的核心种质,分别占全部种质资源的16.25%和9.98%;核心种质包括国内资源442份,国外资源59份;Nei's基因多样度(0.2789)和Shannon-Wiener指数(0.4243)在<0.05概率条件下与初级核心资源(=0.2791,=0.4302)无显著性差异,多态条带百分率(PB,%)、多态条带保留率(PBR,%)、变异系数符合率(VR)、极差符合率(CR)分别为91.25%、95.23%、99.14%、86.85%。方差差异百分率(VD,%)和均值差异百分率(MD,%)均为0。测验结果表明,核心种质的遗传多样性指数与原始种质差异不显著。位点优先取样策略构建的核心种质比对照随机取样策略丢失的多态性位点数少,且同一遗传距离下位点优先取样策略构建的核心种质具有更高的遗传多样性,更能构建一个具有代表性的核心种质,Shannon-Wiener多样性指数比Nei’s多样性指数检测效率高。【结论】基于地理来源分组,组内按表型数据聚类按比例法抽样构建芝麻初级核心种质,再结合SSR分子标记数据,采用SM相似系数进行UPGMA逐步聚类是构建芝麻核心种质较适宜的方法,所构建的核心种质较好地代表了基础种质的遗传多样性。
芝麻;种质资源;核心种质;代表性检验
0 引言
【研究意义】芝麻种质资源是芝麻新品种培育及研究重要的物质基础。在国家芝麻产业技术体系支持下,芝麻种质资源评价岗位团队在埃塞俄比亚、印度和中国辽宁、吉林、江苏、安徽、江西、广西、湖南、贵州等省区110个县(市)考察,共收集各类芝麻资源2 000余份,河南省农业科学院芝麻研究中心已保存有3 000余份,目前,资源总数已达5 210余份,这是对中国自“六五”以来持续开展的全国性芝麻种质资源考察收集的一个重要补充和丰富。这些宝贵的资源虽然为芝麻遗传改良研究提供了大量的材料,但庞大的资源数量又给保存、评价、鉴定和利用带来了困难。核心种质是以最小的资源数量和遗传重复最大程度地代表整个遗传资源的多样性,它的建立既保证了遗传多样性,又减少了资源数量[1]。因此,对保存的芝麻资源建立核心种质是十分迫切和必要的。【前人研究进展】自Frankel等[2]首次提出核心种质(core collection)的概念以来,先后建立了水稻、大豆、小麦等多种农作物的核心种质[3-5]。国内外学者对芝麻核心种质构建方面也做了大量研究工作。Bisht等[6]对印度3 129份芝麻种质资源进行研究,构建了包括343份的印度芝麻核心种质。Zhang等[7]采用分层取样策略,依据来源、品种类别、生态类型3层将中国国家种质资源库保存的“九五”以前收集的4 251份芝麻种质分成14组,按20%的固定比例选取初选核心种质884份,采用离差平方和法分组进行系统聚类,依据核心种质数量为初选核心种质50%的原则,建立了包含453份种质的中国芝麻核心种质。明确了中国保存芝麻种质资源的遗传多样性组成和分布,确定了遗传多样性类型和特点。发现了一批重要性状优良种质,通过多点鉴定明确了其利用价值并提供利用。开展了芝麻株高、含油量、芝麻素、芝麻酚林和茎点枯病抗性等重要性状的关联分析研究[8-14]。随后,Zhang等[15]基于表型性状和分子标记评价了中国芝麻核心种质遗传多样性并构建微核心种质184份。Kang等[16]对韩国RDA(Rural Development Administration)基因库保存的2 246份芝麻种质利用离差平方和法进行聚类分析,从10个农业生态区各自随机选取21%的种质构成韩国芝麻核心种质共475份(占21%)。Mahajan等[17]对以色列保存的2 168份芝麻种质进行了研究,构建了172份(占20%)的芝麻核心种质。Park等[18-19]利用SSR分子标记对韩国RDA基因库中来自4个洲15个国家的2 751份种质随机挑选出277份核心种质进行分子遗传多样性和种群结构评价,聚类结果表明地理位置与种质资源间没有明确的关系。随后,对2 751份芝麻种质资源的5个质量性状和10个数量性状进行评估,选出278份(占总数的10.1%)作为核心资源。【本研究切入点】国内外关于芝麻核心种质构建主要基于表型或分子数据,而采用表型与分子数据相结合的方法构建核心种质的研究还鲜见报道。【拟解决的关键问题】本研究首先基于表型数据按地理来源分组聚类构建初级核心种质,再利用核心SSR分子标记逐步聚类筛选构建核心种质,并验证核心种质的代表性,为芝麻种质资源的研究、保存和提高有效利用性奠定基础。
1 材料与方法
1.1 试验材料
河南省农业科学院芝麻研究中心种质库保存芝麻种质资源5 210份,其中4 700份来源于中国23个省(市、自治区)、493份为国外引种、17份来源不明。本研究是基于均有18个表型性状信息数据的5 020份资源开展(电子版附表1)。
1.2 表型性状鉴定
2013—2015年在河南平舆、驻马店和漯河试验基地分别进行田间种植观察和鉴定。试验采取统一编号,每份材料种植2行,行长5 m,株距0.2 m,行距0.4 m。按照《芝麻种质资源描述规范和数据标准》[20]调查出苗期、成熟期,株型、叶型、叶色、茎秆茸毛量、叶腋花数、花色、蒴果棱数、茎秆成熟色、裂蒴性。每小区随机取样5株,测量株高、始蒴高度、黄稍尖长、单株蒴数、蒴果长度、蒴粒数、粒色和千粒重。
1.3 SSR标记分析
1.3.1 基因组DNA的提取 幼苗期,采幼嫩叶片(10株的混合样),利用CTAB法提取芝麻叶片基因组DNA,用1%的琼脂糖凝胶电泳检测。DNA样品置于-20℃备用。
1.3.2 PCR反应体系及扩增条件 PCR反应体系为10 μL,包括1.0 μL基因组DNA(10 ng·μL-1)、1.0 μL Mg2+(10 × Buffer)、0.2 μL Taq DNA聚合酶(5 U·L-1)、0.2 μL dNTPs(10 mmol·L-1)、上下游引物各0.8 μL和ddH2O 6.0 μL。PCR反应程序为95℃ 3 min;94℃ 30 s,55℃—62℃ 30 s(视不同引物而定),72℃ 1 min,30个循环;72℃ 6 min,4℃保存。
1.3.3 SSR引物筛选及产物检测 从课题组通过全基因组重测序开发及网上发表的2 000对SSR标记中筛选出30对核心SSR引物(电子版附表2)[21-24],用于初级核心种质的基因型分析。SSR标记由生工生物工程(上海)股份有限公司合成。扩增产物经8%非变性聚丙烯酰胺凝胶分离,银染检测。
1.4 芝麻核心种质构建
1.4.1 初级核心种质构建 聚类分析的表型性状共18个。对生育期、株高、腿长、黄稍尖长、单株蒴数、蒴果长度、蒴粒数和千粒重8个数值性状进行10级分类,1级≤X-2δ,10级>X+2δ,中间每级间差0.5δ,X为性状平均值,δ为标准差;对株型、叶腋花数、蒴果棱数、叶型、叶色、花冠颜色、茎秆茸毛量、裂蒴性、茎杆成熟色和籽粒颜色10个描述性状按照电子附表3赋值。参照李自超等[25]的方法,将供试材料按照地理来源分组,组内采用标准化的表型数据按比例聚类抽样的方法,根据聚类图从最低分类的每组二个遗传材料中随机取一个材料进入下一轮聚类,通过抽样材料和原始材料的表型遗传多样性比较筛选确定芝麻初级核心种质。
1.4.2 核心种质构建 利用SSR标记对初级核心种质进行基因型分析,采用人工读带的方式,在相同迁移位置上,有带记为1,无带记为0,建立0,1矩阵。采用SM相似性系数估测遗传距离,根据遗传距离使用UPGMA聚类法,利用NTSYSpc-2.10e软件进行聚类分析。采用逐步UPGMA聚类取样法构建核心种质,在最低分类水平的2个材料中采用位点优先取样策略,优先选择具有最多稀有等位基因数的株系进入下一轮聚类。使用检验检测每次聚类形成的核心种质与初级核心种质的Nei’s基因多样度()和Shannon-Wiener指数(),直到核心种质的遗传多样性与初级核心种质开始有显著差异时,终止多次聚类取样,选择上一个与初级核心资源没有显著差异的核心种质作为最佳核心种质[26]。
1.5 核心种质的评价
借鉴前人研究经验[27-33],基于表型性状和SSR分子标记数据,选择表型保留比例(ratio of phenotype retained,RPR)、多态条带百分率(percentage of polymorphic loci,PB,%)、多态条带保留率(reserved rate of number of polymorphic loci,PBR,%)、变异系数符合率(variable rate of coefficient of variation,VR)、极差符合率(coincidence rate of range,CR)、方差差异百分率(variance difference percentage,VD,%)和均值差异百分率(phenotypic indexes of mean difference percentage,MD,%)等参数作为多样性评价指标。参考Li等[34]、毛钧等[35]的计算公式进行计算。
2 结果
2.1 种质资源表型性状统计分析
对供试种质资源10个描述性状进行统计分析,结果表明,芝麻种质资源中单杆类型和分枝类型分别占供试材料的52.33%和47.67%;叶腋花数以三花为主,占全部材料的64.84%,单花占35.16%;89.36%的芝麻资源蒴果棱数为4棱,6棱、8棱和混生的资源仅占10.64%;叶型以披针形为主,占全部材料的50.25%,柳叶形、椭圆形、卵形和心形叶分别占14.67%、16.09%、12.23%和6.76;叶片颜色以绿色为主,占全部材料的74.13%,浅绿和深绿分别占8.28%和17.59%;籽粒颜色以白色为主,占全部材料的54.21%;其次,黄色、黑色、浅褐和褐色分别占13.87%、12.73%、8.63%和5.45%,灰色、乳白、砖红和橄榄绿分别占1.94%、1.68%、1.39%和0.10%;花冠颜色以粉色为主,占全部材料的55.81%,白色、浅紫和紫色花分别占17.36%、15.26%和11.52%;茎秆茸毛量以少茸毛和中等茸毛为主,分别占全部材料的36.70%和30.39%,无茸毛和多茸毛分别占19.47%和13.44%;裂蒴性以轻裂和裂蒴为主,分别占全部材料的53.15%和46.76%,不裂蒴的仅占0.09%;成熟茎秆色以青绿为主,占全部材料的81.78%,绿黄、黄和紫色的资源分别占4.60%、8.80%和1.82%。
通过对芝麻种质资源数值性状进行统计分析(表1),从表中可以看出,芝麻种质资源数值性状的变异也很丰富,变异系数的大小顺序为黄稍尖长>单株蒴数>始蒴高度>蒴粒数>千粒重>株高>蒴果长>生育期。变异范围最大的是单株蒴数,为9—236个,千粒重的变异范围最小,为1.82—4.74 g;Shannon- Wiener指数最大的是始蒴高度,为2.08,蒴果长的Shannon-Wiener指数最小,为1.92。

表1 芝麻种质资源数值性状统计分析
2.2 芝麻核心种质构建
根据徐海明等[36]大群体采用较低的取样比例(10%—20%)的原则,在初级核心种质取样量达到原始种质16%时,取样量为803份,表型保留比例达到97.37%。补充数值性状极值和稀有描述性状变异类型的13个材料纳入初级核心种质,最终构建的芝麻初级核心种质含816份,占种质资源总数的16.25%,表型保留比例提高到100%。
利用河南省农业科学院芝麻研究中心种质资源研究室筛选的30对核心SSR引物对816份初级核心种质进行分析,使用NTSYS软件对初级核心种质材料进行UPGMA逐步聚类分析,随着剔除样品份数的增多,核心种质的遗传多样性呈下降趋势。当样品个数降为500时,SSR标记基因型频率的Nei’s基因多样度变化与初级核心种质未达到显著差异,而Shannon- Wiener多样性指数在5%概率水平开始出现显著差异,可见,Nei’s基因多样度对样品数变化没有Shannon- Wiener指数敏感。最终确定核心种质的最佳取样量为501份样品(表2),基因多样度为0.2791,Shannon- Wiener指数为0.4302,占全部资源的9.98%。
2.3 芝麻核心种质的代表性检验
根据SSR标记Shannon-Wiener指数出现显著差异前后的取样量501份和500份进行随机取样构建编号为R-501和R-500的2个随机核心种质,芝麻聚类核心种质(ZM-501和ZM-500)和2个随机核心种质(R-501和R-500)分别与初级核心种质进行检验(表3)。
从表中数据可以看出,在相同取样量下,随机核心种质的多样性低于聚类核心种质,且与初级核心种质的遗传多样性差异显著,表明聚类取样优于随机取样;同时在取样量为501个样品时,随机取样的SSR标记Shannon-Wiener指数与初级核心种质差异显著,而聚类取样无显著差异。因此,可认为聚类取样明显优于随机取样。
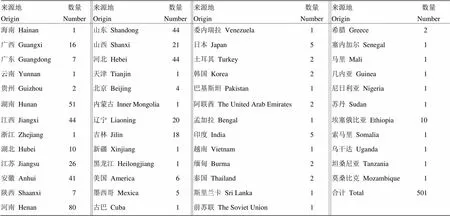
表2 芝麻核心种质的来源及数量
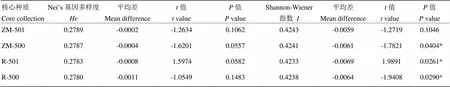
表3 芝麻核心种质SSR遗传多样性t检验
初级核心种质SSR标记的=0.2791,=0.4302。*表示在0.05水平差异显著
For primary core collection by SSR,=0.2791,=0.4302. *mean significance at the 0.05 level
为进一步确认基于SSR标记构建的芝麻核心种质的代表性,对SSR分子标记数据的多态条带数以及生育期、株高、始蒴高度、黄稍尖长、单株蒴数、蒴粒数、蒴果长、千粒重等8个数值性状的变异系数、极差、方差、均值进行了统计(表4和表5),并计算各核心种质的多态条带百分率(PB,%)、多态条带保留率(PBR,%)、变异系数符合率(VR)、极差符合率(CR)、方差差异百分率(VD,%)和均值差异百分率(MD,%)。

表4 芝麻核心种质代表性的分子数据比较
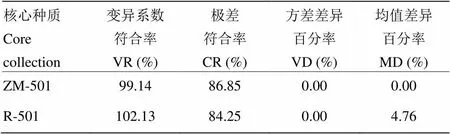
表5 芝麻核心种质代表性的表型数据比较
综上所述,获得的芝麻核心种质ZM-501由501份材料组成,其中国内资源442份,国外资源59份,Nei’s基因多样度(0.2789)和Shannon-Wiener指数(0.4243)在<0.05概率条件下与初级核心资源(分别为=0.2791,=0.4302)无显著性差异,而且与全部种质资源均值差异百分率为0,极差符合率为86.85%,构建的核心种质能够代表原种质遗传多样性,最终确定基于SSR标记数据的SM相似系数逐步聚类方法适用于芝麻核心种质的构建。
3 讨论
3.1 芝麻核心种质构建
本研究借鉴前人的研究经验,基于表型性状和SSR分子标记数据,对5 020份芝麻种质构建了芝麻初级核心种质816份(占全部资源的16.25%)和核心种质501份(占全部资源的9.98%),并进行了代表性验证。结果表明,按照资源的地理来源分组,组内采用表型按比例聚类取样的方法,构建芝麻初级核心种质;利用SSR标记对初级核心种质采用位点优先逐步UPGMA聚类取样法构建核心种质,既兼顾表型数据在种质评价中的作用,又准确反映初级核心种质间的差异,适合芝麻核心种质构建。该方法已用于葡萄[1]、甘蔗[26]、水稻[27]、大豆[37]和小麦[38]的核心种质构建。
核心种质构建中取样比例至关重要。Brown[28]根据中性选择理论推导认为,占整个原始种质资源5%—10%的核心种质样品能够代表整个资源70%以上的遗传变异。李自超等[27]认为取样比例应根据具体物种遗传结构及数量规模状况而定,总资源份数多的物种其核心种质所取比例可小一些,总资源份数较少的物种核心种质所取比例可相对大一些。国内外不同作物构建的核心种质,占原始种质的比例一般为5%—40%,大多数在5%—15%[4,39-40]。本研究根据使用检验检测每次聚类形成的核心种质与初级核心种质的Nei’s基因多样度()和Shannon-Wiener指数(),直到核心种质的遗传多样性与初级核心种质开始有显著差异时,终止多次聚类取样,选择上一个与初级核心资源没有显著差异的核心种质作为最佳核心种质,在核心种质遗传多样性的保留程度和代表性上优于固定比例取样法。
3.2 芝麻核心种质构建中分子标记和遗传参数的选择
本研究利用自主开发和网上公布的覆盖全基因组的2 000余对SSR标记筛选出的30对核心SSR标记构建的核心种质,较早期Zhang等[15]利用表型和从36对SRAP标记和10对SSR标记中筛选出的11对SRAP标记和3对SSR标记构建芝麻微核心种质在代表性和准确性方面有所提高。对核心种质的评价选择Shannon-Wiener指数、Nei’s基因多样度、多态条带百分率、多态条带保留率、变异系数符合率、极差符合率、方差差异百分率和均值差异百分率等参数作为多样性评价指标,发现SSR标记基因型频率的Nei’s基因多样度对样品数变化没有Shannon-Wiener指数敏感,这与毛钧等[35]的研究结果一致。
3.3 芝麻核心种质的管理和利用
核心种质的建立为加强和实现种质资源的有效管理及开发利用提供了一个十分便利的条件和途径。核心种质建立后,要建立完善的繁种、供种及管理体制,以保证核心种质的有效利用。加强对所构建核心种质的后续评价研究,挖掘资源特异基因;同时加强资源应用研究,建立各类专项核心种质,满足生产、育种对不同资源类型的需要。同时,聚类分析发现,芝麻种质资源并未按其地理来源聚在一起,而国外资源遗传多样性丰富。因此广泛引种,深入研究,对核心种质实时进行动态调整,不断补充新搜集的芝麻种质资源,不断优化核心种质结构、使其遗传变异最大化将更有利于其开发利用。目前,课题组正在利用核心种质开展优异种质、基因的筛选与克隆、重要农艺性状的关联分析及重要性状遗传规律等研究。核心种质的构建将有助于加速芝麻分子生物学相关研究进程。
4 结论
基于地理来源分组,组内按表型数据聚类按比例法抽样构建芝麻初级核心种质,结合SSR分子标记数据,采用SM相似系数进行UPGMA逐步聚类是构建芝麻核心种质较适宜的方法。核心种质包含501份芝麻资源,保留了原种质9.98%的样品。Nei’s基因多样度(0.2789)和Shannon-Wiener指数(0.4243)在<0.05概率条件下与初级核心资源(=0.2791,=0.4302)无显著性差异,多态条带百分率(PB,%)、多态条带保留率(PBR,%)、变异系数符合率(VR)、极差符合率(CR)分别为91.25%、95.23%、99.14%、86.85%。方差差异百分率(VD,%)和均值差异百分率(MD,%)均为0。核心种质的遗传多样性指数与原始种质差异不显著,建立的核心种质具有较好的代表性。
References
[1] 郭大龙, 刘崇怀, 张君玉, 张国海. 葡萄核心种质的构建. 中国农业科学, 2012, 45(6): 1135-1143.
Guo D L, Liu C H, Zhang J Y, Zhang G H. Construction of grape core collections., 2012, 45(6): 1135-1143. (in Chinese)
[2] Frankel O H.. Cambridge: Cambridge University Press, 1984: 161-170.
[3] Zhang H, Zhang D, Wang M, Sun J, Qi Y, Li J, Wei X, Han L, Qiu Z,Tang S, Li Z. A core collection and mini core collection ofL. in China., 2010, 122(1): 49-61.
[4] 邱丽娟, 曹永生, 常汝镇, 周新安, 王国勋, 孙建英, 谢华, 张博, 李向华, 许占有, 刘立宏. 中国大豆核心种质构建及其取样方法研究. 中国农业科学, 2003, 36(12): 1442-1449.Qiu L J, Cao Y S, Chang R Z, Zhou X A, Wang G X, Sun J Y, Xie H, Zhang B, Li X H, Xu Z Y, Liu L H. Establishment of Chinese soybean (L.) core collection and sampling strategy., 2003, 36(12): 1442-1449. (in Chinese)
[5] Balfourier F, Roussel V, Strelchenko P, Exbrayat- Vinson F, Sourdille P, Boutet G, Koenig J, Ravel C, Mitrofanova O, Beckert M, Charmet G. A worldwide bread wheat core collection arrayed in a 384-well plate., 2007, 114(7): 1265-1275.
[6] Bisht I S, Mahajan R K, Loknathan T R, Agrawal R C.Diversity in Indian sesame collection and stratification of germplasm accessions in different diversity groups., 1998, 45: 325-335.
[7] Zhang X R, Zhao Y Z, Cheng Y, Feng X Y, Guo Q Y, Zhou M D, Hodgkin T. Establishment of sesame germplasm core collection in China., 2000(47): 273-279.
[8] Wang L H, Zhang Y X, Li P W, Wang X F, Zhang W, Wei W L, Zhang X R. HPLC analysis of seed sesamin and sesamolin variation in a sesame germplasm collection in China.Journal of the American Chemists' Society, 2012, 89(6): 1011-1020.
[9] 车卓, 张艳欣, 孙建, 张秀荣, 尚勋武, 王化俊. 应用SRAP标记分析黑芝麻核心种质遗传多样性. 作物学报, 2009, 35(10): 1936-1941.
Che Z, Zhang Y X, Sun J, Zhang X R, Shang X W, Wang H J. Genetic diversity analysis of black sesame (DC) core collection of china using SRAP markers., 2009, 35(10): 1936-1941. (in Chinese)
[10] 车卓, 张艳欣, 孙建, 张秀荣, 尚勋武, 王化俊. 芝麻核心收集品中育成品种(系)的遗传多样性分析. 植物遗传资源学报, 2009, 10(3): 373-377.
Che Z, Zhang Y X, Sun J, Zhang X R, Shang X W, Wang H J. Analysis of genetic diversity for cultivars released of sesame core collection., 2009, 10(3): 373-377. (in Chinese)
[11] 危文亮, 张艳欣, 吕海霞, 王林海, 黎冬华, 张秀荣. 芝麻资源群体结构及含油量关联分析. 中国农业科学, 2012, 45(10): 1895-1903.
Wei W L, Zhang Y X, Lü H X, Wang L H, Li D H, Zhang X R. Population structure and association analysis of oil content in a diverse set of Chinese sesame (L.) germplasm., 2012, 45(10): 1895-1903. (in Chinese)
[12] 丁霞, 王林海, 张艳欣, 黎冬华, 高媛, 危文亮, 王蕾, 张秀荣. 芝麻核心种质株高构成相关性状的遗传变异及关联定位. 中国油料作物学报, 2013, 35(3): 262-270.
Ding X, Wang L H, Zhang Y X, Li D H, Gao Y, Wei W L, Wang Lei, Zhang X R. Genetic variation and associated mapping for traits related to plant height constitutions in core collections of sesame (L.)., 2013, 35(3): 262-270. (in Chinese)
[13] 王蕾, 黎冬华, 齐小琼, 张艳欣, 丁霞, 王林海, 危文亮, 高媛, 张秀荣. 芝麻核心种质芝麻素和芝麻酚林的关联分析. 中国油料作物学报, 2014, 36(1): 32-37.
Wang L, Li D H, Qi X Q, Zhang Y X, Ding X, Wang L H, Wei W L, Gao Y, Zhang X R. Association analysis of sesamin and sesamolin in the core sesame (L.) germplasm., 2014, 36(1): 32-37. (in Chinese)
[14] 张艳欣, 王林海, 黎冬华, 危文亮, 高媛, 张秀荣. 芝麻茎点枯病抗性关联分析及抗病载体材料挖掘. 中国农业科学, 2012, 45(13): 2580-2591.
Zhang Y X, Wang L H, Li D H, Wei W L, Gao Y, Zhang X R. Association mapping of sesame (L.) resistance to macrophomina phaseolina and identification of resistant accessions., 2012, 45(13): 2580-2591. (in Chinese)
[15] Zhang Y X, Zhang X R, Che Z, Wang L H, Wei W L, Li D H. Genetic diversity assessment of sesame core collection in China by phenotype and molecular markers and extraction of a mini-core collection.,2012, 13: 102.
[16] Kang C W, Kim S Y, Lee S W, Mathur P N, Hodgkin T, Zhou M D, Lee J R. Selection of a core collection of Korean sesame germplasm by a stepwise clustering method., 2006, 56: 85-91.
[17] Mahajan R K, Bisht I S, Dhillon B S. Establishment of a core collection of world sesame (L.) germplasm accessions., 2007, 39(1): 53-64.
[18] Park J H, Suresh S, Cho G T, Choi N G, Baek H J, Lee C W, Chung J W. Assessment of molecular genetic diversity and population structure of sesame (L.) core collection accessions using simple sequence repeat markers., 2013, 12(1): 112-119.
[19] Park J H, Suresh S, Raveendar S, Baek H J, Kim C K, Lee S, Cho G T, Ma K H, Lee C W, Chung J W. Development and evaluation of core collection using qualitative and quantitative trait descriptor in sesame (L.) germplasm., 2015, 60(1): 75-84.
[20] 张秀荣, 冯祥运. 芝麻种质资源描述规范和数据标准. 北京: 中国农业出版社, 2006: 22-37.
Zhang X R, Feng X Y.(L.). Beijing: China Agriculture Press, 2006: 22-37. (in Chinese)
[21] Zhang H Y, Miao H M, Wei L B, Li C, Zhao R H, Wang C Y. Genetic analysis and QTL mapping of seed coat color in sesame (L.)., 2013, 8(5): e63898.
[22] Dixit A, Jin M H, Chung J W, Yu J W, Chung H K, Ma K H, Park Y J, Cho E G. Development of polymorphic microsatellite markers in sesame (L.)., 2005, 5: 736-738.
[23] Wang L H, Zhang Y X, Qi X Q, Gao Y, Zhang X R. Development and characterization of 59 polymorphic cDNA-SSR markers for the edible oil crop(Pedaliaceae)., 2012, 99(10): e394-8.
[24] Pham T D. Analyses of genetic diversity and desirable traits in sesame (L., Pedaliaceae): Implication for breeding and conservation[D]. Swedish University of Agricultural Sciences, 2011.
[25] 李自超, 张洪亮, 曹永生, 裘宗恩, 魏兴华, 汤圣祥, 余萍, 王象坤. 中国地方稻种资源初级核心种质取样策略研究. 作物学报, 2003, 29(1): 20-24.
Li Z C, Zhang H L, Cao Y S, Qiu Z E, Wei X H, Tang S X, Yu P, Wang X K. Studies on the sampling strategy for primary core collection of Chinese ingenious rice., 2003, 29(1): 20-24. (in Chinese)
[26] 刘新龙, 刘洪博, 马丽, 李旭娟, 徐超华, 苏火生, 应雄美, 蔡青, 范源洪.利用分子标记数据逐步聚类取样构建甘蔗杂交品种核心种质库. 作物学报, 2014, 40(11): 1885-1894.
Liu X L, Liu H B, Ma L, Li X J, Xu C H, Su H S, Ying X M, Cai Q, Fan Y H. Construction of sugarcane hybrids core collection by using stepwise clustering sampling approach with molecular marker data., 2014, 40(11): 1885-1894. (in Chinese)
[27] 李自超, 张洪亮, 曾亚文, 杨忠义, 申时全, 孙传清, 王象坤. 云南地方稻种资源核心种质取样方案研究. 中国农业科学, 2000, 33(5): 1-7.
Li Z C, Zhang H L, Zeng Y W, Yang Z Y, Shen S Q, Sun C Q, Wang X K. Study on sampling schemes of core collection of local varieties of rice in Yunnan, China., 2000, 33(5): 1-7. (in Chinese)
[28] Brown A H D. Core collection: a practical approach to genetic resources management., 1989, 31: 818-824.
[29] Ersking W, Muehlbauer F J. Allozyme and morphological variability, out crossing rate and core collection formation in lentil germplasm., 1991, 83: 119-125.
[30] Jansen J, Hintum T. Genetic distance sampling: a novel sampling method for obtaining core collections using genetic distances with an application to cultivated lettuce., 2007, 114: 421-428.
[31] 胡兴雨, 王纶, 张宗文, 陆平, 张红生. 中国黍稷核心种质的构建. 中国农业科学, 2008, 41(11): 3489-3502.
Hu X Y, Wang L, Zhang Z W, Lu P, Zhang H S. Establishment of broomcorn millet core collection in China., 2008, 41(11): 3489-3502. (in Chinese)
[32] 高志红, 章镇, 韩振海, 房经贵. 中国果梅核心种质的构建与检测. 中国农业科学, 2005, 38(2): 363 -368.
Gao Z H, Zhang Z, Han Z H, Fang J G. Development and evaluation of core collection of Japanese apricot germplasms in China., 2005, 38(2): 363-368. (in Chinese)
[33] 齐永文, 樊丽娜, 罗青文, 王勤南, 陈勇生, 黄忠兴, 刘睿, 刘少谋, 邓海华, 李奇伟. 甘蔗细茎野生种核心种质构建. 作物学报, 2013, 39(4): 649-656.
Qi Y W, Fan L N, Luo Q W, Wang Q N, Chen Y S, Huang Z X, Liu R, Liu S M, Deng H H, Li Q W. Establishment ofL. core collections., 2013, 39(4): 649-656. (in Chinese)
[34] Li Z C, Zhang H L, Zeng Y W, Yang Z Y, Shen S Q, Sun C Q, Wang X K. Studies on sampling schemes for the establishment of core collection of rice landraces in Yunnan, China., 2002, 49: 67-74.
[35] 毛钧, 刘新龙, 苏火生, 陆鑫, 林秀琴, 蔡青, 范源洪. 基于表型与分子数据的斑茅核心种质构建. 植物遗传资源学报, 2016, 17(4): 607-615.
Mao J, Liu X L, Su H S, Lu X, Lin X Q, Cai Q, Fan Y H. Constructing core collection ofbased on phenotype and molecular markers., 2016, 17(4): 607-615. (in Chinese)
[36] 徐海明, 邱英雄, 胡晋, 王建成. 不同遗传距离聚类和抽样方法构建作物核心种质的比较. 作物学报, 2004, 30(9): 932-936.
Xu H M, Qiu Y X, Hu J, Wang J C. Methods of constructing core collection of crop germplasm by comparing different genetic distances, cluster methods and sampling strategies., 2004, 30(9): 932-936. (in Chinese)
[37] 赵丽梅, 董英山, 刘宝, 郝水, 王克晶, 李向华. 中国一年生野生大豆核心资源的构建. 科学通报, 2005, 50(10): 989-996.
Zhao L M, Dong Y S, Liu B, Hao S, Wang K J, Li X H. Construction of core resources of annual wild soybean () in China., 2005, 50(10): 989-996. (in Chinese)
[38] 董玉琛, 曹永生, 张学勇, 刘三才, 王兰芬, 游光霞, 庞斌双, 李立会, 贾继增. 中国普通小麦初选核心种质的产生. 植物遗传资源学报, 2003, 4(1): 1-8.
Dong Y S, Cao Y S, Zhang X Y, Liu S C, Wang L F, You G X, Pang B S, Li L H, Jia J Z. Establishment of candidate core collections in chinese common wheat germplasm., 2003, 4(1): 1-8. (in Chinese)
[39] Ortiz R, Ruiz-Tapia E N, Mujica-Sanchez A. Sampling strategy for a core collection ofgermplasm., 1998, 96: 475-483.
[40] Yonezawa K, Nomura T, Morishima H. Sampling strategies for use in stratified germplasm collections//Hodgkin T, Brown A H D, van Hintum T J L, Morales E A V.. USA: John Wiley & Son, 1995: 35-53.
(责任编辑 李莉)
附表1 芝麻种质资源的来源及数量
Schedule 1 The origin and number of sesame germplasm resources

来源地Origin数量Number来源地Origin数量Number来源地Origin数量Number来源地Origin数量Number 海南Hainan6山东Shandong437委内瑞拉Venezuela1希腊Greece11 广西Guangxi175山西Shanxi216日本Japan46塞内加尔Senegal1 广东Guangdong71河北Hebei445土耳其Turkey2马里Mali1 云南Yunnan8天津Tianjin7韩国Korea2几内亚Guinea8 贵州Guizhou22北京Beijing39巴基斯坦Pakistan1尼日利亚Nigeria2 湖南Hunan551内蒙古Inner Mongolia2阿联酋The United Arab Emirates20苏丹Sudan1 江西Jiangxi452辽宁Liaoning192孟加拉Bengal10埃塞俄比亚Ethiopia94 浙江Zhejiang4吉林Jilin182印度India47索马里Somalia1 湖北Hubei95新疆Xinjiang7越南Vietnam3乌干达Uganda1 江苏Jiangsu263黑龙江Heilongjiang4缅甸Burma21坦桑尼亚Tanzania3 安徽Anhui406美国America44泰国Thailand18莫桑比克Mozambique9 陕西Shaanxi65墨西哥Mexico35斯里兰卡Sri Lanka4来源不祥Unknown17 河南Henan963古巴Cuba1前苏联The Soviet Union4合计Total5020
附表2 SSR引物序列
Schedule 2 The primer sequences of SSR markers

引物Primer正向引物序列Forward primer sequence (5’-3’)反向引物序列Reverse primer sequence (5’-3’) HSRC311CACCATGAAATGTTCAGCAAATACCAGATTCAACAGTTTTGGAGTGG HSRC595GAGACCCTTTATCCATTTCTTGAGATCTTGTTGCCCCACCACTAC HSRC639GTGATAACATATCCGATTTTGTTCCCTTCCTTTCATCACTGTCCCG HSRC778GGTCAATTTGCTCCTCCTCTGACATCATCATTCCTCCCTC HSRC813ACATCATTCCCTTTGGCTCTCTGTTTCTCCATCTGCTTCC HSRC950CCTTTCTTCTTCTAAATCTGCC CACCCTAGTTGTCCATGTTCCT HSRC958CTTCCCTAGCAAACCTCTGTAGTCTTTCCACCCATCCATA HSRC1059TCATCGCTGCTTCCACCTTAGACATTTGAACCCAATCCCTCC HSRC1350TGGAGGGAAAAGTCATTAAAGCAGTGGGGTCAAATCAACTCAGCAT HSRC1482TTCCGGGTCTAGTTTTACTTGCATAGTTCCATTTCTTTGTTGATTTGTCT HSRC1767AATGAGAAGTGGAGTAATGATGTTAAGGGTTATGTAGTGTTTGTTGAG HSRC1890CCAAATAGGATTCTACCAGCAACTAAGCCCATCTAAACTGACCAAC HSRC2005TTGGTCCACTTCGCACTCTATTACCTGGAGTCCTCAAAGTGAAAAC HSRC2256ATTTGATGCCCCTATTTTTCTTCTCAGTTCTAATTTCCGTTTGCTC HSRC2678ATAAATACTCCAACGCTCTCCACTGTCACATCTTCAACCACC HSRC2813ACAGCCGACACAATGTAAAGCAAAGCACAGAATTCAAAGGCAAAAG HSRC3459CAGGGCGTAGGGTCATTCGGTTTCTTGTGGGGTTGG HSRC3538GACCCACCCCAAAATCGCTCATCAACAGTCACCCCC HSRC4138TTCCTCCTCCCCTTCTCAAAACCATCAATTCACCACCACATCCT HSRC4345CTTGACATCGGTCCCCTTACGCAATGTTGGTGACGGC HSRC4653GTCCCACTTTTGATAGTTGAGATGCGAAGAAGGGTTTACTTTCCG Y1972CACGGAAGCAGCTCATCATCCTGCCGACATGACTACAAC GBss-sa-72GCAGCAGTTCCGTTCTTGAGTGCTGAATTTAGTCTGCATAG GBssrsa-123GCAAACACATGCATCCCTGCCCTGATGATAAAGCCA SI-ssr30GATTGCAGAAATTGACACCACACTAGGCGAAGAATTCAAGA ZM1079GTCTGAGACTCGCTTTCATAATTACCAATTTCAAGGGTAGC ZM1413 ACACACACACACACACAATTCGTAGATTTCCCACCTCTCTCT Hs02CCATTAAATTCTTGCTCCCCCTGGTCGTATGCAGCATCTT Hs94CATGTGTTCTCTCCCACCACTCTTGACCATGTTTTCCACC Hs189CTCCAACCCCCATAAATCACGCTTCTGGAGAGGAGATTGC
附表3 芝麻种质资源描述性状赋值
Schedule 3 Coden designed for descriptive traits in sesame

性状Traits赋值Coden of descriptive traits 株型Branching pattern1=单杆,2=分枝1= Non branching, 2=Branching 叶腋花数No. of capsules per axil1=单花,2=三花,3=多花1= One flower, 2= Three flowers, 3=More than three flowers 蒴果棱数No. of locules per capsule1=四棱,2=六棱,3=八棱,4=混合1= Four, 2= Six, 3= Eight, 4= Mixed 叶型Leaf shape1=柳叶型,2=披针形,3=椭圆形,4=卵形,5=心形1=Linear, 2=Lanceolate, 3=Elliptic, 4=Ovate,5=Narrowly cordate 叶色leaf colour1=浅绿,2=绿,3=深绿1=Light green, 2=Green, 3=Dark green 花冠颜色Exterior corolla colour1=白色,2=粉红色,3=浅紫色,4=紫色,5=栗色1=White, 2=Pink, 3=Light purple, 4=Purple, 5=Maroon 茎秆茸毛量Stem hairiness0=无,3=少,5=中等,7=多0=None, 3=Sparse, 5=Medium, 7=Dense 裂蒴性Capsule dehiscence at ripening1=不裂,2=轻裂,3=裂1=Non-shattering, 2=Partially shattering, 3=Completely shattering 茎杆成熟色Main stem colour1=黄,2=绿,3=紫绿,4=紫1=Yellow, 2=Green, 3=Purplish green, 4=Purple 籽粒颜色Seed coat colour1=白,2=乳白,3=黄,4=浅褐,5=褐,6=砖红,7=橄榄绿,8=灰,9=黑1=White, 2=Cream, 3=Yellow, 4=Light brown, 5=Brown, 6=Brick red, 7=Olive, 8=Grey, 9=Black
Construction of Core Collection of Sesame Based on Phenotype and Molecular Markers
LIU YanYang1, MEI HongXian1, DU ZhenWei1, WU Ke1, ZHENG YongZhan1, CUI XiangHua2, ZHENG Lei3
(1Sesame Research Center, Henan Academy of Agricultural Sciences, Zhengzhou 450002;2Zhumadian Academy of Agricultural Sciences, Zhumadian 463000, Henan;3Luohe Academy of Agricultural Sciences, Luohe 462300, Henan)
【Objective】The objective of this study was to manage, research and utilization of sesame (L.) germplasm resources more effectively, and to provide excellent genetic resources for sesame breeding.【Method】In this study, 5 020 accessions of sesame germplasm resources were systematically identified. Firstly, the primary core collections were constructed by using proportion strategy and UPGMA clustering sampling method within subgroups according to geographical origins. Then using an allele preferred sampling strategy and stepwise UPGMA clustering sampling approach according to SSR molecular data, these accessions were further screened to form core collections.The Nei’s gene diversity () and the Shannon-Wiener index () of the core collection and the primary one were measured by-test. The cluster sampling was terminated until the genetic diversity of the core collection begun to have a significant difference with the primary one. Then the core collections without a significant difference with the primary core collection were chosen as the best core collections. The representativeness of the core collections was assessed by the Nei’s diversity index, Shannon-Wiener diversity index, percentage of polymorphic bands, polymorphic band retention, variable rate of coefficient of variation, coincidence rate of range, variance difference percentage and mean difference percentage.【Result】The primary core collections containing 816 accessions and core collections with 501 accessions were constructed, accounting for 16.25% and 9.98% of the total germplasm resources, respectively. The core collections consist of 442 Chinese landraces and 59 foreign germplasm resources. The core collection with 0.2989 in Nei’s diversity index and 0.4243 in Shannon-Wiener diversity index, and did not have a significant difference in molecular diversity with primary core collections (=0.2791,=0.4302) at<0.05. The percentage of polymorphic loci and reserved rate of number of polymorphic loci, variable rate of coefficient of variation and coincidence rate of range were 91.25%, 95.23%, 99.14%, 86.85%, respectively. Variance difference percentage and phenotypic indexes of mean difference percentage was 0. results of-test showed that no significant difference was found in genetic diversity indexes between the core collections and original collections. Compared with the random sampling strategy, allele preferred sampling strategy could construct more representative core collections with higher values of genetic diversity indexes and fewer loss of allele. The Shannon-Wiener index performed higher identifying efficiency than Nei’s diversity index. 【Conclusion】The primary core collections were constructed by using proportion strategy and clustering sampling method within subgroups according to geographical origin, and then using an allele preferred sampling strategy and stepwise UPGMA clustering sampling approach according to SSR molecular data to form core collections, which is a suitable method for constructing sesame core collections. The core collections of sesame are well representative of the original collections in the phenotypic and molecular genetic diversity.
sesame; germplasm resources; core collection; representative test
2017-01-03;接受日期:2017-03-09
国家自然科学基金(31301359)、现代农业产业技术体系建设专项资金(CARS-15)、河南省基础与前沿技术研究计划(152300410143,162300410164)
刘艳阳,E-mail:liuyanyang001@163.com。梅鸿献,E-mail:meihx2003@126.com。刘艳阳与梅鸿献为同等贡献作者。通信作者郑永战,E-mail:sesame168@ 163.com

