基于PCR-DGGE技术分析牧区酸马奶中乳酸菌多样性
马俊英,葛武鹏,姜 琪,房若愚,梁秀珍,吴小勇,耿 炜
(1.西北农林科技大学 食品科学与工程学院,陕西杨凌 712100;2.吉林大学 食品科学与工程学院,长春 130062;3.陕西飞天乳业有限公司,陕西宝鸡 721100;4.咸阳市食品药品检测检验中心,陕西咸阳 712000;5.咸阳市产品质量监督检验所,陕西咸阳 712000)
基于PCR-DGGE技术分析牧区酸马奶中乳酸菌多样性
马俊英1,葛武鹏1,姜 琪2,房若愚1,梁秀珍3,吴小勇4,耿 炜5
(1.西北农林科技大学 食品科学与工程学院,陕西杨凌 712100;2.吉林大学 食品科学与工程学院,长春 130062;3.陕西飞天乳业有限公司,陕西宝鸡 721100;4.咸阳市食品药品检测检验中心,陕西咸阳 712000;5.咸阳市产品质量监督检验所,陕西咸阳 712000)
采用聚合酶链式反应和变性梯度凝胶电泳(PCR-DGGE)技术,分析采自新疆和内蒙古6个牧区的混合酸马奶样品中乳酸菌的总DNA,利用PCR扩增其16S rRNA的V3区段,建立由标准菌株组成的参考梯度,对其进行PCR-DGGE,藉此鉴定菌株,对不能鉴定的菌株切胶回收进行测序鉴定,并分析比较优势菌株。结果表明:共鉴定出10种乳酸菌株,包括植物乳杆菌、嗜酸乳杆菌、马乳酒乳杆菌、发酵乳杆菌、詹氏乳杆菌、短乳杆菌、瑞士乳杆菌、乳酸乳球菌、嗜热链球菌及干酪乳杆菌;且嗜热链球菌为所有样品中的共有菌株;马乳酒乳杆菌、短乳杆菌及瑞士乳杆菌是新疆酸马奶中的优势菌株,而内蒙古样品中的优势菌株不尽相同。
PCR-DGGE;乳酸菌;酸马奶;优势菌株
内蒙古和新疆位于中国北方边疆地区,均属温带大陆性气候,是中国牧区的典型代表。该地区独特的自然条件蕴藏着丰富的乳酸菌资源。长期的自然选择使得乳酸菌具有与当地环境相适应的特性。几千年来,牧民沿袭传统的发酵模式制作各种乳制品使得这种特殊的资源得以保存。酸马奶是一种由鲜马奶接种少量已发酵马奶制成的低酒精含量的发酵乳饮料,其生产工艺并非完全模式化,一般通过乳酸菌和酵母菌自然发酵而成。来源于原料奶和环境的乳酸菌是发酵过程的主要菌种,对乳制品风味和质地的形成起着至关重要的作用[1]。牧区传统酸马奶制作工艺开放,乳酸菌资源丰富,酸马奶已成为近年来乳酸菌开发利用的重要资源[2]。然而,传统酸马奶中的微生物组成和数量常因地区、气候、制作方法、季节等的不同而略有差异。因此,对不同地区酸马奶中乳酸菌多样性的研究具有重要意义。
自Ampe等[3]首次将聚合酶链式反应和变性梯度凝胶电泳(Polymerase chain reaction-denatured gradicnt gel electrophoresis,PCR-DGGE)技术应用于食品微生物研究以来[4],该技术已广泛用于传统发酵食品中微生物种群结构和种群动力学研究[5-6]。目前,PCR-DGGE作为研究复杂微生物体系的一项较为成熟的常规技术,可用于分析优势菌种,最大程度地反映样品中微生物菌群。Ma等[7]采用PCR-DGGE方法对含有乳酸菌的乳制品进行菌群组成分析。Porcellato等[8]将高分辨率熔解分析(high-resolution melt analysis)应用到PCR-DGGE条带的鉴定中,快速将乳制品中乳酸菌鉴定到种的水平。因此,本研究采用PCR-DGGE技术对新疆和内蒙古地区传统发酵的酸马奶中乳酸菌的组成进行分析,并鉴定酸马奶中的优势乳酸菌菌种,为其工业化生产提供技术支持。
1 材料与方法
1.1 材 料
1.1.1 样品来源 酸马奶样品:在新疆伊犁地区4个牧区和内蒙古2个牧区采集30份不同牧民家庭自制传统发酵酸马奶,每个牧区样品采集数为5。装入100 mL无菌样品瓶中,使用冰盒运输至实验室,放入4 ℃冰箱,备用。根据采样地区将30份酸马奶混合成6份具有代表性的样品。采样地点分布如表1所示。
标准菌株:试验所用的乳酸菌标准菌株由西北农林科技大学食品科学与工程学院测试中心提供。

表1 新疆和内蒙古地区奶渣样品的采样地分布情况及样品编号Table 1 Distribution of sample sites and number of samples koumiss from Xinjiang and Inner Mongolia
1.1.2 试剂与仪器 试剂:Takara PremixTaq试剂由宝生物工程(大连)有限公司提供;引物由生工生物工程(上海)股份有限公司合成;链霉蛋白酶(Roche,中国上海)、溶菌酶、丙烯酰胺、去离子甲酰胺、甲叉双丙烯酰胺、TEMED(四甲基乙二胺)、过硫酸铵、十二烷基磺酸钠、尿素,试剂均为分析纯,购于北京索莱宝科技有限公司。MRS培养基和LB培养基参照文献[9]进行配制。
仪器:包括ZONKIA HC-3018R高速冷冻离心机(安徽中科中佳科学仪器有限公司)、PCR仪(PTC-200型)、变性梯度凝胶电泳装置(Bio-Rad,USA)、凝胶成像仪E1617-T130 plus(Bio-Rad,USA)、电泳仪DYY-6C(北京六一)、微量移液器(德国 Eppendorf 公司)、生化培养箱(上海森信实验仪器有限公司)、立式压力蒸汽灭菌器 YXQ-LS-50SII-01-00(上海博迅实业有限公司)、电子分析天平 SE-6001F(美国 Ohaus)、超净工作台 SW-CJ-2D 型(苏州净化设备有限公司)。
1.2 方 法
1.2.1 乳酸菌标准菌株的活化及其基因组DNA的提取 为更好地鉴定样品的DGGE条带,用经平板培养法从酸马奶中分离得到的7种主要乳酸菌[2,10-11]标准菌作为参考。这7种乳酸菌分别为植物乳杆菌(Lactobacillusplantarum)ATCC14917,嗜酸乳杆菌(Lactobacillusacidophilus)ATCC43121,发酵乳杆菌(Lactobacillusfermentum)ATCC14931,短乳杆菌(Lactobacillusbreris)ATCC367,瑞士乳杆菌(Lactobacillushelveticus)ATCC15009,嗜热链球菌(Streptococcusthermophiles)ATCC14485和干酪乳杆菌(Lactobacilluscasei)ATCC393。将标准菌接种于MRS液体培养基中,37 ℃厌氧条件下培养,取1.5~2 mL OD600=1纯化后的标准菌株的菌悬液,离心1 min(4 ℃,12 000 r/min),弃去上清液,再加入与培养液等量的生理盐水(w=0.85%) 悬浮菌体,12 000 r/min(4 ℃)下离心1 min(重复2~3次) 后,加入适量无菌生理盐水,振荡混匀即可。将收集的菌体按照Takara Mini BEST Bacteria Genomic DNA Extraction Kit Ver.3.0的说明提取菌株基因组DNA,置于-20 ℃保存,备用。
1.2.2 酸马奶样品中乳酸菌总DNA提取 酸马奶样品总DNA提取参照EL-baradei等[12]的方法,并进行改进。5 mL酸马奶样品与5 mL质量浓度为20 g/L柠檬酸钠溶液振荡混匀后,加10 mg链霉蛋白酶和20 μL β-巯基乙醇,50 ℃水浴加热3 h。菌体经洗涤后,利用溶菌酶结合SDS变性剂裂解菌体,采用酚仿和异丙醇分离纯化总DNA,经8 g/L琼脂糖电泳检测后,于-20 ℃保存,备用[13]。
1.2.3 PCR扩增乳酸菌基因组DNA16S rRNA的V3区段 细菌16S rRNA的可变区序列因不同细菌而异,而恒定区序列基本保守,所以可以利用恒定区序列设计引物将 16S rRNA片段扩增出来,利用可变区序列的差异来对不同菌属、菌种的细菌进行分类鉴定。16S rRNA 的V3区段是其中的高保守区[14]。采用Hao等[15]报道的基因引物进行PCR扩增。用于DGGE分析的样品经过2轮PCR 扩增获得。以第1步扩增的产物为模版进行第2次扩增[16],使得目的片段浓度增大[1]。以W01和W012为引物,以样品总DNA为模板,对包含16S rRNA的V3区段的700 bp的片段进行PCR扩增。25 μL的反应体系中包含Takara PremixTaq12.5 μL、模板2 μL、上下游引物(10 μmol/L) 各0.5 μL,添加ddH2O至25 μL。PCR反应循环条件:96 ℃预变性4 min;96 ℃变性10 s,50 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃延伸2 min。接着以第1轮PCR产物做模板,以带GC夹(GC-Clamp)的引物(表2)GC-HDA1和HDA2对200 bp左右的V3区进行PCR扩增(表2中[AC]和[GT]表示简并引物)。在50 μL反应体系中包含Takara PremixTaq25 μL、模板2 μL、上下游引物(10 μmol /L)各1 μL,添加ddH2O至50 μL。PCR反应循环条件:94 ℃预变性4 min;94 ℃变性30 s,56 ℃退火30 s,68 ℃延伸1 min,30个循环;68 ℃延伸7 min。PCR扩增产物用2 g/L的琼脂糖凝胶电泳检测。

表2 乳酸菌基因扩增引物Table 2 Primers used for cloning of the lactic acid bacteria gene
1.2.4 标准阶梯的建立 以7种标准菌株的DNA为模板,用引物GC-HDA1/HDA2分别对其V3区进行PCR扩增,反应体系和反应程序与样品的PCR扩增相同,再按照闫苗章等[17]的方法,采用Bio-Rad公司的DcodeTM基因突变检测系统对这7种标准菌株PCR产物及其混合物进行DGGE分析,建立标准阶梯。其中凝胶的丙烯酰胺凝胶质量浓度为8 g/L,变性梯度为40%~60%,上样量为25 μL,先在60 ℃下以50 V电泳1 h,再于65 V电泳14 h,核酸染料染色30 min,用Bio-Rad DOC 1000凝胶成像系统检测。
1.2.5 酸马奶样品PCR产物的DGGE分析 按照“1.2.4”建立标准阶梯的方法,对酸马奶样品中乳酸菌第2轮的PCR产物及标准菌株PCR产物的混合物进行DGGE分析。
1.2.6 凝胶回收测序及种属归类 将DGGE图谱中未能与标准阶梯中菌种比对且位置不同的代表条带切下浸泡于100 μL的ddH2O中4 ℃过夜。吸取2 μL浸泡液作为模板,以不带GC夹的HDA1和HDA2为引物进行PCR 扩增。将扩增产物连接到pMD19-T载体,转化到大肠杆菌JM109中,挑取阳性克隆(白色)接种于LB液体培养基中,37 ℃摇菌过夜。菌液送上海生工生物工程技术服务有限公司进行测序。DGGE优势条带的测序结果与NCBI的GenBank数据比对分析[17],进行种属归类。
2 结果与分析
2.1 参考阶梯的建立
为确认参考阶梯中各个条带所代表的菌种,将7种标准菌株的PCR产物分别加入单独泳道(1~7) ,其混合物加入另外一个泳道M。本试验采用的7种标准菌株可分别在DGGE变性胶中得到鉴别。
如图1所示,7种标准菌株的PCR产物在变性胶中清晰地分开。泳道1~7代表7种不同的标准菌株,通过参考阶梯中各个菌种在DGGE图谱中的位置可以确定参考阶梯中各条带代表的菌种。因此,可以用此参考阶梯来确定酸马奶样品中乳酸菌的部分菌种。
2.2 酸马奶样品中乳酸菌DGGE分析
提取酸马奶样品中细菌总DNA,用特异性引物扩增细菌16S rDNA V3区,对不同地区的酸马奶样品的PCR产物进行DGGE电泳(图2),图中不同地区的酸马奶中乳酸菌DGGE图谱有一定的差异。
6个地区的酸马奶样品的DGGE电泳图中(图2)有23个特异性条带,其中7个可以用参考阶梯确定。已确定的菌种包括ATCC14917(a)、ATCC43121(b)、ATCC14931(e)、ATCC367(g)、ATCC15009(i)、ATCC14485(p)和ATCC393(r)。其余的条带(c、d、f、h、j、k、l、m、n、o、q、s、t、u、v、w)切胶回收测序。测序结果登陆NCBI数据库进行BLAST同源性比对分析。
结合DGGE图谱(图2)、Blast同源性比对结果(表3)及表4可以看出,嗜热链球菌 ATCC14485的条带p出现在所有的酸马奶样品中,但样品3和样品6中条带亮度较弱。确定为马乳酒乳杆菌(Lactobacilluskefiranofaciens) 的条带d,短乳杆菌ATCC367的条带g,瑞士乳杆菌ATCC15009的条带i 出现在所有新疆地区的酸马奶样品(1、2、3、4) 中。条带的亮度反映各个菌种在整个菌群中的相对比例[18]。因此,条带p出现的强亮度和高频率表明嗜热链球菌是酸马奶样品中的共有菌种,而马乳酒乳杆菌(条带d),短乳杆菌(条带 g)和瑞士乳杆菌(条带i)是新疆酸马奶中的优势菌种。采自同一地区的样品1和样品2中,嗜热链球菌(条带p)和瑞士乳杆菌(条带i) 是其共有优势菌种,而样品2中条带的数量又反映其样品中乳酸菌较为丰富;采自同一地区的样品3和样品4除含共有优势菌种瑞士乳杆菌(条带i)外,样品4还有其独特的优势菌种嗜热链球菌(条带p) 和发酵乳杆菌(条带e)。
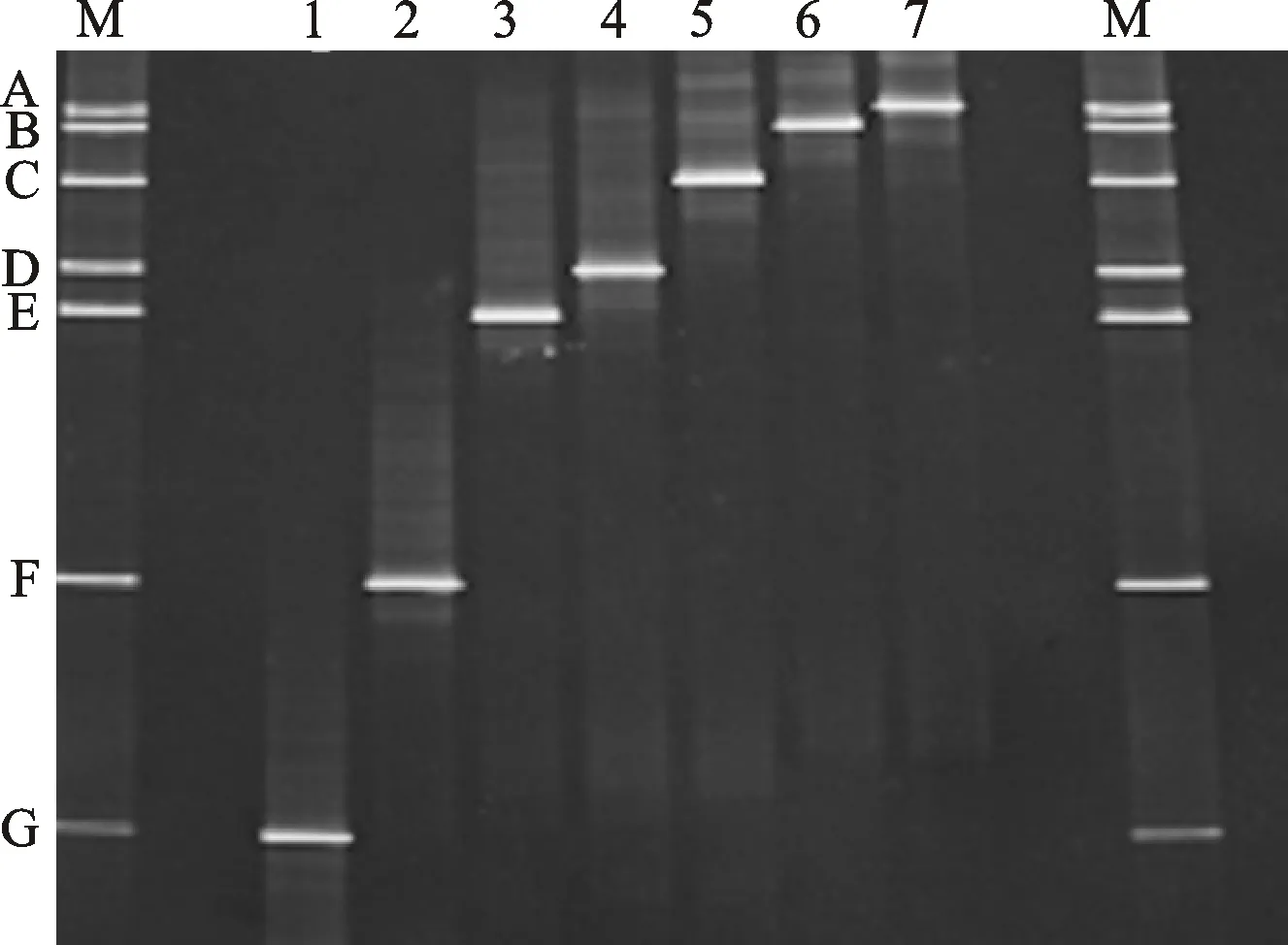
M.参考阶梯Reference ladder 1. ATCC393; 2. ATCC14485; 3. ATCC15009; 4. ATCC367; 5. ATCC14931; 6. ATCC43121; 7. ATCC14917;A~G表示1~7各标准菌在图谱中的位置 A-G represent the location of strain1-7 in profile
图1 建立参考阶梯的标准菌株的DGGE图谱
Fig.1 Denaturing gradient gel electrophoresis profile of strains used in identification ladder for reference
3 讨 论
虽然样品1、2、3、4均来自新疆牧区,但样品1和样品2采自大陆性半干旱气候区的新疆伊犁新源县,而样品3和样品4采自寒温带亚干旱气候区的新疆伊犁昭苏县,气候的差异使优势菌株不同。采自中温带半干旱大陆性气候的样品5中乳酸乳球菌(条带j)和嗜热链球菌(条带p) 是其优势菌种,而采自温带季风型大陆性气候区的样品6中嗜热链球菌则是其优势菌种。样品5和样品6的采样地区虽邻近,但因所处气候区的不同,其优势菌株差异也是很明显的。
这些结果表明,不同地理条件、生产环境的酸马奶有着其特有的菌种。同一地区不同牧民家庭生产的酸马奶中的菌种也会因为各自不同的生产方式及作坊环境而产生差异。此外,酸马奶还会受到马匹品种的影响[19]。

M.参考阶梯 Reference ladder; A.ATCC14917; B. ATCC43121; C.ATCC14931; D. ATCC367; E. ATCC15009; F. ATCC14485; G. ATCC393;1. 新疆伊犁新源县那拉提草原样品 Narat grassland in Xinyuan,Yili,Xinjiang,China;2.新疆伊犁新源县马场样品 Stud-farm in Xinyuan,Yili,Xinjiang,China;3.新疆伊犁昭苏县夏特牧场样品 Xiate pasture in Zhaosu ,Yili,Xinjiang,China;4.新疆伊犁昭苏县阿克达拉乡样品 Akedala in Zhaosu,Yili,Xinjiang,China;5.内蒙古锡林郭勒盟阿巴噶旗样品 Abaga Banner in Xilinguole Inner Mongolia,China;6.内蒙古赤峰市巴林右旗样品 Bahrain right Banner in Chifeng,Inner Mongolia,China.
图2 酸马奶样品中乳酸菌多样性的DGGE图谱
Fig.2 Denaturing gradient gel electrophoresis profiles of the lactic acid bacteria community from koumiss
虽然6个样品中的细菌菌群分布不尽相同,但是采自相同地区的样品的优势菌种却较为相似。由此推测酸马奶中细菌菌群结构上的差异除因地区、气候、季节等的不同而存在差异外,也可能与各家庭生产制作工艺和游牧生活方式的不同有关[15]。孟和毕力格等[20]从内蒙古锡林浩特市的酸马奶样品中鉴定出干酪乳杆菌、植物乳杆菌、嗜酸乳杆菌、副干酪乳杆菌(Lactobacillusparacasei)、棒状乳杆菌棒状亚种(Lactobacilluscoryniformissubsp.coryniformis)、弯曲乳杆菌(Lactobacilluscurvatus)、马乳酒乳杆菌和发酵乳杆菌等乳酸菌。Liu等[10]采用 16S rRNA 基因全序列鉴定内蒙古酸马奶中的优势乳酸菌分别为植物乳杆菌、短乳杆菌、干酪乳杆菌、屎肠球菌、粪肠球菌(Enterococcusfaecalis)、乳酸乳球菌(Lactococcuslactis)、嗜酸乳杆菌、副干酪乳杆菌、德氏乳杆菌保加利亚亚种(Lactobacillusdelbrueckiisubsp.bulgaricus)、瑞士乳杆菌、耐久肠球菌(Enterococcusdurans) ,肠膜明串珠菌(Leuconostocmesenteroides) 及嗜热链球菌;朱丹宇等[21]鉴定新疆酸马奶中的优势乳酸菌有嗜热链球菌、乳酸乳球菌、干酪乳杆菌、植物乳杆菌和发酵乳杆菌;本研究中得出酸马奶中常见的植物乳杆菌,嗜酸乳杆菌,马乳酒乳杆菌,发酵乳杆菌,瑞士乳杆菌,乳酸乳球菌,嗜热链球菌及干酪乳杆菌,此外,还鉴定到詹氏乳杆菌,短乳杆菌这些少有的菌株,嗜热链球菌为所有样品中的共有优势菌株,而马乳酒乳杆菌,短乳杆菌及瑞士乳杆菌为新疆地区酸马奶中的优势菌种。且新疆地区的优势菌株与Hao等[15]利用DGGE结合种特异性PCR的方法分析新疆伊犁牧区酸马奶中乳酸菌的多样性时的研究结果一致。内蒙古地区两份样品的优势菌株差异较大,其优势菌株分别为乳酸乳球菌和嗜热链球菌,并且嗜热链球菌的条带在两样品中的亮度也是迥异。新疆两采样地及内蒙古两采样地的优势菌株也因气候区的不同呈现出差异。

表4 各样品中鉴定出的乳酸菌菌种Table 4 Identification results of lactic acid bacteria in koumiss samples
注:“+”表示有,“-”表示无。
Note:“+” positive,“-” negative.
本研究中,利用DGGE分析检测酸马奶样品中的乳酸菌时,检测到的一些菌株DNA,经培养却不能在选择培养基上发现可见的菌落,这可能是由于这些DNA来源于一些之前就存活于酸马奶样品中但是在低pH环境下凋亡的细菌细胞[15]。也可能是一些乳酸菌因低pH环境处于活的非可培养态(Viable but non-culturable,VBNC)的细胞[22]。采用 PCR-DGGE技术方法分析不同地区酸马奶中乳酸菌多样性,可较为客观地反映出传统工艺制作的酸马奶中乳酸菌的微生物区系,对于全面揭示自然发酵酸马奶优势菌群组成,具有较高的参考价值。通过不同气候区地区的比较发现各地优势菌株的差异,若要系统探明气候区与菌株的相关性,还需扩大研究范围做深入研究。
Reference:
[1] 蒋厚阳,陈芝兰,赵国华,等.PCR-DGGE法分析西藏传统发酵乳制品中乳酸菌的多样性[J].食品科学,2014,35(1):167-173.
JIANG H Y,CHEN ZH L,ZHAO G H,etal.Investigating the diversity of lactic acid bacteria in Tibetan traditional fermented dairy products by PCR-DGGE[J].FoodScience,2014,35(1):167-173(in Chinese with English abstract).
[2] 张明珠.新疆酸马奶中乳酸菌的多态性分析及发酵性能研究[J].食品与发酵科技,2015,51(3):44-48.
ZHANG M ZH.Study on diversity and fermentation characteristic of lactic acid bacteria from koumiss collected in Xinjiang[J].FoodandFermentationTechnology,2015,51(3):44-48(in Chinese with English abstract).
[3] AMPE F,BEN OMAR N,MOIZAN C,etal.Polyphasic study of the spatial distribution of microorganisms in Mexican pozol,a fermented maize dough,demonstrates the need for cultivation-independent methods to investigate traditional fermentations [J].AppliedandEnvironMentalMicrobiology,1999,65(12):5464- 5473.
[4] 励建荣,朱军莉.变性梯度凝胶电泳(DGGE)技术在食品微生物研究中的应用[J].食品科技,2008,33(3):217-220.
LI J R,ZHU J L.Application of DGGE technique in the food microbiology[J].FoodScienceandTechnology,2008,33(3):217-220(in Chinese with English abstract).
[5] OMAR N B,AMPE F.Microbial community dynamics during production of the Mexican fermented maize dough pozol[J].AppliedandEnvironmentalMicrobiology,2000,66(9):3664-3673.
[6] MEROTH C B,HAMMES W P,HERTEL C.Identification and population dynamics of yeasts in sourdough fermentation processes by PCR-denaturing gradient gel electrophoresis [J].AppliedandEnvironmentalMicrobiology,2003,69(12):7453-7461.
[7] MA J X,KONG J,JI M J.Detection of the lactic acid bacteria in commercial yoghurts by PCR-denaturing gradient gel electrophoresis [J].ChineseJournalofApplied&EnvironmentalBiology,2009,15(4):534-539(in English).
[8] PORCELLATO D,GRNNEVIK H,RUDI K,etal.Rapid lactic acid bacteria identification in dairy products by high-resolution melt analysis of DGGE bands[J].LettersinAppliedMicrobiology,2012,54(4):344-51.
[9] 凌代文,东秀珠.乳酸菌的分类鉴定及实验基础[M].北京:中国轻工业出版社,1999:117-129.
LING D W,DONG X ZH.Identification and Experimental Basis of Lactic Acid Bacteria[M].Beijing:China Light Industry Press,1999:117-129(in Chinese).
[10] LIU F,DU L H,DU P,etal.Biodiversity of lactic acid bacteria in home-made koumiss from Inner Mongolia [J].FoodScience,2008,29(2):218-224(in English).
[11] 孙天松,王俊国,张列兵,等.中国新疆地区酸马奶中乳酸菌生物多样性研究[J].微生物学通报,2007,34(3):451-454.
SUN T S,WANG J G,ZHANG L B,etal.The biodiversity of lactic acid bacteria isolated from koumiss-a traditional fermented mare milk product in Xinjiang of China[J].MicrobiolChina,2007,34(3):451-454(in Chinese with English abstract).
[12] EL-BARADEI G,DELACROIX-BUCHET A,OGIER J C.Bacterial biodiversity of traditional Zabady fermented milk[J].InternationalJournalofFoodMicrobiology,2008,121(3):295-301.
[13] 赵 潞,张列兵,张世湘,等.利用种特异性PCR快速鉴定新疆酸马奶优势菌群[J].中国乳品工业,2010,38(1):12-14.
ZHAO L,ZHANG L B,ZHANG SH X,etal.Rapid identification of the dominant LAB in fermented koumiss produced in Xinjiang using species-specific PCR [J].ChinaDairyIndustry,2010,38(1):12-14(in Chinese with English abstract).
[14] 李 鹏,马艳娇,赵 云.16S rRNA、23S rRNA及16S~23S rRNA基因在细菌分离与鉴定中的应用[J].现代畜牧兽医,2008(7):49-52.
LI P,MA Y J,ZHAO Y.Application of 16S rRNA,23S rRNA and 16S-23S rRNA in isolation and identification of bacteria [J].ModernJournalofAnimalHusbandryandVeterinaryMedicine,2008(7):49-52(in Chinese).
[15] HAO Y,ZHAO L,ZHANG H,etal.Identification of the bacterial biodiversity in koumiss by denaturing gradient gel electrophoresis and species-specific polymerase chain reaction [J].JournalofDairyScience,2010,93(5):1926-1933.
[16] TEMMERMAN R,MASCO L,VANHOUTTE T,etal.Development and validation of a nested-PCR-denaturing gradient gel electrophoresis method for taxonomic characterization of bifido bacterial communites [J].AppliedandEnvironmentalMicrobiology,2003,69(11):6380-6385.
[17] 闫苗章,李丽娜,王保莉,等.贵州红壤水稻土淹水培养过程中Fe-氢酶微生物的多样性[J].微生物学报,2013,53(6):577-585.
YAN M ZH,LI L N,WANG B L,etal.Diversity of fe-hydrogenase microbe in Guizhou red paddy soil during flooding incubation[J].ActaMicrobiologicaSinica,2013,53(6):577-585(in Chinese with English abstract).
[18] MUYZER G,WAAL E C D,UITTERLINDEN A G.Profiling of complex microbial populations by denaturing gradient gel electrophoresis analysis of polymerase chain reaction-amplified genes coding for 16S rRNA[J].AppliedandEnvironmentalMicrobiology,1993,59(3):695-700.
[19] WU R,WANG L,WANG J,etal.Isolation and preliminary probiotic selection of lactobacilli from koumiss in Inner Mongolia[J].BasicMicrobiol,2009,49(3):318-326.
[20] 孟和毕力格,乌日娜,王立平,等.不同地区酸马奶中乳杆菌的分离及其生物学特性的研究[J].中国乳品工业,2004,32(11):6-11.
MENGHE BILIGE,WU R N,WANG L P,etal.Isolation and identification of Lactobacillus from koumiss collected in Inner Mongolia and People's Pepublic of Mongolia[J].ChinaDairyIndustry,2004,32(11):6-11(in Chinese with English abstract).
[21] 朱丹宇,王 英,周剑忠,等.酸马奶中乳酸菌的分离和鉴定[J].江苏农业科学,2009(1):245-247.
ZHU D Y,WANG Y,ZHOU J ZH,etal.Isolation and identification ofLactobacillusfrom koumiss[J].JiangsuAgriculturalSciences,2009(1):245-247(in Chinese).
[22] 王亚利,包秋华,王俊国,等.乳酸菌活的非可培养态的研究进展[J].中国乳品工业,2016,44(3):41-45.
WANG Y L,BAO Q H,WANG J G,etal.Progress on the viable but non-culturable(VBNC) state of lactic acid bacteria[J].ChinaDairyIndustry,2016,44(3):41-45(in Chinese with English abstract).
(责任编辑:史亚歌 Responsible editor:SHI Yage)
Diversity of Lactic Acid Bacteria in Koumiss from Xinjiang and Inner Mongolia by PCR - DGGE
MA Junying1,GE Wupeng1,JIANG Qi2,FANG Ruoyu1,LIANG Xiuzhen3,WU Xiaoyong4and GENG Wei5
(1.College of Food Science and Engineering,Northwest A&F University,Yangling Shaanxi 712100,China; 2.College of Food Science and Engineering,Jilin University,Changchun 130062,China; 3.Shaanxi Feitian Dairy Co.Ltd.,Baoji Shaanxi 712000,China; 4.Xianyang Food and Drug Inspection Center,Xianyang Shaanxi712000,China;5. Xianyang Product Quality Supervision and Testing Institute,Xianyang Shaanxi 712000,China)
After total DNA was extracted from koumiss samples from Xinjiang and Inner Mongolia,the V3 region of 16S rRNA gene was amplified by PCR,and a reference ladder derived from amplification products of known lactic acid bacteria was constructed at the same time,then the PCR amplified products were subject to identify the strain by using polymerase chain reaction and denaturing gradient gel electrophoresis(PCR-DGGE),the bands which could not be identified by reference ladder were excised from the gel and sequenced identification,so as to compare and analyze the dominant species.The results showed that 10 dominant species of lactic acid bacteria were identified from the samples.It includedLactobacillusplantarum,Lactobacillusacidophilus,Lactobacilluskefiranofaciens,Lactobacillusfermentum,Lactobacillusjensenii,Lactobacillus brevis,Lactobacillushelveticus,Lactococcuslactis,StreptococcusthermophilusandLactobacilluscasei; moreover,Streptococcusthermophilesappeared in all samples,the dominant species of samples in Xinjiang wereLactobacilluskefiranofaciens,LactobacillusbrevisandLactobacillushelveticuswhile the dominant species were much different from the samples of Inner Mongolia.
Polymerase chain reaction-denaturing gradient gel electrophoresis(PCR-DGGE);Lactic acid bacteria;Koumiss;Dominant species
2016-05-23 Returned 2016-06-12
The Major Product Projects of Strategic Emerging Industries of Shaanxi Province,China(No.2015KTCQ03-08,No.2016KTCQ03-03).
MA Junying,female,master student.Research area:food science.E-mail:winee100@126.com
GE Wupeng,male,Ph.D,associate professor.Research area: food science.E-mail:josephge@sina.com
日期:2017-06-05
2016-05-23
2016-06-12
陕西省科技厅战略性新兴产业重大产品(群)项目(2015KTCQ03-08,2016KTCQ03-03)。 第一作者:马俊英,女,硕士研究生,研究方向为食品科学。E-mail:winee100@126.com 通信作者:葛武鹏,男,博士,副教授,研究方向为食品科学。E-mail:josephge@sina.com
S512.1;S330
A
1004-1389(2017)06-0956-07
网络出版地址:http://kns.cnki.net/kcms/detail/61.1220.S.20170605.1728.042.html

