甘蓝型油菜EMS突变后代材料的分子遗传多样性分析
袁润勤,曲高平,郭 媛,胡胜武
(1.旱区作物逆境生物学国家重点实验室,陕西杨凌 712100;2.西北农林科技大学 农学院,陕西杨凌 712100)
甘蓝型油菜EMS突变后代材料的分子遗传多样性分析
袁润勤1,2,曲高平1,2,郭 媛1,2,胡胜武1,2
(1.旱区作物逆境生物学国家重点实验室,陕西杨凌 712100;2.西北农林科技大学 农学院,陕西杨凌 712100)
旨在利用SSR和SRAP标记从分子水平上分析甘蓝型油菜EMS突变后代材料的遗传差异。选取138个‘中双9号’油菜的EMS突变M3株系(以‘中双9号’为参照),这些材料的叶、花、角果等存在丰富的形态学变异,选用24对扩增条带清晰、多态性高的SRAP引物和20对SSR引物对参试的139个材料进行分析。结果显示,24对SRAP引物共检测到360个位点,其中102个为多态性位点,平均多态性比率为28.33%,平均多态性信息含量PIC为0.60;20对SSR引物共检测到196个位点,其中91个为多态性位点,平均多态性比率为46.43%,PIC为0.59。参试材料之间的遗传相似系数为0.62~0.91,聚类分析和群体结构分析结果显示,参试EMS突变M3株系没有完全按表型突变聚在一起。研究表明,EMS诱导‘中双9号’产生了分子水平上可检测的突变且诱变后代材料之间存在广泛的遗传差异。
甘蓝型油菜;EMS突变后代材料;遗传多样性;SSR标记;SRAP标记
甲基磺酸乙酯(Ethyl methyl sulfonate,EMS)是作物诱变育种中利用最广泛、效果最好的一种化学诱变剂[1]。迄今为止,通过化学诱变育成406个作物品种,其中EMS诱变育成的品种有116个,占化学诱变育成品种的28.6%(http://mvgs.iaea.org/)。甘蓝型油菜是一个比较年轻的物种,近代油菜品质育种(低芥酸、低硫甙)使其经历了2次瓶颈,导致其遗传基础进一步狭窄[2]。通过EMS等诱变处理,是创造油菜新变异、丰富油菜资源的一条重要途径。前人以不同油菜品种为基础材料,通过EMS诱变创建了几个油菜突变体库[3-6],并对突变材料的形态特征、植物学性状及品质等进行了鉴定分析,这些突变体的获得拓宽了油菜遗传变异。
分子标记具有稳定性强、不受环境条件的影响、没有组织或器官的特异性、不受个体发育阶段的影响等特点,广泛应用于作物遗传育种研究。目前,利用SSR分子标记对玉米[7-8]、小麦[9]、大豆[10]等作物EMS突变后代材料进行了检测,发现不同诱变系间存在一定的遗传变异,且这些变异与品质性状、农艺性状相关。这些研究结果为突变体的利用提供了参考依据。然而,目前还未见对油菜EMS突变体后代的分子遗传多样性研究的报道。
西北农林科技大学油菜研究中心生物技术育种课题组利用体积分数为1.0% 的EMS溶液处理‘中双9号’油菜干种子,创建了包括子叶、叶片、花器、株型、角果等多器官变异类型的突变体库[6]。本研究从该突变体库M2代2 558个株系中选择138个不同变异类型材料,种成M3株系,利用SSR和SRAP标记对这138个M3代株系和基础材料‘中双9号’(对照)进行遗传多样性评价,为这些材料在油菜遗传育种中的利用提供参考。
1 材料与方法
1.1 材 料
利用体积分数为1.0% EMS溶液处理甘蓝型油菜‘中双9号’干种子,得到一个变异类型比较丰富的突变体库。从该突变体库M2代2 558个株系中选择138个不同变异类型材料,种成M3株系。这些材料表现出较为广泛的变异,包括25个不育材料,13个花色突变材料,5个花闭合材料,3个无雌蕊突变材料,9个花瓣突变材料,6个其他花变异;3个叶片内卷材料,22个叶色突变材料;15个角果突变材料;17个种子突变材料;5个株型突变材料;还有15个其他类型突变材料(表1)。以基础材料‘中双9号’油菜为对照,进行观察和分析。
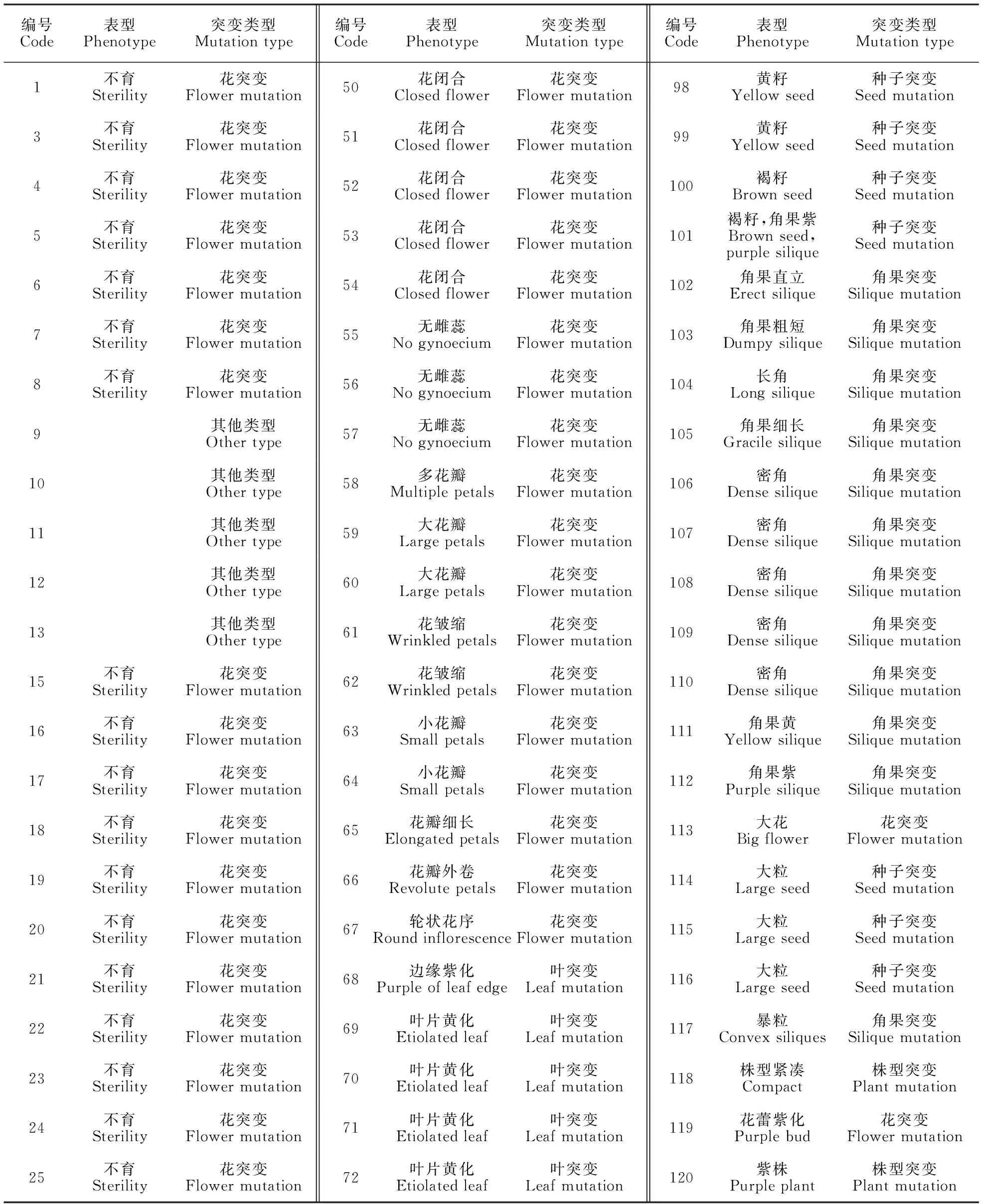
表1 突变材料及其典型特征Table 1 The characteristics of rapeseed mutants
(续表1 Continued table 1)

编号Code表型Phenotype突变类型Mutationtype编号Code表型Phenotype突变类型Mutationtype编号Code表型Phenotype突变类型Mutationtype26不育Sterility花突变Flowermutation73叶片黄化Etiolatedleaf叶突变Leafmutation121紫株Purpleplant株型突变Plantmutation27不育Sterility花突变Flowermutation74叶片黄化Etiolatedleaf叶突变Leafmutation122褐籽Brownseed种子突变Seedmutation28不育Sterility花突变Flowermutation75叶片黄化Etiolatedleaf叶突变Leafmutation123角果紫Purplesilique角果突变Siliquemutation29不育Sterility花突变Flowermutation76黄化-白化Etiolated-albinistcleaf叶突变Leafmutation124角果紫Purplesilique角果突变Siliquemutation30不育Sterility花突变Flowermutation78边缘黄化Etiolatedofleafedge叶突变Leafmutation125角果紫Purplesilique角果突变Siliquemutation31不育Sterility花突变Flowermutation79叶片黄化Etiolatedleaf叶突变Leafmutation127紫株Purpleplant株型突变Plantmutation32不育Sterility花突变Flowermutation80浅绿Lightgreenleaf叶突变Leafmutation128柱头外露Stigmashowing花突变Flowermutation33叶片内卷Involuteleaf叶突变Flowermutation81边缘黄化Etiolatedofleafedge叶突变Leafmutation129墨绿叶Blackishgreenleaf叶突变Leafmutation34叶片内卷Involuteleaf叶突变Flowermutation82不完全黄化Notcompletelyetiolatedleaf叶突变Leafmutation130叶片黄化Etiolatedleaf叶突变Leafmutation35叶片内卷Involuteleaf叶突变Flowermutation83叶片黄化Etiolatedleaf叶突变Leafmutation131花枝异化Sprayalienation花突变Flowermutation36花色浅Flowerwithlightcolor花突变Flowermutation84叶片黄化Etiolatedleaf叶突变Leafmutation132丛生Caespitosebranches株型突变Plantmutation37花色浅Flowerwithlightcolor花突变Flowermutation85不完全黄化Notcompletelyetiolatedleaf叶突变Leafmutation133其他类型Othertype38花色浅Flowerwithlightcolor花突变Flowermutation86浅绿Lightgreenleaf叶突变Leafmutation134其他类型Othertype39花色浅Flowerwithlightcolor花突变Flowermutation87叶片黄化Etiolatedleaf叶突变Leafmutation135其他类型Othertype40花色浅Flowerwithlightcolor花突变Flowermutation88边缘黄化Etiolatedofleafedge叶突变Leafmutation136其他类型Othertype41花色白Whiteflower花突变Flowermutation89黄籽Yellowseed种子突变Seedmutation137其他类型Othertype42花色白Whiteflower花突变Flowermutation90黄籽Yellowseed种子突变Seedmutation138其他类型Othertype43花色白Whiteflower花突变Flowermutation91黄籽Yellowseed种子突变Seedmutation139其他类型Othertype44花色白Whiteflower花突变Flowermutation92黄籽Yellowseed种子突变Seedmutation140其他类型Othertype45花色白Whiteflower花突变Flowermutation93黄籽,角果粗短Yellowseed,dumpysilique种子突变Seedmutation141其他类型Othertype46花色白Whiteflower花突变Flowermutation94黄籽Yellowseed种子突变Seedmutation142其他类型Othertype47花色白Whiteflower花突变Flowermutation95黄籽Yellowseed种子突变Seedmutation143中双9号ZhongshuangNo.9突变基础材料Wildtype48花色白Whiteflower花突变Flowermutation96黄籽Yellowseed种子突变Seedmutation49多花丛生Twoflowerswithonestalk花突变Flowermutation97黄籽Yellowseed种子突变Seedmutation
1.2 方 法
1.2.1 DNA的提取 从每个M3株系中随机选取5个单株,每个单株取等量嫩叶混合,按照Murray等[11]报道的改良CTAB法提取总基因组DNA。
1.2.2 SRAP-PCR SRAP引物参照Li等[12],由上海生工生物工程技术服务有限公司合成,34条上游引物和19条下游引物随机组合成646对引物组合,从中筛选出扩增条带丰富、清晰、稳定的24对引物组合用于对所有参试材料的扩增分析。PCR及产物检测:PCR反应体系为10 μL,其中模板DNA(50 ng/μL)2 μL、ddH2O 2.4 μL、SRAP正反向引物各0.3 μL、2× EsTaqDNA Polymerase(康为世纪生产)5 μL。PCR扩增程序为:94 ℃预变性1 min;94 ℃变性1 min,35 ℃退火1 min,72 ℃延伸1 min, 5个循环;94 ℃变性1 min,50 ℃退火1 min,72 ℃延伸1 min, 35个循环;72 ℃延伸10 min,4 ℃保存。PCR扩增产物用80 g/L非变性聚丙烯酰胺凝胶电泳检测。
1.2.3 SSR-PCR 从380对引物中筛选出20对扩增条带清晰、稳定的多态性引物并用于对所有参试材料的扩增分析。PCR及产物检测:PCR反应体系为10 μL,其中模板DNA(50 ng/μL)2 μL、ddH2O 2.4 μL、SSR正反向引物各0.3 μL、2×EsTaqDNA Polymerase(康为世纪生产)5 μL。PCR扩增程序为:95 ℃预变性4 min;94 ℃变性1 min,60 ℃退火1 min,72 ℃延伸1 min,10个循环,每个循环退火温度下降0.5 ℃;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸60 s,36个循环;72 ℃延伸4 min。PCR扩增产物用80 g/L非变性聚丙烯酰胺凝胶电泳检测。
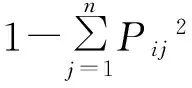
根据0、1矩阵,利用NTSYSpc 2.0软件[13]中的子程序SHAN程序和UPGMA方法进行聚类分析,对139个材料构建聚类图;采用NTsys-pc 2.0软件的Dice计算参试材料的遗传相似系数(GS),遗传距离(GD)=1-GS。利用Structure 2.3.4[14]软件进行群体结构分析。
2 结果与分析
2.1 参试材料的SRAP和SSR分析
从646个SRAP引物组合中筛选出多态性高、条带清晰、重复性好的24对引物对参试的138个M3株系及‘中双9号’(对照)进行分析。结果共扩增出360个条带,其中多态性条带102个,平均每对引物组合检测到4.25个多态性条带,平均多态性比率为28.33%。在选用的24对引物组合中,引物组合Em15+Me11扩增的多态性条带数最多(10),引物组合Em11+Me21为单态位点,各对引物可扩增出1~10个多态性位点。SRAP引物组合PIC为0.17(Em14+Me23)~0.85(Em15+Me11),平均为0.60(表2)。
从380对SSR引物中筛选出20对扩增结果稳定的多态性引物,用于138个M3株系及‘中双9号’(对照)遗传多样性分析。结果共扩增得到196个位点,其中多态性位点91个,平均4.55个,平均多态性比率为46.43%。选用的20对引物可扩增出2~8个多态性位点,其中引物CB10028扩增到8个,引物Ni4-C06和引物Na14-D07仅扩增出2个多态性位点。SSR引物组合PIC为0.21(Ni4-D09)~0.80(CB10028),平均为0.59(表3)。
2.2 聚类分析
基于SRAP和SSR标记数据,139个材料两两组合共得到9 591个遗传相似系数,138个诱变材料与基础材料‘中双9号’的遗传相似系数为0.50~0.82,平均为0.70。将遗传相似系数转化为遗传距离,以0.02为组距进行次数分布分析,139个试验材料间的遗传距离为0.09~0.52,平均为0.29,其中No.33和No.20,No.108和No.20遗传距离最大,为0.52;No.46和No.40,No.41和No.46,No.63和No.85,No.80和No.85,No.121和No.130,No.130和No.131间的遗传距离最小,为0.09。84%的遗传距离数据集中分布在0.21~0.37,遗传距离小于0.21有699个(7.3%),遗传距离大于0.37有840个(8.7%)。97% 的遗传距离分布在0.17~0.42(图1)。
根据SRAP和SSR标记数据,利用UPGMA方法构建聚类图(图2)。结果139个材料被分为5类(Cluster Ⅰ-V):Cluster Ⅰ由44个材料组成,其中25个花突变材料,12个叶突变材料,5个种子突变材料,1个角果突变材料和基础材料‘中双9号’;44个材料间的遗传相似系数为0.74~0.91。Cluster Ⅱ由32个材料组成,其中4个花突变材料,2个叶突变,8个种子突变材料,9个角果突变材料,3个株型突变材料,6个其他类型突变材料;32个材料间的遗传相似系数为0.73~0.91。Cluster Ⅲ由8个材料组成,其中3个花突变材料,1个叶突变材料,2个种子突变材料,1个角果突变材料,1个其他类型材料;8个突变材料的遗传相似系数为0.73~0.85。Cluster Ⅳ由40个突变材料组成,其中21个花突变材料,7个叶突变材料,2个种子突变材料,1个角果突变材料,2个株型突变材料,7个其他类型材料;40个突变材料间的遗传相似系数为0.67~0.88。Cluster Ⅴ包括15个突变材料,其中8个花突变材料,3个叶突变材料,3个角果突变材料, 1个其他类型突变材料;这15个突变材料的遗传相似系数为0.62~0.75。所有供试的138个突变材料没有按照叶突变、花突变、角果突变、种子突变、株型突变和其他突变类型聚类。
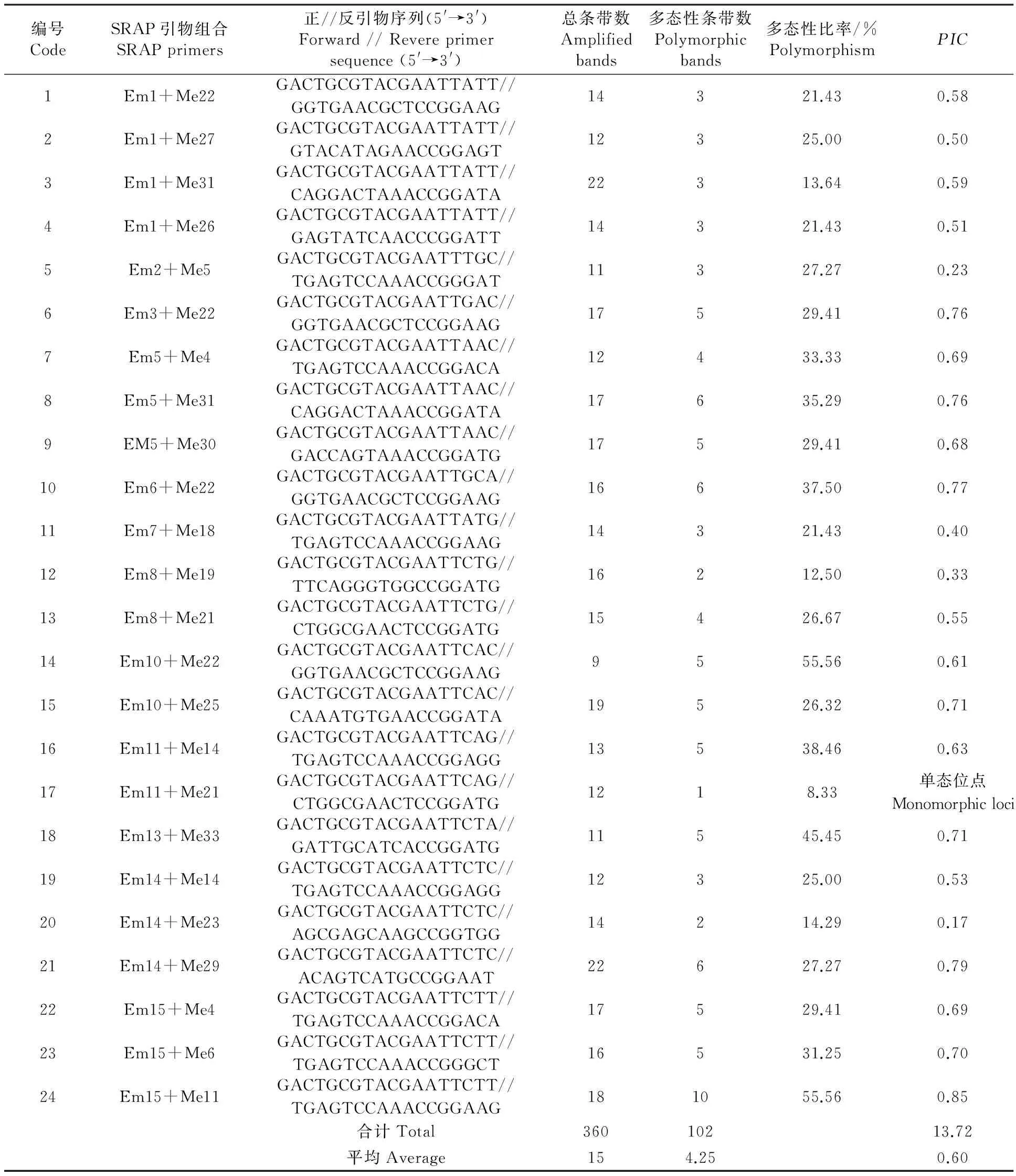
表2 SRAP引物组合及扩增结果Table 2 The SRAP primer combinations and their amplified results
2.3 群体遗传结构分析
根据SRAP和SSR扩增结果,利用STRUCURE 2.2软件对139个材料进行群体结构分析。根据不同k值所对应的LnP(D)值,发现k=5时出现拐点,因此将139个材料分为5个组(GⅠ~GⅤ,图3)。GⅠ包括44个突变材料,其中23个花突变材料,13个叶突变材料,5个种子突变材料,1个角果突变材料,1个株型突变材料,和基础材料‘中双9号’;包括聚类分析Cluster Ⅰ中38个突变材料和基础材料‘中双9号’,Cluster Ⅱ中2个突变材料,Cluster Ⅲ中1个突变材料,Cluster Ⅳ中2个突变材料。G Ⅱ包括16个突变材料,其中5个花突变材料,3个叶突变材料,3个角果突变材料,2个株型突变材料,3个其他类型突变材料;包括聚类分析Cluster Ⅳ中5个突变材料,Cluster Ⅴ中11个突变材料。G Ⅲ包括14个材料,其中4个花突变材料,5个叶突变材料,2个种子突变材料,1角果突变材料,2个其他类型突变材料;包括聚类分析Cluster Ⅲ中的7个突变材料,Cluster Ⅳ中的4个材料,Cluster Ⅴ中的3个突变材料。G Ⅳ包括33个材料,其中4个花突变材料,1个叶突变材料,9个种子突变材料,10个角果突变材料, 2个株型突变材料,7个其他类型的突变材料;包括聚类分析Cluster Ⅱ中的30个突变材料,Cluster Ⅳ中的3个突变材料。G Ⅴ包含32个突变材料,包括25个花突变材料,3个叶突变材料,1个种子突变材料,3个其他类型突变材料;包括聚类分析Cluster Ⅰ中的5个突变材料,Cluster Ⅳ中的26个突变材料,Cluster Ⅴ中的1个突变材料。总体来看,群体结构分析结果与聚类分析结果基本一致,但供试的突变材料还是没有按照叶突变、花突变、角果突变、种子突变、株型突变和其他突变类型聚类。
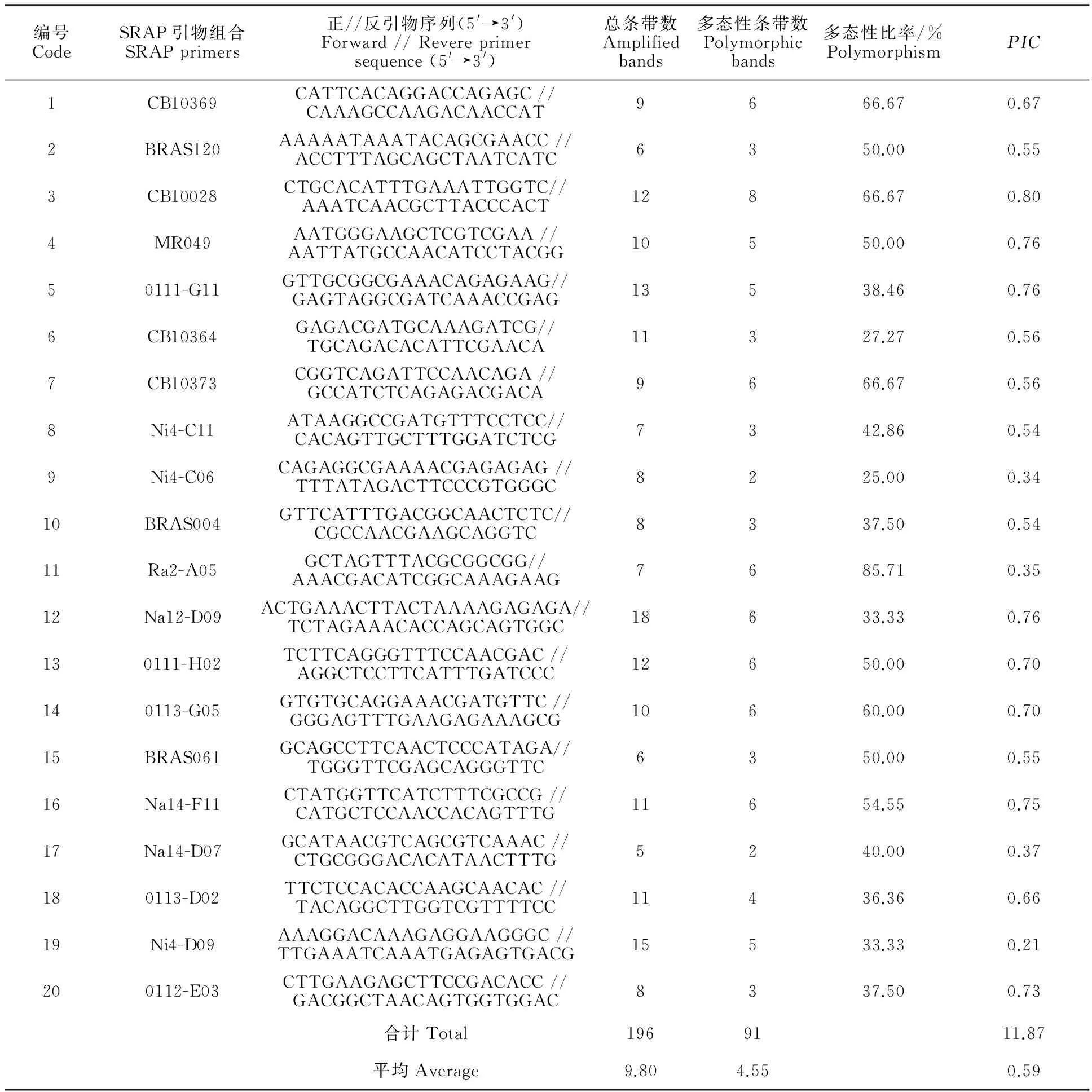
表3 SSR引物信息、扩增带数、多态性带数和PICTable 3 The PIC, total and polymorphic fragments per SSR primer
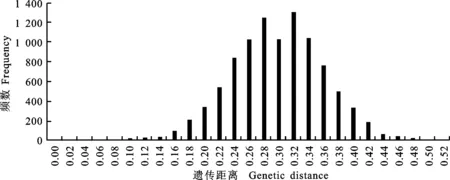
图1 试验材料间遗传距离次数分布直方图Fig.1 The frequency distribution of genetic distance between experimental materials
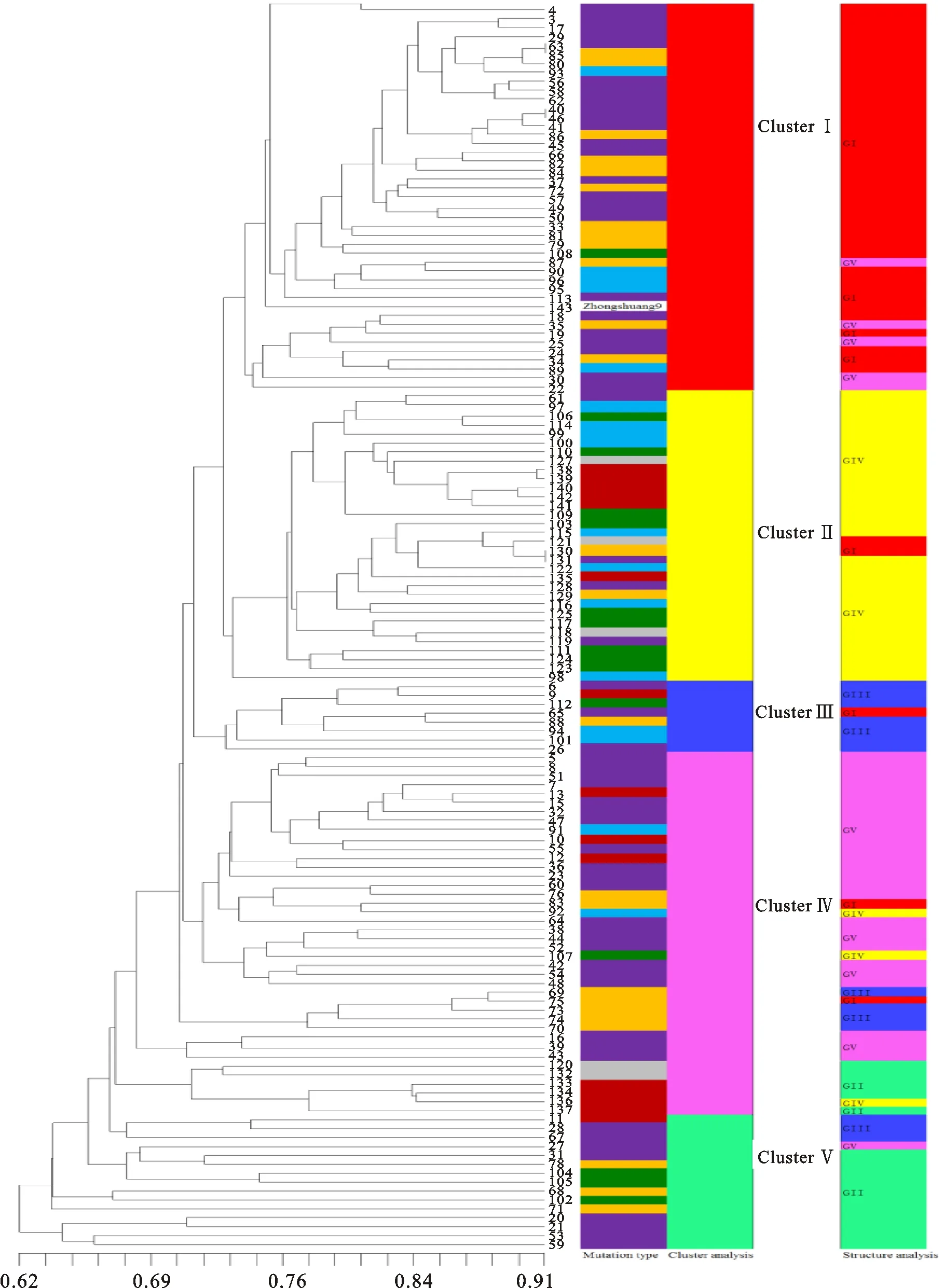
突变类型中紫色为花突变材料 Purple flower mutation;橙色为叶突变材料 Orange leaf mutation;浅蓝色为种子突变 Light blue seed mutation;深绿色为角果突变 Dark green silique mutation;灰色为株型突变材料 Gray plant type mutation;深红色为其他类型 Dark red other types of mutation
图2 基于 SRAP和SSR分子标记构建的139份试验材料的聚类图
Fig.2 Dendrogram of 139 materials generated by SSR and SRAP molecular markers
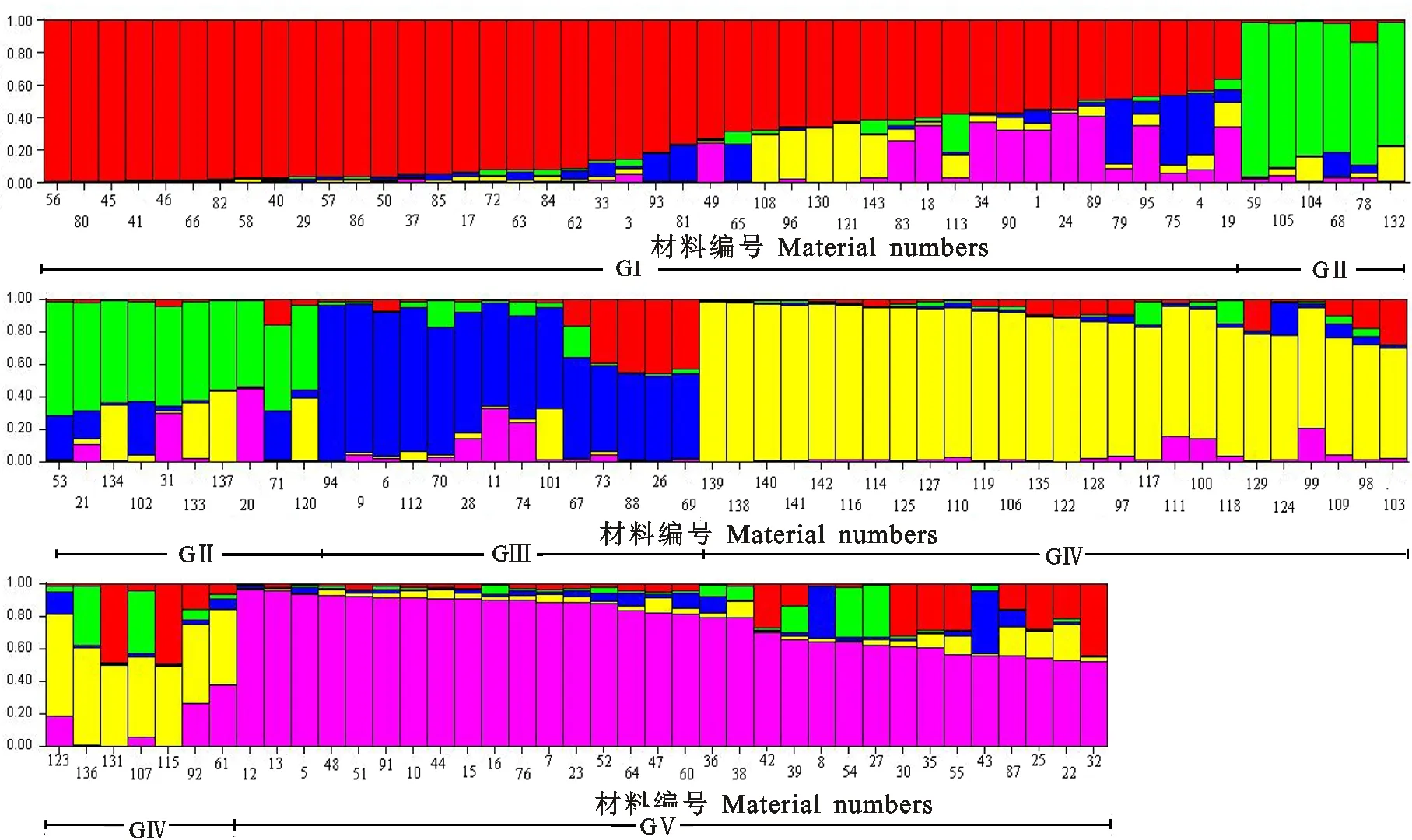
5种颜色代表5个组(GⅠ~GⅤ) Five colors represent five inferred groups;每个柱代表一个突变体 Each bar represents each accession;图中数字为材料编号 The numbers under each bar are the accession numbers as in table 1;每个柱中,不同颜色占柱的百分比,代表突变体遗传物质在该颜色所示组中的比例 The estimated genetic fraction of each accession of each inferred group was indicated in different colors.
图3 SSR和SRAP标记构建的139份试验材料的群体结构图(k=5)
Fig.3 Population structure of the 139 materials by SSR and SRAP molecular markers(k=5)
3 讨 论
与芸薹属其他物种相比,甘蓝型油菜遗传背景狭窄、遗传基础单一[15]。因此,利用理化诱变创造油菜新变异,拓宽其遗传基础,对丰富现有油菜资源具有非常重要的意义。本课题组利用体积分数为0.1% EMS溶液处理‘中双9号’油菜干种子,构建甘蓝型油菜EMS突变体库[6],创造了新的种质资源,分析这些新种质的分子遗传多样性对其在油菜育种中的合理利用具有指导意义。
本研究利用SRAP和SSR标记分析结果表明,138个EMS诱变M3株系与基础材料‘中双9号’的遗传相似系数为0.50~0.82,平均为0.70。Girija等[16]分别用EMS、γ射线、EMS-γ射线诱导豇豆品种‘CO7’,用RAPD标记分析M6代遗传差异,结果显示诱变材料与基础材料的遗传相似系数为0.58~0.71,平均为0.66。李红英等[17]用SSR标记分析玉米自交系AS-9经化学诱变获得的诱变系M1和M4的遗传差异,研究表明诱变系M1和M4与基础材料的遗传相似系数分别为0.32~0.41、0.38~0.48,平均值分别为0.36、0.43。石海春等[7]用EMS-石蜡诱变玉米自交系K305和R08,并用SSR标记分析9个K305诱变系和10个R08诱变系的遗传差异,结果显示,K305诱变系与K305的遗传相似系数为0.79~0.92,平均为0.86,R08诱变系与R08的遗传相似系数为0.71~0.88,平均为0.81。张娜等[18]用EMS处理燕麦品种‘白燕2号’,并分析M2突变体的遗传多样性,结果显示突变体间的遗传相似系数为0.67~0.97。上述结果表明, EMS诱变不同作物产生了分子水平上可检测到的变异,但变异范围因作物及诱变处理不同而异。
基于SRAP和SSR标记结果对139个参试材料进行聚类分析和遗传结构分析,结果显示138个突变体并没有按照突变表型而明显地聚在一起。一方面,不同基因突变可能导致同样的表型,例如前人在拟南芥中发现并报道了多种雄性不育,研究发现它们受控于不同的基因[19-23];另一方面,笔者在对突变体进行表型观察记载时,主要选取最明显直观的表型变异,但突变体间还存在其他变异,只是认为其应用价值不大或忽略记载。因此,导致EMS突变后代没按花、叶、种子、角果、株型突变等聚在一起。建议选择与基础材料遗传相似系数较小的突变材料进行深入研究,可望为油菜遗传改良提供变异更加丰富的材料。
Reference:
[1] 刘 翔.EMS诱变技术在植物育种中的研究进展[J].激光生物学报,2014(3):197-201.
LIU X.Progresses on EMS mutagenesis in plant breeding[J].ActalaserBiologySinica,2014(3):197-201(in Chinese with English abstract).
[2] BUS A,KORBER N,SNOWDONR J,etal.Patterns of molecular variation in a species-wide germplasm set ofBrassicanapus[J].TheoreticalandAppliedGenetics,2011,123(8):1413-1423.
[3] 孙加焱,涂进东,范叔味,等.甘蓝型油菜理化诱变和突变体库的构建[J].遗传,2007,29(4):475-482.
SUN J Y,TU J D,FAN SH W,etal.The screening of mutants induced by physical and chemical factors and construction of mutant population forBrassicanapusL.[J]Heredotas,2007,29(4):475-482(in Chinese with English abstract).
[4] 汪 念.甘蓝型油菜EMS突变体库的构建及TILLING、EcoTILLING技术的应用研究[D].武汉:华中农业大学,2009.
WANG N.Constructing an EMS mutant population and applicating TILLING,EcoTILLING technology inBrassicanapus[D].Wuhan:Huazhong Agriculture University,2009(in Chinese with English abstract).
[5] 石从广,孟华兵,姜宇晓,等.甘蓝型油菜EMS诱变二代农艺与籽粒品质性状的变异与TILLING库的构建[J].核农学报,2010,24(6):1132-1140.
SHI C G,MENG H B,JIANG Y X ,etal.Phenotypic variation of agronomic and grain quality traits in an EMS-induced M2population and the construction of the DNA pool for TILLING analysis[J].JournalofNuclearAgriculturalSciences,2010,24(6):1132-1140(in Chinese with English abstract).
[6] 曲高平,孙妍妍,庞红喜,等.甘蓝型油菜EMS突变体库构建及抗除草剂突变体筛选[J].中国油料作物学报,2014,36(1):25-31.
QU G P,SUN Y Y,PENG H X,etal.EMS mutagenesis and ALS-inhibitor herbicide-resistant mutants ofBrassicanapusL.[J].ChineseJournalofOilCropSciences,2014,36(1):25-31(in Chinese with English abstract).
[7] 石海春,谭义川,夏 伟,等.19份玉米EMS诱变系的遗传差异评价[J].华北农学报,2016,31(1):110-116.
SHI H CH,TAN Y CH,XIA W,etal.Evaluation on genetic differences of 19 maize mutant lines induced by EMS[J].ActaAgriculturaeBoreali-Sinica,2016,31(1):110-116(in Chinese with English abstract).
[8] 覃鸿妮,蔡一林,杨春蓉,等.玉米诱变系的SSR遗传变异分析[J].核农学报,2008(6):750-755,765.
QIN H N,CAI Y L,YANG CH R,etal.Genetic variation of maize(ZeamaysL.) mutants based on SSR analysis[J].JournalofNuclearAgriculturalSciences,2008(6):750-755,765(in Chinese with English abstract).
[9] 王长里.EMS诱导小麦M2代突变的鉴定[D].河北保定:河北农业大学,2008.
WANG CH L.Identification of wheat M2mutations induced by EMS [D].Baoding Hebei:Agricultural University of Hebei,2008(in Chinese with English abstract).
[10] 谢圣男,王宏光,杨 振,等.大豆绥农14突变体库构建及株高性状分析[J].核农学报,2013,27(3):307-313.
XIE SH N,WANG H G,YANG ZH ,etal.Construction of Suinong 14 mutant library and analysis of soybean height mutant[J].JournalofNuclearAgriculturalSciences,2013,27(3):307-313(in Chinese with English abstract).
[11] MURRAY M G,THOMPSON W F.Rapid isolation of high molecular weight plant DNA [J].NucleicAcidsResearch,1980,8(19):4321-4325.
[12] LI G,QUIROS C F.Sequence-related amplified polymorphism(SRAP)a new marker system based on a simple PCR reaction:its application to mapping and gene tagging inBrassica[J].TheoreticalandAppliedGenetics,2001,103(2):455-461.
[13] ROHLF F J.Numerical Taxonomy and Multivariate Analysis System Version 2.0 User Guide[M].Exeter Software Setauket,New York:1998.
[14] PRITCHARD J K,STEPHENS M,DONNELLY P.Inference of population structure using multilocus genotype data[J].Genetics,2000,7(4):574-578.
[15] 伍晓明,许 鲲,王汉中,等.甘蓝型油菜与新疆野生油菜属间杂种的获得与分子鉴定[J].中国油料作物学报,2002,24(4):5-9.
WU X M,XU K,WANG H ZH,etal.Obtained and molecular identification of interspecific hybrids betweenBrassicaand Xinjiang wild rape[J].ChineseJournalofOilCropSciences,2002,24(4):5-9(in Chinese).
[16] GIRIJA M,GNANAMURTHY S,DHANAVEL D.Genetic diversity analysis of cowpea mutant (VignaunguiculataL. Walp) as revealed by RAPD marker [J].InternationalJournalofAdvancedResearch,2013,1(4):139-147.
[17] 李红英,卢存福,兰小中,等.玉米自交系AS-9化学诱变后代SSR遗传变异分析[J].华北农学报,2013,28(3):92-101.
LI H Y,LU C F,LAN X ZH,etal.Genetic variation analysis in maize mutants from AS-9 inbread line based on SSR marker[J].ActaAgriculturaeBoreali-Sinica,2013,28(3):92-101 (in Chinese with English abstract).
[18] 张 娜,杨希文,任长忠,等.白燕2号EMS突变体的形态鉴定与遗传变异分析[J].麦类作物学报,2011,31(3):421-426.
ZHANG N,YANG X W,REN CH ZH,etal.Morphological identification and genetic variation analysis of EMS mutants from Hexaploid Oat (Avenasativa) cultivar Baiyan2[J].JournalofTriticeaeCrops,2011,31(3):421-426 (in Chinese with English abstract).
[19] AARTS M G,DIRKSE W G,STIEKEMA W J,etal.Transposon tagging of a male sterility gene inArabidopsis[J].Nature,1993,363(6431):715-717.
[20] SANDERS P M,WETERINGS B K.Anther developmental defects inArabidopsisthalianamale-sterile mutants[J].SexualPlantReproduction,1998,11(6):297-322.
[21] WILSON Z A,MORROLL S J,SWARUP R,etal.TheArabidopsisMALE STERILITY1 (MS1) gene is a transcriptional regulator of male gametogenesis,with homology to the PHD-finger family of transcription factors[J].PlantJournal,2001,28(1):27-39.
[22] STEINER-LANGE S,UNTE U S,ECKSTEIN L,etal.Disruption ofArabidopsisthalianaMYB26 results in male sterility due to non-dehiscent anthers.[J].PlantJournal,2003,34(4):519-528.
[23] ARIIZUMI T,HATAKEYAMA K,HINATA K R,etal.Disruption of the novel plant protein NEF1 affects lipid accumulation in the plastids of the tapetum and exine formation of pollen,resulting in male sterility inArabidopsisthaliana[J].PlantJournal,2004,39(2):170-181.
(责任编辑:潘学燕 Responsible editor:PAN Xueyan)
Molecular Genetic Diversity in EMS-induced Mutant Progenies inBrassicanapusL.
YUAN Runqin1,2,QU Gaoping1,2,GUO Yuan1,2and HU Shengwu1,2
(1. State Key Laboratory of Crop Stress Biology in Arid Areas,Yangling Shaanxi 712100,China;2. College of Agronomy,Northwest A&F University,Yangling Shaanxi 712100,China )
This study was designed to analyze genetic diversity of EMS-induced mutant progenies inBrassicanapusL. using simple sequence repeat (SSR) and sequence-related amplified polymorphism (SRAP) molecular markers. The selected 138 M3mutant strains ofB.napuscv.‘Zhongshuang No.9’ which served as the control,with abundant morphological variation in leaf,flower and silique organs, was analyzed by 24 SRAP and 20 SSR primer pairs. A total of 360 amplified fragments including 102 polymorphic fragments were detected by 24 SRAP primer combinations,with an average 28.33% of polymorphic fragment percentage and 0.60 of polymorphism information content (PIC) value;whereas,196 amplified fragments containing 91 polymorphic fragments were detected by 20 SSR primer pairs,with an average 46.43% of polymorphic fragment percentage and 0.59 ofPICvalue. The genetic similarity coefficient between 139 experimental materials varied from 0.62 to 0.91. The cluster and population structure analysis exhibited that experimental materials are not exactly classified depending on phenotype mutation. In general,EMS-induced mutations in ‘Zhongshuang No.9’ can be detected at the molecular level and there are extensive genetic differences among the mutant progenies.
BrassicanapusL.;EMS-induced mutant;Genetic diversity;SSR marker;SRAP marker
2016-05-06 Returned 2016-07-06
Earmarked Found for China Agriculture Research System(No.CARS-13).
YUAN Runqin,female,master student. Research area:rapeseed genetics and breeding. E-mail:15091184922@163.com
HU Shengwu,male,Ph.D,professor,doctoral supervisor. Research area:rapeseed genetics and breeding.E-mail:swhu83251@nwsuaf.edu.cn
日期:2017-06-05
2016-05-06
2016-07-06
现代农业产业技术体系建设专项资金(CARS-13)。 第一作者:袁润勤,女,硕士研究生,从事油菜遗传育种研究。E-mail:15091184922@163.com 通信作者:胡胜武,男,博士,教授,博士生导师,从事油菜遗传育种研究。E-mail:swhu83251@nwsuaf.edu.cn
S565.4
A
1004-1389(2017)06-0855-10
网络出版地址:http://kns.cnki.net/kcms/detail/61.1220.S.20170605.1715.016.html

