海南几种大型绿藻18S rRNA基因的克隆与序列分析
姜芳燕+黄海+杨宁+马军

摘 要 对海南沿岸采集到的团集刚毛藻(Cladophora glomerata)、细毛横丝藻(Acrochaete leptochaete)、石莼(Ulva reticulata)和长茎葡萄蕨藻(Caulerpa lentillifera)进行了形态学观察,采用18S rRNA通用引物,通过PCR技术,从这4种绿藻的基因组中扩增到各自的18S rRNA基因序列,并测序。与Genebank中已收录的6种绿藻18 S rRNA基因序列进行比较分析,所得10种绿藻的18S rRNA基因長度为727 bp,变异位点为266个,保守位点为453个。刚毛藻目与蕨藻目的遗传距离最大,为0.376;刚毛藻属内的种间遗传距离最小,为0.010。采用NJ法构建的系统进化树的拓扑结构显示:细毛横丝藻、葡枝横丝藻、石莼、礁膜、羽状尾孢藻和皱溪菜聚为一支;团集刚毛藻与扭曲刚毛藻单独聚为一支;长茎葡萄蕨藻与总状蕨藻聚为一支。说明18S rRNA序列可以作为大型绿藻系统发育研究的分子标记。
关键词 团集刚毛藻 ;细毛横丝藻 ;石莼 ;长茎葡萄蕨藻 ;18S rRNA
中图分类号 Q75 文献标识码 A Doi:10.12008/j.issn.1009-2196.2017.06.011
Cloning and Sequence Analysis of 18S rRNA Genes of
Several Large Green Algae in Hainan Province
JIANG Fangyan1) HUANG Hai1) YANG ning2) MA Jun1)
(1 Hainan Key Laboratory of Tropical Marine Fishery Resources Protection and Utilization,
Hainan Tropical Ocean University, Sanya, Hainan 572022;
2 Sanya Science & Technology Academy of Winter Breeding and Multiplication,
Sanya, Hainan 572000)
Abstract Large green algae Cladophora glomerata, Acrochaete leptochaete, Ulva reticulata, and Caulerpa lentillifera collected from the coast of Hainan province were observed morphologically. With 18S rRNA as universal primer, the genes from these 4 large green algae were amplified to 18 S rRNA gene sequence by PCR. A comparative analysis was made of 18S rRNA gene sequences among the 4 green algae selected and the 6 green algae from the Genebank. The results showed that the 18S rRNA gene sequences of these 10 green algae were 727 bp in length with 266 variable sites and 453 conserved sites. The maximum genetic distance between Cladophrales and Codiales was 0.376. The genetic distance among the species of genus Cladophora was 0.010, the shortest. The topology of phylogenetic tree constructed by NJ method suggested that A. leptochaete, A. repens, U. reticulata, M. nitidum, U. penicilliformis, and P. crispa were clustered into one group; C. glomerata and C. coelothrixwere into another group; C. lentillifera and C. racemosa into another group. This indicated that 18S rRNA sequence could be used as a molecular marker for phylogenetic research of large green algae.
Keywords Cladophora glomerata ; Acrochaete leptochaete ; Ulva reticulata ; Caulerpa lentillifera ; 18S rRNA.
大型海藻是一类重要的海洋生物资源,是海洋生态系统的重要组成部分,在海洋生态系统物质循环和维持海洋生态平衡中发挥着重要作用[1]。丁兰平等[2]在对中国南海多个地区开展野外调查的基础上,通过研究与历史资料整理发现,中国目前绿藻门海藻共计有11目21科48属211个种及变种,其中南海绿藻门有9目16科41属180种及变种、变型,其绿藻多样性占全国的85.31%,表明南海是中国绿藻多样性最丰富的地区。由于生长环境的不同,大型绿藻的藻体形态及结构存在着广泛的变异,因此,单从传统的形态学特征对其进行系统分类有一定的局限性。尤其是石莼目(Ulvales)礁膜科(Monostromaceae)的礁膜属(Monostroma)、石莼科(Ulvaceae)的石莼属(Ulva)和浒苔属(Enteromorpha)3个属[3],相关的物种分类一直存在诸多观点[4]。
20世纪90年代以来,随着分子生物学技术的迅速发展,分子标记技术被广泛应用于海洋藻类的分子鉴定和分子进化研究[5]。杨君等[6]采用RAPD技术对浒苔属藻类进行了初步的分子系统学研究,结果表明,缘管浒苔(Enteromorpha linza)与石莼属有较近的亲缘关系,应深入探究其分类地位。沈颂东和张劲[4]通过对绿藻5.8 S rRNA基因序列扩增及系统发育分析,表明相对于浒苔属的其他物种,浒苔与孔石莼的亲缘关系更近。张耀东等[7]利用18S rRNA序列探讨8种绿藻分子系统发育,研究发现在构建的系统发育树中,浒苔与石莼属物种聚为一类,表现出与石莼属物种更近的亲缘关系。刘岩等[8]利用ITS、rbcL等基因序列以及质体蓝素氨基酸序列,进一步分析了石莼科10种绿藻的序列结构特征和分子系统学关系。研究显示,石莼科浒苔属和石莼属绿藻rbcL和ITS序列差异,未体现出属间分化的差异,显示出在分子进化水平上较为一致的结果。本研究对海南沿岸采集到团集刚毛藻(Cladophora glomerata)、细毛横丝藻(Acrochaete leptochaete)、石莼(Ulva reticulata)和长茎葡萄蕨藻(Caulerpa lentillifera)进行形态学观察,并对这4种绿藻的18S rRNA基因序列进行PCR扩增和测序。然后,选取Genebank收录的6种绿藻的18S rRNA基因序列,以研究这一序列的基本特征,评估其种间变异程度,并探讨这一序列在种类鉴定和分子系统发育等研究中的应用价值,以期为它们的系统进化关系提供分子依据。
1 材料与方法
1.1 材料
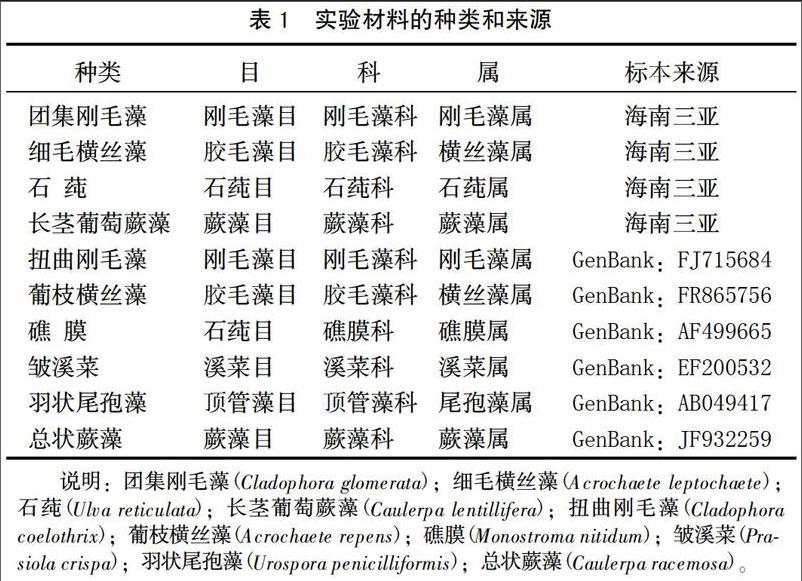
样本从海南省三亚市沿海潮间带采集,选择形态有一定差异且形态完整的藻体进行采集,然后帶回实验室洗净泥沙,编号封存。经形态学特征初步鉴定,所采集的藻样有团集刚毛藻、细毛横丝藻、石莼、长茎葡萄蕨藻。选取GenBank收录的6种常见大型绿藻的18S rRNA基因序列(见表1)。
1.2 方法
1.2.1 样品采集后处理
将采集回来的藻样分别装进1 000 mL大烧杯里,并进行编号。先用高压灭菌过的海水进行清洗,然后用小刷子把藻上多余的杂质进行刷洗,再用高压灭菌的双蒸水漂洗除去盐离子。其中,用于形态特征观察的藻样放在消毒灭菌的平板上,用SONY照相机对藻体的整体外观形态进行拍照。用于DNA提取的藻样,自然晾干后,用干净的封口袋封存,置于-20℃冰箱里备用。
1.2.2 主要分子操作
藻类总DNA提取和PCR产物回收等分子操作参照文献[9]进行。18S rRNA通用引物为18S-F(CCAACCTGGTTGATCCTGCCAGTA)和18S-R(CCTTGTTAACGACTTCACCTTCCTCT)。以4种绿藻基因组为模板,采用通用引物18S-F和18S-R进行PCR扩增,将扩增获得的DNA保守片段,送往深圳华大基因有限公司测序。
1.2.3 数据分析
用Clustal X1.83软件对团集刚毛藻(C. glomerata)、细毛横丝藻(A. leptochaete)、石莼(U. reticulata)、长茎葡萄蕨藻(C. lentillifera)以及从Genebank下载的羽状尾孢藻(U. penicilliformis)、礁膜(M. nitidum)、皱溪菜(P. crispa)、香蕉菜(B. forbesii)、总状蕨藻(C. racemosa)、盾叶蕨藻(C. peltata)的18S rRNA基因序列进行排序,然后用MEGA 7.0软件中的Pairwisedistance工具计算其相对遗传距离,并采用邻接法(Neighbour-Joining,NJ)构建分子进化树,置信度用Bootstrap工具循环1 000次表示检验分子系统树[10]。
2 结果与分析
2.1 4种绿藻的形态学特征
根据观察所采集藻样的形态特征,并查阅相关藻类文献和图片资料,结果表明,所采集的4种藻样分别鉴定为团集刚毛藻、细毛横丝藻、石莼和长茎葡萄蕨藻(图1)。
团集刚毛藻(Cladophora glomerata)属绿藻门(Chlorophyta),刚毛藻目(Cladophrales),刚毛藻科(Cladophoraceae),刚毛藻属(Cladophora)。观察其外观是比较粗糙,分枝规则的丝状体被横隔壁分成多核的节段,常生成簇状或球状,长可达10 cm以上(图1-A)。其基部细胞有假根、或呈假根状,其他细胞呈柱状;植物体呈多细胞分枝的丝状体,在基质上附着生长。
细毛横丝藻(Acrochaete leptochaete)属绿藻门(Chlorophyta),胶毛藻目(Chaetophorales),胶毛藻科(Chaetophoraceae),横丝藻属(Acrochaete)。细毛横丝藻外观深绿色,丝状,藻体呈鲜绿或暗绿色(图1-B),质软,为不分枝的丝状体,外形很像一丛绿绒毛,丝径10~25 μm。藻体是由圆筒状细胞之间相连而成的单列、不分枝的丝状体。大多数都生长在流动的淡水域中,有少数生长在较急的水流中,有小部分种类分布于海水中。
石莼(Ulva reticulata)属绿藻门(Chlorophyta),石莼目(Ulvales),石莼科(Ulvaceae),石莼属(Ulva)。藻体宽而薄,呈片状,颜色呈鲜绿色或黄绿色(图1-C),是由两层细胞构成的绿色膜状体,其长度可达40 cm以上。借基部多细胞的固着器固着在岩石上。
长茎葡萄蕨藻(Caulerpa lentillifera)属绿藻门(Chlorophyta),蕨藻目(Codiales),蕨藻科(Caulerpaceae),蕨藻属(Caulerpa)。藻体呈鲜绿色,匍匐蔓生,有直立茎及匍匐茎之分化,直立茎高约3~8 cm,长出许多密生的小枝,小枝顶端膨大成圆球形,直径约0.1~0.15 cm。匍匐茎圆柱状,平滑,向下长出须状假根。匍匐茎圆柱状,平滑,约0.6~1.0 mm宽,向下长出须状假根(图1-D)。藻体假根必须附着于一定的基质[11]。
2.2 18S rRNA基因序列分析
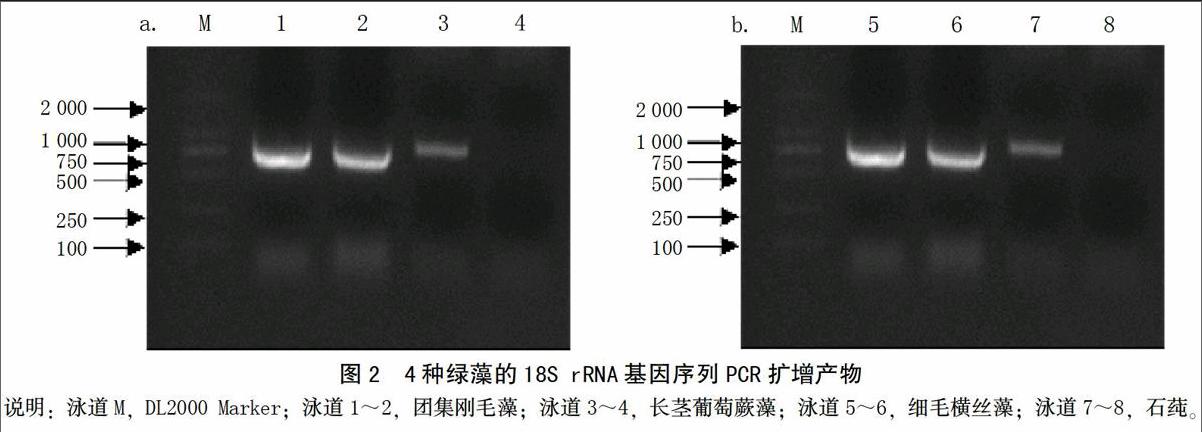
团集刚毛藻、细毛横丝藻、石莼和长茎葡萄蕨藻的18S rRNA基因PCR扩增结果如图2所示。测序结果显示,其基因序列长度约为750 bp。选取Genebank收录的6种绿藻18S rRNA基因序列,对比分析它们之间的亲缘关系。利用MEGA 7.0软件对基因序列间保守位点,变异位点,碱基A、T、C、G的平均含量,遗传距离进行统计分析。结果表明:比对后的10种基因长度为727 bp,共检测到266个核苷酸变异位点,保守位点为453个,其中简约性信息位点为250个。碱基组成T为25.3%,C为20.9%,A为25.9%,G为27.9%,A+T的含量为51.2%,C+G的含量为48.8%,说明碱基组成没有明显的偏向性。
基于Kimura 2-Parameter模型利用MEGA 7.0软件计算出各种之间的遗传距离见(表2)。在这10种藻类中,其中遗传距离最小的是团集刚毛藻与扭曲刚毛藻,为0.010;其次是羽状尾孢藻与礁膜,为0.016,在序列中只有几个碱基的差异。而团集刚毛藻与总状蕨藻的遗传距离最大,其遗传距离为0.376,其次是扭曲刚毛藻与总状蕨藻,其遗传距离为0.374。刚毛藻属内的种间遗传距离最小,为0.010。横丝藻属内的种间遗传距离为0.019,蕨藻属内的种间遗传距离为0.025。石莼目中的石莼和礁膜之间的遗传距离为0.071,而礁膜与顶管藻目中羽状尾孢藻的遗传距离为0.016,礁膜与溪菜目中皱溪菜的遗传距离为0.068,均低于石莼和礁膜之间的遗传距离,说明18 S rRNA分析结果与传统分类结果(表1)仍有一定的分歧。
2.3 系统发育树的构建
基于所得的18S rRNA基因部分序列,采用NJ法构建的系统进化树,如图3所示。其拓扑结构显示,10种藻类利用NJ法所构建的进化树大致形成三分支。细毛横丝藻、葡枝横丝藻、石莼、礁膜、羽状尾孢藻和皱溪菜聚为一支;团集刚毛藻与扭曲刚毛藻单独聚为一支;长茎葡萄蕨藻与总状蕨藻聚为一支。其中,第一分支结果显示,石莼目中的礁膜与顶管藻目的羽状尾孢藻的亲缘关系较近,单独聚为一小分支,而同为石莼目的石莼和礁膜并没有聚为一小分支,相对亲缘关系较远;与核苷酸序列间Kimura双参数距离(表2)结果较一致。大部分的节点自检支持率较高,较清楚地揭示出他们的分化顺序及遗传距离关系。
3 讨论
本研究所涉及的刚毛藻目海藻藻体结构相对简单,易受环境影响,难以通过形态特征加以鉴定。滕林宏[12]通过对31个不同样品的形态观察比较,鉴定了中国海产刚毛藻目3属16个物种。然而,其利用具有高度变异性的ITS序列及相对保守的18 S rDNA序列对上述31个藻样进行分子鉴定,确认了它们应划分为14种及1变种,说明传统的形态特征分类与分子鉴定有一定的分歧。目前对淡水刚毛藻的生态方面有比较全面的研究,而对海产刚毛藻的研究报道较少[12]。本研究的团集刚毛藻,从海南省三亚市沿海潮间带采集获得,其外观较粗糙,分枝规则,基部细胞有假根或呈假根状,单从形态特征鉴定易与其它种混淆。Gubelit[13]调查通过俄罗斯涅瓦海湾的底栖和漂浮的团集刚毛藻的生物量和初级生产力,发现其漂浮种占70%。而传统分类学认为团集刚毛藻(C. glomerata)应为底栖,而脆弱刚毛藻(C. fracta)则为漂浮。但是,Marks和Cummings研究发现,团集刚毛藻和脆弱刚毛藻有非常相似的ITS序列。因此,仅通过观察形态特征和生活方式,区分难度大,有必要结合其他方法辅助形态鉴定。
细毛横丝藻(Acrochaete leptochaete),1892年最早由Huber发现命名。长期以来,有关该藻的分类一直没有定论。先后将细毛横丝藻划分至内皮藻属(Endoderma)、褐友藻属(Phaeophila)、横丝藻属(Acrochaete)和內枝藻属(Entocladia)[15]。直至2007年,Brodie等[16]研究发现,内枝藻属与横丝藻属系同物异名,因此仍沿用Acrochaete leptochaete为其命名。细毛横丝藻常见寄生于刚毛藻属和硬毛藻属物种的细胞外壁,主要生长在海滨地区或是近海地区。目前已报道在欧洲、非洲和印度发现。邓蕴彦[15]在研究硬毛藻及其附生藻的实验分类学时发现,硬毛藻的附生藻为细毛横丝藻,是中国的新记录种,该藻样由山东荣成采集获得。本文研究的细毛横丝藻从海南省三亚市沿海潮间带采集获得,并首次报道了中国南海海域存在细毛横丝藻,但是否为新种还有待进一步研究。
本文通过对几种常见的18 S rRNA基因序列进行分析,从而确定绿藻藻种分类的可信度。根据所建立的系统进化树,说明18 S rRNA序列适合作为大型绿藻系统发育研究的分子标记。
参考文献
[1] 安鑫龙,李雪梅. 大型海藻对近海环境的生态作用[C]. 庆祝中国藻类学会成立30周年暨第十五次学术讨论会摘要集,2009.
[2] 丁兰平,王旭雷,陈善文,等. 南中国海大型绿藻的多样性及刺松藻的环境适应性初步研究[C]. 中国生态学学会海洋生态专业委员会2012年学术年会论文集,2012.
[3] 张学成,马家海,秦 松. 海藻遗传学[M]. 北京:中国农业出版社,2005:108-124.
[4] 沈颂东,张 劲. 大型海洋绿藻5.8 S rRNA基因序列及系统发育分析[J]. 海洋与湖沼,2008,39(4):427-432.
[5] Inagaki Y, Hayashi I Y, Ehara M, et al. Algae or protozoa:phylogenetic position of euglenophytes and dinoflagellates as inferred from mitochondrial sequences[J]. J Mol Evol, 1997, 45(3): 295-300.
[6] 杨 君,安利佳,康晓慧. 石莼属(Ulva)和浒苔属(Enteromorpha)绿藻的RAPD分析[J]. 海洋与湖沼,2000,31(4):408-413.
[7] 张耀东,沈颂东,林阿朋,等. 用 18 S rRNA 序列探讨8种绿藻分子系统发育[J]. 大连水产学院学报,2009,24(1):76-78.
[8] 刘 岩,刘 涛,宫庆礼,等. 常见石莼科绿藻的生物学特征及分子系统学分析[J]. 中国海洋大学学报,2010,40(12):71-80.
[9] 王浩东,张 磊,刘 涛,等. 基于rbcL序列和ITS序列分析的浒苔质体DNA双亲遗传模式证据[J]. 海洋湖沼通报,2013,3:101-107.
[10] Kumar S, Stecher G, Tamura K. MEGA 7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets [J]. Molecular Biology and Evolution, 2016, 33: 1 870-1 874.
[11] 姜芳燕,宋文明,黄 海,等. 长茎葡萄蕨藻的人工养殖技术研究[J]. 热带农业科学,2014,34(8):99-103.
[12] 滕林宏. 中國刚毛藻目海藻的形态及系统发育学研究—兼DN A条形码技术在刚毛藻目的应用初探[D]. 青岛:中国科学院海洋研究所,2011.
[13] Gu belit Y I. Biomass and primary production of Cladophora glomerata (L.) Kutz. in the Neva Estuary [J]. Inland Water Biology, 2009, 2: 300-304.
[14] Marks J C,Cummings M P. DNA sequence variation in the ribosomal internal transcribed spacer region of fresh water Cladophora species (Chlorophyta) [J]. Journal of Phycology, 1996, 32: 1 035-1 042.
[15] 邓蕴彦. 硬毛藻及其附生藻的实验分类学[D]. 青岛:中国科学院海洋研究所,2010.
[16] Brodie J, Maggs C A, John D M. Green seaweeds of Britain and Ireland[M]. London: British Phycologycal Society, 2007: 242.

