嗜热链球菌KLDSSM胞外多糖生物合成途径的分析
,,,
(东北农业大学乳品科学教育部重点实验室,黑龙江哈尔滨 150030)
嗜热链球菌KLDSSM胞外多糖生物合成途径的分析
丁秀云,蒙月月,李柏良,霍贵成*
(东北农业大学乳品科学教育部重点实验室,黑龙江哈尔滨 150030)
为了从遗传水平上探究嗜热链球菌KLDS SM合成胞外多糖(EPS)的能力,本研究从糖被转运进入胞内、糖核苷酸合成及EPS基因簇三方面所涉及的基因进行了相关分析。结果发现,该菌能够转运葡萄糖、乳糖、蔗糖、海藻糖及甘露糖5种糖,具有代谢葡萄糖、乳糖、果糖、蔗糖、海藻糖与甘露糖形成糖酵解中间代谢产物的酶类,拥有合成EPS前体物质UDP-葡萄糖、UDP-半乳糖、UDP-N-乙酰基葡糖胺、UDP-呋喃半乳糖与dTDP-鼠李糖的潜力。同时序列比对分析发现该菌具有一个与高产EPS的菌株ASCC 1275一致性极高的EPS基因簇,且EPS初步产量测定约为331 mg/mL,表明该菌是一株高产EPS潜力菌株,具有较大的应用潜能。
嗜热链球菌KLDS SM,糖转运系统,糖核苷酸,EPS基因簇,生物信息分析
作为一种重要的乳品发酵剂,嗜热链球菌常与保加利亚乳杆菌应用于工业乳制品生产中,如酸奶与奶酪[1-2]。一些嗜热链球菌可以产生胞外多糖(EPS),EPS是菌体在生长代谢中分泌到培养基中的一类多糖类化合物,分子量多在104~6.0×106之间[3-4],为微生物次级代谢产物。EPS可作为增粘剂、稳定剂、乳化剂、胶凝剂应用于食品工业中,改善发酵乳制品的持水性、质构、口感及流变性[5-7],某些EPS还有抗肿瘤[8],降低胆固醇[9],调节免疫系统[10-11],抗氧化[12-13],为肠道菌群提供底物[5],抗致病性微生物[14]等多种生理功能。多糖的生物合成是一个需能的过程,乳酸菌产能有限,很大程度上限制了EPS的产量。相对于保加利亚乳杆菌,嗜热链球菌所产生的EPS对酸奶品质的影响占主导地位。EPS的生物合成过程分为前体糖核苷酸的合成及EPS基因簇指导重复单元合成、链长、聚合与输出。嗜热链球菌产生的杂多糖主要由D-半乳糖、D-葡萄糖和L-鼠李糖聚合而成。
嗜热链球菌KLDS SM分离于中国内蒙古传统发酵酸奶。实验发现该菌可以快速产酸,缩短凝乳时间,并且高产黏。为了从基因水平上分析该菌产生EPS的潜力,对该菌进行了全基因组测序,分析其胞外多糖合成的相关基因。
1 材料与方法
1.1材料与仪器
嗜热链球菌KLDS SM 东北农业大学乳品科学教育部重点实验室工业微生物菌种保藏中心(KLDS-DICC);M17肉汤培养基、细菌基因组提取试剂盒 北京天根生物技术有限公司;脱脂乳粉 Nordmilchag公司;溶菌酶、琼脂粉、透析袋 Solarbio公司;葡萄糖 美国Sigma公司;无水乙醇 天津市天力化学试剂有限公司;三氯乙酸 天津市大茂化学试剂厂;重蒸苯酚 北京博奥拓达科技有限公司;浓硫酸 哈尔滨理工化学试剂有限公司。
LDZF-50KB-Ⅱ立式蒸汽灭菌器 上海申安医疗器械厂;CJ-2D超净工作台 天津泰斯特仪器有限公司;DHP-927型电热恒温培养箱 上海一恒科技有限公司;GL-20G-II离心机 上海安亭科学仪器厂;GL-21M离心机 上海市离心机械研究所;DYY-10C电泳仪 北京六一仪器厂。
1.2实验方法
1.2.1 基因组DNA提取 将冻干保藏的嗜热链球菌KLDS SM以1%(v/v)接种至M17液体培养基中培养24 h以活化菌体,两次转接至M17液体培养基后提取基因组,提取的步骤按照细菌基因组提取试剂盒说明书进行。取5 μL DNA样品进行1%琼脂糖凝胶电泳,检测基因组提取质量及完整性。
1.2.2 基因组的测序、组装与注释 基因组的测序与组装由深圳华大基因科技服务有限公司完成。嗜热链球菌KLDS SM全基因组采用二代Illumina Hiseq 2500(500 bp,PE125)与三代Pacbio RSII(20 K)平台联合测序。过滤除去低质量reads。使用SMRT Analysis v2.3.0流程中的RS_HGAP_Assembly3[15]软件将PacBio数据中的reads从头组装成完整的连续的contig。基于Illumina Hiseq 2500数据,采用GATK分析流程对contig进行第一轮单碱基纠错,SOAPsnp v1.05[16]与SOAPindel v1.08对第一轮结果进行第二轮纠错。Contig成环判断得到完成图水平的完整序列。采用NCBI原核基因组注释流程(PGAP,http://www.ncbi.nlm.nih.gov/books/NBK174280)进行全基因组注释。
1.2.3 生物信息分析 PTS转运系统、ABC型糖转运系统、糖代谢形成糖酵解中间产物及糖核苷酸合成的路径参照KEGG pathway数据库(http://www.genome.jp/kegg/pathway.html),本地Blastp确认路径中涉及基因在该菌的存在情况。下载epsA基因序列,建库,本地Blastp寻找EPS基因簇5′端高度保守的epsA,确认基因簇的位置。Blastx(nr)对基因簇上的各基因进行人工校正,以最优比对作为相应基因的注释。基因组圈图由CGView Server[17]构建。ISfinder[18]用来分析EPS基因簇中存在的转座酶。分析中所涉及的基因的氨基酸序列从Uniprot数据库获得。目前13株已完成全基因组测序的嗜热链球菌:LMG 18311(CP000023.1),S9(CP013939.1),CNRZ1066(CP000024.1),LMD-9(CP000419.1),ND03(CP002340.1),JIM 8232(FR875178.1),MN-ZLW-002(CP003499.1),ASCC 1275(CP006819.1),SMQ-301(CP011217.1),MN-BM-A02(CP010999.1),MN-BM-A01(CP012588.1),CS8(CP016439.1),KLDS 3.1003(CP016877.1)。基因组序列在NCBI(ftp)下载。同样确定EPS基因簇序列,与菌株KLDS SM的序列比较分析(megablast)。
1.2.4 EPS的提取及测定 绘制葡萄糖标准曲线:取葡萄糖适量,(105±5)℃烘干至恒重。精确称取0.06 g,定容至100 mL。分别取0、5、10、15、20、25、30、35、40 μL于EP管中,加蒸馏水定容至60 μL。采用苯酚硫酸法[19]测定糖含量,步骤参照姜琼等[20]的方法,在标准液中加入180 μL由5%苯酚与98%浓硫酸以1∶5体积比配成的显色液,沸水浴25 min后用酶标仪测定490 nm处的吸光度,每个样品重复3次,绘制葡萄糖标准曲线。
EPS的提取及测定:将嗜热链球菌KLDS SM在M17肉汤培养基中培养24 h,传代培养18 h后以4%(v/v)接种量接种至脱脂乳中,42 ℃发酵,凝乳后放于4 ℃。接种24 h后提取粗多糖,步骤参照Shao[21]的方法,稍有改动:发酵乳经沸水浴10 min,冷却后于4 ℃ 9000 r/min离心20 min,去除沉淀取上清,加入80%(w/v)三氯乙酸至7%(w/v)沉淀蛋白,4 ℃放置10 h,4 ℃ 9000 r/min离心20 min后取上清,减压浓缩至原体积的1/3,加入三倍体积的无水乙醇,4 ℃放置24 h后于4 ℃ 9000 r/min离心30 min,去离子水溶解沉淀,装于透析袋(7 kDa)内于4 ℃透析3 d,每天换3次水。苯酚硫酸法测定EPS的含量(校正系数0.9)。
1.3数据处理
实验中每个样品生物学重复三次,采用Excel软件绘制葡萄糖标准曲线。
2 结果与分析
2.1嗜热链球菌KLDS SM基因组的一般特征
嗜热练球菌KLDS SM全基因组序列上传到Genbank数据库,登录号为:CP016026。嗜热链球菌KLDS SM基因组由一个1856787bp的环状染色体组成,G+C含量为39.08%,构建的基因组圈图如图1所示。同绝大多数已测序的嗜热链球菌一样,KLDS SM基因组中并不存在质粒(http://www.ncbi.nlm.nih.gov/genome/genomes/420?)。共预测出1950个基因,由1732个蛋白编码基因,18个rRNA基因,67个tRNA基因,4个ncRNA以及129个假基因组成。

图1 嗜热链球菌KLDS SM基因组圈图Fig.1 Circular genome map of Streptococcus thermophilus KLDS SM注:从外至里,第1和第2圈分别表示正负链上的基因,包括CDS、rRNA、tRNA及其他基因;第3圈表示GC含量;第4圈表示GC skew,浅灰色代表G-C>0的区域,深灰色代表G-C<0的区域,浅灰色与深灰色交界处分别是复制起始点与终止点。
2.2糖转运系统及糖核苷酸的合成

图2 嗜热链球菌KLDS SM糖核苷酸的合成Fig.2 Synthesis of sugar nucleotides of Streptococcus thermophilus KLDS SM
2.2.1 糖转运系统 糖类物质的转运方式一般有三种:磷酸转移酶系统(PTS)、ABC型糖转运系统以及糖渗透酶[22]。其中PTS为主要的转运方式,其组成为:磷酸载体蛋白HPr和EI,负责糖类物质的磷酸化;具有底物特异性的EII,通常由IIA、IIB与IIC(甘露糖存在IID)构成。Blastp分析表明,嗜热链球菌KLDS SM基因组中有一个编码HPr(A9497_02385)和EI(A9497_02380)组成的操纵元。10个PTS系统相关的EII,分别为转运蔗糖的IIABC(A9497_04525),葡萄糖的IIA(A9497_07135)、IIBC(A9497_07130;A9497_07130),果糖的IIA(A9497_02785)、IIABC(A9497_08150),甘露糖的EIIAB(A9497_07770)、IIC(A9497_07765)、IID(A9497_07760),海藻糖的IIA(A9497_07135)、IIBC(A9497_05305)。其中基因(A9497_08150)因缺失而失去转运果糖的功能。ABC型糖转运系统由两个底物结合蛋白、两个渗透酶与一个ATP结合蛋白组成,转运过程消耗ATP。菌株KLDS SM基因组上只有2个基因(A9497_00495;A9497_00205)分别编码ABC型糖转运系统的渗透酶及ATP结合蛋白,无法转运糖类物质。除此之外,菌株KLDS SM有一编码乳糖渗透酶的基因(A9497_02960),用于乳糖的转运,基因组上未发现其他糖渗透酶。
2.2.2 糖核苷酸的合成 嗜热链球菌KLDS SM的糖核苷酸合成途径如图2所示,涉及的酶列于表1。分析表明菌株KLDS SM有利用葡萄糖、乳糖、蔗糖、果糖、海藻糖与甘露糖相关的基因,同时有合成EPS前体糖核苷酸UDP-葡萄糖、UDP-半乳糖、UDP-N-乙酰基葡糖胺、UDP-呋喃半乳糖与dTDP-鼠李糖的潜力。该菌有Leloir途径相关的酶,可以利用半乳糖形成葡萄糖-1-磷酸。在合成过程中,不同的酶在染色体上拷贝数不同。有趣的是,转运进入细胞的糖转化成糖酵解的中间产物相应的酶拷贝数较多。海藻糖-6-磷酸水解酶水解海藻糖-6-磷酸生成葡萄糖与葡萄糖-6-磷酸,在染色体上有2个拷贝。甘露糖-6-磷酸异构酶可以转化甘露糖-6-磷酸为果糖-6-磷酸,高达3个拷贝。推断这种多拷贝现象可能利于细胞快速、高效利用糖类物质。同时,作用于UDP-葡萄糖与UDP-半乳糖相互转变的尿苷二磷酸葡萄糖4-表异构酶高达3个拷贝,利于二者的快速转变,以供重复单元合成过程中对其需求。
2.3 EPS基因簇生物信息分析
比对发现,菌株KLDS SM染色体上存在一个合成EPS的基因簇,如表2与图3所示,该基因簇由22个基因组成(A9497_01480-A9497_01580)。基因簇中epsA-D(A9497_01565 - A9497_01580)编码与EPS生物合成调控、链长决定及输出相关的高度保守基因。epsE(A9497_01560)编码重复单元形成的起始引导糖基转移酶,负责将UDP-半乳糖转移到异戊二烯醇磷酸酯(脂载体)上,同时基因(A9497_01520、A9497_01535、A9497_01545、A9497_01555*、A9497_01555)编码糖基转移酶,转移相应的糖核苷酸指导EPS重复单元的形成。其中A9497_01555*与A9497_01555因截短成为假基因无法发挥转移酶功能,比对发现A9497_01555*与乳酸乳球菌的基因一致性很高,且其下游编码ISLll1转座酶(IS982家族)的A9497_01550与乳酸乳球菌的转座酶同源,推断该糖基转移酶由转座酶转移获得,插入A9497_01555使其成为假基因。A9497_01550在进化过程中因缺失成为假基因,表明基因在较早时期转移至该基因簇上。A9497_01525与A9497_01530与链长决定相关基因A9497_01565和A9497_01570有相似的功能结构域,同样因为截短失去功能,且两者同源于植物乳杆菌的基因。基因A9497_01510与A9497_01515编码翻转酶参与重复单元的输出,A9497_01510因移码突变而失去功能。

表1 嗜热链球菌KLDS SM糖核苷酸合成相关的酶Table 1 Enzymes related to sugar nucleotide synthesis of Streptococcus thermophilus KLDS SM
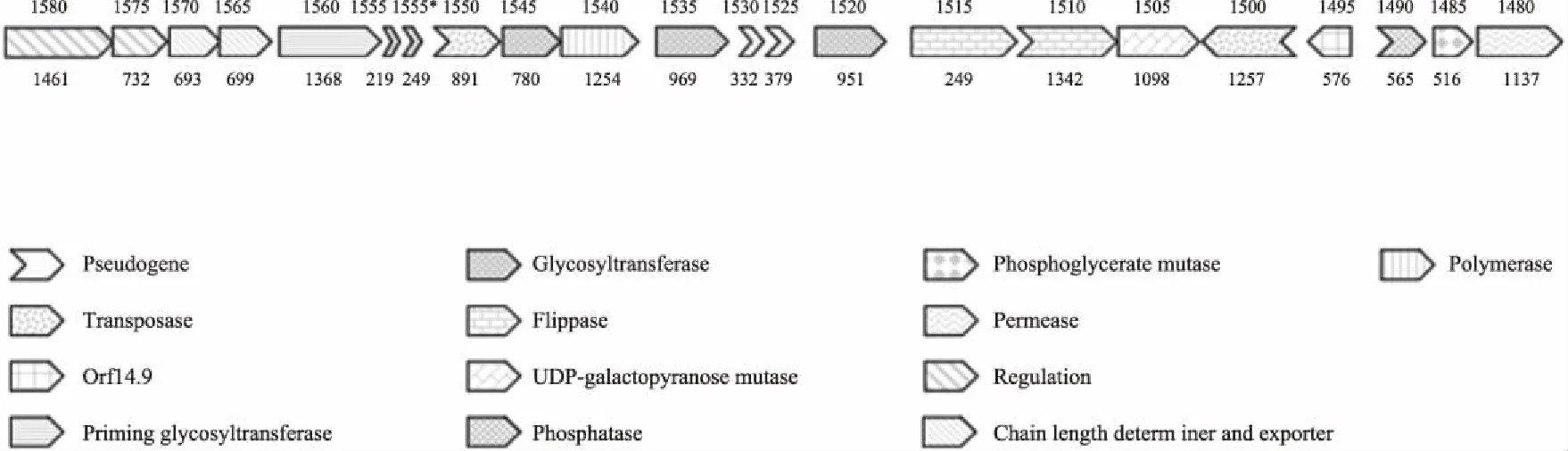
图3 嗜热链球菌KLDS SM EPS基因簇Fig.3 EPS gene cluster of Streptococcus thermophilus KLDS SM注:从5′(左)到3′(右),编码不同产物的基因用不同的图案表示。基因位点与长度(bp)标记于基因上下。

表2 嗜热链球菌KLDS SM EPS基因簇Table 2 EPS gene cluster of Streptococcus thermophilus KLDS SM
注:已用Blastx校正基因簇中的各基因。“*”为校正后出现的基因。基因A9497_01505编码UDP-吡喃半乳糖变位酶负责UDP-吡喃半乳糖的形成。基因簇中的另一个转座酶A9497_01500为ISL3家族的ISSmu2转座酶,同样因基因突变失去功能。基因A9497_01495编码与嗜热链球菌生长相关的ORF14.9[23]。基因A9497_01490 - A9497_01480分别编码磷酸酶(假基因)、磷酸甘油酸变位酶和渗透酶。在EPS基因簇5′上游同样发现编码嘌呤核苷磷酸化酶的高度保守基因deoD(A9497_01590)[23-24]。同时在deoD与epsA之间存在一个反向的IS256家族的IS1191转座酶。
嗜热链球菌KLDS SM EPS基因簇与13株已测序的嗜热链球菌的EPS序列比对(megablast),发现其与菌株ASCC 1275、MN-BM-A02核苷酸序列的一致性最高,而菌株ASCC 1275高产EPS[25]。推测菌株KLDS SM是一株高产EPS的菌株。
2.4 EPS含量测定
以葡萄糖(0.6 mg/mL)作为标准品绘制标准曲线如图4所示。线性回归方程为y=0.0046x-0.0083,R2=0.9981,线性关系良好。经苯酚硫酸法测定菌株KLDS SM中粗多糖总量约为331 mg/mL。

图4 葡萄糖标准曲线Fig.4 Glucose standard curve
3 结论
通过生物信息分析嗜热链球菌KLDS SM的糖转运情况,该菌主要通过PTS系统特异性的转运葡萄糖、蔗糖、海藻糖与甘露糖。仅存在一种糖渗透酶,即乳糖渗透酶转运乳糖。不存在完整的ABC型糖转运系统,可能因为该系统消耗ATP不适合该菌的生理代谢。糖核苷酸合成分析中,发现该菌有利用葡萄糖、乳糖、果糖、蔗糖、海藻糖与甘露糖进入中心代谢的酶类,并且海藻糖与甘露糖代谢相关的酶类有多个拷贝,推测其与糖类物质快速、高效进入碳代谢及能量代谢有关。基因组分析表明该菌有合成EPS的前体物质UDP-葡萄糖、UDP-半乳糖、UDP-N-乙酰基葡糖胺、UDP-呋喃半乳糖与dTDP-鼠李糖的潜力。同时在菌株KLDS SM染色体上存在一个指导EPS生物合成的基因簇,其上特异性的糖基转移酶指导重复单元的形成。EPS序列比对发现菌株KLDS SM与菌株ASCC 1275、MN-BM-A02基因簇高度相似,而菌株ASCC 1275高产EPS,同时初步测定菌株KLDS SM的粗多糖产量约为331 mg/mL,可以推测该菌是一株高产EPS的潜力菌株。嗜热链球菌产生的EPS可以作为天然的食品增稠剂,可提高发酵乳制品的品质,某些类型的EPS有益生作用。因此在后续研究中优化培养条件提高EPS的产量,研究EPS的组成、结构及调控是非常有意义的。
[1]Liu M,Siezen R J,Nauta A. In silico prediction of horizontal gene transfer events inLactobacillusbulgaricusandStreptococcusthermophilusreveals protocooperation in yogurt manufacturing[J]. Applied and Environmental Microbiology,2009,75(12):4120-4129.
[2]Settachaimongkon S,Nout M J R,Antunes Fernandes E C,et al. Influence of different proteolytic strains ofStreptococcusthermophilusin co-culture withLactobacillusdelbrueckiisubsp. bulgaricus on the metabolite profile of set-yoghurt[J]. International Journal of Food Microbiology,2014,177:29-36.
[3]De Vuyst L,De Vin F,Vaningelgem F,et al. Recent developments in the biosynthesis and applications of heteropolysaccharides from lactic acid bacteria[J]. International Dairy Journal,2001,11(9):687-707.
[4]Hassan A N. ADSA foundation scholar award-possibilities and challenges of exopolysaccharide-producing lactic cultures in dairy foods[J]. Journal of Dairy Science,2008,91(4):1282-1298.
[5]Caggianiello G,Kleerebezem M,Spano G. Exopolysaccharides produced by lactic acid bacteria-from health-promoting benefits to stress tolerance mechanisms[J]. Applied Microbiology and Biotechnology,2016,100(9):3877-3886.
[6]Patel S,Majumder A,Goyal A. Potentials of exopolysaccharides from lactic acid bacteria[J]. Indian Journal of Microbiology,2012,52(1):3-12.
[7]Zannini E,Waters D M,Coffey A,et al. Production,properties,and industrial food application of lactic acid bacteria-derived exopolysaccharides[J]. Applied Microbiology and Biotechnology,2016,100(3):1121-1135.
[8]Li W,Ji J,Tang W,et al. Characterization of an antiproliferative exopolysaccharide(LHEPS-2)fromLactobacillushelveticusMB2-1[J]. Carbohydrate Polymers,2014,105:334-340.
[9]Welman A D,Maddox I S. Exopolysaccharides from lactic acid bacteria-perspectives and challenges[J]. Trends in Biotechnology,2003,21(6):269-274.
[10]Makino S,Ikegami S,Kano H,et al. Immunomodulatory effects of polysaccharides produced byLactobacillusdelbrueckiissp.bulgaricusOLL1073R-1[J]. Journal of Dairy Science,2006,89(8):2873-2881.
[11]Schepetkin I A,Faulkner C L,Nelson-Overton L K,et al. Macrophage immunomodulatory activity of polysaccharides isolated from Juniperus scopolorum[J]. International Immunopharmacology,2005,5(13-14):1783-1799.
[12]Ye S,Liu F,Wang J,et al. Antioxidant activities of an exopolysaccharide isolated and purified from marinePseudomonasPF-6[J]. Carbohydrate Polymers,2012,87(1):764-770.
[13]Li S,Huang R,Shah N P,et al. Antioxidant and antibacterial activities of exopolysaccharides fromBifidobacteriumbifidumWBIN03 andLactobacillusplantarumR315[J]. Journal of Dairy Science,2014,97(12):7334-7343.
[14]Santos A,San Mauro M,Sanchez A,et al. The antimicrobial properties of different strains ofLactobacillusspp. isolated from Kefir[J]. Systematic and Applied Microbiology,2003,26(3):434-437.
[15]Chin C,Alexander D H,Marks P,et al. Nonhybrid,finished microbial genome assemblies from long-read SMRT sequencing data[J]. Nature Methods,2013,10(6):563-569.
[16]Li R,Li Y,Fang X,et al. SNP detection for massively parallel whole-genome resequencing[J]. Genome Research,2009,19(6):1124-1132.
[17]Grant J R,Stothard P. The CGView Server-a comparative genomics tool for circular genomes[J]. Nucleic Acids Research,2008,36(suppl 2):W181-W184.
[18]Siguier P. ISfinder-the reference centre for bacterial insertion sequences[J]. Nucleic Acids Research,2006,34(suppl 1):D32-D36.
[19]DuBois M,Gilles K A,Hamilton J K,et al. Colorimetric method for determination of sugars and related substances[J]. Analytical Chemistry,1956,28(3):350-356.
[20]姜琼,谢妤. 苯酚-硫酸法测定多糖方法的改进[J]. 江苏农业科学,2013,41(12):316-318.
[21]Shao L,Wu Z,Zhang H,et al. Partial characterization and immunostimulatory activity of exopolysaccharides fromLactobacillusrhamnosusKF5[J]. Carbohydrate Polymers,2014,107:51-56.
[22]Aleksandrzak-Piekarczyk T,Polak J,Jezierska B,et al. Genetic characterization of the CcpA-dependent,cellobiose-specific PTS system comprising CelB,PtcB and PtcA that transports lactose inLactococcuslactisIL1403[J]. International Journal of Food Microbiology,2011,145(1):186-194.
[23]Wu Q,Tun H M,Leung F C,et al. Genomic insights into high exopolysaccharide-producing dairy starter bacteriumStreptococcusthermophilusASCC 1275[J]. Scientific Reports,2014,4.
[24]Broadbent J R,McMahon D J,Welker D L,et al. Biochemistry,Genetics,and Applications of Exopolysaccharide Production inStreptococcusthermophilus:A Review1[J]. Journal of Dairy Science,2003,86(2):407-423.
[25]Zisu B,Shah N P. Effects of pH,temperature,supplementation with whey protein concentrate,and adjunct cultures on the production of exopolysaccharides byStreptococcusthermophilus1275[J]. Journal of Dairy Science,2003,86(11):3405-3415.
GeneticanalysisandpreliminarydeterminationofproductionofexopolysaccharideofStreptococcusthermophilusKLDSSM
DINGXiu-yun,MENGYue-yue,LIBai-liang,HUOGui-cheng*
(Key Laboratory of Dairy Science,Ministry of Education,Northeast Agricultural University,Harbin 150030,China)
In order to explore the ability of exopolysaccharide synthesis ofStreptococcusthermophilusKLDS SM based on gene level,three aspects of sugar transport systems,sugar nucleotide synthesis and exopolysaccharide gene cluster structure were analyzed in detail. The results showed that the bacteria could transport glucose,lactose,sucrose,trehalose and mannose,had the enzymes for glucose,lactose,fructose,sucrose,trehalose and mannose metabolism. The bacteria had the potential for exopolysaccharide precursor synthesis,including UDP-glucose,UDP-galactose,UDP-N-acetyl glucosamine,UDP-galactofuranose and dTDP-rhamnose. Furthermore,sequence alignment analysis showed that the exopolysaccharide sequence of this bacteria had a high identity to that of strain ASCC 1275,and preliminary determination of exopolysaccharide yield was about 331 mg/mL. The results showed that the strain was a potential EPS producing strain and had great potential in industry.
StreptococcusthermophilusKLDS SM;sugar transport system;sugar nucleotide;exopolysaccharide gene cluster;bioinformatics analysis
2017-01-03
丁秀云(1990-),女,硕士研究生,研究方向:乳酸菌生物技术,E-mail-aidingxiuyun@outlook.com。
*通讯作者:霍贵成(1958-),男,博士,教授,研究方向:乳品加工与食品微生物,E-mail-gchuo58@126.com。
农业部公益性行业(农业)科研专项(201203009)。
TS201.3
:A
:1002-0306(2017)12-0151-06
10.13386/j.issn1002-0306.2017.12.028

