致病菌与糖的特异性识别及其分析应用研究
崔飞云,徐 溢,赵 斌,车玉兰,刘露露
(1.重庆大学 化学化工学院,重庆 400044;2.重庆大学 光电工程学院微系统研究中心,重庆 400044;3.重庆大学 新型微纳器件与系统技术国防重点学科实验室,重庆 400044;4.重庆大学 微纳系统及新材料技术国际研发中心,重庆 400044)
综 述
致病菌与糖的特异性识别及其分析应用研究
崔飞云1,3,4,徐 溢1,2,3,4*,赵 斌1,3,4,车玉兰1,3,4,刘露露1,3,4
(1.重庆大学 化学化工学院,重庆 400044;2.重庆大学 光电工程学院微系统研究中心,重庆 400044;3.重庆大学 新型微纳器件与系统技术国防重点学科实验室,重庆 400044;4.重庆大学 微纳系统及新材料技术国际研发中心,重庆 400044)
致病菌往往通过凝集素-糖特异性识别来实现对宿主细胞的粘附,进而感染宿主组织,引起病变。 因此,研究致病菌与糖的特异性识别有利于进一步了解感染性疾病的致病机制,为致病菌的特异性检测和感染性疾病的治疗提供新的策略。该文总结了致病菌-糖特异性识别的相关机制机理;介绍了目前主要的研究方法和技术,特别评述了荧光光谱、表面等离子体共振、电化学阻抗谱及石英晶体微天平等技术在该研究中的应用现状,并对这4种技术与微流控芯片平台的结合进行了探讨;针对致病菌检测特异性差、耐药性严重等难题,重点综述了致病菌-糖的特异性识别在细菌分离、富集、检测、鉴别、生物膜抑制及抗菌糖类药物筛选方面的应用。最后对致病菌-糖特异性识别基础和应用研究进行了展望。
糖-凝集素相互作用;多效价相互作用;细菌粘附;细菌检测;综述
糖类是生物体进行新陈代谢所需能量的主要来源,同时,它还调节着一系列生命活动,具有重要的生物学功能[1]。糖的生物学功能主要是通过与蛋白质发生特异性识别来实现。可特异性识别糖的蛋白质主要包括单克隆抗体、酶、糖转运蛋白和凝集素[2]。其中,糖-凝集素特异性识别相关的生理学及病理学过程有致病菌或病毒感染、细胞信号传导、细胞凋亡、受精、癌症转移以及免疫调节等[3]。致病菌感染研究在感染性疾病治疗和细菌特异性检测方面具有重要的意义。所以,本文重点综述了致病菌表面凝集素与宿主细胞表面聚糖特异性识别相关的机制机理、分析检测技术和实际应用,并结合本课题组的工作特色[4-5],着重总结其在细菌检测方面的最新应用进展。最后对致病菌-糖特异性识别基础与应用研究进行展望。
1 致病菌-糖特异性识别的相关机制
1.1 致病菌的粘附机制
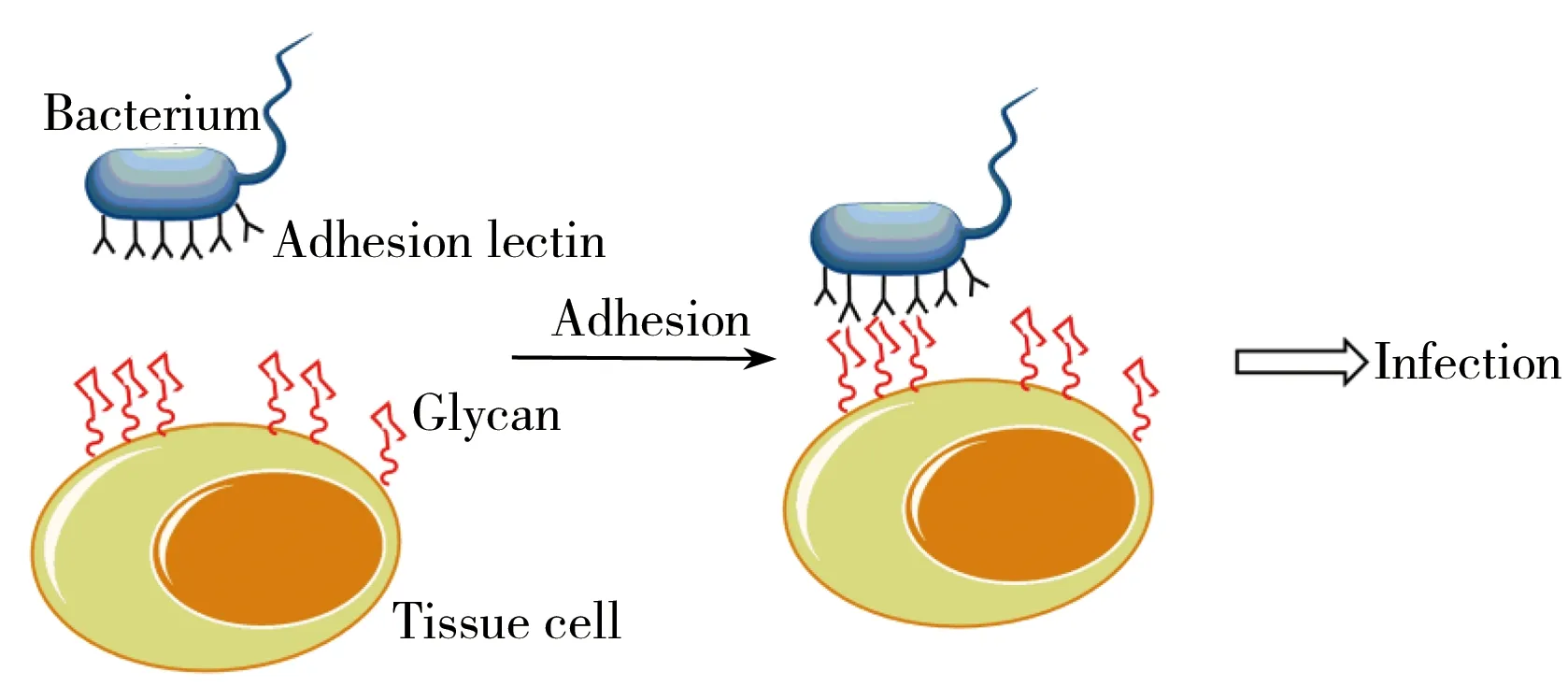
图1 细菌粘附过程原理示意图Fig.1 The principle of bacterial adhesion as a prelude to infection
细菌粘附是细菌感染人畜的先决条件,它受到多种理化因素和生物因素的制约,是多种粘附素和受体共同作用的结果,具有宿主特异性、组织特异性和细胞特异性[6-7]。作为毒力因子之一的菌毛,一般会参与致病菌对宿主细胞的特异性粘附过程[8],其主要途径是多个菌毛凝集素与宿主细胞表面多个聚糖发生特异性相互作用(图1)。
与细菌凝集素特异性结合的聚糖(Glycan)是宿主细胞膜表面糖复合物的糖基部分。聚糖一般分为3大类:连接脂质的聚糖、通过氮原子与蛋白质连接的聚糖(N-连接聚糖)以及通过氧原子与蛋白质连接的聚糖(O-连接聚糖)。聚糖中最常见的组分是己糖。正常情况下,己糖通过5-羟基与1-醛基反应形成半缩醛,以六元环吡喃糖构型形式出现。聚糖中常出现的己糖包括D-半乳糖(Gal)、D-葡萄糖(Glc)、D-甘露糖(Man)、N-乙酰-D-半乳糖胺(GalNAc)、N-乙酰-D-葡糖胺(GalNAc)等。此外,还有其他一些单糖,如葡糖醛酸(GlcA)、木糖(Xyl)、岩藻糖(Fuc)和N-乙酰神经氨酸(NeuAc)。
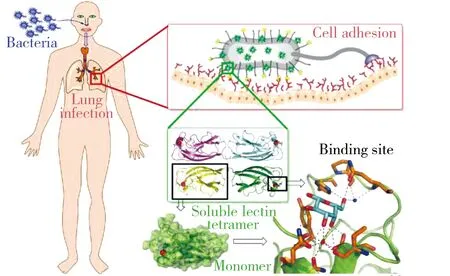
图2 铜绿假单胞菌粘附人体肺部上皮细胞过程示意图 [9]Fig.2 Schematic description of adhesion and lung infection by P.aeruginosa[9]
铜绿假单胞菌(Pseudomonasaeruginosa)又称绿脓杆菌,是目前研究较多的一种革兰氏阴性菌,会引起菌血症、慢性肺部感染、尿道感染以及急性溃疡性角膜炎等多种疾病。该菌表面有两种凝集素LecA(PA-IL)和LecB(PA-IIL),它们均为四聚物单体。LecA主要与人体肺部上皮细胞表面糖脂的α-Gal残基特异性结合,LecB则与多种组织上皮细胞表面的岩藻糖基、甘露糖基以及路易斯酸结合[9]。通过这两类特异性结合,铜绿假单胞菌粘附到宿主细胞表面,进而感染细胞和组织,引发病变(图2)。
微需氧型的革兰氏阴性菌幽门螺旋杆菌(Helicobacterpylori)能引起胃炎、胃溃疡以及胃癌。目前发现,其表面具有BabA(特异性识别Lewis b)、LabA(特异性识别N,N-二乙酰基氨基乳糖苷,LacdiNAc)和SabA(即H-1型,特异性识别唾液酸化的Lewis a和Lewis x) 3种凝集素[8,10-12]。这3种凝集素-糖相互作用介导了H.pylori对胃粘膜的粘附。Magalhães等[13]证明了缺乏岩藻糖转移酶的小鼠受H.pylori侵染的几率大大降低,其原因是岩藻糖转移酶的缺失导致了BabA特异性识别的Lewis a和Lewis x生物合成受阻。
革兰氏阴性菌大肠杆菌(Escherichiacoli)是常见的肠道、尿道致病菌,其粘附过程主要由菌毛凝集素与肠道上皮细胞表面聚糖的特异性相互作用来介导。大肠杆菌菌毛凝集素主要分为1型、P型、S型及F1C型[14]。在1型菌毛末端,具有可与α-Man特异性识别的FimH凝集素。在P型菌毛上,具有与Galα1-4Gal(Galabiose)特异性识别的PapG凝集素。具有P型菌毛的大肠杆菌,如E.coliO157∶H7能引起出血性肠炎、溶血性尿毒综合症,甚至死亡。S型和F1C型凝集素分别与唾液酸化的半乳糖和GalNAcβ1-4Gal发生特异性识别。正是由于这些菌毛凝集素的存在,才使大肠杆菌特异性粘附人体的不同组织,从而引发不同程度的疾病。
以上3种致病菌均为革兰氏阴性菌,也有研究发现,一些革兰氏阳性菌也可通过其表面的凝集素来粘附宿主细胞,如猪链球菌(Streptococcussuis)通过识别半乳二糖来粘附猪的肺组织、脑组织等,从而引发脑膜炎、败血症及肺部感染。 表1总结了文献[15-17]中细菌凝集素-糖特异性作用的例子。
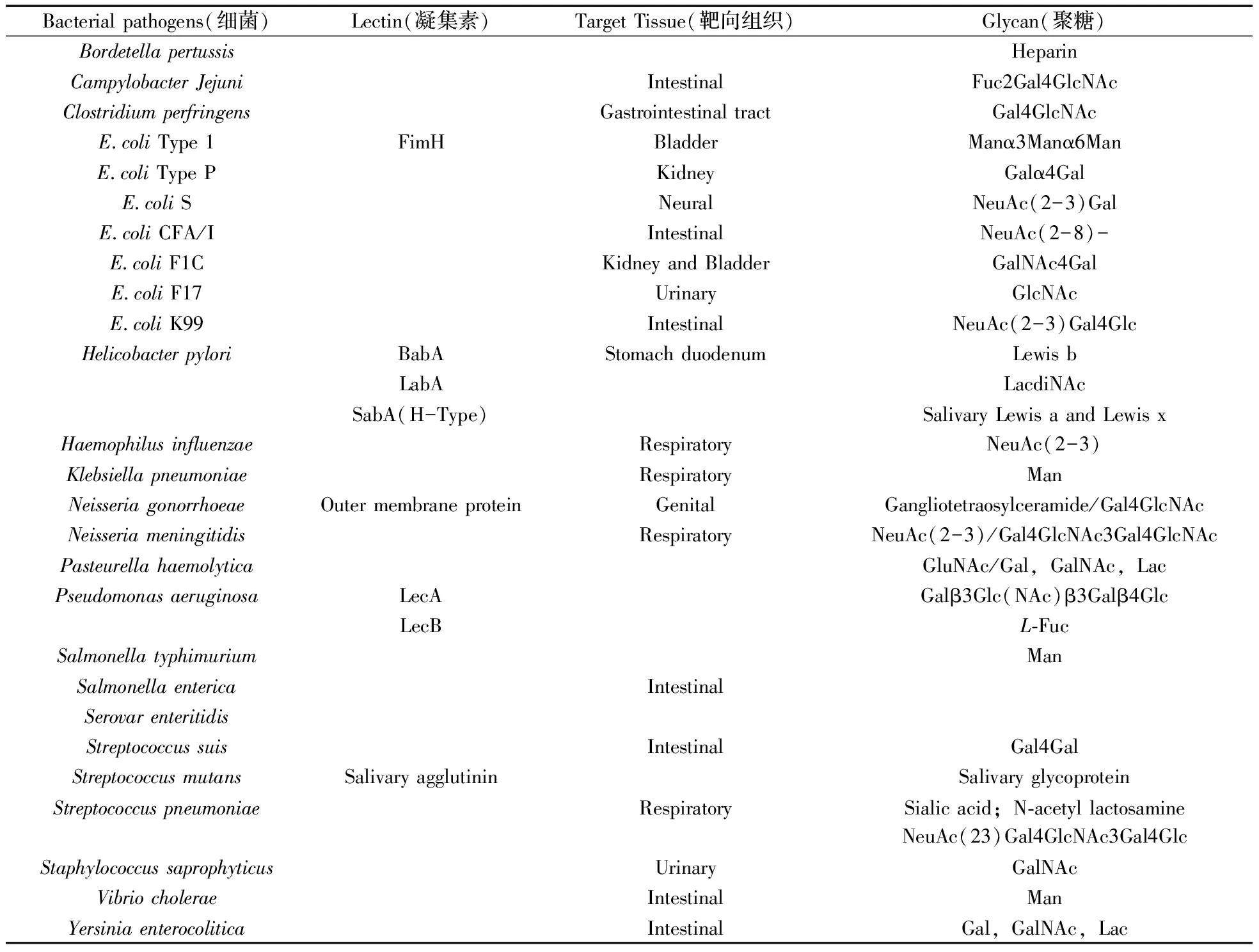
表1 以糖为识别受体的致病菌[15-17]
1.2 凝集素-糖的特异性识别机制
从分子水平来看,致病菌与糖的特异性识别实质为凝集素与糖的相互作用。凝集素与糖之间的相互作用包括氢键、金属配位键、范德华力和疏水性作用力等。有研究指出,糖分子上的 CH 基团与凝集素分子中芳环类残基之间的 CH- π 作用也扮演着重要角色[18]。
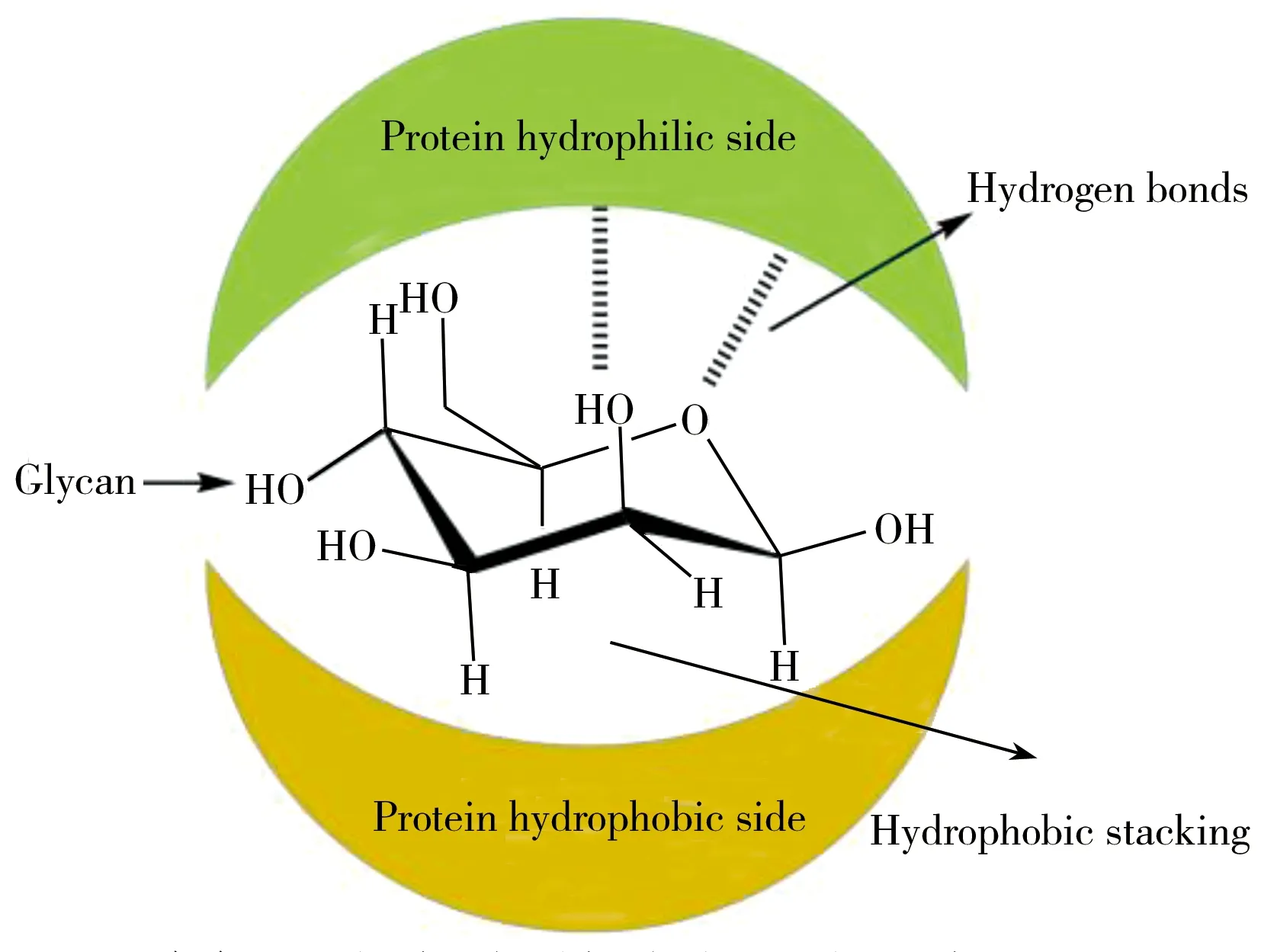
图3 凝集素-糖结合方式示意图Fig.3 Contact points between carbohydrate and lectin
由于糖分子具有多个羟基,凝集素分子存在多个氨基和羧基,所以氢键广泛存在。Ca2+,Mn2+等二价阳离子通过形成配位健来直接或间接地参与糖与凝集素的相互作用过程。例如,伴刀豆凝集素ConA(与α-D-甘露糖和α-D-葡萄糖发生特异性识别)的单体含有1个过渡金属离子结合位点S1(一般为Mn2+),1个Ca2+结合位点S2以及1个糖类结合位点[19]。P.aeruginosa表面的凝集素PA-IL和PA-IIL也需要在Ca2+调节下分别与Gal和Fuc特异性识别[20]。除了以上两种与亲水性相关的作用力外,疏水作用力在糖-凝集素识别中起着重要的作用,如Gal与凝集素的芳香烃残基之间的作用(图3)。
为了在分子水平上深入理解凝集素-糖相互作用,越来越多的学者从晶体结构学的角度来阐明其结合机制。Hung等[21]从晶体结构学的角度解释了大肠杆菌凝集素FimH与D-甘露糖的相互作用,分析结果表明,FimH表面的糖结合域可与单糖Man完美紧密的结合。甘露三糖与甘露戊糖等寡糖与它的结合亲和力之所以增强,是多余糖链的疏水面与FimH的“疏水脊”重叠的结果。在糖链上修饰上其它疏水性基团(如苯环)也可以增加其结合亲和力。沙门氏菌1型菌毛凝集素也可与Man特异性识别,但甘露寡糖或疏水基团修饰的甘露糖并不能增加其结合亲和力,可能是因为其凝集素表面不具有“疏水脊”区域[22]。
1.3 多效价相互作用机制
一个粒子(小分子、寡糖、蛋白质、核酸或一些分子的集合体、细胞膜、细胞器、病毒、细菌或细胞)与其它粒子相互作用时,独立结合点的个数称为效价(Valent)[23-24]。凝集素与糖单效价相互作用的结合亲和常数Ka一般为103~104L/mol,多效价相互作用的结合亲和常数Ka一般大于106L/mol[25-26]。生物体中,一般由多个糖配体与多个凝集素受体或一个凝集素的多个结合域进行结合,通过这种多效价相互作用的方式增加结合亲和力和结合特异性,这种效应称为“多效价效应”或“簇集效应”[27-28]。例如:流感病毒与单个唾液酸结合的Kd在mmol/L数量级,然而在生理条件下流感病毒与宿主细胞受体(唾液酸)的Kd却达到nmol/L数量级[29],这是多效价相互作用的结果。
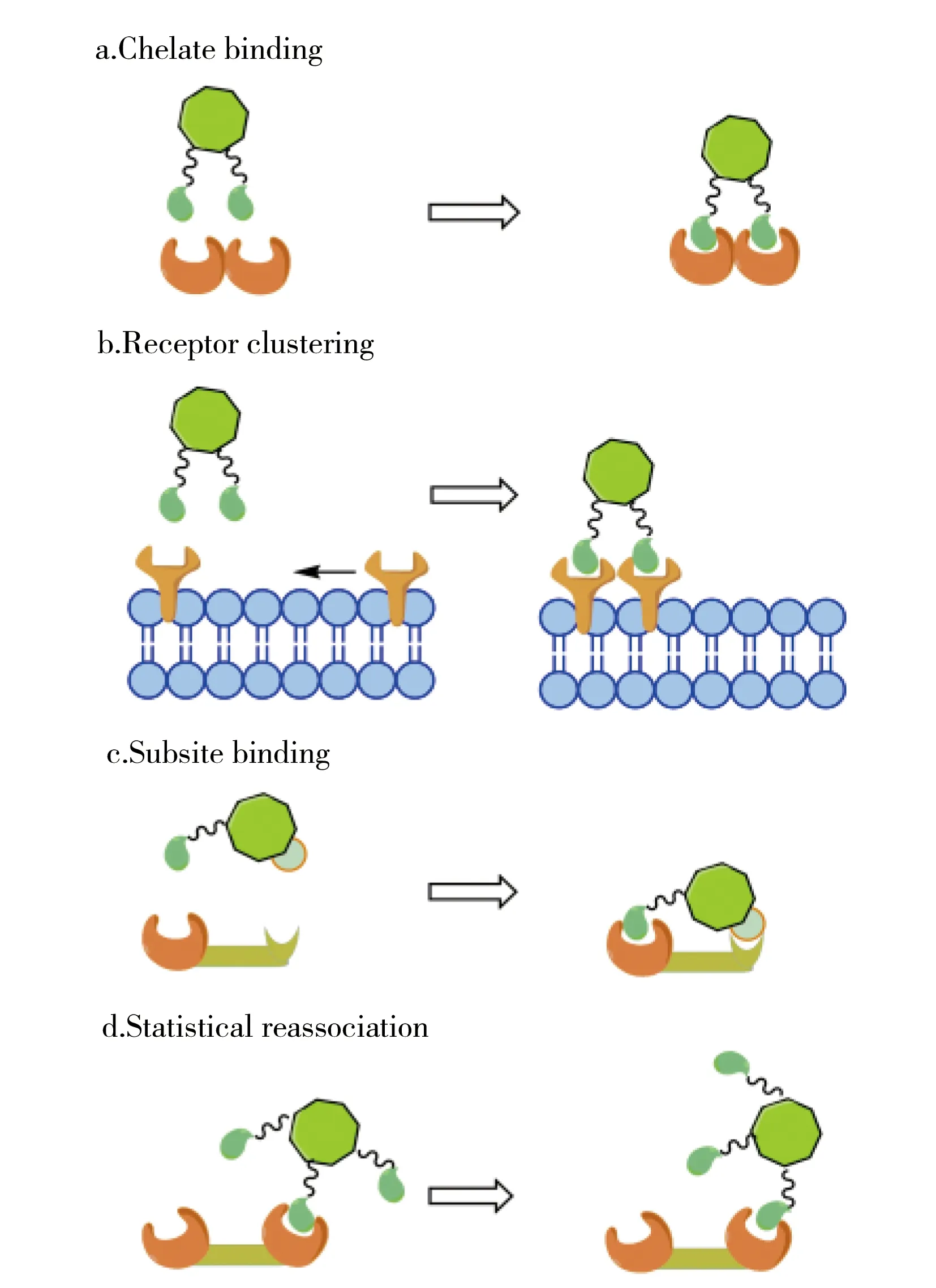
图4 受体-配体单多效价相互作用的模式图Fig.4 Mechanisms diagram of interaction between multivalent ligands and multivalent receptors
在致病菌-糖特异性识别的过程中,一个致病菌往往由多个菌毛同时参与粘附,同一个菌毛上的凝集素往往会与多个糖配体进行结合,以增加结合亲和力和结合特异性[15,30]。正是由于多个糖-凝集素的单效价相互作用同时发生,才表现出整体较强的多效价相互作用,但多效价相互作用不等于单效价相互作用的叠加。 研究表明,多效价相互作用的机制比较复杂,其主要有4种结合模式(图4),即螯合结合(Chelate binding)、受体簇集结合(Receptor clustering)、亚位点结合(Subsite binding)以及统计学上的重新结合(Statistical reassociation)[31]。不同的相互作用模式产生不同的生物响应。螯合结合模式是指1个同源多聚体凝集素的n个结合位点同时与1个多糖基分子的m个配体发生相互作用。这种结合模式可以大大增强受体-配体之间的结合亲和力。Zhu等[32]在研究E.coli与Man的相互作用时,揭示了E.coliORN178可同时与10~11个Man分子通过螯合结合模式发生多效价相互作用,与之前文献提到的通过亚位点结合模式提高结合亲和力不同。受体簇集模式是指当多效价的配体存在时,细胞膜表面的单效价受体通过在脂质双分子层中的扩散作用聚集,使其发生多效价相互作用。亚位点结合模式是指1个非均匀二效价配体与单效价受体结合后,其另外1个亚识别位点与受体以不同的结合亲和力和特异性发生的相互作用,亚单位相互作用力主要为疏水作用力,一般特异性很弱。统计学意义上重新结合概率大的模式主要是指,一个配体周围有高密度的受体,一旦一个受体与配体解离,则周围其它的受体会很快会再与配体结合,维持了受体-配体动态的结合,从而增加了结合亲和力。
2 致病菌-糖特异性识别的研究方法与技术
目前,研究凝集素-糖或细菌-糖特异性识别的方法和技术有很多,高分辨率核磁共振、X射线晶体衍射和计算机分子模拟技术通常用来研究凝集素-糖特异性结合的分子机制[2];亲和色谱、亲和毛细管电泳、等温滴定量热是经典的检测生物分子之间相互作用的方法,通常用来计算细菌/凝集素-糖结合亲和力[26];质谱、原子力显微镜、扫描电子显微镜、激光共聚焦成像、紫外-可见光谱、荧光光谱、循环伏安法、方波伏安法和差分脉冲伏安法等主要用于以糖为识别受体的细菌检测研究。
近年来,基于荧光光谱、表面等离子体共振、石英晶体微天平和电化学阻抗等检测技术的生物传感在研究细菌/凝集素-糖特异性识别方面展现出优异的性能,它们既可用来计算细菌/凝集素-糖结合亲和力常数,也可应用到细菌检测、糖类抗粘附药物筛选等研究领域,得到研究者的广泛关注,且该类生物传感与微阵列芯片、微流控芯片结合的研究更是该领域的前沿与热点。
2.1 荧光光谱
荧光光谱是研究生物分子相互作用的重要手段。在溶液体系中,利用荧光光谱研究细菌-糖的特异性识别主要采用荧光竞争性结合试验(Fluorescence competition binding assay)或荧光猝灭试验。Abdelhamid等[33]将壳聚糖(Chitosan)固定在CdS QDs上,得到CdS@CTS,并选择能与壳聚糖特异性识别的铜绿假单胞菌、金黄色葡萄球菌作为CdS@CTS的猝灭剂。当CdS@CTS与细菌结合后,其荧光强度降低,基于荧光强度的变化值,利用Stern-Volmer方程计算得到CdS@CTS与两种细菌的结合常数均在105数量级。
糖芯片(Carbohydrate microarray)是将多个不同的糖分子通过共价或非共价方式固定在基质(玻璃、硅片等)上,进而对凝集素或细菌等待测样品进行测试、分析的手段,其检测技术一般为荧光光谱[34]。糖芯片最鲜明的特征是样品用量少和通量高。Gade等[35]将Man和Fuc修饰的β-环糊精-二茂铁化合物固定在玻璃上,用于E.coliORN 178和P.aeruginosa检测。Flannery等[36]评述了糖芯片用于探索细菌-糖相互作用的现状,并指出糖芯片是目前未充分利用但获得较大关注的一种检测技术,它在细菌种类鉴别、细菌感染治疗方面将得到更广阔的应用。
当然,糖芯片存在一定的局限。一方面,糖芯片表面固定的糖分子缺乏移动性,而这种移动性对于细菌与糖的多效价相互作用至关重要。在细胞表面,糖一般以糖脂或糖蛋白的形式存在,它们所处的环境是可以移动的脂质双分子层;另一方面,在基质表面糖的固定密度难以控制。而糖的固定密度决定了细菌-糖之间发生单效价相互作用还是多效价相互作用。针对这两个问题,Zhu等[32]将含有甘露糖的微小单层囊泡(Small unilamellar vesicle,SUV)固定在修饰有胆固醇-聚乙二醇(cholesteryl-PEG)的玻璃基底上,形成支撑脂质双分子层(Supported lipid bilayers,SLBs)。通过该方法制备的糖芯片充分模拟了细胞表面的微环境,使得Man具有移动性,同时,可以通过控制SUV中Man的量来控制Man在SLBs上的密度。将E.coliORN178在该糖芯片上进行粘附实验,利用显微镜观察Man不同密度区域细菌粘附的数量,然后利用Langmuir等温吸附模型计算它们的解离常数,当Man的界面密度θM=0.2 nm-2时,E.coliORN178与Man的单效价亲和结合常数为KD1=(1.0±0.2)×10-22mol/L,多效价亲和结合常数KD2=(1.3±0.3)×10-27mol/L,此时多效价相互作用占主导;当Man的界面密度θM=0.02 nm-2时,E.coliORN178与Man的单效价亲和结合常数为KD1=1.0×10-21mol/L,多效价亲和结合常数KD2=5.2×10-17mol/L,此时单效价相互作用占主导。由此,揭示了一种新的E.coli粘附机制,即随着可移动性Man密度的增加,凝集素FimH与Man的相互作用由单效价转变为多效价,这种多效价相互作用进一步激发了E.coli的多菌毛粘附,显著增强了它们之间的结合力。
2.2 表面等离子体共振
表面等离子体共振(Surface plasmon resonance,SPR)是一种可以实时跟踪生物分子相互作用的光学检测技术,其优点是响应快、免标记。首先,将糖共价键合在金膜表面构建生物传感界面,然后将凝集素、细菌等分析物溶液以恒定流速流过传感界面,当两者发生亲和作用时,会引起生物传感界面上质量增加,从而导致界面折射率变化,并通过共振角信号的改变来显示。反射率的变化(RI)与结合在金属表面的生物大分子质量成正相关,1 000 RU的变化表示传感界面上1 ng/mm2的质量变化[37-38]。目前,利用该方法研究糖-凝集素结合亲和力的较多[39-41],而用来研究糖-细菌结合亲和力的较少。Bulard等[42]在金表面固定了岩藻糖(Fuc)、唾液酸(Neu5Ac)等7种糖,利用SPR来检测这7种糖与大肠杆菌不同血清型K-12/O157∶H7/O157∶H7-/O55∶H7/O145∶H28-相互作用力的大小,依据亲和力大小差异鉴别出E.coliO157∶H7,E.coliO157∶H7-和E.coliO145∶H28-这3种不同的菌株。
表面等离子体共振成像(SPR imaging)利用电荷耦合器件(Charge coupled device,CCD)记录样品反射光的图像,从而进行生物分子相互作用的动态或静态分析[43]。Smith等[44]以SPRi为检测技术,并依据弗鲁姆基等温吸附方程(Frumkin isotherm),计算了凝集素jacalin在Gal界面上的吸附系数KADS=(2.2±0.8)×107L/mol和凝集素ConA在Man界面上的吸附系数KADS=(5.6±1.7)×106L/mol。与SPR相比,SPRi最大的优势是易于与微阵列芯片结合,可以实现大量无需标记样品的原位、实时检测。
2.3 电化学阻抗谱
电化学阻抗谱(Electrochemical impedance spectroscopy,EIS)技术可以通过测量多个参数对电极表面生物分子间的相互作用过程和动力学机制加以分析。在检测凝集素-糖相互作用方面,EIS展现出较高的灵敏度,Tkac等[45]利用阻抗糖生物传感检测流感病毒血凝素,检测限可达amol/L数量级。此外,阻抗检测具有无需标记的优点,且越来越多的研究表明,基于阻抗检测的生物传感器件更易实现自动化、便携化及高通量检测。在国内,鞠熀先课题组和朱俊杰课题组在该领域做了很出色的工作[46-48]。他们将不同的凝集素固定在电极表面,用EIS来检测癌细胞表面糖基的动态表达。近年来,EIS开始应用到细菌-糖特异性识别相关的研究中。Guo等[49]将巯基化的甘露糖分子通过自组装单分子层(SAMs)的方式固定在金电极表面,用电化学阻抗谱法来特异性检测E.coliORN 178(特异性表达野生1型菌毛),检测限为102CFU/mL,但其构建的甘露糖传感界面在含有0.1 mol/L K3[Fe(CN)6]/K4[Fe(CN)6](1∶1)的PBS(pH 7.2)检测液中5 h甚至2 d才能达到稳定状态,耗时过长,易使传感界面失去生物活性,所以,糖传感界面的研究有待深入,电化学阻抗检测液有待优化。
由于电分析元件可以与微加工技术(MEMS)和微流控芯片平台很好地融合,这使得它易于制备经济的现场检测器件(POCT)[50-53]。La Belle等[54]制备了集成微电极的微流控芯片,利用电化学阻抗法检测糖-凝集素的相互作用,取得了良好效果。在微流控芯片检测单元中,待检测样品具有流动性的特点,可以更加有效地模拟生理条件,且能缩短检测时间。因此,将基于阻抗检测的糖生物传感与微流控芯片相结合,有望成为分析细菌-糖相互作用的有力工具。
2.4 石英晶体微天平
石英晶体微天平(Quartz crystal microbalance,QCM)是一种可检测传感器表面吸附物质质量变化的技术。在液相环境下,QCM 在测定生物样品如蛋白质、核酸、病毒、细菌及细胞等方面具有特色,测定共振频率比较容易达到较高的精确度,目前已有在细菌-糖特异性识别中的应用研究。Wang等[55]利用QCM研究了铜绿假单胞菌P.aeruginosaPAO1和大肠杆菌E.coliK-12与4种含有半乳糖(Gal)的高分子聚合物的相互作用,结果表明含有C-型凝集素的PaeruginosaPAO1在Ca2+的调节下可以与含有Gal的高分子较好的特异性识别。该课题组又在基于石英晶体微天平的生物传感界面上固定了葡萄糖(Glu)、半乳糖(Gal)及疏水性分子功能化的聚N-异丙基丙烯酰胺,通过检测P.aeruginosaPAO1与不同界面之间的相互作用,证明了凝集素与糖的特异性相互作用在铜绿假单胞菌对宿主细胞粘附过程中起主导作用[56]。Ma等[57]在金电极表面设计了甘露糖(Man)和甘露糖-凝集素ConA(Man/ConA)两种可以特异性检测细菌的传感界面,通过QCM检测E.coliW1485,检测限分别为1.7×104CFU/mL和50 CFU/mL。但QCM不易实现多组分检测,且非特异性吸附干扰严重,因此干扰信号的消除有待进一步解决。
3 致病菌-糖特异性识别的应用研究进展
3.1 细菌的分离与富集
细菌表面凝集素与糖具有较强的特异性识别亲和力,所以利用糖基修饰的磁性纳米粒子对细菌进行分离富集有可能得到更佳的效果。El-Boubbou等[58]首次将Man和Gal修饰的二氧化硅包裹的四氧化三铁磁性纳米粒子用于E.coliORN178的分离。Hatch等[59]比较了几种糖复合物修饰的磁珠与商业化多克隆抗体修饰的磁珠对污水中大肠杆菌的分离效率,发现糖复合物修饰的磁珠具有更好的选择特异性和分离效果。Behra等[60]制备了Man修饰的多孔聚乙二醇(PEG)磁性微球,该微球可以选择性结合溶液中特定的大肠杆菌菌株。由于PEG具有亲水性,大大降低了该磁性微球与细菌的疏水相互作用,提高了结合特异性。另外,多孔磁性微球的捕获效率比无孔磁性微球大3~4倍,当细菌与该磁性微球的浓度比小于30∶1时,捕获效率可达70%。
3.2 细菌的检测与鉴别
在细菌检测中,选择稳定性好、识别亲和力高、易于固定在传感界面上或纳米粒子表面的特异性识别受体成为一大研究热点[61-64]。细菌特异性识别受体主要有抗体(Abs)[65]、抗菌肽(AMPs)[66]、核酸适配体[67]、凝集素[68]、噬菌体[69]和分子印迹聚合物(MIP)[70]等。本课题组前期采用抗体检测了沙门氏菌[71]。首先将第一抗体固定在微流控芯片的检测区用于对沙门氏菌的捕获,然后加入修饰有第二抗体的量子点,利用荧光法得到检测限为37 CFU/mL。同时,本课题组设计并制备了碳量子点-适配体复合物纳米粒子,利用适配体对鼠伤寒沙门氏菌进行检测[72],检测限达到50 CFU/mL。然而,抗体存在对pH值和温度的要求较高、不能区别死活细菌以及成本高等缺点;而适配体易被核酸酶降解,所以需寻找新的细菌检测特异性识别受体。
由于致病菌表面凝集素与糖可以特异性识别,且它们之间多效价相互作用的亲和力很高,所以糖成了细菌检测领域很有开发潜力的特异性识别受体。与抗体相比,糖分子不会在温度和pH值改变的情况下因变性而失去活性,且糖分子较抗体分子小,有利于在传感界面上高密度固定,促进多效价识别,增加特异性识别亲和力,进而提高细菌检测灵敏度。Nilsson等[73]首次尝试以二糖Galα1-4Galβ作为生物传感器的识别元件,用来检测大肠杆菌(表达P型菌毛),实验得到S/N=75,充分证明了糖分子可以作为细菌特异性检测的识别受体。Jelinek等[74]在早期综述了糖生物传感器(Carbohydrate biosensors)用于致病菌检测的原理及应用,但其综述的内容主要集中在致病菌与糖或糖纳米粒子相互作用的研究,真正用于致病菌检测的较少,针对大肠杆菌的检测研究检测限均在104CFU/mL以上。Guo等[49]通过自组装单分子层(SAMs)的方式,将巯基化的甘露糖分子固定在金电极表面,用电化学阻抗法来特异性检测E.coliORN 178(特异性表达野生1型菌毛),检测限达到102CFU/mL。Ma等[57]将Man通过苯酚-聚噻吩连接链分子共价固定在金表面,通过Man与ConA的相互作用将ConA固定在界面上,得到了SM/TQ/Man/ConA传感界面,以方波伏安法为检测方法,E.coliW1485的检测限达25 CFU/mL。Yazgan等[75]在Man上修饰了苯基,采用表面等离子体共振生物传感器(SPR Biosensors),对E.coliATCC25922的检测限低至2.5 CFU/mL,这标志着以糖为识别受体的生物传感体系可以获得与抗体或适配体相当甚至更低的细菌检测灵敏度。表2总结了1994~2015年以糖为识别受体检测致病菌的相关文献。
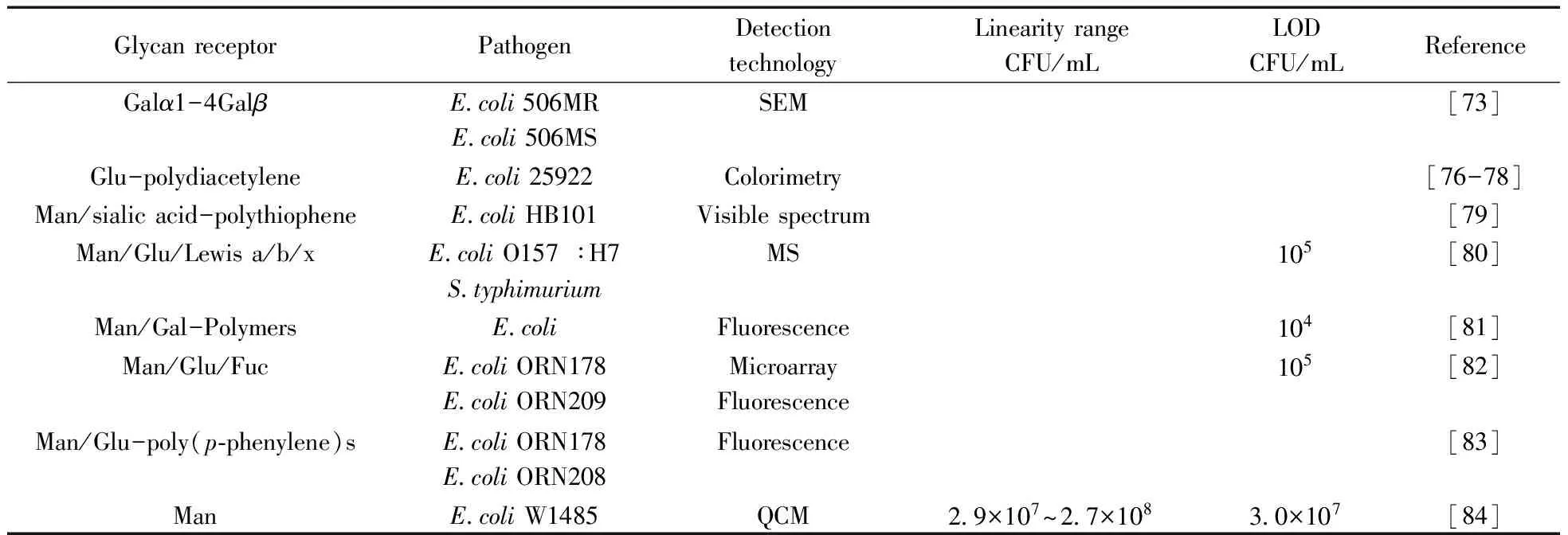
表2 以糖为识别受体的细菌检测
(续表2)
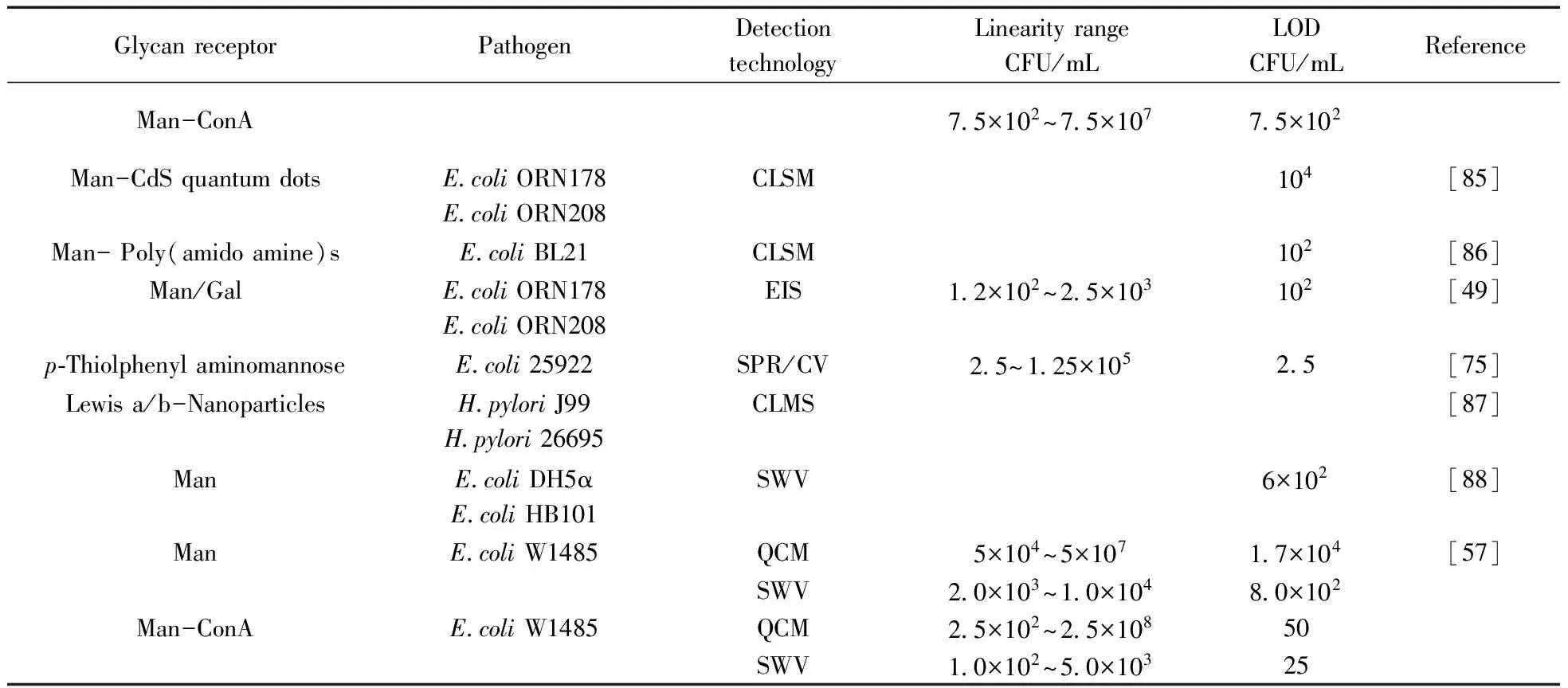
GlycanreceptorPathogenDetectiontechnologyLinearityrangeCFU/mLLODCFU/mLReferenceMan-ConA7 5×102~7 5×1077 5×102Man-CdSquantumdotsE coliORN178E coliORN208CLSM104[85]Man-Poly(amidoamine)sE coliBL21CLSM102[86]Man/GalE coliORN178E coliORN208EIS1 2×102~2 5×103102[49]p⁃ThiolphenylaminomannoseE coli25922SPR/CV2 5~1 25×1052 5[75]Lewisa/b-NanoparticlesH pyloriJ99H pylori26695CLMS[87]ManE coliDH5αE coliHB101SWV6×102[88]ManE coliW1485QCM5×104~5×1071 7×104[57]SWV2 0×103~1 0×1048 0×102Man-ConAE coliW1485QCM2 5×102~2 5×10850SWV1 0×102~5 0×10325
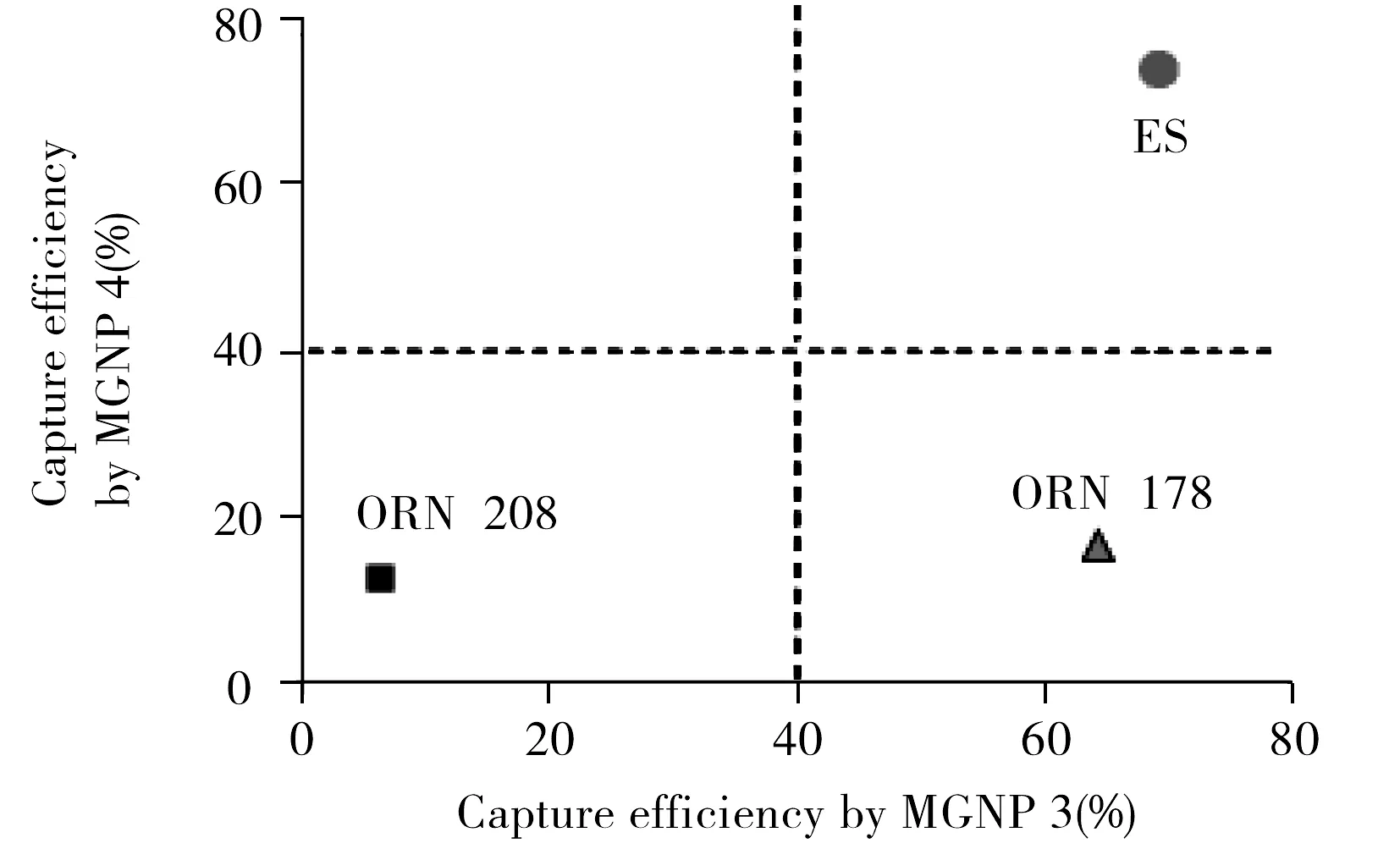
图5 MGNP3(Man)和MGNP4(Gal)鉴别3种E.coli 菌株的结果图[59]Fig.5 E.coli strain differentiation by MGNP3(Man) and MGNP4(Gal)[59]
在二维空间和三维空间上增加糖密度,可增加有效结合位点,增大发生多效价相互作用的几率,是提高糖生物传感检测灵敏度的主要途径。Wang等[89]系统综述了金属纳米材料、碳纳米材料、量子点、磁性纳米材料及硅纳米材料等在糖生物传感构建中的功能,并介绍了其在细菌检测方面的应用。用糖作为识别受体来检测细菌的主要挑战之一是糖具有交叉特异性(Cross-specificity)或称为广谱特异性(Broad-specificity),如Man既可与表达1型菌毛的大肠杆菌特异性识别,又可与鼠伤寒沙门氏菌、肺炎克雷伯菌特异性识别,从而影响了糖生物传感检测细菌的特异性。因此,还需要科研工作者进一步发掘与单一细菌特异性识别的糖链。化学合成法可以根据需要在糖链上修饰其它功能性基团,使得糖的特异性增强。另外,通过定性检测不同致病菌或同一致病菌不同菌株亚型与几种不同糖受体的结合亲和力的大小,可以鉴别不同的细菌菌株[90-91]。El-Boubbou等[58]根据Man磁性糖纳米粒子和Gal磁性纳米粒子对E.coliORN178/ORN208/ES 3种菌株的不同捕获率鉴别了这3种菌株,其结果如图5所示。细菌-糖特异性识别的定量研究,将进一步推动糖识别配体对细菌特异性检测和鉴别的研究进程。
3.3 细菌生物膜形成的抑制
细菌生物膜(Bacterial biofilm)是附着在生物惰性或活性材料表面上的微生物群体。当细菌处于生物膜状态时,内部的细菌能够逃避抗生素或机体免疫系统的作用,造成机体持续性感染或免疫性疾病,严重威胁人类健康,同时,造成了巨大的经济损失[92-93]。
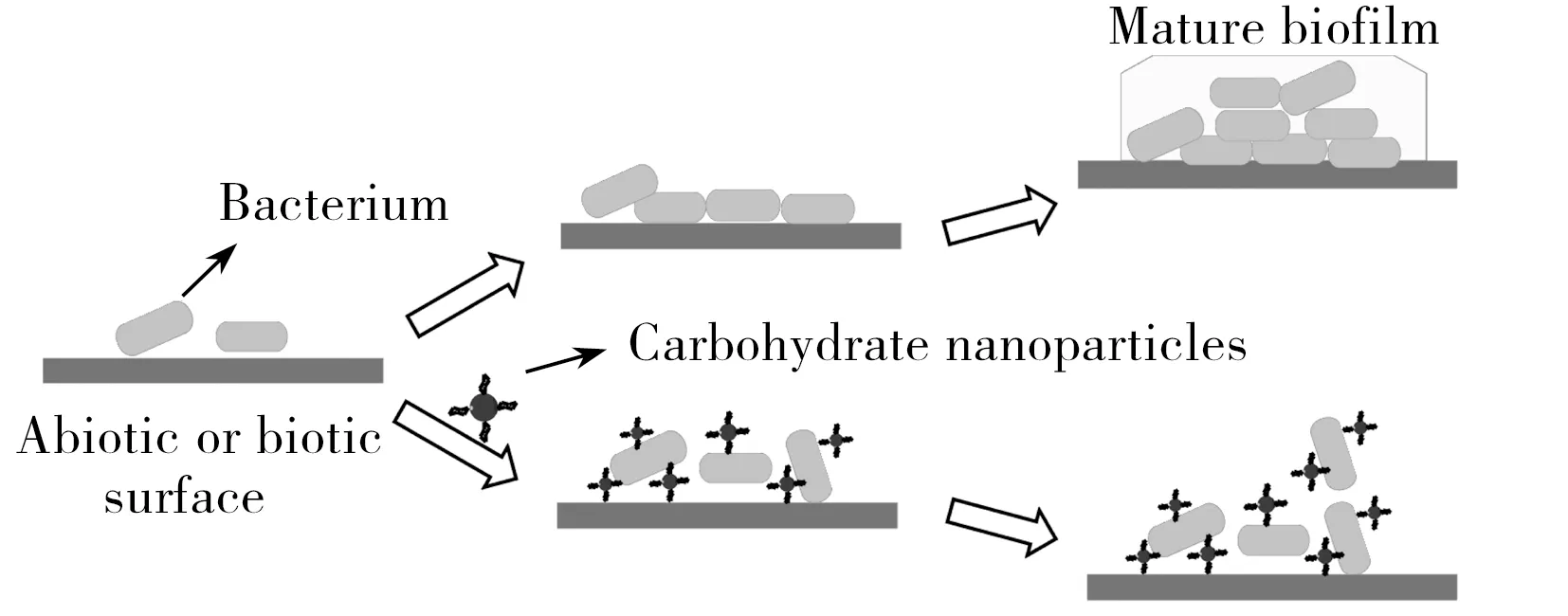
图6 糖纳米粒子抑制细菌生物膜原理示意图Fig.6 Schematic representation of ND-mannose ability to inhibit of biofilm formation
最近研究表明,糖纳米粒子可以通过抑制细菌粘附来扰乱生物膜的形成(图6)。Barras等[94]和Khanal等[95]分别合成了Man修饰的金刚石纳米粒子ND-Man和ND-Man3,利用吸光度法测试其对E.coli在96孔板聚氯乙烯(PVC)表面上培养24 h后生物膜形成状况的影响。结果表明,当ND-Man的浓度为80 μmol/L,ND-Man3的浓度为21 μmol/L时,可以显著抑制E.coil生物膜的形成。这可能是由于糖纳米粒子阻止了菌毛FimH粘附蛋白对PVC的粘附。将细菌先在96孔板上培养24 h,待生物膜形成后,再加入该糖纳米粒子,则不再影响细菌生物膜的状态。
P.aeruginosa生物膜的形成与其表面的凝聚素LecA和LecB相关。Johansson等[96]筛选出2种针对LecB的岩藻糖多肽树枝状大分子FD2(C-Fuc-LysProLeu)4(LysPheLysIle)2LysHisIleNH2和PA8(OFuc -LysAlaAsp)4(LysSerGlyAla)2LysHisIleNH2,其中FD2 可以有效抑制P.aeruginosa生物膜的形成(IC50=10 μmol/L),并有效驱散已经形成的生物膜。这说明LecB的多效价糖类抑制剂可以作为抑制和驱散生物膜的有效试剂。Reymond课题组制备了多种半乳糖苷修饰的多肽树枝状大分子,通过抑制LecA来抑制或驱散P.aeruginosa生物膜[97-99],发现多效价的糖配体对于生物膜形成抑制至关重要。Boukerb等[9]制备了四效价的半乳糖苷(Gal)和岩藻糖苷(Fuc)修饰的芳烃硼酸大分子,并利用吸光度法检测其对P.aeruginosa在96孔板上培养24 h后生物膜形成状况的影响,发现当其浓度为5 mmol/L时,能明显减少P.aeruginosaPAO1生物膜的形成,而单效价的βGalOMe和αFucOMe对生物膜的形成无显著影响。对照组实验中,四效价甘露糖苷和葡糖糖苷却不能有效抑制生物膜形成,表明糖苷抑制细菌生物膜形成具有特异性。多效价糖簇之所以能抑制生物膜的形成,可能是由于细菌表面的凝集素参与或能促进生物膜的形成,但具体的参与机制以及多效价糖簇抑制生物膜生成的机制尚需进一步研究。
3.4 糖类抗菌药物的筛选
致病菌对传统抗生素的耐药性日益增强,所以研发新的作用模式或针对细菌新靶点的抗菌药物具有巨大意义。天然多糖、糖聚合物、糖簇、糖树枝状大分子及糖纳米粒子等可以通过抑制细菌的粘附来预防和治疗细菌感染性疾病,具有取代抗生素的潜能[100]。体外及动物实验表明,能特异性识别细菌菌毛凝集素的可溶性多糖可以阻止相应细菌对哺乳动物细胞的粘附,且糖类抗粘附药物并不是杀死细菌,这使得细菌的突变速度减慢,耐药性降低[101]。所以糖类抗粘附药物在感染性疾病治疗方面展现出光明的前景。
拟糖物(Glycomimetic)是人工合成的多效价糖化合物分子,与天然聚糖相比,具有代谢稳定性。同时,它可与致病菌凝集素发生高选择性、高亲和力的特异性识别,是抗粘附药物筛选的重要对象[102]。拟糖物包括糖树枝状大分子、糖复合物、糖聚合物和糖纳米粒子等,其设计与合成需要考虑的因素有糖的密度、糖的空间排列、支架、连接链以及均匀多效价还是非均匀多效价等。关于这方面的内容,已有相关综述报道[103-104]。
针对E.coli引起的尿道感染性疾病,Appeldoorn等[105]合成了系列多效价的甘露糖化合物,测试了其抗粘附特性,为抗粘附药物的研究提供了基础。Hartmann等[106]合成5种带有不同官能团的甘露糖苷,检测了它们对E.coliPKL1162粘附哺乳动物结肠细胞的影响,结果显示修饰有苯环和β-内酰胺环的甘露糖苷具有优异的抗粘附特性且细胞毒性很低。Chaudhary等[107]制备了一系列不同形貌的糖基化金纳米粒子并检测它们的抗粘附和抗感染性能,结果表明,甘露糖基化的金纳米粒子呈棒状结构时比球状或星状具有更强的抗感染能力。
4 总结与展望
糖组学是继基因组学、蛋白质组学后,生物化学领域的又一大科学研究前沿。由于糖链的结构与功能比核苷酸链和多肽链更加丰富和复杂,所以,糖组学的研究是一个非常庞大而又繁重的任务。致病菌-糖特异性识别是该庞大任务中很小但很重要的一个研究点,它可为致病菌的特异性分离、富集、检测、鉴别、生物膜抑制以及抗生素替代药物的筛选等提供新的解决方案。
本文总结了致病菌-糖特异性识别的机制机理及新型的研究方法和技术,重点归纳总结了近20年来相关的应用研究进展。致病菌之所以能够粘附宿主细胞表面进而侵染细胞引起疾病,主要是因为其表面的凝集素与宿主细胞表面的聚糖发生多效价特异性识别。为了进一步阐明致病菌-糖特异性识别的机制,并将该类机制应用到细菌检测以及相关疾病的治疗中,强有力的研究方法和技术必不可少。本文重点介绍了既能用于机制研究、结合亲和力计算,又能用于细菌检测、药物筛选的4种检测技术,即荧光光谱、表面等离子体共振、电化学阻抗谱和石英晶体微天平。荧光技术在生物成像、标志物追踪方面具有独特优势。其余3种技术均具有无需标记的优点,可以尽可能的避免外来物质的干扰,使检测体系变得更加简单且接近于生物体系本身。同时,这4种检测技术都易于集成到微阵列芯片或微流控芯片平台上,通过它们的结合,研究快速、灵敏、高通量、便携化的检测新方法将是目前乃至今后一段时间的研究热点。
以糖作为致病菌检测、鉴别的特异性识别受体,以糖类化合物或糖纳米粒子作为抑制细菌感染和生物膜形成的替代药物是基于致病菌-糖特异性识别机制研究的具体应用,但目前还停留在实验研究阶段,并未进行实际应用。主要面临的挑战有以糖作为识别受体具有交叉特异性,使得检测特异性降低。再者,致病菌的同一条染色体所携带的基因会表达多种凝集素,当其所表达的凝集素不同时,糖类药物抑制其感染的效果会变差。同时,单糖对致病菌凝集素的亲和力很低,并不能有效抑制其感染,所以了解致病菌凝集素的表达规律,设计并合成结合亲和力强、生物相容性好的糖化合物或糖微米/纳米粒子是目前该领域研究的重点。
[1] Bertozzi C R,Kiessling L L.Science,2001,291(5512):2357-2364.
[2] Huang Y,Huang J H,Xie Q J,Yao S Z.Prog.Chem.(黄毅,黄金花,谢青季,姚守拙.化学进展),2008,20(6):942-950.
[3] Walker D B,Joshi G,Davis A P.Cell.Mol.LifeSci.,2009,66(19):3177-3191.
[4] Jiang Y,Xu Y,Wang R J,Su X,Dong C Y.Prog.Chem.(蒋艳,徐溢,王人杰,苏喜,董春燕.化学进展),2015,27(9):1240-1250.
[5] Zhao H Z,Wang C Y,Xu Y,Wang R J,Jiang Y,Liao X,Zheng X Q.J.Instrum.Anal.(赵华宙,王春艳,徐溢,王蓉,蒋艳,廖鑫,郑祥权.分析测试学报),2015,34(3):268-275.
[6] Pera N P,Pieters R J.Med.Chem.Commun.,2014,5(8):1027-1035.
[7] Ielasi F S,Alioscha-Perez M,Donohue D,Claes S,Sahli H,Schols D,Willaert R G.mBio,2016,7(4):1-7.
[8] Linke D,Goldman A.BacterialAdhesion:Chemistry,BiologyandPhysics.Dordrecht:Springer,2011:227-240.
[9] Boukerb A M,Rousset A,Galanos N,Méar J,Thépaut M,Grandjean T,Gillon E,Cecioni S,Abderrahmen C,Faure K,Redelberger D,Kipnis E,Dessein R,Havet S,Darblade B,Matthews S E,de Bentzmann S,Guéry B,Cournoyer B,Imberty A,Vidal S.J.Med.Chem.,2014,57(24):10275-10289.
[10] Gonçalves I C,Magalhães A,Costa A M S,Oliveira J R,Henriques P C,Gomes P,Reis C A,Martins M C L.ActaBiomater.,2016,33:40-50.
[11] Fei Y Y,Schmidt A,Bylund G,Johansson D X,Henriksson S,Lebrilla C,Solnick J V,Borén T,Zhu X D.Anal.Chem.,2011,83(16):6336-6341.
[12] Shu J,Yu H J,Liu X W,Zhang D D,Li Z.Prog.Biochem.Biophys.(舒健,于汉杰,刘夏薇,张旦旦,李铮.生物化学与生物物理进展),2016,43(5):449-460.
[13] Magalhães A,Gomes J,Ismail M N,Haslam S M,Mendes N,Osório H,David L,Le Pendu J,Haas R,Dell A,Borén T,Reis C A.Glycobiology,2009,19(12):1525-1536.
[14] Zourob M,Elwary S,Turner A.PrinciplesofBacterialDetection:Biosensors,RecognitionReceptorsandMicrosystems.New York:Springer,2008:659-687.
[15] Imberty A,Varrot A.Curr.Opin.Struct.Biol.,2008,18(5):567-576.
[16] Lindhorst K T,Oscarson S.ImplicationsandReflections:GlycoscienceandMicrobialAdhesion.Berlin Heidelberg:Springer,2009:17-65.[17] Haseley S R.Anal.Chim.Acta,2002,457(1):39-45.
[18] Li M M,Xiong Y T,Qing G Y,Sun T L.Prog.Biochem.Biophys.(李闵闵,熊雨婷,卿光焱,孙涛垒.生物化学与生物物理进展),2016,43(2):115-127.
[19] Zeng X Q,Andrade C A S,Oliveira M D L,Sun X.Anal.Bioanal.Chem.,2012,402(10):3161-3176.
[20] Imberty A,Mitchell E P,Wimmerová M.Curr.Opin.Struct.Biol.,2005,15(5):525-534.
[21] Hung C,Bouckaert J,Hung D,Pinkner J,Widberg C,Defusco A,Auguste C G,Strouse R,Langermann S,Waksman G,Hultgren S J.Mol.Microbiol.,2002,44(4):903-915.
[22] Firon N,Ofek I,Sharon N.Infect.Immun.,1984,43(3):1088-1090.
[23] Mammen M,Choi S K,Whitesides G M.Angew.Chem.Int.Ed.,1999,30(9):2754-2794.
[24] Muller C,Despras G,Lindhorst T K.Chem.Soc.Rev.,2016,45(11):3275-3302.
[25] Zeng X Q,Qu K,Rehman A.AccountsChem.Res.,2016,49(9):1624-1633.
[26] Liang P H,Wang S K,Wong C H.J.Am.Chem.Soc.,2007,129(36):11177-11184.
[27] Lundquist J J,Toone E J.Chem.Rev.,2002,102(2):555-578.
[28] Jayaraman N.Chem.Soc.Rev.,2009,38(12):3463-3483.
[29] Hanson J E,Sauter N K,Skehel J J,Wiley D C.Virology,1992,189(2):525-533.
[30] Karlsson K A.Curr.Opin.Struct.Biol.,1995,5(5):622-635.
[31] Cecioni S,Imberty A,Vidal S.Chem.Rev.,2015,115(1):525-561.
[32] Zhu X Y,Holtz B,Wang Y N,Wang L X,Orndorff P E,Guo A.J.Am.Chem.Soc.,2009,131(38):13646-13650.[33] Abdelhamid H N,Wu H F.J.Mater.Chem.,2013,1(1):6094-6106.
[34] Park S,Gildersleeve J C,Blixt O,Shin I.Chem.Soc.Rev.,2013,42(10):4310-4326.
[35] Gade M,Paul A,Alex C,Choudhury D,Thulasiram H V,Kikkeri R.Chem.Commun.,2015,51(29):6346-6349.[36] Flannery A,Gerlach J Q,Joshi L,Kilcoyne M.Microarrays,2015,4(4):690-713.
[37] Safina G.Anal.Chim.Acta,2012,712:9-29.
[38] Wang X,Xu J Y,Chen Y.Prog.Chem.(王霄,许吉英,陈义.化学进展),2015,27(5):550-558.
[39] Geuijen K P M,Halim L A,Schellekens H,Schasfoort R B,Wijffels R H,Eppink M H.Anal.Chem.,2015,87(16):8115-8122.
[41] Bhattarai J K,Sharma A,Fujikawa K,Demchenko A V,Stine K J.Carbohyd.Res.,2015,405:55-65.
[42] Bulard E,Bouchetspinelli A,Chaud P,Roget A,Calemczuk R,Fort S,Livache T.Anal.Chem.,2015,87(3):1804-1811.
[43] Liu X J,Zhang Q Q,Tu Y,Zhao W F,Gai H W.Anal.Chem.,2013,85(24):11851-11857.
[44] Smith E A,Thomas W D,Kiessling L L,Corn R M.J.Am.Chem.Soc.,2003,125(20):6140-6148.
[45] Hushegyi A,Bertok T,Damborsky P,Katrlik J,Tkac J.Chem.Commun.,2015,51(35):7474-7477.
[46] Ding L,Cheng W,Wang X J,Xue Y D,Lei J P,Yin Y B,Ju H X.Chem.Commun.,2009,(46):7161-7163.
[47] Ding L,Ji Q J,Qian R C,Cheng W,Ju H X.Anal.Chem.,2010,82(4):1292-1298.
[48] Cao J T,Hao X Y,Zhu Y D,Sun K,Zhu J J.Anal.Chem.,2012,84(15):6775-6782.
[49] Guo X F,Kulkarni A,Doepke A,Halsall H B,Iyer S,Heineman W R.Anal.Chem.,2012,84(1):241-246.
[50] Rackus D G,Shamsi M H,Wheeler A R.Chem.Soc.Rev.,2015,44(15):5320-5340.
[51] Pumera M,Merkoçi A,Alegret S.TrAC,TrendsAnal.Chem.,2006,25(3):219-235.
[52] Labib M,Sargent E H,Kelley S O.Chem.Rev.,2016,116(16):9001-9090.
[53] Oja S M,Wood M,Zhang B.Anal.Chem.,2013,85(2):473-486.
[54] La Belle J T,Gerlach J Q,Svarovsky S,Joshi L.Anal.Chem.,2007,79(18):6959-6964.
[55] Wang Y N,Narain R,Liu Y.Langmuir,2014,30(25):7377-7387.
[56] Wang Y N,Kotsuchibashi Y,Liu Y,Narain R.ACSAppl.Mater.Interfaces,2015,7(3):1652-1661.
[57] Ma F,Rehman A,Liu H Y,Zhang J T,Zhu S L,Zeng X Q.Anal.Chem.,2015,87(3):1560-1568.
[58] El-Boubbou K,Gruden C,Huang X F.J.Am.Chem.Soc.,2007,129(44):13392-13393.
[59] Hatch D M,Weiss A A,Kale R R,Iyer S S.Chem.Biol.Chem.,2008,9(15):2433-2442.
[60] Behra M,Azzouz N,Schmidt S,Volodkin D V,Mosca S,Chanana M,Seeberger P H,Hartmann L.Biomacromolecules,2013,14(6):1927-1935.
[61] Ngundi M M,Kulagina N V,Anderson G P,Taitt C R.ExpertRev.Proteomic.,2006,3(5):511-524.
[62] Vikesland P J,Wigginton K R.Environ.Sci.Technol.,2010,44(10):3656-3669.
[63] Templier V,Roux A,Roupioz Y,Livache T.TrAC,TrendsAnal.Chem.,2016,79:71-79.
[64] Foudeh A M,Fatanat D T,Veres T,Tabrizian M.LabChip,2012,12(18):3249-3266.
[65] Barreiros Dos Santos M,Agusil J P,Prieto-Simón B,Sporer C,Teixeira V,Samitier J.Biosens.Bioelectron.,2013,45:174-180.
[66] Silva R R,Avelino K Y P S,Ribeiro K L,Franco O L,Oliveira M D L,Andrade C A S.Front.Microbiol.,2014,5(443):1-7.
[67] Hernández R,Vallés C,Benito A M,Maser W K,Rius F X,Riu J.Biosens.Bioelectron.,2014,54:553-557.
[68] Yang H Y,Zhou H F,Hao H Y,Gong Q J,Nie K.Sens.ActuatorB,2016,229:297-304.
[69] Mejri M B,Baccar H,Baldrich E,Del Campo F J,Helali S,Ktari T,Simonian A,Aouni M,Abdelghani A.Biosens.Bioelectron.,2010,26(4):1261-1267.
[70] Dickert F L,Hayden O,Halikias K P.Analyst,2001,126(6):766-771.
[71] Wang R J,Ni Y,Xu Y,Jiang Y,Dong C Y,Chuan N.Anal.Chim.Acta,2015,853:710-717.
[72] Wang R J,Xu Y,Zhang T,Jiang Y.Anal.Methods,2015,7(5):1701-1706.
[73] Nilsson K G I,Mandenius C.Nat.Biotechnol.,1994,12(12):1376-1378.
[74] Jelinek R,Kolusheva S.Chem.Rev.,2004,104(12):5987-6016.
[75] Yazgan I,Noah N M,Toure O,Zhang S,Sadik O A.Biosens.Bioelectron.,2014,61:266-273.
[76] Ma Z F,Li J R,Liu M H,Cao J,Zou Z Y,Tu J,Jiang L.J.Am.Chem.Soc.,1998,120(48):12678-12679.
[77] Ma Z F,Li J R,Jiang L,Cao J,Boullanger P.Langmuir,2000,16(20):7801-7804.
[78] Su Y L,Li J R,Jiang L,Cao J.J.ColloidInterf.Sci.,2005,284(1):114-119.
[79] Baek M,Stevens R C,Charych D H.BioconjugateChem.,2000,11(6):777-788.
[80] Bundy J L,Fenselau C.Anal.Chem.,2001,73(4):751-757.
[81] Disney M D,Zheng J,Swager T M,Seeberger P H.J.Am.Chem.Soc.,2004,126(41):13343-13346.
[82] Disney M D,Seeberger P H.Chem.Biol.,2004,11(12):1701-1707.
[83] Xue C H,Jog S P,Murthy P,Liu H Y.Biomacromolecules,2006,7(9):2470-2474.
[84] Shen Z H,Huang M C,Xiao C D,Zhang Y,Zeng X Q,Wang P G.Anal.Chem.,2007,79(6):2312-2319.
[85] Mukhopadhyay B,Martins M B,Karamanska R,Russell D A,Field R A.TetrahedronLett.,2009,50(8):886-889.[86] Yang W,Pan C Y,Luo M D,Zhang H B.Biomacromolecules,2010,11(7):1840-1846.
[87] Park S,Kim G,Park S,Pai J,Rathwell D,Park J,Kang Y,Shin I.J.Am.Chem.Soc.,2015,137(18):5961 -5968.
[88] Dechtrirat D,Gajovic-Eichelmann N,Wojcik F,Hartmann L,Bier F F,Scheller F W.Biosens.Bioelectron.,2014,58:1-8.
[89] Wang Y Z,Qu K,Tang L H,Li Z L,Moore E,Zeng X Q,Liu Y,Li J H.TrAC,TrendsAnal.Chem.,2014,58:54-70.
[90] Mader A,Gruber K,Castelli R,Hermann B A,Seeberger P H,Rädler J O,Leisner M.NanoLett.,2012,12(1):420-423.
[91] Richards S,Fullam E,Besra G S,Gibson M I.J.Mater.Chem.B,2014,2(11):1490-1498.
[92] Joachim I,Rikker S,Hauck D,Ponader D,Boden S,Sommer R,Hartmann L,Titz A.Org.Biomol.Chem.,2016,14(33):7933-7948.
[93] Ligeour C,Vidal O,Dupin L,Casoni F,Gillon E,Meyer A,Vidal S,Vergoten G,Lacroix J,Souteyrand E,Imberty A,Vasseur J,Chevolot Y,Morvan F.Org.Biomol.Chem.,2015,13(31):8433-8444.
[94] Barras A,Martin F A,Bande O,Baumann J,Ghigo J,Boukherroub R,Beloin C,Siriwardena A,Szunerits S.Nanoscale,2013,5(6):2307-2316.
[95] Khanal M,Larsonneur F,Raks V,Barras A,Baumann J,Martin F A,Boukherroub R,Ghigo J,Ortiz Mellet C,Zaitsev V,Garcia Fernandez J M,Beloin C,Siriwardena A,Szunerits S.Nanoscale,2015,7(6):2325-2335.
[96] Johansson E M V,Crusz S A,Kolomiets E,Buts L,Kadam R U,Cacciarini M,Bartels K,Diggle S P,Cámara M,Williams P,Loris R,Nativi C,Rosenau F,Jaeger K,Darbre T,Reymond J.Chem.Biol.,2008,15(12):1249-1257.
[97] Kadam R U,Bergmann M,Hurley M,Garg D,Cacciarini M,Swiderska M A,Nativi C,Sattler M,Smyth A R,Williams P,Cámara M,Stocker A,Darbre T,Reymond J.Angew.Chem.Int.Ed.,2011,50(45):10631-10635.
[98] Reymond J,Bergmann M,Darbre T.Chem.Soc.Rev.,2013,42(11):4814-4822.
[99] Bergmann M,Michaud G,Visini R,Jin X,Gillon E,Stocker A,Imberty A,Darbre T,Reymond J.Org.Biomol.Chem.,2016,14(1):138-148.[100] Bernardi A,Jimenez-Barbero J,Casnati A,De Castro C,Darbre T,Fieschi F,Finne J,Funken H,Jaeger K,Lahmann M,Lindhorst T K,Marradi M,Messner P,Molinaro A,Murphy P V,Nativi C,Oscarson S,Penades S,Peri F,Pieters R J,Renaudet O,Reymond J,Richichi B,Rojo J,Sansone F,Schaffer C,Turnbull W B,Velasco-Torrijos T,Vidal S,Vincent S,Wennekes T,Zuilhof H,Imberty A.Chem.Soc.Rev.,2013,42(11):4709-4727.[101] Sharon N.BiochimicaetBiophysicaActa(BBA)-GeneralSubjects,2006,1760(4):527-537.
[102] Sattin S,Bernardi A.TrendsBiotechnol.,2016,34(6):483-495.
[103] Michaud G,Visini R,Bergmann M,Salerno G,Bosco R,Gillon E,Richichi B,Nativi C,Imberty A,Stocker A,Darbre T,Reymond J.Chem.Sci.,2016,7(1):166-182.
[104] Hartmann M,Lindhorst T K.Eur.J.Org.Chem.,2011,2011(20/21):3583-3609.
[105] Appeldoorn C C M,Joosten J A F,Ait El Maate F,Dobrindt U,Hacker J,Liskamp R M J,Khan A S,Pieters R J.Tetrahedron:Asymmetry,2005,16(2):361-372.
[106] Hartmann M,Papavlassopoulos H,Chandrasekaran V,Grabosch C,Beiroth F,Lindhorst T K,Röhl C.FEBSLett.,2012,586(10):1459-1465.
[107] Chaudhary P M,Sangabathuni S,Murthy R V,Paul A,Thulasiram H V,Kikkeri R.Chem.Commun.,2015,51(86):15669-1567.
Specific Recognition of Pathogenic Bacterium towards Glycan and Its Applications in Analytical Chemistry
CUI Fei-yun1,3,4,XU Yi1,2,3,4*,ZHAO Bin1,3,4,CHE Yu-lan1,3,4,LIU Lu-lu1,3,4
(1.School of Chemistry and Chemical Engineering,Chongqing University,Chongqing 400044,China;2.Microsystem Research Center,School of Optoelectronic Engineering,Chongqing University,Chongqing 400044,China; 2.Defense Key Disciplines Lab of Novel Micro-Nano Devices and System Technology,Chongqing University, Chongqing 400044,China;4.International R&D Center of Micro-Nano Systems and New Materials Technology,Chongqing University,Chongqing 400044,China)
Pathogen adhesion to host tissue is a prerequisite for a majority of infectious diseases.A common way for accomplishing adhesion is specific recognition of bacterium cellular lectins towards glycan on the surface of the host cells.Therefore,research on pathogenic bacteria-glycan interactions is beneficial to further understand the pathogenic mechanism of infectious diseases.Meanwhile,it could provide new strategies for specific detection on pathogenic bacteria and treatment of infectious diseases,and the mechanisms related with adhesion of pathogenic bacteria,carbohydrate-lectin interactions and multivalent interactions were summed up.The analysis technologies of pathogenic bacteria-glycan interactions were introduced,especially,the latest development and research of surface plasmon resonance,quartz crystal microbalances,fluorescence spectrum and electrochemical impedance spectroscopy were summarized.Special attention was paid to the combination of those detecting technologies and microfluidic chip platforms.In view of the serious problems of antibiotics resistance and poor specificity of pathogenic bacteria detection,the applications of bacterial isolation,enrichment,detection,identification,biofilm inhibition and the screening of antimicrobial agents were reviewed.Finally,the future development for basic research and application of pathogenic bacteria-glycan interactions were prospected.
carbohydrate-lectin interactions;multivalent interactions;bacterial adhesion;bacterial detection;review
2016-12-15;
2017-02-07
国家自然科学基金项目(21375156);国家高技术研究发展计划(863)项目(2015AA021104);重庆市前沿研究重点项目(cstc2015jcyjBX0010);重庆市科技惠民项目(cstc2015shmszx00014)
10.3969/j.issn.1004-4957.2017.06.021
O657.1;R379
A
1004-4957(2017)06-0817-12
*通讯作者:徐 溢,博士,教授,研究方向:微流控芯片生化分析,Tel:023-65111022,E-mail:xuyibbd@sina.com

