盐生植物小花碱茅CYP86A基因的RNAi载体构建
王沛,崔彦农,高丽,王锁民*
盐生植物小花碱茅CYP86A基因的RNAi载体构建
王沛1,2,崔彦农1,高丽1,王锁民1*
(1.草地农业生态系统国家重点实验室,兰州大学草地农业科技学院,甘肃 兰州 730020;2.西南民族大学青藏高原研究院,四川 成都 610041)
拒盐型盐生植物小花碱茅根系具有高度木栓化的内皮层可能是其抵御盐胁迫的重要策略之一。本研究以盐胁迫下小花碱茅幼苗的根为材料,采用RT-PCR方法扩增到木栓质生物合成关键基因CYP86A的RNAi靶片段,以中间载体pHANNIBAL和植物表达载体pART27为基础,采用酶切连接结合In-Fusion的克隆方法成功构建了以35S启动子驱动,含PtCYP86A基因片段反向重复序列的RNAi植物表达载体pARC,为利用RNAi技术深入探究根系木栓化在小花碱茅拒盐中的功能奠定基础。
小花碱茅;木栓质;PtCYP86A;RNAi载体构建
盐生植物在长期适应逆境的过程中形成了较甜土植物更为强大的耐盐结构和生理机制,蕴藏着更丰富高效的抗逆基因资源。小花碱茅(Puccinelliatenuiflora)是一种优良的牧草,同时因具有很强的耐盐碱能力在改良作物抗性方面拥有巨大的潜力[1-3]。本课题组前期研究表明,限制根部Na+单向内流以减少Na+的净积累,从而维持很高的K+、Na+选择性是小花碱茅主要的耐盐机制[4]。此外,Peng等[5]通过对小花碱茅根横切面的X射线微区分析发现,Na+主要分布在内皮层细胞间隙,而K+主要分布在细胞内。解剖学研究也发现小花碱茅在盐胁迫下内皮层明显加厚[6-8]。因此,其高度木栓化的内皮层可能是阻断Na+从皮层进入中柱的关键。
木栓质在内皮层细胞壁的内表面沉积形成木栓层(suberin lamella),是植物根系两种重要的质外体屏障之一[9-10]。木栓质能有效阻止水分和溶质离子从质外体直接运输到内皮层原生质体,因此影响离子在根中跨细胞途径的径向运输,其屏障作用可能比凯氏带更有效[11-13]。C16~C20的ω-羟基化脂肪酸和α,ω-双羧基脂肪酸(简称α,ω-二酸)是构成木栓质的主要单体,是脂肪酸在细胞色素P450 ω-羟化酶(P450)的催化下形成的衍生物[14-19]。在拟南芥(Arabidopsisthaliana)和马铃薯(Solanumtuberosum)中分别证实了CYP86A1及其同源基因CYP86A33在木栓质单体合成中的重要作用[20-21]。
鉴于此,本研究在已克隆到的PtCYP86A基因序列的基础上,构建了以PtCYP86A基因为靶标的RNAi植物表达载体,以期为深入分析PtCYP86A在小花碱茅拒盐中的功能与作用奠定基础。
1 材料与方法
1.1 实验材料
植物材料为4周龄小花碱茅幼苗,种子、中间载体pHANNIBAL菌保、植物表达载体pART27菌保为兰州大学牧草逆境生理与基因工程实验室保存。试验时间为2013年5-10月。
1.2 主要试剂
RNAprep Pure植物总RNA提取试剂盒、DNA marker购自北京天根,M-MuLV第一链cDNA的合成试剂盒、UNIQ-10柱式DNA胶回收试剂盒、SanPrep柱式质粒小量抽提试剂盒购自上海生工,Phusion高保真DNA聚合酶购自美国Thermo公司,普通Taq酶、In-Fusion HD Cloning Kit、pMD19-T simple vector、T4 DNA连接酶以及各种限制性内切酶购自大连宝生物,大肠杆菌DH5α感受态细胞购自北京全式金,其他生化试剂均为进口或国产分析纯产品。
1.3 研究方法
1.3.1 材料培养 小花碱茅种子播撒于铺有吸水纸的纱网架上,置于白瓷盘中进行暗培养,待种子发芽整齐后浇灌1/2 Hoagland营养液[4]。约5 d更换一次营养液。培养室温度为25 °C,光照16 h/d,光强约为600 μmol/(m2·s),空气相对湿度控制在60%~80%。
1.3.2 总RNA的提取及cDNA第一链的合成 用150 mmol/L NaCl处理4周龄小花碱茅幼苗24 h,取其根系,加入液氮磨至粉末状,按照RNAprep Pure植物总RNA提取试剂盒的操作说明书提取总RNA,按照M-MuLV第一链cDNA合成试剂盒进行反转录反应。
1.3.3PtCYP86A基因RNAi靶位片段的扩增 参照Serra等[21]RNAi 靶片段的选择以及invitrogen网站的设计软件,选取1083~1620 bp (共538 bp)区段作为RNAi靶位区段,设计一对特异性引物并在上游引物上分别引入酶切位点XhoⅠ (C1-F)和XbaⅠ (C2-F),下游引物上分别引入酶切位点KpnⅠ(C1-R)和Hind Ⅲ(C2-R),分别进行PCR扩增。引物序列如下:
C1-F:CCGCTCGAGCTTCGACGAGCTGGACCG
C1-R:GGGGTACCCGCAGTGGTTGTCGGGAT
C2-F:GCTCTAGACTTCGACGAGCTGGACCG
C2-R:CCCAAGCTTCGCAGTGGTTGTCGGGAT
其中C1为正向插入片段,C2为反向插入片段(下划线为酶切位点序列)。
使用UNIQ-10柱式DNA胶回收试剂盒回收PCR扩增产物,将回收产物连接到pMD19-T simple载体,转化大肠杆菌E.coliDH5α感受态,蓝白斑筛选得到的阳性克隆经质粒PCR鉴定确认后,送至上海生工测序,使用DNAMAN 6.0对序列进行分析。
1.3.4 以PtCYP86A为靶标的RNAi载体构建 用XhoⅠ/KpnⅠ双酶切连有正向片段C1的pMD19-T simple vector载体,回收小片段(C1),与同样双酶切线性化的pHANNIBAL载体连接,转化E.coliDH5α感受态,得到重组质粒pHC1;然后用XbaⅠ/Hind Ⅲ双酶切连有反向片段C2的pMD19-T simple vector载体并回收小片段(C2),连接到XbaⅠ/Hind Ⅲ双酶切后的pHC1载体,得到以pHANNIBAL载体的内含子(PDK intron)部位作为中心、两侧含有反向重复序列的表达盒pHC1C2 (图1)。
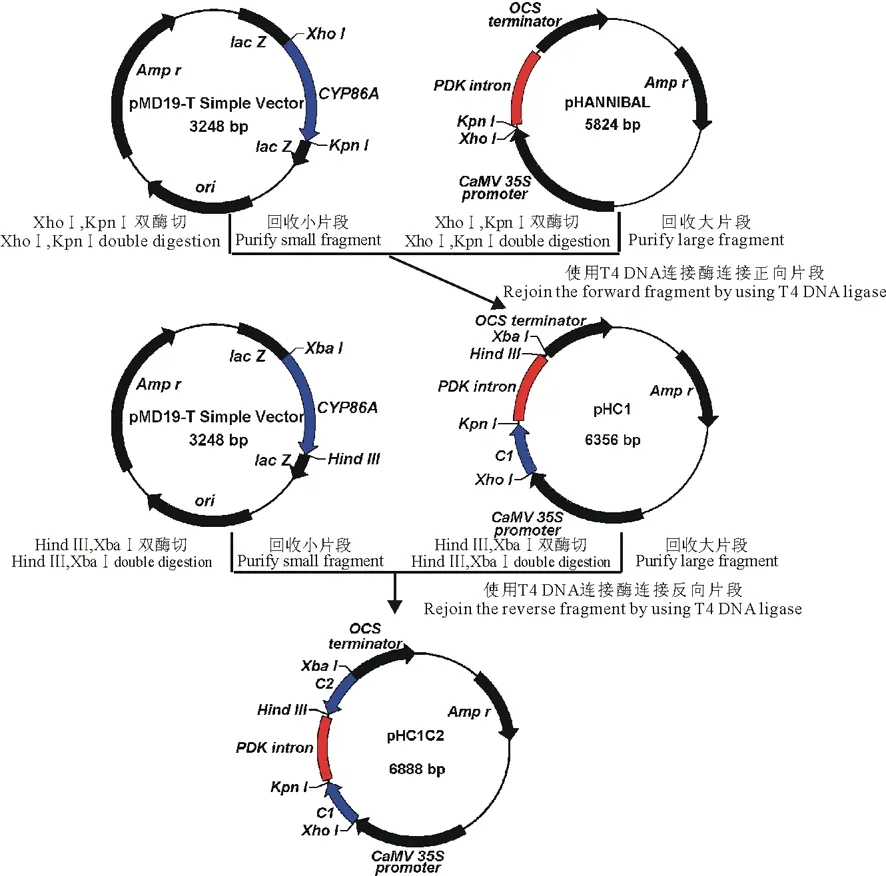
图1 中间载体pHC1C2的构建流程Fig.1 Flow diagram of construction of intermedium vector pHC1C2
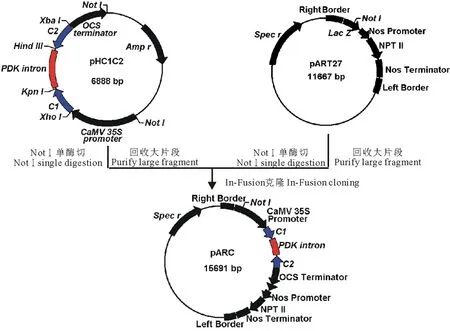
图2 PtCYP86A-RNAi表达载体pARC的构建流程Fig.2 Flow diagram of construction of PtCYP86A-RNAi expression vector pARC
用NotⅠ单酶切线性化pART27载体;同时用NotⅠ单酶切pHC1C2并回收纯化4044 bp的大片段,设计该片段的全长引物,并分别在上下游引物的5′端引入一段与线性化pART27载体末端互补的15 bp扩展序列,最终的引物序列如下:
P1:GACCTGCAGGCGGCCGCTCGACGAATTAATTCCAATCCC
P2:ATCACTAGTGCGGCCGCAGATTTAGGTGACACTATAG
采用高保真酶扩增目的片段,回收纯化,按照In-Fusion HD Cloning Kit说明书,将该片段和线性化pART27载体连接,得到具有小花碱茅CYP86A反向重复序列的RNAi重组载体pARC (图2)。
2 结果与分析
2.1 总RNA的提取及检测
以150 mmol/L NaCl处理下小花碱茅根系为材料提取的总RNA通过电泳检测,结果显示28S RNA和18S RNA条带清晰,前者亮度约是后者的2倍(图略),说明所提取的RNA完整性较好;经NanoDrop1000核酸蛋白检测仪测得OD260/OD280平均值为1.97,OD260/OD230平均值为2.13,表明RNA纯度较高,可用于RT-PCR扩增。
2.2PtCYP86A基因RNAi靶片段的扩增
以总RNA反转录所得到的第一链cDNA为模板,用特异引物C1和C2分别进行PCR扩增,得到了预期538 bp的特异条带(图3)。将该条带回收纯化后连接到pMD19-T simple载体上,转化E.coli.DH5α感受态。经蓝白斑筛选后提取质粒,通过PCR检测筛选阳性克隆。测序后分析表明,这两个序列与PtCYP86A基因相应区段同源性为100% (结果未显示),说明已成功扩增到目的片段。
2.3 以PtCYP86A为靶标的RNAi 载体构建
按照PtCYP86ARNAi植物表达载体的构建流程(图1和图2),通过酶切连接结合In-Fusion的方法,构建了具有PtCYP86A反向重复序列的RNAi植物表达载体pARC。通过质粒PCR检测鉴定出阳性克隆后(图4),根据pARC载体上的限制性酶切位点对其进行酶切分析,结果发现,经XbaⅠ/XhoⅠ双酶切后,在1893 bp处出现特异条带(图5,泳道2),与C1+C2+PDK intron大小相符;经XbaⅠ/KpnⅠ双酶切后,在1349 bp处出现特异条带(图5,泳道3),与C2+PDK intron片段大小相符。以上结果表明,以PtCYP86A为靶标的RNAi载体已构建成功,可用于农杆菌的转化。

图3 RT-PCR产物凝胶电泳图Fig.3 Agarose gel electrophoresis of RT-PCR products
M为DNA标记;1,2为RT-PCR产物。M indicates DNA marker;1,2 indicate RT-PCR products.
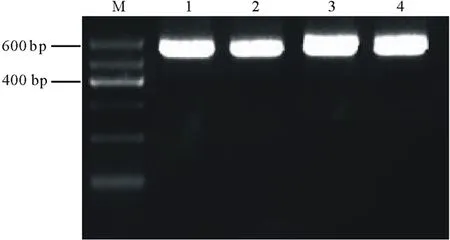
图4 pARC重组质粒阳性克隆的PCR鉴定Fig.4 PCR identification of the positive clones of pARC recombinant plasmids
M为DNA标记;1,2,3,4为阳性克隆。M indicates DNA marker;1,2,3,4 indicate positive clones.
3 讨论
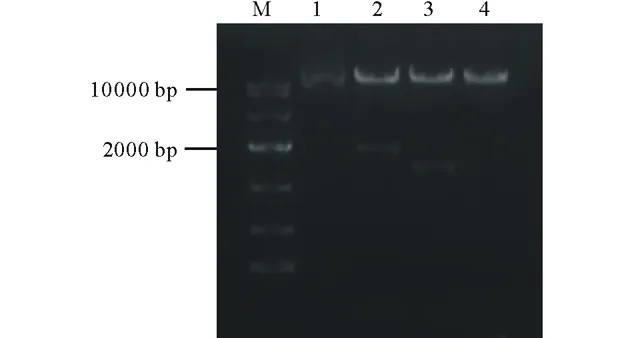
图5 pARC重组质粒的酶切验证Fig.5 Restrict digestion validation of the pARC recombinant plasmidM为DNA标记;1为环状质粒;2为XbaⅠ/XhoⅠ双酶切;3为XbaⅠ/KpnⅠ双酶切;4为线性质粒。M indicates DNA marker;1 indicates circular plasmid;2 indicates double digestion by XbaⅠ/XhoⅠ;3 indicates double digestion by XbaⅠ/KpnⅠ;4 indicates linearized plasmid.
RNAi技术与其他基因表达抑制技术相比,具有特异、高效、稳定等优点,是基因功能研究中最常用且重要的手段之一。该技术主要通过少量小干扰RNA (small-interference RNA,siRNA) 或双链RNA (double-stranded RNA,dsRNA)介导,特异降解目标序列的靶mRNA,从而抑制相应基因表达,属于典型的转录后基因沉默(post-transcriptional gene silencing,PTGS)[22]。在植物中,最长效稳定的手段是构建表达载体并通过农杆菌侵染的方法将dsRNA 转入植物体内,载体中连入的具有反向重复基因序列的结构在植物体内转录为mRNA后,自我互补形成hpRNA[23],进而导致植物内同源基因编码的mRNA 特异性降解。
本研究中,使用T4 DNA连接酶始终无法将NotⅠ酶切pHClC2后得到的大片段连接至NotⅠ单酶切后的双元载体pART27,甚至对线性化的pART27载体进行磷酸化处理后,仍得不到阳性克隆,最终通过In-Fusion的方法,对NotⅠ酶切pHClC2后得到的大片段进行PCR扩增,使用In-Fusion酶将纯化后的PCR产物克隆到目标载体pART27上,得到了阳性克隆。In-Fusion克隆方法与传统的T4连接酶相比,具有操作简便、连接效率高、适用性广等特点,其原理是向目的片段的引物5′端引入与线性化载体末端互补的15 bp同源序列,通过In-Fusion酶特异性识别该序列进而将目的片段与载体融合在一起,可以较轻松实现大片段的克隆。
靶基因干扰片段的选择是RNAi能否成功的关键,有研究表明,干扰效率具有长度依赖性,dsRNA 越长效果越明显:400~500 nt>200~300 nt>50~100 nt,内源性基因片段越短,对dsRNA的耐受性越高[24]。外源基因片段太短不利于载体的构建操作,过短的dsRNA 也不容易被核酸内切酶(Dicer)识别;而过长的片段在构建操作中易引起重组,并且结构并不稳定。因此,所选干扰片段长度应尽量控制在200~600 bp[25]。Elbashir等[26]认为靶基因序列应在基因转录起始位点下游100个核苷酸以后,且该段DNA的GC含量在40%~70%的范围内。为了防止沉默到其他非标靶基因,本研究参照Altschul等[27]以及Serra等[21]的设计原则,设计选择了小花碱茅CYP86A基因靠近3′端538 bp的非保守序列作为干扰片段,GC含量为66%,满足高效RNAi载体的条件,理论上将对PtCYP86A基因产生较好的沉默效果。
4 结论
本研究通过酶切连接结合In-Fusion的方法,成功构建了盐生植物小花碱茅木栓质合成关键基因PtCYP86A的RNAi载体,为研究木栓质在小花碱茅拒盐中的作用奠定了基础。
References:
[1] Wang S M,Zhu X Y,Shu X X.Studies on the characteristics of ion absorption and distribution inPuccinelliatenuiflora.Pratacultural Science,1994,3(1):39-43.王锁民,朱兴运,舒孝喜.碱茅离子吸收与分配特性的研究.草业科学,1994,3(1):39-43.
[2] Zhu X Y,Wang S M,Yan S G,etal.The research progresses of plant resistance to salt and its mechanism of resistance in the genusPuccinellia.Pratacultural Science,1994,11(3):9-15.朱兴运,王锁民,阎顺国,等.碱茅属植物抗盐性与抗盐机制的研究进展.草业科学,1994,11(3):9-15.
[3] Wang S M,Zheng W J,Ren J Z,etal.Selectivity of various types of salt-resistant plants for K+over Na+.Journal of Arid Environment,2002,52(4):457-472.
[4] Wang C M,Zhang J L,Liu X S,etal.Puccinelliatenuifloramaintains a low Na+level under salinity by limiting unidirectional Na+influx resulting in a high selectivity for K+over Na+.Plant Cell and Environment,2009,32(5):486-496.
[5] Peng Y H,Zhu Y F,Mao Y Q,etal.Alkali grass resists salt stress through high [K+] and endodermis barrier to Na+.Journal of Experimental Botany,2004,55:939-949.
[6] Stelzer R,Läuchli A.Salt- and flooding tolerance ofPuccinelliapeisonis.II.Structural differentiation of the root in relation to function.Zeitschrift für Pflanzenphysiologie,1977,84:95-108.
[7] Stelzer R,Läuchli A.Salt- and flooding tolerance ofPuccinelliapeisonis.III.Distribution and localization of ions in the plant.Zeitschrift für Pflanzenphysiologie,1978,88:437-448.
[8] Zhu Y J,Zhang Y,Hu Z Z,etal.Studies on the microscopic structure ofPuccinelliatenuifloraroots under different salinity stress.Grassland of China,2001,23(1):37-40.朱宇旌,张勇,胡自治,等.小花碱茅根适应盐胁迫的显微结构研究.中国草地,2001,23(1):37-40.
[9] Enstone D E,Peterson C A,Ma F.Root endodermis and exodermis:structure,function,and responses to the environment.Journal of Plant Growth Regulation,2002,21(4):335-351.
[10] Schreiber L.Transport barriers made of cutin,suberin and associated waxes.Trends in Plant Science,2010,15(10):546-553.
[11] Schreiber L,Hartmann K,Skrabs M,etal.Apoplastic barriers in roots:chemical composition of endodermal and hypodermal cell walls.Journal of Experimental Botany,1999,50:1267-1280.
[12] Ranathunge K,Schreiber L,Franke R.Suberin research in the genomics era-new interest for an old polymer.Plant Science,2011,180(3):399-413.
[13] Martinka M,Dolan L,Pernas M,etal.Endodermal cell-cell contact is required for the spatial control of Casparian band development inArabidopsisthaliana.Annals of Botany,2012,110(2):361-371.
[14] Werck-Reichhart D,Feyereisen R.Cytochromes P450:a success story.Genome Biology,2000,1(6):1-9.
[15] Nawrath C.The biopolymers cutin and suberin.The Arabidopsis Book,2002,1:e0021.
[16] Franke R,Briesen I,Wojciechowski T,etal.Apoplastic polyesters inArabidopsissurface tissues-a typical suberin and a particular cutin.Phytochemistry,2005,66(22):2643-2658.
[17] Beisson F,Li-Beisson Y,Bonaventure G,etal.The acyltransferase GPAT5 is required for the synthesis of suberin in the seed coat and root of Arabidopsis.Plant Cell,2007,19(1):351-368.
[18] Franke R,Schreiber L.Suberin-a biopolyester forming apoplastic plant interfaces.Current Opinion in Plant Biology,2007,10(3):252-259.
[19] Pollard M,Beisson F,Li Y,etal.Building lipid barriers:biosynthesis of cutin and suberin.Trends in Plant Science,2008,13(5):236-246.
[20] Höfer R,Briesen I,Beck M,etal.TheArabidopsiscytochrome P450CYP86A1 encodes a fatty acid omega-hydroxylase involved in suberin monomer biosynthesis.Journal of Experimental Botany,2008,59(9):2347-2360.
[21] Serra O,Soler M,Hohn C,etal.CYP86A33-targeted gene silencing in potato tuber alters suberin composition,distorts suberin lamellae,and impairs the periderm’s water barrier function.Plant Physiology,2009,149(2):1050-1060.
[22] Fire A,Xu S,Montgomery M K,etal.Potent and specific genetic interference by double-stranded RNA inCaenorhabditiselegans.Nature,1998,391:806-811.
[23] Waterhouse P M,Graham M W,Wang M B.Virus resistance and gene silencing in plants can be induced by simultaneous expression of sense and antisense RNA.Proceedings of the National Academy of Sciences,1998,95(23):13959-13964.
[24] Hammond S M,Bernstein E,Beach D,etal.An RNA directed nuclease mediates post-transcriptional gene silencing inDrosophilacells.Nature,2000,404:293-296.
[25] Burch-Smith T M,Miler J L.PTGS approaches to large-scale functional genomics in plants[M]//Hannon G,ed.RNAi:A Guide to Gene Silencing.New York:Cold Spring Harbor Laboratory Press,2003:243-263.
[26] Elbashir S M,Lendeckel W,Tuschl T.RNA interference is mediated by 21- and 22- nucleotide RNAs.Genes and Development,2001,15(2):188-200.
[27] Altschul S F,Gish W,Miller W,etal.Basic local alignment search tool.Journal of Molecular Biology,1990,215(3):403-410.
Construction of RNAi expression vector ofCYP86Agene in halophytePuccinelliatenuiflora
WANG Pei1,2,CUI Yan-Nong1,GAO Li1,WANG Suo-Min1*
1.StateKeyLaboratoryofGrasslandAgro-ecosystems,CollegeofPastoralAgricultureScienceandTechnology,LanzhouUniversity,Lanzhou730020,China;2.InstitutionofQinghai-TibetanPlateau,SouthwestMinzuUniversity,Chengdu610041,China
The salt-excluding halophytePuccinelliatenuifloraexhibits a highly suberized endodermis in the root,which may contribute to its salt tolerance.In this study,the RNAi target fragment ofCYP86A,a key gene in suberin biosynthesis,was amplified by RT-PCR from roots ofP.tenuifloraseedlings under salt stress.Then based on the intermediate vector pHANNIBAL and the plant expression vector pART27,the RNAi plant expression vector pARC,which contained inverted repeats ofPtCYP86Aand was driven by the 35S promoter,was constructed successfully by restriction enzyme digestion,ligation and In-Fusion cloning.The work might be a good basis for further investigating the roles of roots suberization in Na+exclusion inP.tenuifloraby RNAi technique.
Puccinelliatenuiflora;suberin;PtCYP86A;construction of RNAi vector
10.11686/cyxb2017076 http://cyxb.lzu.edu.cn
王沛,崔彦农,高丽,王锁民.盐生植物小花碱茅CYP86A基因的RNAi载体构建.草业学报,2017,26(6):105-110.
WANG Pei,CUI Yan-Nong,GAO Li,WANG Suo-Min.Construction of RNAi expression vector ofCYP86Agene in halophytePuccinelliatenuiflora.Acta Prataculturae Sinica,2017,26(6):105-110.
2017-03-02;改回日期:2017-04-10
国家自然科学基金(31470503),教育部博士点基金优先发展领域项目(20130211130001)和甘肃省科技支撑项目(144FKCA058)资助。
王沛(1987-),男,甘肃庆阳人,在读博士。E-mail:wp_10@lzu.edu.cn
*通信作者Corresponding author.E-mail:smwang@lzu.edu.cn

