磁性富集荧光法检测大肠杆菌
杨宇,牛承岗,曾光明
磁性富集荧光法检测大肠杆菌
杨宇,牛承岗,曾光明
(湖南大学环境科学与工程学院,环境生物与控制教育部重点实验室,湖南长沙,410082)
为研究大肠杆菌的高灵敏快速检测方法,建立一种基于萘酰亚胺标记的DNA探针和磁性四氧化三铁氧化石墨烯分离的大肠杆菌O157:H7检测方法。将标记萘酰亚胺的ssDNA(单链DNA)作为捕获探针吸附在磁性氧化石墨烯表面,当目标ssDNA存在时,捕获探针与之部分杂交,利用磁场将目标ssDNA捕获并富集。然后加入释放探针完成杂交,使标记荧光的捕获探针从磁性氧化石墨烯的表面释放出来,通过测定溶液荧光强度可以实现大肠杆菌O157:H7的高灵敏检测。实验结果表明:在一定条件下,O157:H7数量的对数值和荧光强度与空白值荧光强度的比值(/0)呈线性关系,线性范围为150~1.5×106个/mL,经过富集后检出限可达100 个/mL。该方法灵敏度高,检出限低,耗时较短,操作简单,为致病菌的高灵敏检测提供新思路。
大肠杆菌O157:H7;磁性氧化石墨烯;富集;DNA杂交;萘酰亚胺
肠出血性大肠杆菌(,EHEC)是人类食源性以及水源性病原体,食用被该菌污染的食物会导致腹泻和肠炎,严重时将导致出血性结肠炎和溶血性尿毒症综合症,甚至死亡[1]。EHEC以大肠杆菌O157:H7血清型为代表菌株[2]。建立一种快速、准确检测大肠杆菌方法,对环境监测和临床医学流行病学诊断有现实意义[3]。目前,检测大肠杆菌的方法主要有传统方法、免疫学检测方法和分子生物学方法。传统方法依赖于观察表型特性,该方法耗时长,不适用于快速检测[4]。免疫学检测法主要有酶联免疫吸附测定(ELISA)等方法,ELISA能对进行准确、快速定量,但阳性结果需用培养法确认[5]。分子生物学方法主要根据大肠杆菌DNA中的特异性基因来进行定性定量分析,分为聚合酶链式反应技术(PCR技术)和DNA探针检测等方法[6−7]。PCR技术可以将部分DNA序列进行复制,信号放大,故灵敏度较高[8]。近几年,磁性纳米材料发展迅速,其中,磁性氧化石墨烯纳米材料不仅具有优良的磁学性能、良好的生物相容性以及水溶性等优点,而且可以强烈地吸附ssDNA,且具有较强的荧光猝灭能力[9−11]。为了进一步研究DNA探针检测方法在检测细菌方面的应用,提高检测方法的灵敏度,本实验结合磁性氧化石墨烯和DNA探针检测方法的优点,采用水溶性较好的N-羟乙基-4-N(氨乙基)-1,8-萘酰亚胺(4-N- aminoethyl-N-hydroxyethyl-1,8-naphthalimide,AHA)作为荧光物质标记大肠杆菌O157:H7的一段特异性ssDNA,将其作为捕获探针C*,吸附在磁性四氧化三铁/氧化石墨烯(MGO)表面。通过捕获探针先后与目标ssDNA和将捕获探针从MGO表面释放出来的ssDNA(释放探针R)杂交,达到对O157:H7捕获、富集和检测的目的。实验建立了用于实际样品中大肠杆菌O157:H7快速检测方法,并对实验条件进行了优化,该方法具有灵敏度高、快速简便、经济、特异性好等优点。
1 实验
1.1 仪器与试剂
仪器为:高压蒸汽灭菌器(LDZX−30 FBS,上海申安医疗器械厂);酸度计(pHS−3B,上海仪电科学仪器股份有限公司);荧光分光光度计(FluoroMax−4 Spetroflurometer,法国HORIBA Jobin Yvon公司)。
试剂为:PBS(phosphate buffered saline)缓冲液配制:3.05 g NaH2PO4·12H2O,8.00 g NaCl,0.20 g KH2PO4和0.20 g KCl,溶解于1 L超纯水中(pH=7.2)。
营养肉汤培养基配制:10.0 g蛋白胨,5.0 g牛肉膏,5.0 g NaCl溶解于1 L超纯水中(pH为7.0~7.2)。实验所用培养基、缓冲液和超纯水在使用前均灭菌。
1.2 捕获探针,释放探针和目标ssDNA碱基序列
参考GenBank公布的大肠杆菌O157:H7的特异性基因片段及相关文献介绍,本实验设计了3条探针[12]。DNA序列采用软件Primer Premier 5.0进行设计,并用Ribosomal Database Project II 和NCBI Blast 2数据库提供的Probe Match软件检测序列的特异性。设计的DNA序列如下,捕获探针C(capture probe C):5′COOH-TTTTTTTTTTCATAGAACGCTAATAAGAAGTCCAGTG-3′;释放探针R(release probe R):5′-AAAAAAAAAA-3′;目标碱基序列T(target DNA T):5′-CACTGGACTTCTTATTACCGTTCTATG-3′。其中,捕获探针C分为2段:一段与目标碱基序列T互补,另一段与释放探针R互补。所有DNA序列委托上海生物工程股份有限公司合成。
1.3 磁性氧化石墨烯的合成
本文所采用的MGO分2步合成:第1步合成GO,第2步GO与FeCl3在一定条件下反应合成MGO。
GO根据Hummers方法制备[13]。
MGO的制备:称取0.5 g用上述方法制备的GO,加入到80 mL乙二醇中超声分散3 h。在室温下加入1.6 g FeCl3和3.2 g醋酸钠,搅拌30 min,将溶液转移到100 mL聚四氟乙烯内衬的不锈钢高压釜中200 ℃反应6 h,然后自然冷却到室温。离心得到黑色固体,用乙醇洗涤数次,真空干燥得MGO[14−15]。
1.4 捕获探针的荧光标记
N-羟乙基-4-N(氨乙基)-1,8-萘酰亚胺(AHA)由本课题组合成,该化合物具有强烈的荧光、较好的水溶性和热稳定性。首先,将1.3 mg AHA溶于适量PBS缓冲液中,然后加入50 nmol/L探针C和200 mg EDC,接着添加PBS缓冲液将溶液定量至10 mL。震荡摇晃10 min后加入120 mg NHS,继续震荡摇晃2 h。接着放置在冰箱4 ℃过夜,让未反应的EDC水解失活。最后透析数次,除去未连接的AHA,得荧光标记的捕获探针C*[16]。
1.5 菌株的培养与细菌数量
大肠杆菌O157:H7菌株(No. NCTC 12900)购于中国典型培养物保藏中心(武汉,中国),在营养肉汤培养基中37 ℃恒温培养24 h。
本实验使用平板菌落计数法对培养24 h的O157:H7进行定量检测。将液体培养基中的O157:H7稀释10,1×102,1×103,1×104,1×105,1×106,1×107和1×108倍,分别取100 μL溶液涂布在已灭菌的伊红美蓝培养基(EMB)表面,恒温培养24 h,之后进行菌落计数,确定细菌数量。
1.6 大肠杆菌O157:H7DNA提取
使用GenerayTM基因提取试剂盒提取经过24 h培养的O157:H7的DNA。DNA提取出来后,用PBS缓冲液将其稀释为若干个浓度备用,为后面的杂交试验做准备。
1.7 杂交试验及荧光检测
本实验用合成的T对各实验条件进行了优化,并且探讨了实验的可行性。在最佳实验条件下,用大肠杆菌提取得到的DNA验证了该方法的实际应用效果。首先,50 nmol/L 捕获探针C*与0.18 g/L MGO混合1 h,得C*-MGO溶液。同时,将用试剂盒提取出来的DNA加热到95 ℃保持5 min,然后迅速浸泡在冰水里5 min,以打开双链,得到目标ssDNA T。接着将不同浓度的T分别加到C*-MGO溶液中,放置在50.6 ℃恒温培养箱中培育2.5 h完成部分杂交。接着放置在磁场中10 min,收集磁性颗粒,移除原溶液,加入200 nmol/L 释放探针R,此时溶液体积为原体积的1/4,相当于富集4倍,在室温下杂交2 h,完成杂交试验。杂交后探针C*从MGO表面释放,磁性分离石墨烯,通过检测荧光变化,可对O157:H7进行定量分析。
所有实验均作3个平行样,灭菌超纯水代替T为空白对照。荧光仪的仪器参数:激发波长ex=450 nm,发射波长em=540 nm,激发狭缝和发射狭缝均为10 nm。
2 结果与讨论
2.1 实验原理
在MGO溶液中,由于MGO对ssDNA强烈的吸附作用,当捕获探针C*存在时,C*吸附于MGO表面。此时标记于C*一端的AHA与MGO发生荧光能量共振转移,荧光猝灭,如图1(a)所示。接着加入目标ssDNA T,由于捕获探针C*的一段DNA序列与T互补,C*与T完成部分杂交,双链部分脱离MGO表面,如图1(b)所示。接着利用磁场收集磁性颗粒,移除原溶液,再加入少量释放探针R溶液进行杂交。C*完全脱离MGO表面,荧光恢复。磁性分离石墨烯后通过检测溶液荧光变化对O157:H7的进行定量分析。如图1(c)所示。
2.2 MGO对捕获探针的吸附
图2所示为不同质量浓度的MGO下荧光猝灭光谱。在本实验中,MGO作为一种荧光猝灭剂,其对捕获探针C*的吸附情况可以根据荧光强度的变化来判断。本实验研究了不同质量浓度MGO对荧光强度猝灭情况,并对吸附时间做出优化。捕获探针C*浓度取50 nmol/L,当MGO质量浓度在0~0.27 g/L之间变化时,荧光猝灭效果随浓度增大而增强,如图2所示。图3显示当质量浓度为0.18 g/L时,猝灭效率高达96 %。本实验选取0.18 g/L为最佳吸附质量浓度。
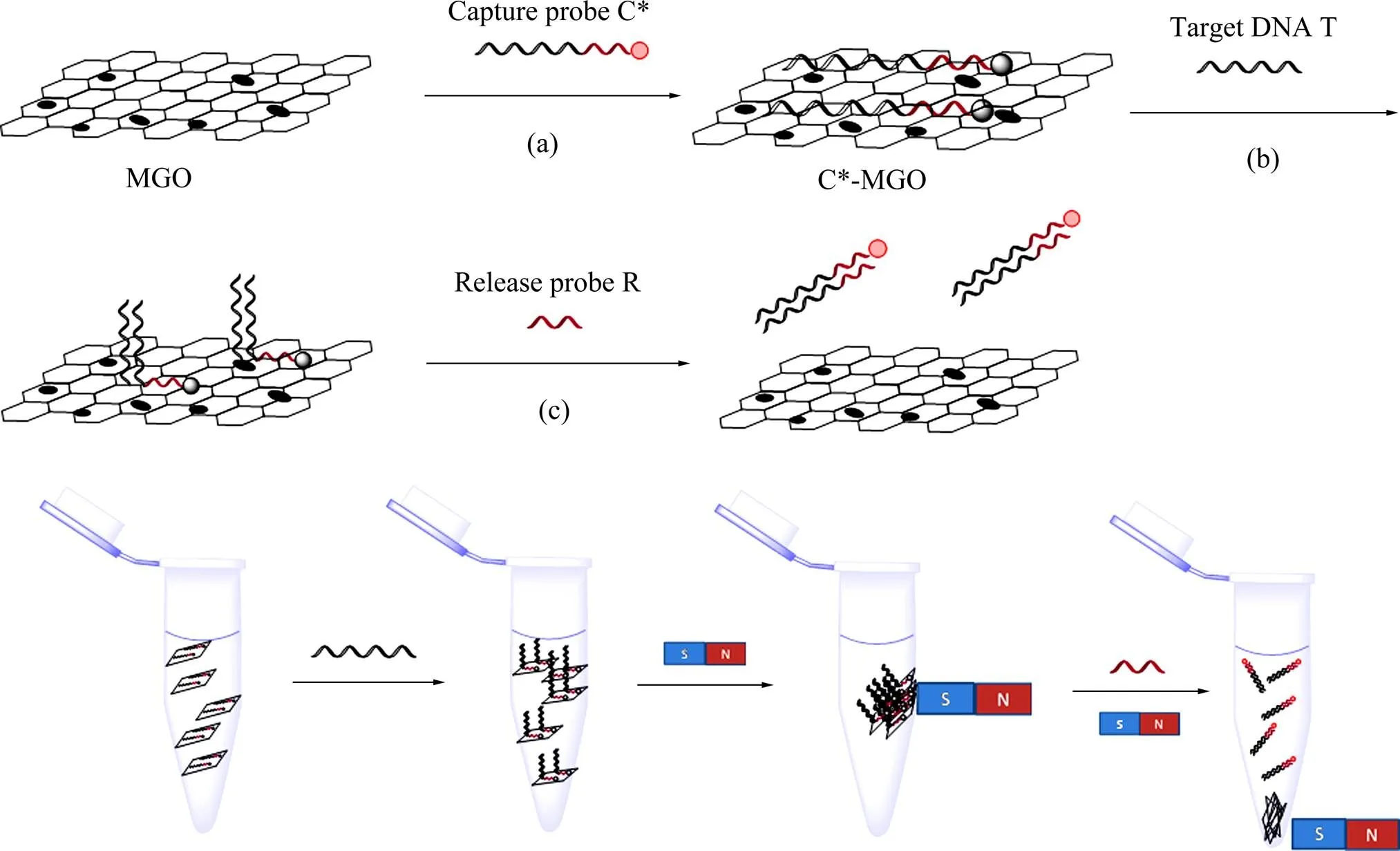
图1 基于N-羟乙基-4-N(氨乙基)-1,8-萘酰亚胺荧光探针与磁性氧化石墨烯检测大肠杆菌O157:H7示意图

质量浓度/(mg∙mL−1): 1—0; 2—0.03; 3—0.05; 4—0.12; 5—0.18; 6—0.21。

图3 不同质量浓度MGO下荧光猝灭效率图
本实验还探讨MGO对捕获探针C*吸附效果达到最优所需要的时间。将0.18 g/L MGO与50 nmol/L C*混合,记录荧光强度,结果见图4。在30 min以内,荧光强度迅速减小。30 min到1 h阶段,荧光强度缓慢减小到4%,此后基本保持不变。本实验选取1 h为最佳捕获探针C*吸附时间。

图4 荧光强度随吸附时间的变化
2.3 杂交温度与时间的优化
实验首先对杂交温度进行了优化。捕获探针C*与目标单链T杂交的温度从最适复性温度(or=m− 25)、苛刻复性温度s=m−10或s=m−15及非苛刻复性温度ns=m−30或ns=m−35中选择。本实验中研究的探针m=60.6 ℃,故or=35.6 ℃,s=50.6 ℃以及ns= 25.6 ℃。实验在对上述3种温度下的杂交情况进行研究,MGO质量浓度为0.18 mg/mL,杂交时间为2.5 h,富集倍数为4,C*-MGO浓度为50 nmol/L,T浓度为10 nmol/L。实验结果见图5,在50.6 ℃荧光强度达到最高。原因可能是低温下高分子量的DNA难以很好地展开,导致不能完全杂交配对[17]。

图5 杂交温度优化
杂交时间决定着杂交是否完全,是杂交反应的重要影响因子之一。本实验将10 nmol/L T与50 nmol/L C*-MGO在50.6 ℃培育杂交,每隔0.5 h记录荧光强度(富集倍数为4)。如图6所示,反应开始后,荧光强度随反应时间快速上升,2.0 h后增长变缓,2.5 h后,荧光强度基本不变。而对照的空白实验中,荧光值一直较为平稳。故本实验选取杂交时间为2.5 h较为 合适。

1—实验组;2—空白对照组。
2.4 富集倍数对实验结果的影响
由于DNA含量低,检测困难,本实验利用MGO将待测目标物捕获并富集,通过对待测目标物的富集,进而放大荧光强度,提高灵敏度。本实验对富集倍数进行了优化。将50 nmol/L C*-MGO与10 nmol/L T在50.6 ℃杂交2.5 h。检测富集1倍、2倍、3倍、4倍、5倍的荧光强度,结果见图7。由图7可见:随着富集倍数增加,荧光强度显著增加,空白值缓慢增加。空白值略有增加的原因可能是随着富集倍数增加,溶液体积缩小,背景荧光物质浓度增加,检测出的荧光强度也随之增加。图8所示为实验组的荧光强度与空白对照组荧光强度的比值(/0)变化情况。由图5可见:在4倍富集时,达到最优。

图7 荧光强度随富集倍数的变化

图8 荧光强度比值(F/F0)随富集倍数的变化
2.5 检测方法的线性范围与灵敏度
本实验对从上海生工购买的目标DNA序列和从本实验培养的O157:H7提取出的DNA进行了研究,所有实验均在最优实验条件下进行。图9和图10所示为随着浓度增大(0~500 nmol/L),目标序列ssDNA T的/0变化情况。实验在T浓度为50 pmol/L到30 nmol/L呈现出良好的线性关系(=0.229+1.729,2=0.989 8,其中,为目标DNA的浓度,为/0)。本实验空白组相对标准偏差为4.4%,根据空白值3倍的标准偏差计算[18],检出限为50 pmol/L。
实验对培养的O157:H7进行了检测。在最优条件下,测得的荧光强度比(/0)随相应O157:H7数量的变化曲线如图11所示。随着O157:H7数量的增加,荧光强度逐渐增大。实验结果表明:O157:H7数量在150~1.5×106个/mL范围内,O157:H7数量的对数lg(个/mL)和/0呈良好的线性关系,线性方程为=1.947 9−2.798 1(为细菌数量的对数,为/0),2=0.991 0,空白值相对标准偏差为3.5%,检出限为100 个/mL。

浓度/(nmol∙L−1): 1—0; 2—0.5; 3—4; 4—17; 5—35; 6—100; 7—200; 8—500。
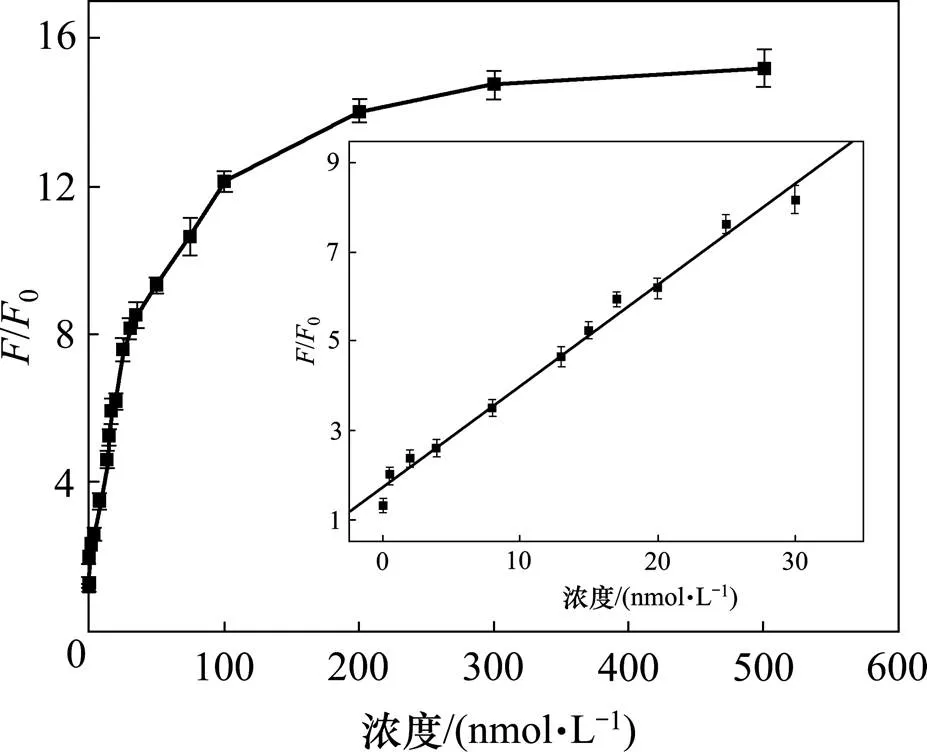
图10 不同浓度T和荧光强度比值(F/F0)的关系

O157:H7含量/(个∙mL−1): 1—1.5×107; 2—1.5×106; 3—1.5×1074; 4—1.5×103; 5—15。
3 结论
1) 磁性富集荧光法检测大肠杆菌的体系中,最优试验条件如下:MGO质量浓度为0.18 g/L,吸附时间为1.0 h,DNA探针杂交时间2.5 h,杂交温度50.6 ℃,富集倍数为4倍。
2) 在检测大肠杆菌O157:H7的实际应用中,该方法灵敏度高、检出限低、耗时较短,不需要PCR扩增,不需要通过其他手段可实现信号放大,可在5.0 h内检出细菌含量为100 个/mL。
3) 该方法为O157:H7的检测提供了一种新方法,为其他致病菌的检测提供了新思路,在环境监测和食品卫生领域具有潜在的应用前景。
[1] SCOTER S, ALDRIDGE M. Validation of a method for the detection of E.coli 0157:H7 in foods[J]. Food Control, 2000, 1l(2): 85−95.
[2] WIRTH T, FALUSH D, LAN R T, et al. Sex and virulence in Escherichia coli: an evolutionary perspective[J]. Molecular Microbiology, 2006, 60(5): 1136−1151.
[3] HUANG Jin, WU Yanrong, CHEN Yan, et al. Pyrene-excimer probes based on the hybridization chain reaction for the detection of nucleic acids in complex biological fluids[J]. Angewandte Chemie International Edition, 2011, 50(2): 401−404.
[4] SIDARI R, CARIDI A. Methods for detecting enterohaemorrhagic escherichia coli in food[J]. Food Reviews International, 2011, 27(2): 134−153.
[5] FENG Min, YONG Qianqian, WANG Wenbing, et al. Development of a Monoclonal antibody-based ELISA to detect Escherichia coli O157:H7[J]. Food and Agricultural Immunology, 2013, 24 (4): 481−487.
[6] SONG Zhiling, ZHAO Xuhua, LIU Weina, et al. Magnetic graphitic nanocapsules for programmed DNA fishing and detection[J]. Small, 2013, 9(6): 951−957.
[7] 蒋绍妍, 王文星, 薛向欣, 等. 利用PCR-DGGE分析茂名油页岩矿区土壤细菌群落组成[J]. 中南大学学报(自然科学版), 2015, 46(12): 4719−4724. JIANG Shaoyan, WANG Wenxing, XUE Xiangxin, et al. Composition of bacteria in soil from oil shale mine in Maoming using PCR-DGGE technique[J]. Journal of Central South University (Science and Technology), 2015, 46(12): 4719−4724.
[8] VO V G, THUY T N, THI K O N, et al. A novel multiplex PCR method for the detection of virulence-associated genes of Escherichia coli O157:H7 in food[J]. 3 Biotech, 2015, 6(5): 5−13.
[9] GEIM A K. Graphene: status and prospects[J]. Science, 2009, 324: 1530−1534.
[10] 肖建田, 胡大胜, 陈灵娜, 等. 多空位缺陷和硼氮杂质对锯齿型石墨烯纳米带电子结构的影响[J]. 中南大学学报(自然科学版), 2012, 43(11): 4361−4366. XIAO Jiantian, HU Dasheng, CHEN Lingna, et al. Effect of defect and boron/nitrogen doping on electronic properties of zigzag graphene nanoribbons[J]. Journal of Central South University (Science and Technology), 2012, 43(11): 4361−4366.
[11] HUANG Dawei, NIU Chenggang, WANG Xiaoyu, et al. “Turn-on” fluorescent sensor for Hg2+based on single-stranded DNA functionalized Mn:CdS/ZnS quantum dots and gold nanoparticles by time-resolved mode[J]. Analytical Chemistry, 2013, 85(2): 1164−1170.
[12] NICOLE T P, GUY P, VALERIE B, et al. Genome sequence of enterohaemorrhagic Escherichia coli O157:H7[J]. Nature, 2001, 409: 529−533.
[13] HUMMERS J W S, OFFEMAN R E. Preparation of graphitic oxide[J]. Journal of the American Chemical Society, 1958, 80(6): 1339−1339.
[14] MA Xinxing, TAO Huiquan, YANG Kai, et al. A functionalized graphene oxide-iron oxide nanocomposite for magnetically targeted drug delivery, photothermal therapy, and magnetic resonance imaging[J]. Nano Research, 2012, 5(3): 199−212.
[15] SUN Hongmei, CAO Linyuan, LU Lehui. Magnetite/reduced graphene oxide nanocomposites: one step solvothermal synthesis and use as a novel platform for removal of dye pollutants[J]. Nano Research, 2011, 4(6): 550−562.
[16] FAHLMAN R P, SEN D. DNA conformational switches as sensitive electronic sensors of analytes[J]. Journal of the American Chemical Society, 2002, 124(17): 4610−4616.
[17] RUAN Min, NIU Chenggang, ZENG Guangming, et al. Rapid detection of Staphylococcus aureus via a sensitive DNA hybridization assay based on a long-lifetime luminescent europium marker[J]. Microchimica Acta, 2011, 175(1/2): 105−112.
[18] MAO Xiaole, YANG Liju, SU Xiaoli, et al. A nanoparticle amplification based quartz crystal microbalance DNA sensor for detection of Escherichia coli O157:H7[J]. Biosens Bioelectron, 2006, 21(7): 1178−1185.
(编辑 陈爱华)
Fluorescent detection ofbased on magnetic enrichment
YANG Yu, NIU Chenggang, ZENG Guangming
(Key Laboratory of Environmental Biology Pollution Control, Ministry of Education, School of Environmental Science Engineering, Hunan University, Changsha 410082, China)
A novel sensitive assay was investigated for the highly sensitive and rapid detection ofO157:H7. It was based on magnetic Fe3O4/graphene oxide (MGO) and DNA probes which was modified by 4-N-aminoethyl-N- hydroxyethyl-1,8-naphthalimide (AHA). First, the ssDNA labelled with AHA was employed as a capture probe and adsorbed on the surface of MGO. When the target ssDNA appeared, capture probe hybridized with it in part. Then target ssDNA was fished and enriched via external magnetization. Afterwards, a solution of release probe was added to the magnetic particles to complete the hybridization which resulted in separating the AHA from MGO. The results show that the linear relationship is found between logarithm value of E.coli and the ratio of fluorescence intensities (/0) from 150 to 1.5×106/mL. The detection limit is 100 cfu/mL. The highly sensitive, rapid and simple means is demonstrated to be a noble tool for the detection of pathogenic bacteria.
O157:H7; magnetic Fe3O4/graphene oxide; enriching; DNA hybridization; 4-N-aminoethyl- N-hydroxyethyl-1,8-naphthalimide
10.11817/j.issn.1672-7207.2017.05.002
X831;X839.2
A
1672−7207(2017)05−1134−07
2016−07−22;
2016−09−04
国家自然科学基金资助项目(51541801) (Project(51541801) supported by supported by the National Natural Science Foundation of China)
牛承岗,博士,教授,从事分析化学和水污染处理研究;E-mail: cgniu@hnu.edu.cn

