MicroRNA通用探针法的建立及布鲁氏菌病患者血浆microRNA-146a的检测
程慧敏,魏青青,张 欢,刘金玲,韩小虎,王大力,王兴龙
MicroRNA通用探针法的建立及布鲁氏菌病患者血浆microRNA-146a的检测
程慧敏1,2,魏青青2,张 欢2,刘金玲2,韩小虎2,王大力3,王兴龙1
目的 建立一种通用、敏感、特异的microRNA检测方法,用于检测布鲁氏菌病患者血浆microRNA-146a的表达,探讨其作为诊断标识的可能性。方法 以microRNA-146a模拟物为待检物,通过正交实验设计,对退火温度、探针浓度、试剂盒选择进行优化,建立特异敏感的通用检测方法;从布病患者和正常健康人血浆样本提取RNA,应用通用探针法进行检测,比较布病患者血浆microRNA-146a的表达变化。结果 建立了优化的通用探针法,与染料法相比特异性更强、扩增范围更大,具有通用检测能力。应用通用探针法检测布鲁氏菌病患者血浆microRNA-146a,发现布病患者血浆中microRNA-146a的表达被抑制(P<0.01),表明其可能作为布病诊断的分子标识。结论 经优化的通用探针法是一种通用、敏感、特异的检测方法,可用于microRNA的检测,microRNA-146a可能作为布病诊断的标识分子。
通用探针法;正交设计;microRNA-146a;布鲁氏菌病
microRNA (miRNA)是一类由内源基因编码的长度约为19-22 个核苷酸的非编码单链RNA分子,可以作为疾病特征性标识[1]。但因其片段较短,易降解且表达量不高,多采用荧光定量法进行检测。不同microRNA同源性较高[2],探针法的特异性远高于染料法,但是荧光定量探针法在检测目的基因时,需要针对每个microRNA设计探针进行检测,实验成本高,操作要求高[3],在临床检测中受到一定限制。本研究在TaqMan荧光定量PCR的基础上建立一种通用探针法,通过设计新的通用探针和引物,使用正交设计法对多因素进行优化,不仅能扩大检测范围和灵敏性,也为荧光定量法microRNA检测条件的优化提供参考[4-6]。最后应用通用探针法对布病患者血浆中的microRNA-146a进行检测,探讨其作为诊断标识的可能性。
1 材料与方法
1.1 模拟物合成与样本准备 模拟物由上海吉玛公司合成,序列为Has-microRNA-146a-5p(5′-UGAGAACUGAAUUCCAUGGGUU-3′),12 000 r/min离心5 min,将模拟物稀释至0.2 μmol/L。布鲁氏菌病患者血浆样本来源于黑龙江农垦总局总医院,试管凝集试验进行血清学检测,抗体滴度在1∶50以上视为阳性,健康志愿者血浆样本来源于中国疾病预防控制中心鼠疫布病防治基地。收集清晨空腹全血样本2 mL,采用枸橼酸钠抗凝管,4 ℃静置4 h后4 000 g/min离心5 min,小心分离上层血浆后保存于-80 ℃。
1.2 RNA提取与反转录 RNA提取采用传统trizol法(Invitrogen,TRIzol○R Reagent),取200 μL血浆,加入相应体积trizol后,震荡混匀,静置5 min。加入40 μL氯仿,震荡,静置5 min后4 ℃ 12 000 g 25 min。吸去上层至新管,加200 μL异丙醇-20 ℃过夜,4 ℃ 12 000g离心 25 min弃上清。用200 μL 75%酒精洗涤RNA沉淀一次,使RNA重悬,4 ℃ 12 000 g离心15 min。弃上清,倒置控干酒精,加15 μL DEPC水溶解,-80 ℃保存。超微量紫外分光光度计(NanoDrop2000)测定RNA浓度均>100 ng/μL,OD268/OD280=1.8-2.0。
将20份健康志愿者血浆样本RNA各取2 μL制成混合RNA样本。将混合RNA样本、布鲁氏菌病患者血浆RNA样本和健康志愿者血浆RNA样本各20份分别加Poly(A)尾,布鲁氏菌病患者和健康志愿者加尾产物逆转录,逆转录严格按照天根生化科技(北京)有限公司miRcute miRNA cDNA第一链合成试剂盒操作说明书进行。逆转录引物序列:5′-CAG TGC AGG GTC CGA GGT CAG AGC CAC CTG GGC AAT TTT TTT TTT TVN-3′,由Takara公司合成。反应在金属浴(卡尤迪H2O3-100C)中进行,完成后cDNA保存于-20 ℃。
1.3 通用探针法建立与条件的优化 建立通用探针法,microRNA-146a上游引物序列:5′-TGA GAA CTG AAT TCC ATG GGT T-3′,通用下游引物序列:5′-CAG TGC AGG GTC CGA GGT-3′,通用探针:5′-FAM-CAG AGC CAC CTG GGC AAT TT-BHQ1-3′[7],序列均由Takara公司合成。
采用正交设计,对通用探针法的反应体系和反应程序进行优化,主要包括试剂盒、退火温度和探针浓度。与普通PCR不同,多数荧光定量PCR试剂盒多将Taq酶,buffer,dNTPs,MgCl2等制成混合溶液,因为混合溶液配比不同,Taq酶不同等,故扩增效率也不同。本实验将不同试剂盒混合溶液作为一个可变因素[8],结合温度、探针浓度共3因素,选定4个水平。以microRNA-146a为例,对浓度为0.2×10-2μmol/L模拟物microRNA-146a进行扩增,产物长66 bp。设计因素及其水平,见表1。[L16(4)3]正交设计表,见表2。实时荧光定量PCR反应液购于康维世纪的GoldStar TaqMan Mixture (Low ROX),Takara公司的Premix Ex Taq(Probe qPCR)试剂盒,TRANSGEN biotech公司的2×EasyTaq PCR SuperMix试剂盒,天根生化科技公司的SuperReal PreMix(Probe)。将反应体系缩小至10 μL,反正程序按照试剂盒说明书进行。扩增反应在美国Bio-Rad公司的IQ5实时定量PCR仪上进行,每次试验进行3次独立重复实验。
表1 正交设计因素及其水平
Tab.1 Influential factors and counts of orthogonal design

水平Counts因素Factors试剂盒MasterMix退火温度(℃)AnnealingTemperature探针浓度(μmol/L)Probeconcentration1GoldStarTaqManMixture620.22PremixExTaq60.70.43EasyTaqPCRSuperMix58.40.64SuperRealPreMix550.8
表2 [L16(4)3]正交设计表
Tab.2 [L16(4)3]Orthogonal design table
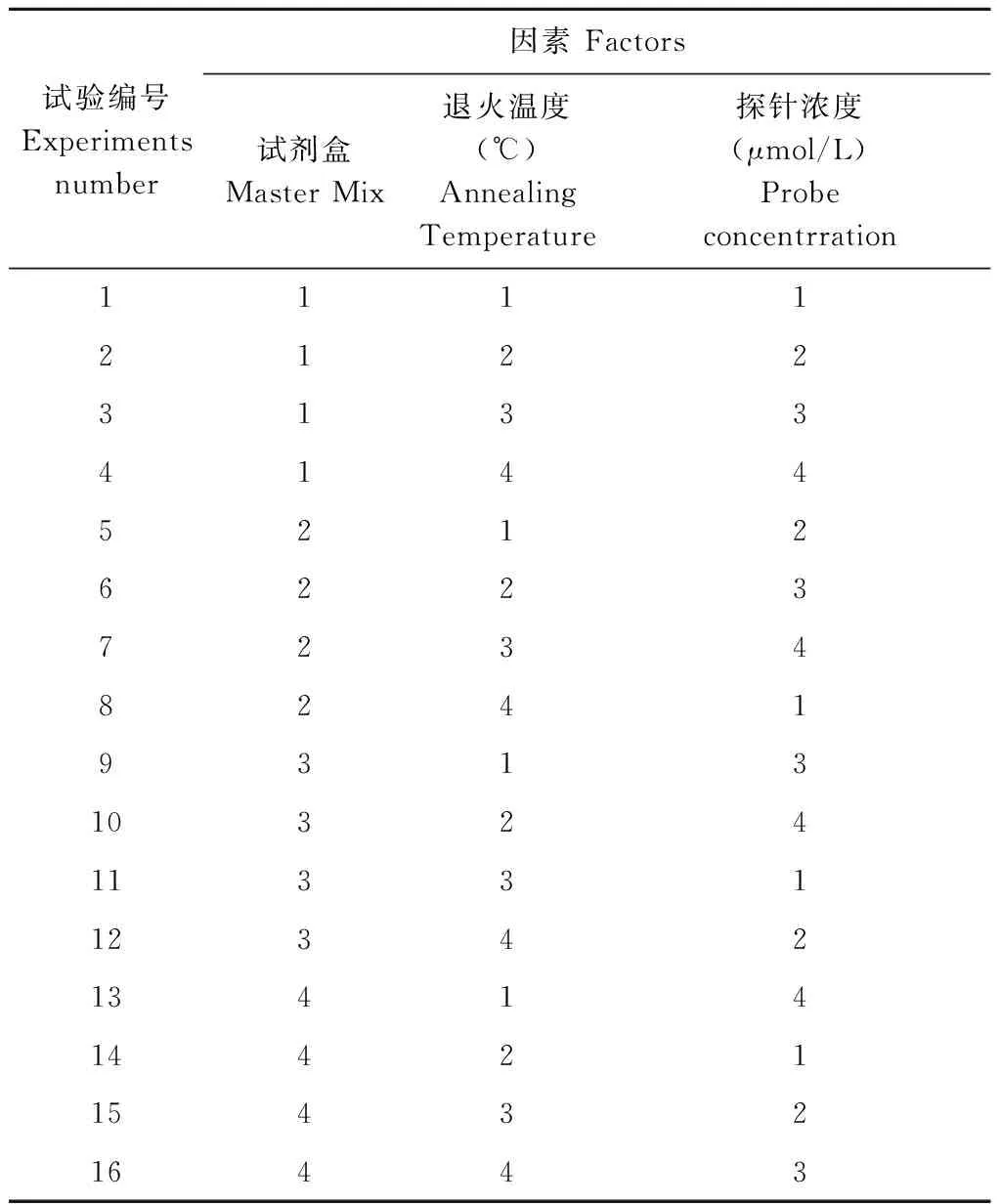
因素Factors试验编号Experimentsnumber试剂盒MasterMix退火温度(℃)AnnealingTemperature探针浓度(μmol/L)Probeconcentrration11112122313341445212622372348241931310324113311234213414144211543216443
1.4 通用探针法评价 将通用探针法与SYBR GreenI染料法进行对比,在临床样本中对通用探针检测方法进行评价。评价包括性能和通用性两方面。性能的评价是将混合的RNA样本加Poly(A)尾,分别取4、2、0.4 μL加尾产物用于逆转录,使用通用探针法和SYBR Green I染料法扩增血浆样本中的microRNA-146a、microRNA-122和microRNA-155,比较通用探针法和SYBR GreenI染料法的性能方面的差别。通用性评价是将混合的RNA样本加poly(A)尾,取4 μL加尾产物用于逆转录,使用通用探针法和SYBR Green I染料法扩增血浆样本中的microRNA-155、microRNA-146b、microRNA-223、microRNA-181c、microRNA-27a、microRNA-29a、microRNA-29b-1、microRNA-146a共8条microRNA,探索通用探针法对不同microRNA扩增的通用性。实验均以RNase free H2O作为阴性对照。
1.5 通用探针法的应用 将模拟物进行10倍梯度稀释,共5个浓度梯度(0.2-0.2×10-4μmol/L),应用最佳反应条件进行荧光定量PCR,绘制microRNA-146a标准曲线。将20例布鲁氏菌病患者和20例健康志愿者分为患病组和健康组,统计样本信息。应用通用探针法,根据microRNA-146a标准曲线计算临床样本microRNA-146a浓度。
1.6 数据统计方法 应用SPSS 17.0软件和Microsoft Excel 2007进行数据统计分析,采用两独立样本t检验。P≤0.05为差异具有统计学意义。
2 结 果
2.1 通用探针法的建立和条件的优化 通用探针法的原理是在加Poly(A)尾之后,用与含有通用探针互补序列的引物进行逆转录,产物用通用引物、通用探针和microRNA特异引物进行扩增,从而实现一次逆转录可检测多个不同的microRNA分子,而优化的扩增体系是确保扩增效率的关键。以microRNA-146a为例,设计通用引物和探针,并对通用探针法的体系进行摸索和优化。通过统计学分析,试剂盒种类、退火温度以及探针浓度三因素相互独立,P>0.05。分析3次独立重复实验标准差, σ<0.5。荧光定量扩增Ct值见表3,对结果进行直观分析[9]。第1、4、9、11、14次试验,扩增效果极差,循环数大于40。第7和12次实验,标准差σ>0.5,实验重复性差,结果不可靠。在其余9次实验中,第3次实验结果扩增曲线完整平滑,见图1。3次独立重复实验σ=0.14,Ct=31.28,实验重复性好,经过优化设计Ct值提高了,检测浓度下限提高10倍,见图1和图2。在第3次实验中,使用2×GoldStar TaqMan Mixture,探针浓度为0.6 μmol/L,退火温度为58.4 ℃。比较多次实验可以得出最佳反应程序:第一阶段95 ℃预变性10 min;第二阶段95 ℃变性15 s,58.4 ℃退火和延伸40 s,收集荧光信号,进行40次循环。最佳反应体系见表4。通过正交设计优化通用探针法反应条件,提高了检测的灵敏性,扩大了检测范围。
表3 [L16(4)3]正交设计结果
Tab.3 [L16(4)3] Orthogonal design result

试验编号Experimentnumber重复1Repeat1重复2Repeat2重复3Repeat3标准差σStandarddeviationCt平均值CtMeanvalue135.79N/A6.19——232.132.5231.580.47088632.06667331.3431.1231.390.14364331.283334N/AN/AN/A——534.0834.0534.080.01732134.07632.9233.0832.990.08020832.99667733.04N/A31.431.13844232.235832.7232.6632.620.05033232.666679N/AN/AN/A——1035.4834.9736.660.86685335.7033311N/A37.57N/A——1236.6134.1233.231.7519834.653331334.0534.0934.190.07211134.1114N/AN/AN/A——1535.0434.1734.530.4371534.581634.2333.9734.790.41904734.33

图1 优化前试验的扩增曲线Fig.1 Amplification curve of non-optimizational real-time PCR reaction

图2 正交设计第3次试验的扩增曲线Fig.2 Amplification curve of optimizational real-time PCR reaction
表4 优化的血浆microRNA-146a荧光定量PCR体系
Tab.4 Optimizational real-time PCR reaction of plasma microRNA-146a

反应成分Reactioncomponents反应体系/μLReactionsystem终浓度Finalconcentration2×GoldStarTaqManMixture51×10×buffer11×Primer(F)0.20.2μmol/LPrimer(P)0.20.2μmol/LProbe0.60.6μmol/LRNAfreeH2O3—Total10—
2.2 通用探针法的敏感性和特异性 在临床样本检测中,通用探针法的性能明显优于SYBR Green I染料法,见表5。将两种检测方法进行纵向对比,当加尾产物量不同时,通用探针法中加尾产物量越高,循环数越小,即通用探针法可以准确的区分出血浆样本中目标microRNA的含量,含量越高,越容易检测。但是SYBR Green I染料法检测microRNA-122时,加尾产物量越高反而循环数越小,对于血浆样本中的目标microRNA浓度的检测并不准确,在检测microRNA-155时,阴性对照也产生了荧光信号,表明SYBR Green I染料法只有在microRNA浓度较高时才能检测到,灵敏性差。将两种检测方法进行横向对比,在检测同一条microRNA的相同加尾产物量时,通用探针法的灵敏性均高于染料法,且阴性对照没有荧光信号,不存在干扰,在进行大量样本筛查时具有较大优势。
表5 SYBR Green I染料法和通用探针法性能比较结果
Tab.5 Efficiency comparison results of SYBR Green I assay and universal probe assay
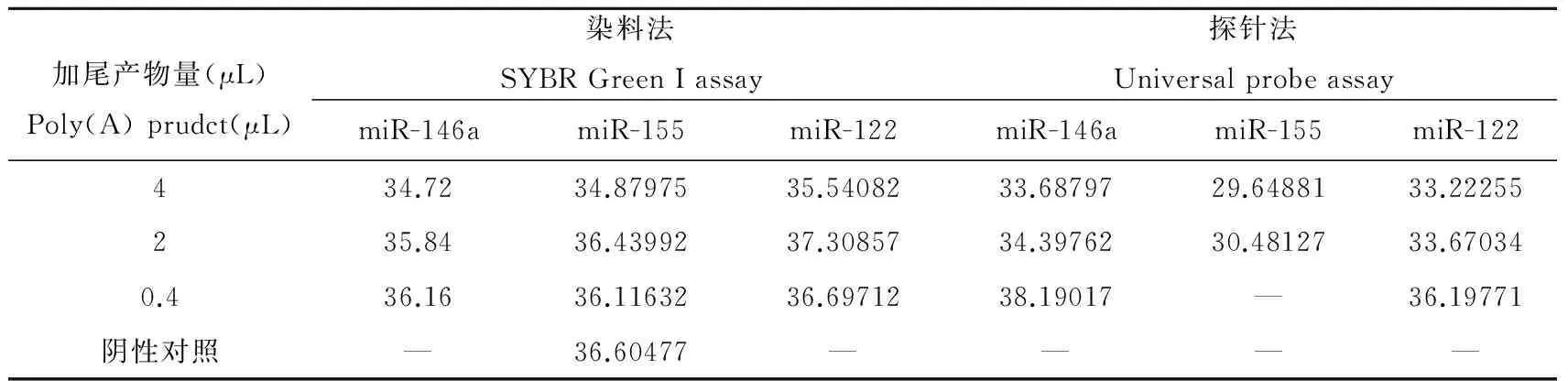
加尾产物量(μL)Poly(A)prudct(μL)染料法SYBRGreenIassay探针法UniversalprobeassaymiR-146amiR-155miR-122miR-146amiR-155miR-122434.7234.8797535.5408233.6879729.6488133.22255235.8436.4399237.3085734.3976230.4812733.670340.436.1636.1163236.6971238.19017—36.19771阴性对照—36.60477————
2.3 通用探针法通用性 在临床样本检测中,使用通用探针法和SYBR GreenI染料法对随机挑选的8条microRNA进行扩增,结果见表6。通用探针法可对8条microRNA进行检测,除microRNA-146a,其余血浆样本中microRNA循环数均在35个循环及以下,阴性对照均没有扩增。虽然通用探针法在扩增microRNA时循环数较大,但是SYBR Green I染料法阴性对照均产生了荧光信号,对目标microRNA存在干扰,如microRNA-223和microRNA-155,血浆样本和阴性对照循环数接近,结果难以判断。综上,说明通用探针法具有良好的特异性和通用性。
表6 SYBR Green I染料法和通用探针法通用性比较结果
Tab.6 Universal comparison results of SYBR Green I assay and universal probe assay

Ct值CtvaluemiR-146amiR-146bmiR-223miR-155miR-29amiR-181cmiR-27amiR-29b-1染料法SYBRGreenIassay样本Sample33.9029.8832.9433.5831.8735.3932.3734.64阴性Control37.2533.3233.5234.0735.4337.5939.6736.73探针法Universalprobeassay样本Sample37.6931.1432.6432.0934.6735.6935.6035.73阴性Control————————
2.4 通用探针法检测布鲁氏菌病患者血浆microRNA-146a 将模拟物microRNA-146a进行10倍梯度稀释,应用通用探针法,得到microRNA-146 a扩增曲线,对数图见图3。根据microRNA-146a扩增曲线绘制microRNA-146a标准曲线,见图4。该标准曲线包括5个数量级,下限可至20 pmol/L。R2=0.9816,K=-3.112,E=1.09,表明具有良好的线性关系,各稀释度间相关性好、误差小。成功建立microRNA-146a标准曲线,可进一步对每一例血浆样本中的microRNA-146a进行绝对定量分析。
将通用探针法应用于临床样本的检测,比较布鲁氏菌病患者和健康志愿者血浆样本中的microRNA-146a表达水平。统计样本基本信息,患病组年龄在4到71岁之间,20人中女性有7人,健康组年龄在16到83岁之间,20人中女性有9人,患病组和健康组年龄和性别差异不具有统计学意义(P>0.1)。患病组者抗体滴度均大于50 IM/mL,健康组试管凝集试验呈阴性。根据microRNA-146a标准曲线,采用绝对定量分析方法[10]计算样本中microRNA-146a的浓度。患病组血浆microRNA-146a浓度从14.74 nmol/L到8 447.26 nmol/L(95%CI:23.45-1 799.86),健康组microRNA-146a浓度从97.99 nmol/L-140 192.41 nmol/L(95%CI:17 013.24-48 218.79)。患病组和健康组样本基本信息及数据结果,见表7。
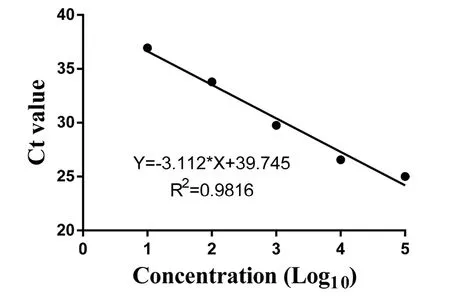
图4 模拟物microRNA-146a标准曲线Fig.4 Standard curve of microRNA-146a mimics
表7 样本信息及数据结果
Tab.7 Information and result of plasma sample

特征信息Characteristics患病组(n=20)Brucellosis健康组(n=20)HealthyvolunteerP样本信息Sampleinformation平均年龄Meanage46.0547.85>0.1年龄95%CIAge95%CI37.48-54.6239.62-56.08性别(女,%)Sex(Female,%)35%45%>0.1抗体滴度范围(IΜ/mL)SATtiter(IM/mL)50-800——数据结果Resultofanalysis浓度平均值(nmol/L)MeanConcentration911.6632616.02<0.01标准差σStd.Deviation1897.8233338.24抽样标准误差Std.ErrorofMean424.37 7454.66浓度95%CIConcentration95%CI23.45-1799.8617013.24-48218.79浓度最大值(nmol/L)Upperconcentration(nmol/L)8447.26140192.41浓度最小值(nmol/L)Lowerconcentration(nmol/L)14.7497.99
比较患病组和健康组血浆microRNA-146a表达水平,进行统计学分析,Levene检验方差不齐,P<0.05,两独立样本t检验P<0.01,患病组和健康组血浆microRNA-146a表达水平差异具有统计学意义。患病组和健康组血浆microRNA-146a表达水平差异有统计学意义,患病组血浆microRNA-146a表达水平低于健康组,绘制柱状散点图,见图5。

图5 患病组与健康组血浆microRNA-146a表达差异Fig.5 Expression of plasma microRNA-146a in patients group and control group
3 讨 论
MicroRNA作为一类基因表达调控分子,在基因表达调控中发挥着非常重要的作用[11]。越来越多的研究发现,microRNA可作为疾病的诊断标志物用于疾病诊断[12-13],因此,有关microRNA的检测方法的文章呈指数级增长。目前的检测方法主要有两大类,一是基于染料法的通用检测方法,一次加尾逆转录后可以检测很多个靶标分子[14-15],另一个是基于特定microRNA序列的探针法,一次只能检测一个靶标分子[16-17]。从原理上看,前者通用性好,但特异性不足,后者特异性较好,但通用性不足,成本高,检测过程复杂。而通用探针法则是结合两者的优点,一步逆转录后可以用探针实现多个靶标分子的检测。
本研究中,我们对通用探针法进行了系统的优化,设计了新的通用探针,对影响探针法的因素采用正交设计的方法进行了系统的优化,从而建立了敏感特异的检测方法。从优化过程的数据看,引物探针浓度、退火温度和所用检测试剂均会影响检测效率。采用正交设计方法比单个因素逐步优化的优势在于,能够寻找到多个因素相互作用及同步优化的作用[18]。所以,尽管设计的优化实验反应数大,正交设计优化能够获得最优的反应条件,这也为其它检测方法的建立和优化提供借鉴。同时对通用探针法的性能和通用性进行了讨论,发现该方法与常用的SYBR GreenI染料法相比具有良好的性能和通用性,在检测临床样本时,对于靶标microRNA反应灵敏高效,可同时可对多条microRNA进行检测,且特异性好、没有干扰。
布鲁氏菌病是一种慢性感染性疾病,布鲁氏菌感染宿主后会抑制宿主免疫,使细菌能够在宿主内生存繁殖[19]。因此,感染布鲁氏菌的患者会表现一定的免疫抑制,而这种免疫状态的改变可能作为诊断的标记。MicroRNA-146a是一类与免疫调控相关的microRNA分子,已经证实其参与了很多免疫反应的调节[20-21]。本研究中,我们比较分析了布病患者血浆中microRNA-146a的表达,并与健康人进行比较,结果发现,感染布鲁氏菌的患者血浆中microRNA-146a的表达水平被显著抑制,这提示microRNA分子可能作为一个布病诊断的分子标记。但是,由于本研究中只比较分析了数量较少的样本,这种规律还需要进一步扩大样本进行验证。另一方面,microRNA-146a作为一种调控分子,会因各种因素的影响发生变化,所以这种变化不是布鲁氏菌感染所特异的,因此,在作为诊断标识的过程中,还需考虑其它因素。
[1] Gustafson D,Tyryshkin K,Renwick N. MicroRNA-guided diagnostics in clinical samples[J]. Best Pract Res Clin Endocrinol & Metabol,2016,30(5): 563-575.DOI: 10.1016/j.beem.2016.07.002
[2] Bartel DP. MicroRNAs: genomics,biogenesis,mechanism,and function[J]. Cell,2004,116(2): 281-297. DOI:10.1016/S0092-8674(04)00045
[3] Dupouey J,Ninove L,Ferrier V,et al. Molecular detection of human rhinoviruses in respiratory samples: a comparison of TaqMan probe-,SYBR green I-and BOXTO-based real-time PCR assays[J]. Virol J,2014,11(1): 1.DOI: 10.1186/1743-422X-11-31.
[4] Wang GL,Qiu YX,Fan HY. Esatblishiment and application of PCR detection system of genetically modified maize in laboratory[J]. Genom Appl Biol,2015,34(8):1778-1783.(in Chinese)
王国莉,丘裕雄,范红英. 转基因玉米实验室 PCR 检测体系的优化及应用[J]. 基因组学与应用生物学,2015,34(8):1778-1783.
[5] Du QX,Sun JH,Wang XW,et al.Application of orthogonal design to optimize real-time quantitative PCR condition[J]. Chin J of Forensic Med,2008.23(2):111-115.(in Chinese)
杜秋香,孙俊红,王小伟,等. 正交设计在实时定量 PCR 检测条件优化中的应用[J]. 中国法医学杂志,2008,23(2): 111-114.
[6] Maurin M. Real-time PCR as a diagnostic tool for bacterial diseases[J]. Expert Rev Mol diagnost,2012,12(7): 731-754. DOI: 10.1586/erm.12.53
[7] Kang K,Zhang X,Liu H,et al. A novel real-time PCR assay of microRNAs using S-Poly (T),a specific Oligo (dT) reverse transcription primer with excellent sensitivity and specificity[J]. PLoS One,2012,7(11):e48536. DOI: 10.1371/journal.pone.0048536
[8] Buzard GS,Baker D,Wolcott M J,et al. Multi-platform comparison of ten commercial master mixes for probe-based real-time polymerase chain reaction detection of bioterrorism threat agents for surge preparedness[J]. Forens Sci Int,2012,223(1): 292-297. DOI: 10.1016/j.forsciint.2012.10.003
[9] He ZW,Liu YS,Chen LH,et al.Optimization of PCR condition by orthogonal design visual analysis [J]. Acta Acad Med Hubei;1998,23(4):403-404.(in Chinese)
何正文,刘运生,陈立华,等. 正交设计直观分析法优化 PCR 条件[J]. 湖南医科大学学报,1998,23(4): 403-404.
[10] Formisano-Tréziny C,de San Feliciano M,Gabert J. Development of plasmid calibrators for absolute quantification of miRNAs by using real-time qPCR[J]. J Mol Diagnost,2012,14(4): 314-321. DOI: 10.1016/j.jmoldx.2012.02.008
[11] Zhou X,Yang PC. MicroRNA: a small molecule with a big biological impact[J]. MicroRNA,2012,1(1):1-1. DOI: 10.2174/2211536611201010001
[12] Krauskopf J,Verheijen M,Kleinjans J C,et al. Development and regulatory application of microRNA biomarkers[J]. Biomarkers in medicine,2015,9(11): 1137-1151. DOI: 10.2217/bmm.15.50
[13] Xu L,Qi X,Duan S,et al. MicroRNAs: potential biomarkers for disease diagnosis[J]. Bio-medical materials and engineering,2014,24(6): 3917-3925. DOI: 10.3233/BME-141223
[14] MJ Mentzel C,Skovgaard K,Córdoba S,et al. Wet-lab tested microRNA assays for qPCR studies with SYBR-Green and DNA primers in pig tissues[J]. MicroRNA,2014,3(3): 174-188. DOI:10.2174/2211536604666141226194231
[15] Huang GL,Zhang XH,Guo GL,et al. Clinical significance of miR-21 expression in breast cancer: SYBR-Green I-based real-time RT-PCR study of invasive ductal carcinoma[J]. Oncology Reports,2009,21(3): 673. DOI:10.3892/or_00000270
[16] Wong W,Farr R,Joglekar M,et al. Probe-based real-time PCR approaches for quantitative measurement of microRNAs[J]. J Visualiz Exper,2015 (98): e52586-e52586. DOI: 10.3791/52586.
[17] Wang YN,Yu L,Zhao XS,et al. Establishment of plasma microRNA detection method by using TaqMan probe based quantitative reverse transcription PCR[J]. Cellul Mol Biol,2014,61(3): 51-56.
[18] Liu RJ,Zhang YW,Wen CW,et al.Study on the design and analysis methods of orthogonal experiment[J]. Experimental technology and management,2010,27(9):52-55. DOI:10.3969/j.issn.1002-4956.2010.09.016.(in Chinese)
刘瑞江,张业旺,闻崇炜,等. 正交试验设计和分析方法研究[J]. 实验技术与管理,2010,27(9): 52-55.
[19] de Figueiredo P,Ficht TA,Rice-Ficht A,et al. Pathogenesis and Immunobiology of Brucellosis: Review of Brucella-Host Interactions[J]. Am J Pathol,2015,185(6): 1505-1517. DOI: 10.1016/j.ajpath.2015.03.003
[20] Saba R,Sorensen DL,Booth SA. MicroRNA-146a: a dominant,negative regulator of the innate immune response[J]. Front Immunol,2014,5: 578. DOI: 10.3389/fimmu.2014.00578
[21] Xu WD,Lu MM,Pan HF,et al. Association of MicroRNA-146a with autoimmune diseases[J]. Inflammation,2012,35(4): 1525-1529. DOI: 10.1007/s10753-012-9467-0
Development of a universal probe based microRNA assay and detection of plasma microRNA-146a of human brucellosis
CHENG Hui-min1,2,WEI Qing-qing2,ZHANG Huan2,LIU Jin-ling2,HAN Xiao-hu2, WANG Da-li3,WANG Xing-long1
(1.InstituteofMilitaryVeterinary,AcademyofMilitaryMedicalSciences,Changchun130122,China; 2.ShenyangAgricultureUniversity,Shenyang110866,China; 3.PestisandBrucellosisTherapeuticBase,ChineseCentersforDiseaseControlandPrevention,Baicheng137000,China)
We developed a universal probe based microRNA detection assay and applied it to detect microRNA-146a in human brucellosis,testing the possibility of using it as diagnosis signature. By using orthogonal design,the annealing temperature,probe concentration and commercial kits were optimized and the assay was developed. Total RNAs were isolated from plasma of human brucellosis and healthy control,and microRNA-146a was detected and compared. Results reveal that the optimized universal probe assay was established,which was more specific than the SYBR GreenI assay,and had a wider range of amplification. Compared with healthy control,the application of universal probe assay for the detection of serum microRNA-146a in patients with brucellosis was significantly inhibited (P<0.01). Implying the potential of microRNA-146a as biomarker in diagnosis of brucellosis.It is suggested that universal probe based assay is a universal,specific and sensitive method for microRNA detection. MicroRNA-146a represents a potential biomarker for human brucellosis diagnosis.
universal probe assay; orthogonal design; microRNA-146a; human brucellosis
Wang Xing-long,Email: wangxl-2006@163.com
10.3969/j.issn.1002-2694.2017.05.011
王兴龙,Email:wangxl-2006@163.com
1.军事兽医研究所,军事医学科学院,长春 130122; 2.沈阳农业大学,沈阳 110866; 3.中国疾病预防控中心鼠疫布病防治基地,白城 137000
R378.5
A
1002-2694(2017)05-0441-08
2016-07-21 编辑:王晓欢
- 中国人兽共患病学报的其它文章
- 中国西北四省(区)结核分枝杆菌分离株一线药物耐药状况及其影响因素分析
- 美国《Emerging Infectious Diseases》2017年第3期有关人兽共患病论文摘译
- 龟头包皮炎患者淋病奈瑟菌分离与鉴定及其耐药机制研究
- miR-16在金黄色葡萄球菌脓毒症中的表达及意义探讨
- Identification of non-tuberculosis mycobacteria speciesof clinical isolates from patients clinically diagnosed with tuberculosis in Fujian Province,China
- 四对结核分枝杆菌毒素-抗毒素系统基因功能的初步研究

