大麦不同组织成熟过程中DNA甲基化的MSAP分析
熊肖,李博龚强,徐延浩
(长江大学农学院,湖北荆州434025;湖北省涝渍灾害与湿地农业重点实验室( 长江大学) ,湖北荆州434025;主要粮食作物产业化湖北省协同创新中心( 长江大学) ,湖北荆州434025)
大麦不同组织成熟过程中DNA甲基化的MSAP分析
熊肖,李博龚强,徐延浩
(长江大学农学院,湖北荆州434025;湖北省涝渍灾害与湿地农业重点实验室( 长江大学) ,湖北荆州434025;主要粮食作物产业化湖北省协同创新中心( 长江大学) ,湖北荆州434025)
DNA甲基化参与调控生长植物发育。使用甲基化敏感扩增多态性(methylation sensitive amplification polymorphism,MSAP)方法并结合毛细管自动电泳分析仪,对大麦(HordeumvulgareL)的种子、根、茎、叶等4种组织在成熟过程中的DNA甲基化修饰进行了分析。结果表明,大麦不同组织在成熟过程中基因组总甲基化水平改变较小,但 44.90%~65.17% 的CCGG位点甲基化模式在不同组织在成熟过程中发生了改变。在大麦种子成熟过程中,CCGG位点半甲基化水平急剧升高,达到65.40%,全甲基化位点显著下降仅为13.39%,与其他组织的甲基化特征差异显著。结果显示,在大麦组织成熟的过程中,DNA甲基化修饰发生了巨大的变化。
大麦(HordeumvulgareL);DNA甲基化;甲基化敏感扩增多态性(MSAP);组织类型;发育
发生在胞嘧啶位点上的甲基化修饰是表观遗传修饰的一种重要修饰形式,在调控基因时空表达、基因组防御、胚胎发育、基因组印迹等方面起着重要的作用[1]。陆光远等[2]研究表明,在油菜幼苗形成过程中,从胚根到下胚轴到子叶,DNA甲基化水平逐渐增加。玉米穗位叶、雌穗苞叶、雄穗3 个组织中基因组CCGG位点胞嘧啶甲基化水平逐渐降低[3]。类似的现象在鸡等[4~6]其他动植物中也有报道。由此可见,基因组DNA甲基化水平对调控动植物组织分化起着重要的作用[1,7]。DNA甲基化也是植物应答基因组胁迫的一种重要调控方式[8]。Xu等[9]研究表明,在基因组加倍过程中,芸薹花与叶片基因组DNA甲基化调控模式存在差异。比较和分析生物不同组织基因组甲基化水平,是解析DNA甲基化调控器官发育及组织特异逆境应答的基础。
检测DNA甲基化的方法主要有色谱法、酶切法、测序法、甲基化PCR检测法、芯片法等多种方法[10]。以甲基化修饰敏感性不同限制性内切酶和PCR 为基础的甲基敏感扩增片段多态性 (Methylation sensitive amplified polymorphism,MSAP) 灵敏度高、不受基因组序列信息限制、检测位点多、成本低并能同时分析多个样品。目前[9,11~13],MSAP已应用在杂种优势分析、基因组加倍、逆境应答等多个领域的研究中。但传统的MSAP分析依赖于聚丙烯酰胺凝胶电泳,其操作自动化水平较低,严重地制约了分析通量。对MSAP分析自动化与高通量的研究是改进MSAP分析方法的主要方向[6]。
大麦(HordeumvulgareL)是酿造工业重要的原料和饲料作物,其基因组已完成测序[14]。作为一个典型的二倍体植物,大麦是细胞遗传学的模式生物,目前正成为麦类作物功能基因组学的一个代表物种[15]。与玉米、油菜等作物相比,缺乏对大麦不同组织基因组DNA甲基化水平的比较分析。本研究使用MSAP方法并结合毛细管自动电泳仪对大麦不同组织基因组DNA甲基化水平进行比较分析,为大麦表观遗传研究积累更多资料。
1 材料与方法
1.1 试验材料及处理
供试品种为长江大学农学院保存的大麦鄂啤2号(HordeumvulgareL.cv.Epi 2)。材料于2015年11月4日播种在长江大学农学院试验田,田间管理按常规方法进行。试验田行长1.5m,株距10cm,行距25cm,单穴点播,每个小区7行,共设置3个小区,按小区分别取样、保存。2015年12月下旬采集幼苗的叶片和茎秆;2016年3月下旬采集抽穗后的旗叶和茎秆、并挖取地下根;2016年4月下旬采集乳熟期的种子。种子成熟后分小区脱粒、晾晒,2016年9月底作为成熟种子样品使用。2015年11月4日在人工气候箱中(上海佳语JYC-412)萌发种子,7d后收集幼根保存备用。
1.2 DNA提取
用CTAB法(2% CTABw/v,100mmol/L Tris-HCl pH 8.0,20mmol/L EDTA pH 8.0,2.8mol/L NaCl)提取各样品基因组总DNA。粗提的DNA加入RNAase A消化过夜,用酚-氯仿-异戊醇(25∶24∶1)抽提后用氯仿-异戊醇(24∶1)抽提1~2次,用无水乙醇沉淀DNA。DNA干燥后溶于TE溶液。用1% 的琼脂糖凝胶电泳检测基因组DNA 的完整性,用Nano Drop 2000检测样品DNA浓度。
1.3 MSAP的酶切与连接
双酶切反应体系总体积20μL,其中DNA 400ng,NEB 10×buffer 2μL,HpaII 20U或MspI 10 U,EcoRI 10 U,100×BSA(10mg/mL)0.2μL。37℃温浴5h,75℃ 10min中止反应。双酶切反应体系中加入EcoRI接头5pmol,HpaII/MspI接头50pmol(表1),T4 DNA连接酶 1μL(NEB),ATP 1mmol/L。14℃ 水浴12h。75℃ 10min中止反应。

表1 MSAP接头、引物信息
1.4 MSAP的预扩增、选择性扩增
预扩增使用50ng (3μL)酶切连接产物作为模板,25μL PCR反应体系含有0.2μmol/LEcoRI和HpaII/MspI预扩增引物(EcoRI + 0,HpaII/MspI + 0) (表1),1× PCR buffer,1.5mmol/L MgCl2,0.2mmol/L dNTP ,2 U Taq酶(Fermentas)。PCR 扩增程序为 94℃ 3min; 然后 94℃ 30s,56℃ 40s,72℃ 60s共 23个循环;72℃ 延伸 10min。
预扩产物用ddH2O溶液稀20倍,取1μL作为选择扩增的模板DNA。选择性扩增引物对采用在预扩引物的序列基础上加上几个碱基组成(EcoRI + 3个选择性碱基和HpaII/MspI +2~4个选择性碱基,表2)。选择性扩增PCR反应体系和预扩增PCR反应体系相同。PCR扩增程序为 94℃ 60s,接下来 94℃ 30s,65℃ 30s,72℃ 60s,每个循环退火温度降低 0.7℃,执行 12 个循环;接着在 94℃ 30s,56℃ 30s,72℃ 60s 扩增23个循环;最后72℃ 延伸10min。
1.5 MSAP的扩增片段的自动电泳与数据读取分析
利用毛细管自动电泳仪(Fragment Analyzer FSv2-CE,美国AATI)对MSAP选择性扩增PCR产物进行电泳分离,分离试剂盒为DNF-900 dsDNA Reagent Kit 35~500bp(AATI,美国),该试剂盒能区分35~500bp的片段,比较适用于MSAP扩增片段分离。毛细管电泳分离条件:8.0kV预电泳30s后7.5kV 10s吸取内参DNA分子;接着7.5kV 10s吸取样品DNA分子后,8.0kV电泳95min分离MSAP扩增片段。
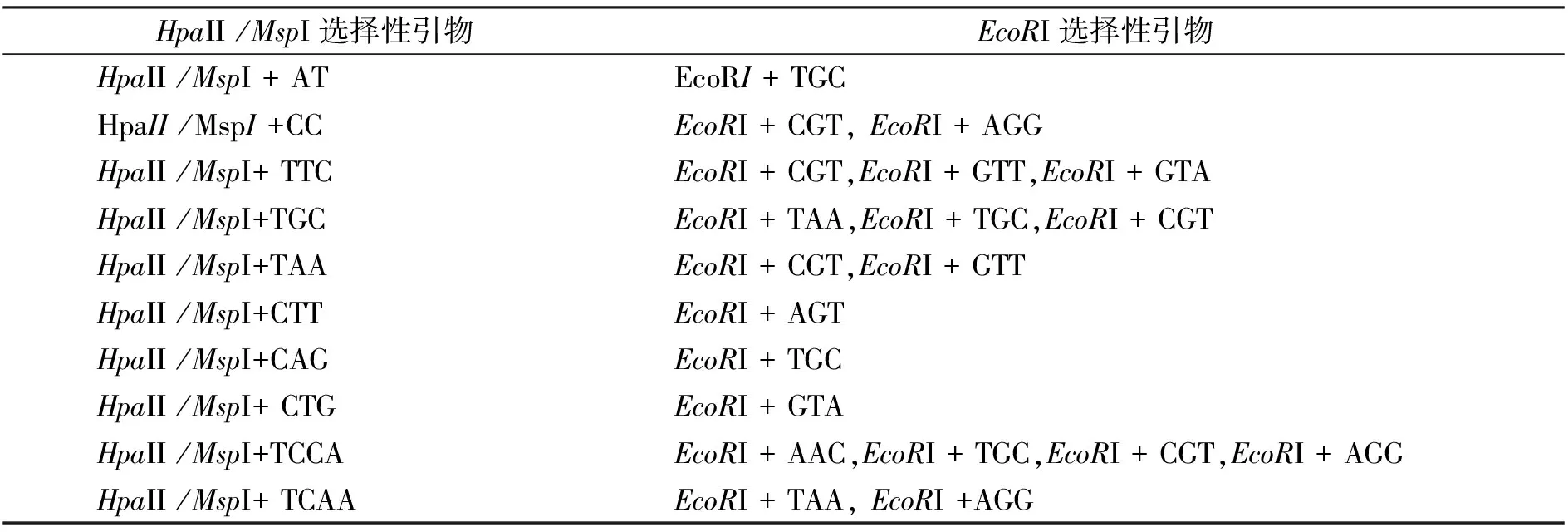
表2 MSAP选择性引物组合信息
电泳结束后用PROSize 2.0软件(AATI,美国)矫正内参DNA标记,将所有分子量相同的内参DNA标记校准到同一水平位置后读取分离片段的分子量并导出所有分析样本数据。由于MSAP目标条带较多,使用Excel将目标分离片段转化为0/1矩阵进行甲基化率、甲基化模式比较分析。
2 结果与分析
2.1 大麦不同组织基因组DNA甲基化水平比较
HpaII酶切未甲基化和单链外侧C甲基化的CCGG位点,MspI酶切未甲基化和双链内侧C甲基化的CCGG位点。当样品在HpaII酶切扩增产物中有带而在MspI酶切扩增产物中无带,则说明该位点发生了单链外侧的胞嘧啶甲基化;反之,则说明该位点发生了双链内侧胞嘧啶甲基化;两者扩增产物中均有带,说明该位点未发生甲基化(表3)。
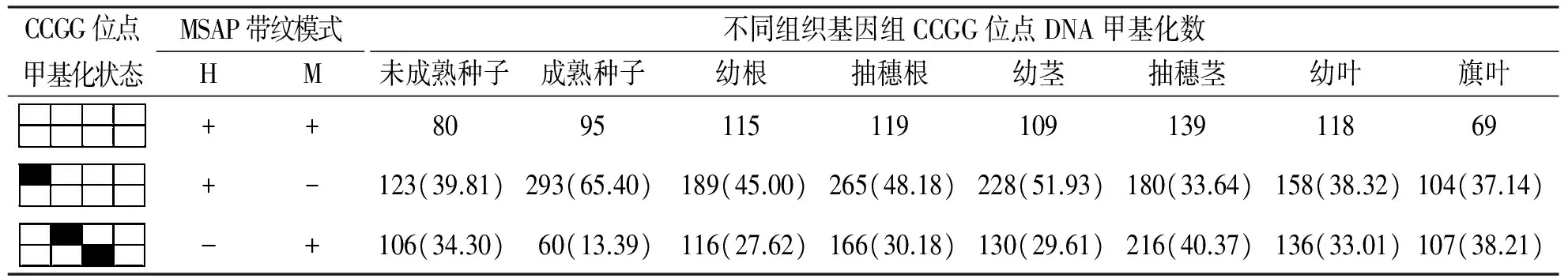
表3 大麦不同组织基因组CCGG位点DNA甲基化水平
注:1)格子代表CCGG双链,黑色的格子代表甲基化的位点。2)H代表EcoRI-HpaII酶切,M代表EcoRI-MspI酶切;+表示有带,-表示无带。3)括号中数据代表该类型所占比例(%)。
大麦乳熟期种子CCGG位点总甲基化水平为74.11%,成熟种子甲基化水平为78.79%,在种子成熟过程中,甲基化水平上升4.68%;大麦幼根CCGG位点总甲基化水平为72.62%,抽穗后根系总甲基化水平为78.36%,随着发育成熟,根系甲基化水平升高5.74%;大麦幼茎CCGG位点总甲基化水平为76.65%,抽穗后茎秆总甲基化水平为74.02%,甲基化水平下降2.64%;大麦幼叶CCGG位点总甲基化水平为71.36%,旗叶总甲基化水平为75.35%,随着发育成熟,叶片甲基化水平升高3.99%(表3)。总的来说,在大麦发育成熟的过程中,不同器官基因组总甲基化水平维持在75%左右,改变程度在5%左右。在大麦种子成熟过程中,CCGG位点半甲基化水平急剧升高,达到65.40%,增加25.60%;全甲基化位点下降20.91%,仅为13.39%;但从幼嫩茎到成熟茎的过重中,CCGG位点半甲基化水平下降15.17%,而全甲基化水平提高12.54% (表3)。但在根和叶成熟过程中,CCGG位点半甲基化水平和全甲基化水平改变程度较小(表3)。
2.2 大麦不同组织在成熟过程中DNA甲基化模式的改变
虽然在大麦不同组织成熟过程中基因组总甲基化水平改变程度较低,但不同位点的甲基化修饰模式还需要进一步分析。20对选择性引物组合在4组组织中共扩增得到3235条带(图1)。根据MSAP带纹在每组幼嫩组织和成熟组织中的相对模式,MSAP带纹模式可以分3大类,第一类在组织成熟过程中没有发生DNA甲基化状态改变的位点,根据其带纹模式再分为无甲基化位点、单链外侧甲基化位点、双链内侧甲基化位点等3个类型(A1~A3);第二类是在组织成熟过程中发生了超甲基化的位点,依据其带纹模式,共包括5个类型(B1~B5);第三类是在组织成熟过程中发生了去甲基化的位点,依据其带纹模式,共分为5个类型(C1~C5)(表4)。
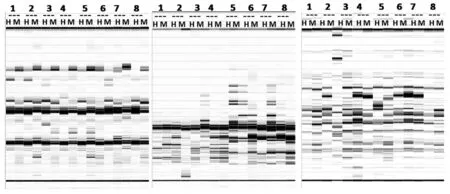
注:1)1.新种子样品;2.老种子样品;3.幼根样品;4.幼茎样品;5.幼叶样品;6.拔节根样品;7.拔节茎样品;8.拔节叶样品。2)H表示EcoRI-HpaII酶切扩增电泳带,M表示EcoRI-MspI酶切扩增电泳带。3)选择性引物组合依次为HpaII/MspI+TTC×EcoRI+CGT,HpaII/MspI+TAA×EcoRI+GTT,HpaII/MspI+CTG×EcoRI+GTA。图1 大麦不同组织的MSAP电泳带纹模式

带纹类型带纹亚类幼嫩组带型HM成熟组带型HM不同器官扩增带纹数量种子根茎叶ABC总计A11111123222229231A210107510413762A3010158889385小计256414459378B11110411513B2110182145B3010099919988B4100052857592B5110028221217小计191211205215C1011112874C210112667C30001801109592C4001167121421C500101278947100小计288225169224735850833817
注:1)A.DNA甲基化未改变位点;B.超甲基化位点;C.去甲基化位点。2)H代表EcoRI-HpaII酶切,M代表EcoRI-MspI酶切;1代表有扩增带纹,0代表无扩增带纹。
对MSAP结果(表4)的分析表明,在种子成熟过程中,65.17%检测的CCGG位点发生了甲基化状态的改变,其中25.99%的位点发生了超甲基化,39.18%的位点发生了去甲基化;在根系成熟的过程中,51.29%检测的CCGG位点发生了甲基化状态的改变,其中24.82%的位点发生了超甲基化,26.47%的位点发生了去甲基化;在茎成熟的过程中,44.90%检测的CCGG位点发生了甲基化状态的改变,其中24.61%的位点发生了超甲基化,20.29%的位点发生了去甲基化;在叶片成熟的过程中,53.73%检测的CCGG位点发生了甲基化状态的改变,其中26.32%的位点发生了超甲基化,27.41%的位点发生了去甲基化。说明虽在大麦组织成熟的过程中,可能经历过基因组甲基化状态的重排。
3 讨论
本研究使用毛细管自动电泳仪分离MSAP片段,一次能分离96个样品,并且自动读取条带的分子量,避免了人工读带的主观误差,保留了毛细管电泳适应自动化、通量高、分辨率高的特点,又保留了MSAP成本相对较低的特点,为大麦DNA甲基化分析提供了一个快速、高通量的分析技术手段[6]。
MSAP仅能分析CCGG位点双链内侧全甲基化和单链外侧半甲基化修饰,植物基因组中还存在CG、CHG、CHH位点的甲基化修饰,这些位点的甲基化状态在组织成熟过程中的变化还需要使用其他研究手段进一步去分析[16]。通过MSAP分析,发现大麦不同组织在成熟过程中基因组CCGG位点DNA甲基化水平相对比较稳定,改变程度大约在5%。这与在芸薹属[9]、菘蓝[12]、玉米[3]等多种作物中的研究结果类似。这说明一定程度的基因组甲基化水平对维持基因组的稳定性具有重要作用。
但在大麦不同组织成熟过程中,超过一半的CCGG位点的甲基化模式发生了重排,其中超甲基化事件与去甲基化事件基本相当。一般认为DNA甲基化是调控基因表达的一种重要表观遗传修饰,DNA超甲基化能关闭或降低基因的表达,而去甲基化则可以启动或增强基因的表达,但具体哪些基因发生了时空转录的变化,还需要通过转录组和甲基化测序来进一步确定。
DNA甲基化修饰是植物响应逆境胁迫的一种重要方式。对大麦干旱胁迫下的叶片和根系甲基化组的研究表明,叶片在干旱胁迫下发生更多的去甲基事件,而根系发生更多的甲基化事件[13]。这与本研究观察到的不同组织器官成熟过程中具有组织特异的甲基化改变模式一样。与本研究相比,菘蓝[11]、油菜[9]等不同植物器官[17]的甲基化模式改变程度及其在逆境下的甲基化重排程度都要小得多。这可能是因为大麦基因组较大,基因组中大部分是重复序列和反转座子序列,而这些位点是甲基化修饰和调控的热点区域[13]。
基因组内DNA甲基化受到DNA甲基化酶、染色质域甲基化转移酶、组蛋白甲基转移酶等酶的作用,而去甲基化修饰则主要与沉默抑制因子(ROS1)、转葡萄糖基酶、氧化去甲基化、水解去甲基化等机制有关[1,7,16]。RNA介导的DNA甲基化修饰也是调控DNA甲基化的重要机制[18]。这些机制在大麦组织成熟和分化过程中是如何调控基因组DNA甲基化的修饰是一个值得深入研究的问题。
[1]Satyaki P R V,Gehring M.DNA methylation and imprinting in plants:machinery and mechanisms[J].Crit Rev Biochem Mol Biol,2017,52:163~175.
[2]陆光远,伍晓明,陈碧云,等.油菜种子萌发过程中DNA甲基化的MSAP分析[J].科学通报,2005,50(24):2750~2756.
[3]Lu Y,Rong T,Cao M.Analysis of DNA methylation in different maize tissues[J].Journal of Genetics and Genomics,2008,35:41~48.
[4]徐青,张沅,孙东晓,等.应用MSAP方法检测鸡不同组织基因组的甲基化状态[J].遗传,2011,33(6):620~626.
[5]Osabe K,Clement J D,Bedon F,etal.Genetic and DNA methylation changes in cotton (Gossypium) genotypes and tissues[J].PLoS One,2014,9:e86049.
[6]李金龙,唐韶青,邹智元,等.北京油鸡MSAP毛细管电泳荧光检测技术的建立[J].遗传,2014,36(5):495~502.
[7]李亚娇,郭九峰,王淑妍,等.DNA甲基化与植物生长发育的表观遗传调控研究进展[J].生态科学,2016,(4):192~198.
[8]Deleris A,Halter T,Navarro L:DNA methylation and demethylation in plant immunity[J].Annual Review of Phytopathology,2016,54:579~603.
[9]Xu Y,Zhao Q,Mei S,etal. Genomic and transcriptomic alterations following hybridisation and genome doubling in trigenomic allohexaploidBrassicacarinata×Brassicarapa[J].Plant Biology,2012,14(5):734~744.
[10]邢宝松,郭启祥,马强,等.DNA甲基化检测方法研究进展[J].河南农业科学,2007,36(11):17~19.
[11]杨飞,徐延浩.温度对菘蓝基因组DNA甲基化的影响[J].中国实验方剂学杂志,2013,19(6):113~116.
[12]杨飞,徐延浩.四倍体菘蓝基因组DNA甲基化的甲基化敏感扩增多态性分析[J].中草药,2013,44(3):344~348.
[13]Chwialkowska K,Nowakowska U,Mroziewicz A,etal.Water-deficiency conditions differently modulate the methylome of roots and leaves in barley (HordeumvulgareL.)[J].Journal of Experimental Botany,2016,67:1109~1121.
[14]Mayer K F,Waugh R,Brown J W,etal.A physical,genetic and functional sequence assembly of the barley genome[J].Nature,2012,491:711~716.
[15]Feuillet C,Stein N,Rossini L,etal.Integrating cereal genomics to support innovation in the Triticeae[J].Functional & Integrative Genomics,2012,12:573~583.
[16]王瑞娴,徐建红.基因组DNA甲基化及组蛋白甲基化[J].遗传,2014,36(3):191~199.
[17]Alonso C,Pérez R,Bazaga P,etal. MSAP markers and global cytosine methylation in plants:a literature survey and comparative analysis for a wild-growing species[J].Molecular Ecology Resources,2015,16:80~90.
[18]Matzke M A,Mosher R A:RNA-directed DNA methylation:an epigenetic pathway of Increasing complexity[J].Nature Reviews Genetics 2014,15:394~408.
[编辑] 余文斌
2017-03-16
长江大学青年基金资助项目(2015cqr16);湿地生态与农业利用教育部工程中心开放课题(KF201601)。
熊肖(1988-),男,硕士生,研究方向为植物分子育种。通信作者:徐延浩,xyh09@yangtzeu.edu.cn。
Q78;S512.3
A
1673-1409(2017)10-0029-05
[引著格式]熊肖,李博,龚强,等.大麦不同组织成熟过程中DNA甲基化的MSAP分析[J].长江大学学报(自科版),2017,14(10):29~33,42.

