玉米FLA蛋白家族的生物信息学分析
钟静+胡颖+陈亚波+宋群星


摘要:类成束阿拉伯半乳糖蛋白(fasciclin-like arabinogalactan proteins,简称FLAs)是一类广泛分布于植物体内的富含羟脯氨酸的糖蛋白,在植物生长发育和形态构建中发挥着重要作用。基于已公布的玉米蛋白数据库确定了26个FLA蛋白,并对其理化性质、系统发生树、蛋白结构和功能域等进行了分析。结果表明,FLA蛋白氨基酸长度在249~682个之间,理论等电点在4.99~10.76之间,主要定位在质膜上,多数为疏水性蛋白;二级结构由α-螺旋、无规则卷曲和延伸链等元件组成;部分蛋白空间结构具有较强保守性。
关键词:玉米;FLA;蛋白家族;基因家族;生物信息学;理化性质;结构特征
中图分类号: Q943.2文献标志码: A文章编号:1002-1302(2017)07-0023-05
阿拉伯半乳糖蛋白(arabinogalactan proteins,简称AGPs)是一类富含羟脯氨酸的糖蛋白[1]。根据核心蛋白骨架差异,AGPs被分为经典AGPs(classical AGPs)、非经典AGPs(non-classical AGPs)、赖氨酸富集AGPs(lysine-rich AGPs)、AG多肽(AG peptides)、类成束蛋白AGPs(fasciclin-like arabinogalactan proteins,简称FLAs)、类木质形成素AGPs(xylogen-like AGPs,简称XLAs)和类植物蓝素AGPs(phytocyanin-like AGPs,简称PLAs)等7类[2-4]。AGPs的蛋白骨架一般包括N端信号肽和1段长度可变且富含脯氨酸(Pro)、丙氨酸(Ala)、丝氨酸(Ser)、苏氨酸(Thr)的区域(PAST)[1]。经典AGPs还包含1个C端GPI锚定信号。FLAs作为AGPs的1个亚类,除具有AGPs的典型结构之外,还含有1~2个成束蛋白结构域(fasciclin domains,简称FAS)[5]。
FLAs在植物生长发育过程中发挥重要作用。有研究显示,FLAs参与了茎、侧根、花粉小孢子发育,以及棉花纤维细胞的伸长等过程[6-8],此外还可影响植株茎的强度和弹性[9]。对其作用机制的研究表明,FLAs可能是通过影响细胞壁的形成从而发挥其生物学功能[10-13]。目前已经在拟南芥、油菜、水稻、小麦中分别鉴定出了21、33、27、34个FLAs[3,5,14-15],但是在重要的粮食和经济作物玉米中还未见关于FLAs的报道。因此,本研究采用生物信息学方法,对玉米FLAs蛋白家族成员进行全面的鉴定和系统进化分析,并进一步分析其蛋白质产物的理化性质、结构特征和功能域等特点,以期为进一步揭示植物FLAs的结构和功能奠定基础。
1材料与方法
1.1序列来源
分别从Pfam(http://pfam.sanger.ac.uk/)、Interpro(http://www.ebi.ac.uk/interpro/)数据库中下载玉米中含有FAS结构域的蛋白序列[16-17]。将获得的序列提交到maizeGDB(http://www.maizegdb.org/)数据库中进行比对,获得目标蛋白质的全长序列。从拟南芥官方数据库TAIR(http://www.arabidopsis.org/)中下载获得拟南芥FLAs基因和蛋白序列数据。
1.2序列分析
利用Compute pI/Mw tool(http://web.expasy.org/compute_pi/)計算相对分子量及理论等电点。利用SignalP 4.1(http://www.cbs.dtu.dk/services/SignalP/)、Plant- mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)分析蛋白信号肽和亚细胞定位[18]。利用ProtScale(http://web.expasy.org/cgi-bin/protscale/protscale.pl)分析蛋白亲水性/疏水性。利用NPS(http://npsa-pbil.ibcp.fr/cgi-bin/npsa_automat.plpage=/NPSA/npsa_hnn.html)分析蛋白质的二级结构。利用SWISS-MODEL(http://swissmodel.expasy.org/)分析蛋白质的三级结构。
1.3模体识别
利用MEME program3(http://meme-suite.org/)模体搜索工具识别FLAs家族相关蛋白质所共有的模体,并对相关参数进行修改,将可找到的模体数最大值调整为15个,每个模体的最大宽度调整为100,其他均为默认值[19]。
1.4系统进化树的构建
应用多序列比对工具ClustalX 1.83对玉米、拟南芥FLAs氨基酸全序列进行比对[20]。采用MEGA7.0软件中的邻接法(neighbor-joining,简称NJ)构建系统发育树[21]。通过随机逐步比较的方法搜索最佳系统进化树,对生成的系统树进行Bootstrap校正。
2结果与分析
2.1玉米FLAs家族成员的鉴定和理化性质分析
分别从Pfam、Interpro数据库下载玉米中含有FAS结构域的蛋白序列,并将获得的序列分别提交到maizeGDB数据库中进行比对,获得其全长序列。对来源于2个数据库中的蛋白质全长进行交叉比对,去除重复序列。进一步将获得的蛋白序列提交到SignalP 4.1检测N端信号肽,不含有信号肽的不属于AGPs家族。对含有信号肽的序列手动查找其中富含PAST的区域,并将蛋白序列提交到PSORT检测C端糖基磷脂酰肌醇(GPI)锚定信号,最终获得了26个玉米FLAs并分别命名为 ZmFLA1~ZmFLA26(表1)。
对这26个ZmFLAs进行氨基酸理化性质分析表明,氨基酸序列长度在249~682个之间,分子量在25 175.47~72 410.16 u 之间,理论等电点在4.99~10.76之间(表1)。亚细胞定位分析显示,绝大多数ZmFLAs都定位在质膜上,其中 ZmFLA8、ZmFLA14除定位在质膜上外,还可能定位在核中,而ZmFLA5则仅定位在核上(表1)。亲水性/疏水性分析显示,ZmFLA2/6/24 亲水区域明显大于疏水区域,说明这3个 ZmFLA 蛋白亲水性较强,为亲水性蛋白;其余23个 ZmFLA 蛋白为疏水性蛋白(表1)。N-糖基化位点检测结果显示,所有 ZmFLAs 都至少含有1个N-糖基化位点,ZmFLA22 甚至包含7个N-糖基化位点(表1),说明ZmFLAs蛋白骨架可能具有较高的糖基化修饰率。
2.2ZmFLAs系统进化和序列特异性分析
对玉米、拟南芥中的FLAs进行系统进化分析显示,26个ZmFLAs、21个AtFLAs被聚类在8个独立的进化分支中(图1-A)。分支Ⅷ仅含有2个AtFLAs,其余7个分支都同时有ZmFLAs、AtFLAs的分布。但是在同一分支中的ZmFLAs、AtFLAs 并无明显交叉分布,来源于不同物种的FLAs通常分别聚类在小的分支中。例如分支Ⅱ含有2個拟南芥FLAs、5个玉米FLAs,其中AtFLA1、AtFLA2聚类在1个小分支中,而ZmFLA4/10/19聚类在另1个小分支中。进一步的序列特异性分析显示,尽管玉米、拟南芥的FLAs都含有模体1,但是不同的FLAs含有其他不同的模体。同时,处于同一个进化分支中的FLAs往往具有类似的模体。例如进化分支Ⅰ中,AtFLA3/5/14、ZmFLA14都含有模体1、模体7、模体8、模体11,ZmFLA12/16/25则含有模体1、模体8、模体11;模体10仅出现在分支Ⅱ、Ⅲ的蛋白质序列中,而模体4、模体5、模体9仅出现在分支Ⅴ中(图1-B)。
2.3ZmFLAs蛋白结构分析
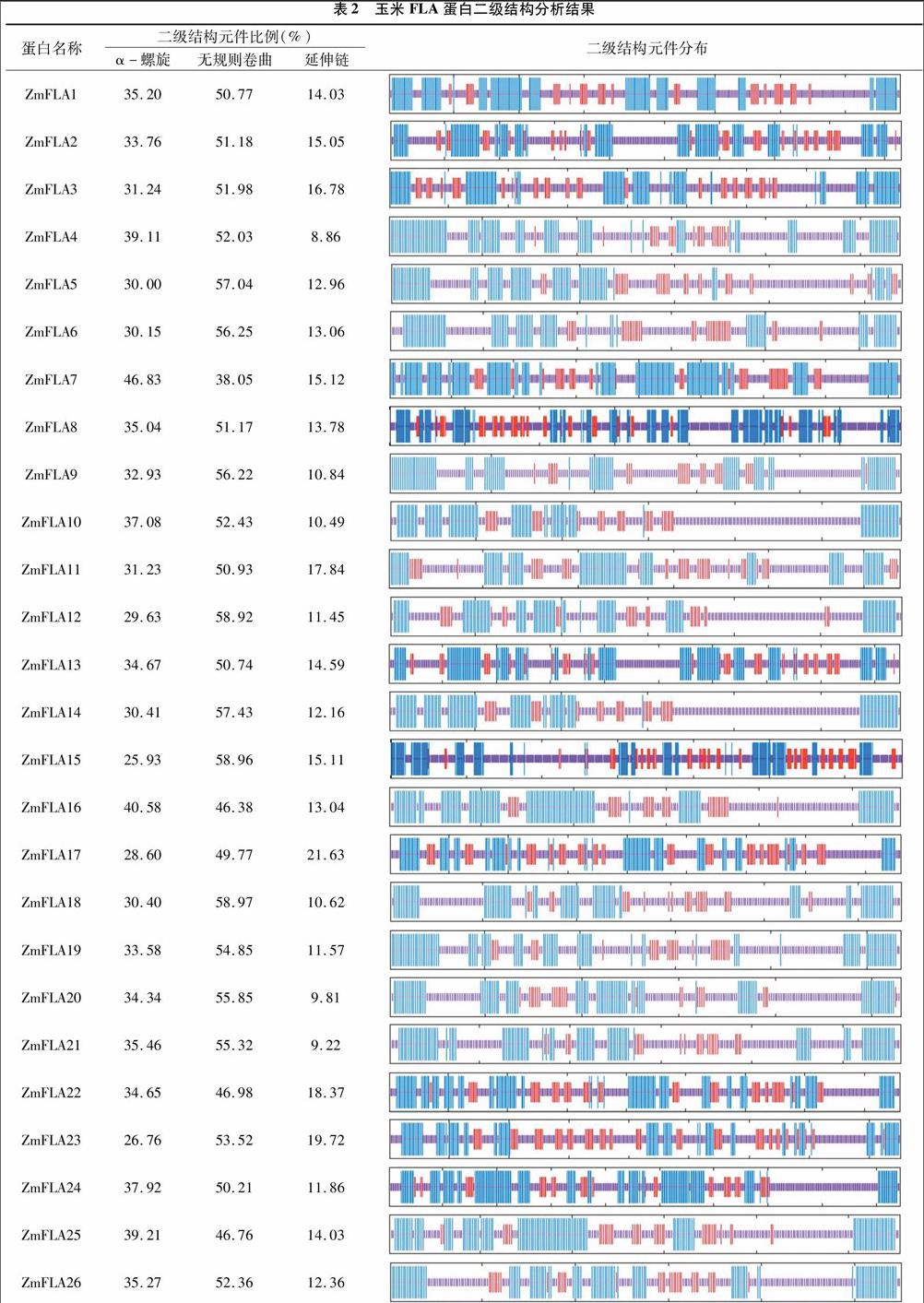
通过NPS程序对这26个ZmFLAs蛋白序列进行二级结构分析表明,蛋白质均由α-螺旋、无规则卷曲和延伸链等结构元件组成,但是这3种元件的比例和分布存在差异。其中无规则卷曲所占比例最高,在38.05%~58.97%之间;其次为 α-螺旋,在25.93%~46.83%之间;延伸链所占比例最低,在8.86%~21.63%之间(表2)。进一步利用Swiss-Model同源建模的方法预测这26个ZmFLAs蛋白的三级结构。从图2可以看出,三级结构主要是由α-螺旋、无规则卷曲和延伸链等元件组成,其中ZmFLA1/2/3/7/13/17/22/23/24空间结构较为保守;ZmFLA8/12/14/16/18/19/25与这些蛋白类似,但是空间结构明显较为松散;其余ZmFLAs的结构则与上述蛋白存在明显差异,其中ZmFLA5/10/11/20结构较类似。
3结论与讨论
本研究通过多种数据库和在线软件,对玉米FLA蛋白基因家族进行了全面的鉴定和分析。结果显示,从玉米全基因组中共鉴定出了26个FLAs,对其理化性质的研究显示,大多数ZmFLAs都是疏水性氨基酸。亚细胞定位预测结果显示,绝大多数ZmFLAs定位在质膜上,这可能与FLAs参与细胞间信号转导有关[13]。系统进化分析结果显示,玉米、拟南芥FLAs被聚类在8个独立的进化分支中,每个进化分支中都有玉米、拟南芥FLAs蛋白的分布,暗示在单子叶、双子叶植物中,该家族成员可能拥有共同的祖先。但是,在每个进化分支中,不同物种来源的FLAs蛋白并没有呈现交叉分布,这说明该蛋白基因家族的扩张可能是发生在单子叶、双子叶植物进化之后,并且其扩张在单子叶、双子叶植物中是独立进行的。
目前认为,蛋白质的一级结构决定二级结构,二级结构又决定着三级结构,而蛋白质的功能往往取决于其空间结构。本研究对26个ZmFLAs的结构进行了分析。二级结构分析结果表明,ZmFLAs蛋白包含α-螺旋、无规则卷曲和延伸链等元件。其中无规则卷曲所占比例最高,α-螺旋次之,延伸链比例最低。同时,各个ZmFLAs中不同元件的比例和分布也存在差异。进一步的三级结构分析表明,尽管部分 ZmFLAs 的空间结构十分保守或者类似,但是其拓扑结构和聚合角度也存在明显差异,这暗示不同的ZmFLAs可能具有各自独特的功能。
已有关于FLAs的报道主要集中在双子叶模式植物拟南芥中,鲜见对单子叶经济作物中FLAs生物学功能的相关报道。本研究应用生物信息学的方法鉴定了玉米FLAs蛋白家族成员,并对FLAs的基本理化性质、亚细胞定位、系统进化、二级结构和三级结构等进行了详细分析,以期为深入探讨ZmFLAs的生物学功能提供理论基础。
参考文献:
[1]Ellis M,Egelund J,Schultz C,et al. Arabinogalactan-proteins:key regulators at the cell surface?[J]. Plant Physiology,2010,153(2):403-419.
[2]Schultz C,Johnson K,Currie G,et al. The classical arabinogalactan protein gene family of arabidopsis[J]. The Plant Cell,2000,12(9):1751-1768.
[3]Ma H,Zhao J. Genome-wide identification,classification,and expression analysis of the arabinogalactan protein gene family in rice (Oryza sativa L.)[J]. Journal of Experimental Botany,2010,61(10):2647-2668.
[4]馬浩力,余礼,梁荣洪,等. 高等植物阿拉伯半乳糖蛋白的功能研究[J]. 中国科学:生命科学,2015,45(2):113-123.
[5]Johnson K L,Jones B J,Bacic A,et al. The fasciclin-like arabinogalactan proteins of Arabidopsis. A multigene family of putative cell adhesion molecules[J]. Plant Physiology,2003,133(4):1911-1925.
[6]Li J,Yu M,Geng L,et al. The fasciclin-like arabinogalactan protein gene,FLA3,is involved in microspore development of Arabidopsis[J]. The Plant Journal:for Cell and Molecular Biology,2010,64(3):482-497.
[7]Johnson K,Kibble N,Bacic A,et al. A fasciclin-like arabinogalactan-protein (FLA) mutant of Arabidopsis thaliana,fla1,shows defects in shoot regeneration[J]. PLoS One,2011,6(9):e25154.
[8]Huang G,Gong S,Xu W,et al. A fasciclin-like arabinogalactan protein,GhFLA1,is involved in fiber initiation and elongation of cotton[J]. Plant Physiology,2013,161(3):1278-1290.
[9]Macmillan C,Mansfield S,Stachurski Z,et al. Fasciclin-like arabinogalactan proteins:specialization for stem biomechanics and cell wall architecture in Arabidopsis and Eucalyptus[J]. The Plant Journal:for Cell and Molecular Biology,2010,62(4):689-703.
[10]Shi H,Kim Y,Guo Y,et al. The Arabidopsis SOS5 locus encodes a putative cell surface adhesion protein and is required for normal cell expansion[J]. The Plant Cell,2003,15(1):19-32.
[11]Persson S,Wei H,Milne J,et al. Identification of genes required for cellulose synthesis by regression analysis of public microarray data sets[J]. Proceedings of the National Academy of Sciences of the United States of America,2005,102(24):8633-8638.
[12]Seifert G,Xue H,Acet T. The Arabidopsis thaliana fasciclin like arabinogalactan protein 4 gene acts synergistically with abscisic acid signalling to control root growth[J]. Annals of Botany,2014,114(6):1125-1133.
[13]Wang H,Jiang C,Wang C,et al. Antisense expression of the fasciclin-like arabinogalactan protein FLA6 gene in Populus inhibits expression of its homologous genes and alters stem biomechanics and cell wall composition in transgenic trees[J]. Journal of Experimental Botany,2015,66(5):1291-1302.
[14]Faik A,Abouzouhair J,Sarhan F. Putative fasciclin-like arabinogalactan-proteins (FLA) in wheat (Triticum aestivum) and rice (Oryza sativa):identification and bioinformatic analyses[J]. Molecular Genetics and Genomics,2006,276(5):478-494.
[15]Showalter A,Keppler B,Lichtenberg J,et al. A bioinformatics approach to the identification,classification,and analysis of hydroxyproline-rich glycoproteins[J]. Plant Physiology,2010,153(2):485-513.
[16]Finn R,Mistry J,Schuster-Bckler B,et al. Pfam:clans,web tools and services[J]. Nucleic Acids Research,2006,34:D247-D251.
[17]Quevillon E,Silventoinen V,Pillai S,et al. InterProScan:protein domains identifier[J]. Nucleic Acids Research,2005,33:W116-W120.
[18]Petersen T N,Brunak S,von Heijne G,et al. SignalP 4.0:discriminating signal peptides from transmembrane regions[J]. Nature Methods,2011,8(10):785-786.
[19]Bailey T L,Williams N,Misleh C,et al. MEME:discovering and analyzing DNA and protein sequence motifs[J]. Nucleic Acids Research,2006,34:W369-W373.
[20]Thompson J D,Gibson T J,Plewniak F,et al. The CLUSTAL_X Windows interface:flexible strategies for multiple sequence alignment aided by quality analysis tools[J]. Nucleic Acids Research,1997,25(24):4876-4882.
[21]Kumar S,Stecher G,Tamura K. MEGA7:molecular evolutionary genetics analysis version 7.0 for bigger datasets[J]. Molecular Biology and Evolution,2016,33(7):1870-1874.

