脑源性神经营养因子启动子区甲基化与孤独症谱系障碍关系初探☆
周家秀丁明崔冬张琳琳周甄会秦尚够廖汝琴王艳何福生杨峰王明帮
·论 著·
脑源性神经营养因子启动子区甲基化与孤独症谱系障碍关系初探☆
周家秀*丁明**崔冬***张琳琳*周甄会*秦尚够*廖汝琴*王艳△何福生△杨峰****王明帮△
目的 通过比较孤独症谱系障碍(autism spectrum disorders,ASD)患者和正常对照脑源性神经营养因子(brain derived neurotrophic factor,BDNF)基因启动子Ⅰ区和Ⅳ区各CpG单元甲基化率,探讨ASD可能的发病机制。 方法 选取ASD患者12例及正常对照12名,利用飞行时间质谱法检测全血中BDNF基因启动子Ⅰ和Ⅳ区各CpG单元甲基化率,并分析其相关性距离、进化关系,比较两组各单元甲基化率。 结果 在BDNF启动子Ⅰ区和Ⅳ区分别检测到17个和8个CpG单元的甲基化率。ASD患者组BDNF启动子Ⅰ区中CpG单元4、7、10、35,以及BDNF启动子Ⅳ区CpG单元11.12、14相关性距离较近,聚类成比较小的分支。ASD患者BDNF启动子Ⅰ区CpG单元5.6甲基化率低于对照组(P<0.05),Ⅳ区CpG单元3和15甲基化率高于对照组(P<0.05)。 结论 ASD患者BDNF启动子Ⅰ区CpG单元5.6和Ⅳ区CpG单元3和15甲基化率在ASD患者组和对照组差异显著,提示BDNF启动子甲基化可作为ASD潜在的生物标志物深入研究。
孤独症谱系障碍 脑源性神经营养因子 启动子 甲基化
遗传因素和非遗传因素在抑郁症、精神分裂症和双相情感障碍等精神疾病的发生发展中均扮演重要角色[1-3]。非遗传因素目前研究较多的是表观遗传,包括DNA甲基化、组蛋白修饰、非编码RNA调控等。脑源性神经营养因子(brain-derived neurotrophic factor,BDNF)可调节轴/树突发生、突触形成等[4],研究显示,它在精神疾病发病中起重要作用[5]。环境应激会引起DNA异常甲基化,从而影响中枢神经系统中BDNF基因的表达,并影响突触可塑性和记忆的形成,可能对精神疾病的发生有一定影响[6]。目前,关于孤独症谱系障碍(autism spectrum disorders,ASD)BDNF基因表达的研究较多,如ZHENG等[7]对14篇文献进行meta分析,发现ASD患者外周血清和血浆中BDNF的表达水平明显高于正常对照。但直接研究ASD患者BDNF基因启动子的报道较少。BDNF甲基化异常与精神性疾病相关[8],并且血液BDNF DNA甲基化可反映大脑BDNF DNA甲基化情况[9]。本研究通过检测ASD患者外周血中BDNF启动子Ⅰ区和Ⅳ区各CpG单元的甲基化水平,比较其与正常对照间的差异,探讨ASD可能的发病机制。
1 对象与方法
1.1 研究对象 患者组来自深圳市儿童医院心理科及言语治疗科门诊ASD患儿,收集时间为2015 年7~12月。纳入标准:①根据国际最新诊断标准,即《美国精神障碍诊断与统计手册第5版》(Diagnostic and Statistical Manual of Mental Disorders,5th Edition,DSM-5),诊断为ASD“需要非常多支持”的患儿;②年龄<14岁;③性别不限。排除标准:①患有其他精神疾病(如精神分裂症等);②患有其他神经发育障碍疾病;③患有遗传代谢性疾病;④患有严重神经系统疾病以及颅脑损伤史等重大躯体疾病史。共收集12例患儿,男7例,女5例;年龄2~6岁,平均(3.3±1.4)岁。
对照组来自深圳市儿童医院儿保科体检儿童。纳入标准:①无精神疾病,身体健康;②与患者组年龄及性别匹配。排除标准同患者组。共收集12名对照儿童,男7名,女5名;年龄1~6岁,平均(3.1±2.3)岁。
两组性别、年龄差异无统计学意义(P>0.05)。所有入组儿童家长对本研究知情并签署知情同意书。本研究经深圳市儿童医院伦理委员会审批。
1.2 血液采集和DNA提取 患者组和对照组儿童均于清晨抽取空腹静脉血3 mL于EDTA抗凝管(BD公司,美国)中,保存于-80℃冰箱,然后使用血液DNA提取试剂盒OMEGA D3392(Omega Bio-Tek公司,美国)提取全血中的DNA。
1.3 甲基化检测及质控 通过UCSC网站(http:// genome.ucsc.edu/)查询BDNF基因启动子Ⅰ区和Ⅳ区的序列,其中Ⅰ区位置为chr11:27 744 025-27 744 279,共计255 bp,Ⅳ区位置为chr11:27 723 096-27 723 467,共计 372 bp。然后利用 Epi-Designer软件(Sequenom公司,美国)设计引物,BDNF基因启动子Ⅰ区正向和反向引物分别为5’-TGGGTTATGGTTTTTTAAATGTGAG-3’和5’-CC CTCTATTAAACACTCCAAAAAAA-3’,Ⅳ区正向和反向引物分别为5’-GGTAGAGGTAGGGAGATTTTATGTT-3’和5’-CCTACCCCCACTCTAATTAAAA CAA-3’,确定可检测的区段,并进行引物合成(Thermo Fisher公司,美国)。甲基化检测之前,先利用Qubit dsDNA HS Assay Kit(Thermo Fisher公司,美国)进行DNA定量检测,并统一将DNA稀释至 50 ng/μL,加入 96孔板。利用 EZ DNA Methylation Gold Kit(Zymo Research公司,美国),参照试剂盒说明对待测DNA样本进行亚硫酸氢盐(bisulfite)处理和PCR扩增。PCR为5 μL反应体系,包括ddH2O 1.42 μL,配有20 mmol/L MgCl2的10×PCRBuffer0.50μL,dNTP0.040μL,酶0.04μL,正反向引物2 μL,待检测DNA 1 μL。反应条件为95℃预变性4 min;95℃变性20 s,56℃退火30 s,72℃延伸60 s,共45个循环;72℃保持3 min;25℃静置。得到的PCR产物通过Mass ARRAY(Sequenome公司,美国)进行DNA甲基化定量检测,其原理是基于基质辅助激光解析电离飞行时间质谱(MALDI-TOF-MS)反应,检测CpG位点甲基化发生情况,然后通过MassARRAY系统自带的Epi-Typer软件v1.2得到甲基化率数据。具体过程:首先,EpiTyper软件会根据该区域的CpG数量,分别编号,即得到CpG位点CpG_1,_2,…,如果多个CpG位点不能被检测到,或者检测到的CpG位点是连续的,即编号为CpG单元,如CpG_1.2,然后根据质谱峰图判断CpG单元甲基化率。能检测出CpG单元甲基化率的样本占所有样本的比例为检出率。质谱数据质控利用EpiTyper软件v1.2(Sequenome公司,美国)完成,检出率低于90%的CpG单元为低质量CpG单元,不纳入后续统计分析。
1.4 统计学方法 使用R语言包进行统计分析。针对ASD患者组和对照组检测到的BDNF基因启动子Ⅰ区和Ⅳ区各CpG单元甲基化率水平,利用两独立样本单边秩和检验(Wilcoxon检验)进行组间比较。参考患者和对照BDNF启动子Ⅰ区和Ⅳ区CpG单元的甲基化率数值,利用Heatmap函数绘制BDNF启动子Ⅰ区和Ⅳ区CpG单元甲基化模式热图。所有被试BDNF启动子Ⅰ区和Ⅳ区CpG单元甲基化率数据的相关性距离分析采用Pearson相关分析,具体过程是,先通过(1-甲基化单元之间的Pearson相关系数)做转换,然后用R 的dist函数计算距离,即dist(1-cor),其中cor为甲基化单元之间的Pearson相关系数,然后进行Heatmap展示,BDNF启动子Ⅰ区和Ⅳ区各CpG单元的进化关系通过所有样本的相关性距离数值进行聚类分析。检验水准α=0.05。
2 结果
2.1 BDNF启动子Ⅰ区和Ⅳ区CpG单元检测率BDNF基因启动子Ⅰ区90%以上检出率的CpG单元数为17个;BDNF启动子Ⅳ区90%以上检出率的CpG单元数为8个。甲基化率情况见表1。
2.2 BDNF启动子Ⅰ区和Ⅳ区各CpG单元甲基化率相关性距离分析 BDNF启动子Ⅰ区和Ⅳ区各CpG单元甲基化率相关性分析结果见图1。BDNF启动子Ⅰ区中,CpG单元4、7、10、35相关性距离较近,CpG单元3、11相关性距离也较近;BDNF启动子Ⅳ区中,CpG单元1.2,11.12相关性距离较近,CpG单元11.12和5.6相关性距离较近。
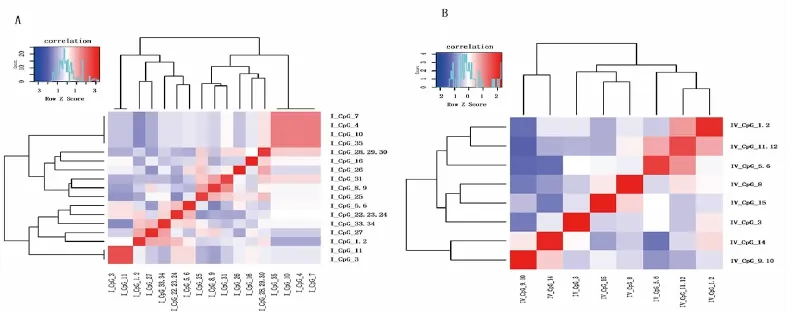
图1 BDNF启动子Ⅰ区和Ⅳ区各CpG单元相关性距离热图 A为BDNF启动子Ⅰ区各CpG单元甲基化率相关性距离,B为BDNF启动子Ⅳ区各CpG单元甲基化率相关性距离。各图左上为相关性距离变量的Z尺度标准化;热图中红色代表近的相关性距离,蓝色代表远的相关性距离;一个方格代表一个相关性距离数值。
2.3 BDNF启动子Ⅰ区和Ⅳ区CpG单元甲基化进化关系图 BDNF启动子Ⅰ区和Ⅳ区各CpG单元聚类分析结果见图2。BDNF启动子Ⅰ区中CpG单元聚成3个大的分支,如CpG单元4、7、10、35聚为一个分支,CpG单元22.23.24、5.6、11和3聚为一个大的分支,CpG单元 8.9、31、25、26、16、28.29.30、33.34、1.2和27聚为另一个大的分支。而BDNF启动子Ⅳ区中各CpG单元没有聚类成大的分支,但可以看出,CpG单元11.12与1.2聚为一个小的分支,CpG单元9.10与14聚为一个小的分支,CpG单元11.12与1.2聚为一个小的分支,CpG单元15与18聚为一个小的分支。
2.4 患者组和对照组BDNF启动子Ⅰ区和Ⅳ区CpG单元甲基化差异 BDNF启动子Ⅰ区CpG单元5.6甲基化率患者组低于对照组(W=43,P= 0.049),其他各CpG单元在患者组和对照组间差异均无统计学意义(P>0.05);BDNF启动子Ⅳ区中,CpG单元3(W=103,P=0.039)和CpG单元15(W=102,P=0.041)在ASD患者组和对照组间差异有统计学意义。见表1。
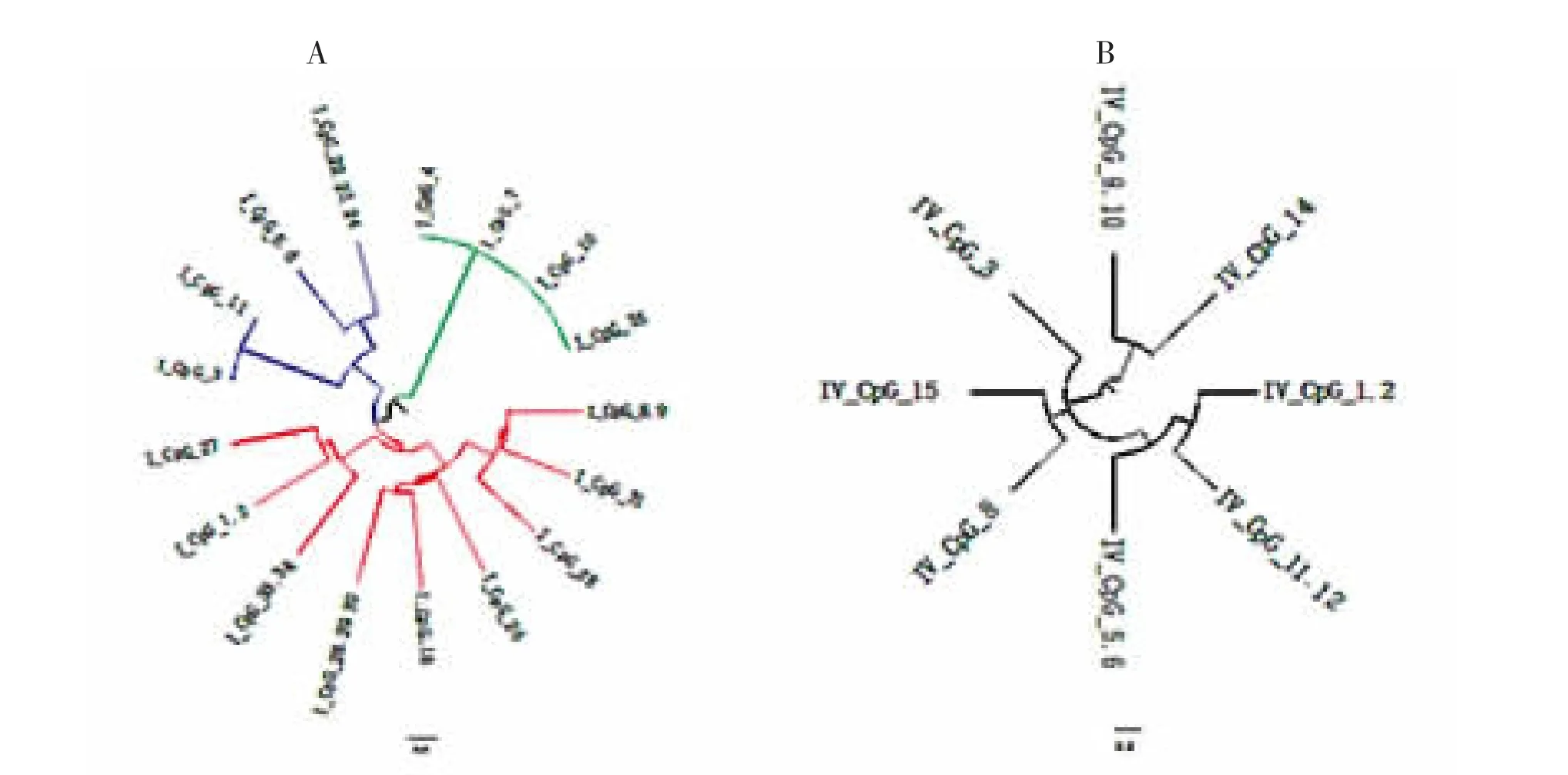
图2 BDNF启动子Ⅰ区和Ⅳ区CpG各单元甲基化进化关系图 A为BDNF启动子Ⅰ区各CpG单元甲基化率进化关系,3种不同的颜色代表3个大的分支;B为BDNF启动子Ⅳ区各CpG单元甲基化率进化关系,未发现明显大的分支,只标注为黑色。
表1 患者组和对照组BDNF启动子Ⅰ区和Ⅳ区各CpG单元甲基化率(±s)
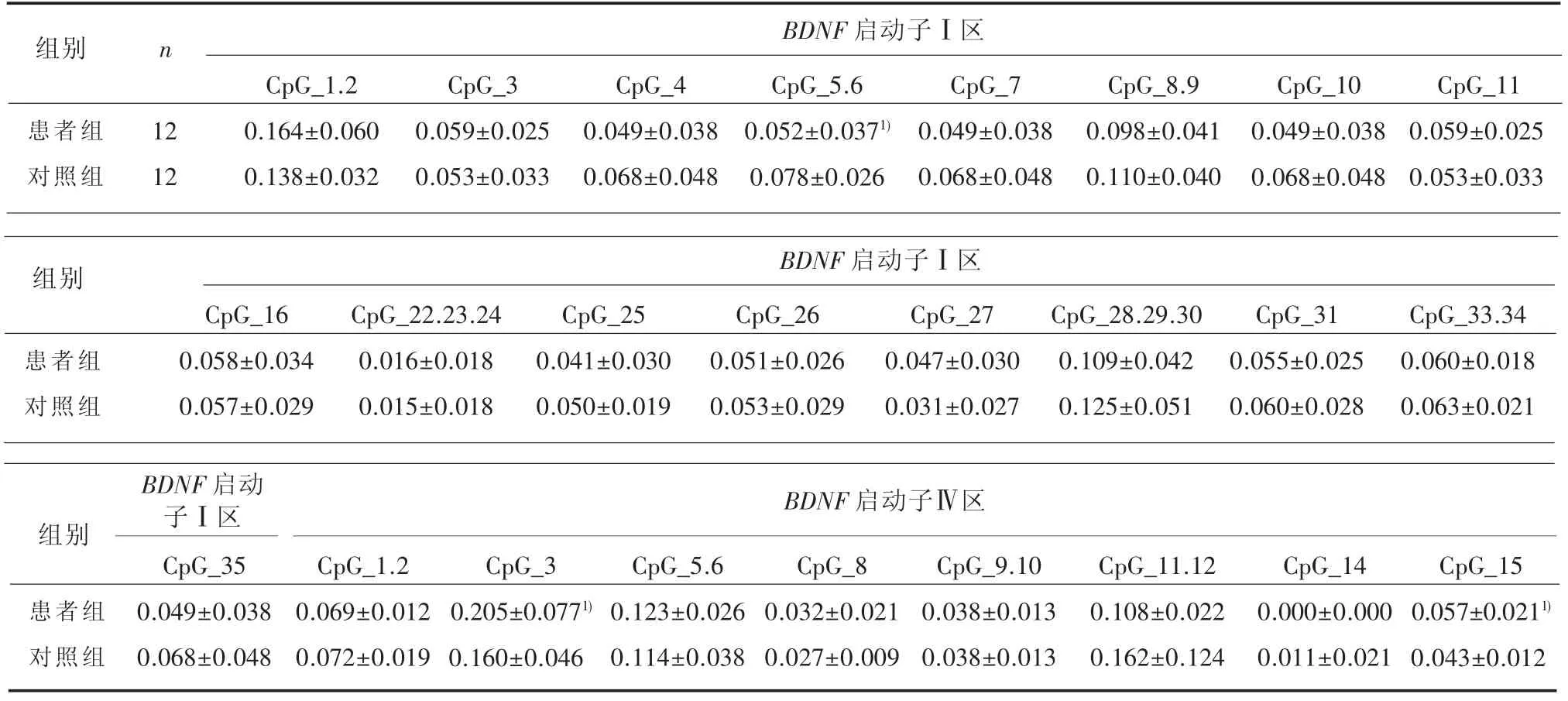
表1 患者组和对照组BDNF启动子Ⅰ区和Ⅳ区各CpG单元甲基化率(±s)
1)与对照组比较,经两独立样本单边秩和检验(Wilcoxon检验),P<0.05
BDNF启动子Ⅰ区组别 n患者组对照组12 12 CpG_1.2 0.164±0.060 0.138±0.032 CpG_3 0.059±0.025 0.053±0.033 CpG_4 0.049±0.038 0.068±0.048 CpG_5.6 0.052±0.0371)0.078±0.026 CpG_7 0.049±0.038 0.068±0.048 CpG_8.9 0.098±0.041 0.110±0.040 CpG_10 0.049±0.038 0.068±0.048 CpG_11 0.059±0.025 0.053±0.033 BDNF启动子Ⅰ区组别患者组对照组CpG_16 0.058±0.034 0.057±0.029 CpG_22.23.24 0.016±0.018 0.015±0.018 CpG_25 0.041±0.030 0.050±0.019 CpG_26 0.051±0.026 0.053±0.029 CpG_27 0.047±0.030 0.031±0.027 CpG_28.29.30 0.109±0.042 0.125±0.051 CpG_31 0.055±0.025 0.060±0.028 CpG_33.34 0.060±0.018 0.063±0.021 BDNF启动子Ⅳ区组别BDNF启动子Ⅰ区患者组对照组CpG_35 0.049±0.038 0.068±0.048 CpG_1.2 0.069±0.012 0.072±0.019 CpG_3 0.205±0.0771)0.160±0.046 CpG_5.6 0.123±0.026 0.114±0.038 CpG_8 0.032±0.021 0.027±0.009 CpG_9.10 0.038±0.013 0.038±0.013 CpG_11.12 0.108±0.022 0.162±0.124 CpG_14 0.000±0.000 0.011±0.021 CpG_15 0.057±0.0211)0.043±0.012
3 讨论
BDNF是一个重要的神经系统发育调控因子,其表达异常与多种神经和精神性疾病相关[10]。大量的研究表明,ASD患者BDNF水平较高[11]。本研究通过MassArray检测12例ASD患者和12名对照BDNF启动子Ⅰ区和Ⅳ区的甲基化率,发现BDNF启动子Ⅰ区CpG单元4、7、10、35,及BDNF启动子Ⅳ区中CpG单元1.2、11.12甲基化率相关性距离较近,同时也能很好地聚类到一个小的分支,说明可能其接受比较相似的调控。本研究发现ASD患者BDNF启动子Ⅰ区CpG单元5.6甲基化率显著低于对照组,Ⅳ区CpG单元3和15甲基化率显著高于对照组。ZHENG等[7]大规模的meta分析发现ASD患者血清或者血浆中存在较低的BDNF表达水平,本研究很好地支持了该结果。BDNF基因启动子区甲基化水平,是抑郁症诊断的标志物,如FUCHIKAMI等[12]研究发现,抑郁症患者外周血BDNF启动子Ⅰ区的35个CpG单元中有29个甲基化水平显著低于正常对照。同时,BDNF启动子区甲基化水平与精神疾病治疗效果相关,D'ADDARIO等[13]研究发现重性抑郁障碍患者BDNF启动子Ⅰ甲基化水平高于正常对照,当患者使用心境稳定剂后,其BDNF启动子Ⅰ甲基化水平则显著降低。但是关于ASD患者BDNF启动子甲基化研究不多,本研究发现在ASD患者和对照BDNF启动子区中也存在差异显著的CpG单元,说明ASD患者和对照组可能BDNF基因表达有差异,并且提示可以深入评估如BDNF启动子Ⅰ区CpG单元5.6及Ⅳ区CpG单元3和15是否可作为ASD潜在的标志物。
本研究采用的MassArray技术能进行单碱基水平甲基化检测,有很高的精度性和准确性。但本研究也有一定的局限性,如样本量较小,这个主要是由于MassArray甲基化检测技术成本相对较高所致。下一步可以对发现差异的甲基化单元,如将BDNF启动子Ⅰ区CpG单元5.6甲及Ⅳ区CpG单元3和15在大样本中进行验证,或者进行功能研究,如利用细胞模型或者动物模型来研究BDNF启动子Ⅰ区和Ⅳ区甲基化是否影响BDNF基因的表达,以探索BDNF甲基化参与ASD发病的具体机制。
[1]ABDOLMALEKY HM,ZHOU JR,THIAGALINGAM S.An update on the epigenetics of psychotic diseases and autism[J]. Epigenomics,2015,7(3):427-449.
[2]卢天兰,伍智镠,阮燕燕,等.孤独症谱系障碍核心家系染色体核型分析研究[J].中国神经精神疾病杂志,2016,42(3): 150-155.
[3]井绪秀,王庆中,禹顺英.NLGNs基因在孤独症谱系障碍中的研究进展[J].中国神经精神疾病杂志,2015,41(7):445-448.
[4]LEWIN GR,BARDE YA.Physiology of the neurotrophins[J]. Annu Rev Neurosci,1996,19:289-317.
[5]ANGELUCCI F,BRENE S,MATHE AA.BDNF in schizophrenia,depression and corresponding animal models[J].Mol Psychiatry,2005,10(4):345-352.
[6]ROTH TL,LUBIN FD,FUNK AJ,et al.Lasting epigenetic influence of early-life adversity on the BDNF gene[J].Biol Psychiatry,2009,65(9):760-769.
[7]ZHENG Z,ZHANG L,ZHU T,et al.Peripheral brain-derived neurotrophic factor in autism spectrum disorder:a systematic review and meta-analysis[J].Sci Rep,2016,6:31241.
[8]KANG HJ,KIM JM,BAE KY,et al.Longitudinal associations between BDNF promoter methylation and late-life depression [J].Neurobiol Aging,2015,36(4):e1761-1767.
[9]KUNDAKOVIC M,GUDSNUK K,HERBSTMAN JB,et al.DNA methylation of BDNF as a biomarker of early-life adversity[J]. Proc Natl Acad Sci U S A,2015,112(22):6807-6813.
[10]DUCLOT F,KABBAJ M.Epigenetic mechanisms underlying the role of brain-derived neurotrophic factor in depression and response to antidepressants[J].J Exp Biol,2015,218(Pt 1):21-31. [11]QIN XY,FENG JC,CAO C,et al.Association of Peripheral Blood Levels of Brain-Derived Neurotrophic Factor With Autism Spectrum Disorder in Children:A Systematic Review and Meta-analysis[J].JAMA Pediatr,2016,70(11):1079-1086.
[12]FUCHIKAMI M,MORINOBU S,SEGAWA M,et al.DNA methylation profiles of the brain-derived neurotrophic factor (BDNF)gene as a potent diagnostic biomarker in major depression[J].PLoS One,2011,6(8):e23881.
[13]D'ADDARIO C,DELL'OSSO B,GALIMBERTI D,et al.Epigenetic modulation of BDNF gene in patients with major depressive disorder[J].Biol Psychiatry,2013,73(2):e6-7.
The study on the association of the methylation in the promoter region of brain derived neurotrophic factor with autism spectrum disorders.
ZHOU Jiaxiu,DING Ming,CUI Dong,ZHANG Linlin,ZHOU Zhenhui,QIN Shanggou, LIAO Ruqin,WANG Yan,HE Fusheng,YANG Feng,WANG Mingbang.Autism Genome Research Center,Shenzhen Following Precision Medical Research Institute,Guangdong,518001,China.Tel:0755-82636223.
Objective The aim of present study was to detect methylation rate of CpG unit of brain derived neurotrophic factor (BDNF)promoter and to study the epigenetic mechanism of autism spectrum disorders(ASD). Methods Total of 12 ASD patients and 12 healthy controls were recruited.The methylation rate of CpG unit in BDNF promoterⅠandⅣwere detected using Sequenom MassArray method.The methylation model,correlationship,evolutionary relationship of CpG units in BDNF promoterⅠandⅣwere detected and compared between ASD patients and healthy controls.Results The methylation rate was identified in 17 and 8 CpG units in BDNF promoterⅠand BDNF promoter Ⅳ.A close correlation distance was detected in BDNF promoterⅠCpG units 4,7,10,35,and BDNF promoterⅣCpG units 11.12,14.BDNF promoterⅠ CpG units 4,7,10,35,and BDNF promoterⅣ CpG units 11.12,14 could beclustered.ASD patients had a significant lower methylation rate in BDNF promoterⅠCpG unit 5.6 andⅣCpG units 3 and 15 compare with healthy controls(P<0.05).Conclusions The DNA methylation rate in BDNF promoterⅠCpG unit 5.6 andⅣCpG units 3 and 15 may be used as potential biomarkers of ASD.
Autism Spectrum Disordersbrain derived neurotrophic factorPromoterMethylation rate
R749.93
A
2016-10-23)
(责任编辑:肖雅妮)
10.3969/j.issn.1002-0152.2017.02.008
☆深圳市科创委基础研究项目(编号:JCYJ20130401114111453);深圳市科技创新委员会基础研究项目(编号:JCYJ20140416141331555)
* 广东省深圳市儿童医院心理科(深圳518038)
** 广东省深圳市儿童医院科教科
*** 广东省深圳市儿童医院儿科研究所
**** 广东省深圳市儿童医院言语治疗科
△ 深圳市众循精准医学研究院,自闭症基因组研究所

