日本鳗鲡IFN-γ 基因的鉴定、表达模式及启动子活性分析
彭喜霞黄 贝段利朋李春艳梁 英聂 品,黄文树,
(1. 集美大学水产学院, 厦门 361021; 2. 中国科学院水生生物研究所淡水生态与生物技术国家重点实验室, 武汉 430072; 3. 鳗鲡现代产业技术教育部工程研究中心, 厦门 361021; 4. 福建省海洋生物资源开发利用协同创新中心, 厦门 361005)
日本鳗鲡IFN-γ 基因的鉴定、表达模式及启动子活性分析
彭喜霞1黄 贝1段利朋1李春艳1梁 英1聂 品1,2黄文树1,3,4
(1. 集美大学水产学院, 厦门 361021; 2. 中国科学院水生生物研究所淡水生态与生物技术国家重点实验室, 武汉 430072; 3. 鳗鲡现代产业技术教育部工程研究中心, 厦门 361021; 4. 福建省海洋生物资源开发利用协同创新中心, 厦门 361005)
为揭示鱼类IFN-γ的生物学功能, 研究从日本鳗鲡(Anguilla japonica)中克隆获得了IFN-γ基因, 命名为AjIFN-γ。AjIFN-γ具有脊椎动物IFN-γ的典型特征: 包括4外显子/3内含子的基因结构、C端的IFN-γ特征性氨基酸基序和1个核定位信号, 以及6个α-螺旋反向平行构成的二级结构。AjIFN-γ在日本鳗鲡所有组织中均低水平转录表达, 其中肝脏中表达量最高, 其次是皮肤和头肾。Poly I:C刺激和迟缓爱德华氏菌感染均可显著诱导AjIFN-γ在鳃、头肾、体肾和(或)脾脏中的转录表达, 表明AjIFN-γ能够参与日本鳗鲡抗菌和抗病毒的免疫过程。此外, 研究还克隆了AjIFN-γ基因的5′调控区序列共1536 bp, 并构建了一系列AjIFN-γ 5′调控区删节突变体, 分析其启动子活性, 结果表明, 上游–240/+136区域中含有起始AjIFN-γ转录的关键启动子调控元件,–1062/–814区域存在转录的正调控元件, 而–1252/–1062区域存在转录的负调控元件。上述结果进一步丰富了鱼类IFN-γ的基础知识。
IFN-γ; 日本鳗鲡; 转录表达; 启动子
在哺乳动物中, IFN-γ (interferon-γ, 干扰素γ)是II型IFN的唯一成员, 主要由CD4+辅助T淋巴细胞、CD8+细胞毒性T细胞、NK细胞以及APC细胞产生[1—3]。IFN-γ在进化中高度保守, 其基因组均由4个外显子和3个内含子结构组成, 氨基酸序列的C端含有IFN-γ特征基序和由4个连续的赖氨酸或精氨酸组成的核定位基序(Nuclear localization site; NLS)。IFN-γ不仅可调节前炎症细胞因子和趋化因子的产生、激活巨噬细胞的吞噬作用、刺激具有抗菌活性的氧中间体和一氧化氮的产生和释放, 还可促进Th0细胞分化为Th1细胞, 并诱导MHCⅠ、MHCⅡ类分子和抗原提呈细胞上协同刺激分子的表达以及B细胞中免疫球蛋白类型转换[4], 在宿主抵抗病毒和胞内细菌感染的过程中发挥着重要的作用。
与哺乳动物不同, 鱼类基因组中发现两个紧密连锁的IFN-γ基因。第一个IFN-γ是Zou等[5]通过基因共线性分析从河鲀(Takifugu rubripes)中首先发现。2006年, Igawa等[6]发现在斑马鱼(Danio rerio)和绿河鲀(Tetraodon nigrovirdis)基因组IFN-γ的附近发现另一个IFN-γ同源基因, 即IFN-γrel (IFN-γ related gene), 随后, 从斑点叉尾(Ictalurus punctatus)[7]、鲤(Cyprinus carpio)[8]、金鱼(Carassius auratus auratus)[9]和草鱼(Ctenopharyngodon idellus)[10,11]等鱼类克隆了该基因。研究发现该两个IFN-γ基因均为4个外显子/3个内含子结构, 其氨基酸序列的C-端都含有一段保守的IFN-γ特征基序, 然而, IFN-γ的C-端有1个NLS(由4个精氨酸或赖氨酸组成), 而IFN-γrel则无。近年, Shibasaki等[12]在鲫(Carassius auratus langsdorfi)和金鱼中再克隆出一个IFN-γrel基因, 并提出根据其氨基酸序列的C端是否具有NLS将鲤科鱼类IFN-γrel分为两类, 即IFN-γrel1的C端有一段富含碱性氨基酸的NLS, 在金鱼和鲫中均为“KHHHR”, 而IFN-γrel2则缺乏该NLS。根据该分类特征, 目前已发现的斑马鱼和草鱼IFN-γrel均属于IFN-γrel1, 而鲤IFN-γrel则属于IFN-γrel2。除绿河鲀外, 目前克隆到的鱼类IFN-γrel基因均源自鲤形总目(1971年拉斯分类系统), 无法确定以上IFN-γrel的特征是鱼类共有, 还是鲤形总目鱼类所特有的。
IFN-γ与哺乳动物IFN-γ具有类似的功能。首先, 鱼类IFN-γ参与鱼类抗病毒反应: Poly I:C可显著诱导虹鳟(Oncorhynchus mykiss)头肾细胞、斑马鱼的鳃和肠、大西洋鳕(Gadus morhua)的头肾以及大黄鱼(Pseudosciaena crocea)多个组织(肾、脾脏、鳃、头肾、皮肤和胸腺)中IFN-γ的转录表达[6,13—15];鲤科疱疹病毒三型(Cyprinid herpesvirus-3, CyHV-3)可显著诱导鲤IFN-γ和IFN-γrel的转录表达[16]。重组表达的鱼类IFN-γ可诱导抗病毒基因GBP (Guanylate-Binding Protein)、ISG (Interferon-Stimulated Gene)和Mx (Myxovirus-resistant)等的表达[17—19]; 鲫IFN-γ能够显著提高浸泡感染了CHNV (Crucian Carp Hematopoietic Necrosis Virus)鲫胸腺细胞(GTS9细胞)的存活率[12], 大西洋鲑IFN-γ具有抗IPNV (Infectious Pancreatic Necrosis Virus)和SAV3 (Salmonid Alphavirus 3)的生物活性[18]。其次, 鱼类IFN-γ也参与抗菌免疫反应: LPS刺激可上调斑马鱼和鲤IFN-γrel基因的表达量[6,8], 鳗弧菌(Vibrio anguillarum)及哈维氏弧菌(Vibrio harveyi)可分别诱导IFN-γ基因在大西洋鳕头肾组织和大黄鱼多个组织(肝脏、脾脏和头肾等)中的表达[14,15]。与哺乳动物IFN-γ相似, 鱼类IFN-γ也可激活巨噬细胞和诱导MHC II类分子表达[4,9,13,20,21]。此外, IFN-γ还可参与鱼类抗寄生虫感染的免疫过程: 刺激隐核虫(Cryptocryon irritans)感染可诱导大黄鱼脾脏、肠和皮肤等组织中IFN-γ的表达[15], 血液寄生的拟锥虫(Trypanoplasma borreli)感染也可上调鲤IFN-γ的表达[8]。
日本鳗鲡(Anguilla japonica)属于鳗鲡目鳗鲡科, 是重要的经济鱼类。在养殖过程中, 细菌、寄生虫、真菌和病毒性疾病时有发生, 给鳗鲡养殖业带来巨大的损失[22]。然而, 目前有关日本鳗鲡抗病基因相关研究仍较为薄弱。本文首先从日本鳗鲡中克隆获得IFN-γ基因(命名AjIFN-γ), 并研究AjIFN-γ在健康状态以及病原感染情况下的表达模式, 初步分析了AjIFN-γ 5′-调控区的转录调控活性。本研究将目前鱼类IFN-γ的研究领域延伸到鳗鲡总目, 将有助于更全面地了解鱼类IFN-γ的结构、功能以及进化过程, 同时, 为深入探索鳗鲡的免疫防御机制提供基础资料。
1 材料与方法
1.1 实验材料
日本鳗鲡[质量(203±53) g]购于福建省集美大学水产养殖基地, 养殖水温为(28±2)℃。迟缓爱德华氏菌(Edwardsiella tarda)为本实验室保种。AJSB细胞株为本实验室构建, 未发表。
1.2 AjIFN-γ cDNA全长序列的扩增
根据cDNA文库中获得的IFN-γEST序列, 设计引物AjIFN-gF/AjIFN-gR, 以日本鳗鲡肠道cDNA为模板, PCR扩增获得AjIFN-γcDNA部分序列。反应体系(25 μL)为: HSTMReaction Mix 12.5 μL(HSTMkit, 东盛生物), ddH2O 9.25 μL, 上下游引物各1.0 μL, HSTMTaq聚合酶0.25 μL(HSTMkit, 东盛生物), 模板1.0 μL。反应程序为: 94℃ 3min; 94℃ 30s, 62℃30s, 72℃ 40s, 运行38个循环; 72℃ 10min。然后根据获得的部分cDNA序列, 参照SMARTerTMRACE cDNA Amplification Kit(Clontech, 美国)试剂盒原理, 设计合成3′-和5′-RACE特异性引物(表 1)。以肠道3′-和5′-SMART cDNA为模板, 进行巢式PCR。拼接RACE获得的3′-和5′-末端序列与cDNA部分序列, 得到AjIFN-γ基因的cDNA全长序列。再根据拼接获得的cDNA全长序列, 在UTR区设计引物(表1), 通过PCR验证拼接的cDNA全长序列。PCR产物经电泳、回收、连接、转化后将阳性克隆子送测序, 具体操作参照本课题组前期研究[23]。
1.3 AjIFN-γ基因序列生物信息学分析
利用NCBI网站中的Blast (http://blast.ncbi.nlm. nih.gov/Blast.cgi)进行序列比对, 使用Primer Premier 6软件设计引物, 利用ExPASy的翻译工具(http:// web.expasy.org/translate/)获得对应的氨基酸序列, ExPASy (http://web.expasy.org/compute_pi/)预测蛋白分子量和pI值, 用SignalP 4.1 Server程序(http:// www.cbs.dtu.dk/services/SignalP/)分析信号肽, 使用NetNGlyc 1.0程序(http://www.cbs.dtu.dk/services/ NetNGlyc/)预测潜在的N-糖基化位点, 分别使用PSIPRED程序(http://bioinf.cs.ucl.ac.uk/psipred/)和Phyre2程序(http://www.sbg.bio.ic.ac.uk/phyre2/)预测蛋白的二级和三级结构。用ClastalW进行氨基酸序列多重比对; 使用MEGA 6.0中的邻接法构建系统进化树, Bootstrap置换1000次。
1.4 AjIFN-γ的表达模式分析
迟缓爱德华氏菌菌液制备: 取迟缓爱德华氏菌菌株划线于LB琼脂平板, 37℃培养16h, 挑取单克隆于1 mL LB液体培养基中, 37℃, 180 r/min培养4h。取100 μL菌液按1:100 (体积比)加入LB液体培养基, 37℃, 180 r/min培养至菌液A600达到0.5—0.7时, 收菌。取500 μL菌液, 梯度稀释后各取100 μL涂布在LB琼脂平板上, 37℃培养16h后计数菌落单位, 推算原菌液的浓度, 用PBS缓冲液洗涤并稀释至使用浓度。
正常养殖鳗鲡不同组织中的表达差异: 采集健康日本鳗鲡(黑仔鳗)血液、鳃、心脏、肝脏、肠、胃、脾脏、头肾、体肾、性腺、鳔和皮肤共12个组织/器官, 使用Lightcycler 480 SYBR Green I试剂盒(Roche, 德国)在Ligthcycler 480 II PCR仪(Roche,德国)上, 参照Wang等[24]方法进行实时荧光定量PCR, 研究AjIFN-γ在不同组织中的转录表达量差异。β-actin和AjIFN-γ的定量引物的荧光信号采集温度分别为80℃、82℃和76℃。通过Excel工具计算AjIFN-γ与β-actin的拷贝数比值, 以确定AjIFN-γ在mRNA水平相对β-actin的表达量, 使用DPS 7.0数据处理系统进行统计分析, 使用GraphPad Prism 6.0软件绘图。
不同免疫物刺激后的表达差异: 实验设PBS对照组、Poly I:C刺激组(1 mg/100 g鱼, Sigma)和迟缓爱德华氏菌刺激组(2×107cfu/100 g鱼), 免疫方法是腹腔注射。在注射后8h、16h、24h和72h分别采集脾脏、鳃、头肾及体肾, 每个时间点、每条鱼的每个组织作为单独样品, 进行实时荧光定量PCR检测。定量数据经β-actin校正后, 以相应时间点、相同组织的PBS对照组中靶基因的表达量为基准计算AjIFN-γ相对表达量的倍数变化。结果用Excel软件进行计算, 用单因素变量方差分析法(ANOVA)分析刺激组与PBS对照组之间的表达差异的显著性。
1.5 AjIFN-γ基因启动子活性分析
根据日本鳗鲡基因组数据设计引物AjIFN-γ ProF/AjIFN-γProR, 以日本鳗鲡肌肉基因组为模板,PCR扩增获得AjIFN-γ基因上游约2000 bp的调控序列。利用TSSG程序(http://linux1.softberry.com)预测其启动子转录起始位点(Transcriptional Start Site, TSS)及TATA-box。其调控区的转录因子结合位点(Transcription Factor Binding Site, TFBS)的预测利用Alibaba 2程序(http://www.gene-regulation.com/ pub/programs/alibaba2/)和PAPIA程序(http://mbs. cbrc.jp/papia/)。

表 1 本文中所用引物序列Tab. 1 Primers used in the present study
根据克隆的5′调控序列以及预测的TFBS, 设计5′调控区删节引物(表 1)。上、下游引物5′-端分别添加限制性内切酶MluⅠ和Bgl Ⅱ的酶切位点。以pMD19-T-AjIFN-gPro质粒作为模板, PCR扩增系列5′调控区删节片段: P1 (–1400/+136)、P2 (–1252/+ 136)、P3 (–1062/+136)、P4 (–814/+136)、P5 (–543/+ 136)和P6 (–240/+136), 经限制性内切酶MluⅠ和BglⅡ酶切后, 连接至pGL3-basic载体(Promega, 美国),构建删节突变体质粒。使用E.Z.N.A Endo-Free Plasmid DNA Mini Kit (Omega, 美国)提取相应质粒并测序验证。
将AjIFN-γ基因5′调控区删节突变体质粒转染AJSB细胞, 具体操作如下: 将AJSB细胞(1.5×105cell/mL)接种到24孔细胞培养板中, 28℃培养48h至贴壁后,使用Lipofectamine 3000 (Invitrogen, 美国)进行转染。每孔加入相应删节突变体质粒0.5 μg和内参质粒pRL-TK (Promega, 美国)0.005 μg, 对照组转染pGL3-basic质粒和pRL-TK质粒。转染24h后, 用Dual-Luciferase Reporter Assay System (Promega, 美国)检测细胞裂解产物中的萤火虫荧光素酶和海肾荧光素酶活性。每组设3个重复。
2 结果
2.1 AjIFN-γ基因序列分析
根据EST序列设计引物, PCR扩增获得cDNA序列, 再设计RACE引物, 经克隆、拼接和PCR验证, 获得AjIFN-γ的全长cDNA序列, GenBank登录号为KU950362。AjIFN-γcDNA序列全长共912 bp, 开放阅读框546 bp, 可编码181个氨基酸(aa)的多肽, 预测该多肽N-端21 aa为信号肽。同时, 本实验室还克隆了日本鳗鲡IFN-γrel (AjIFN-γrel)的cDNA序列(登录号为KU950363), AjIFN-γrelcDNA全长为888 bp,开放阅读框为483 bp可编码160个aa的多肽, 比AjIFN-γ少21 aa, 其N-端23 aa为信号肽。AjIFN-γ cDNA的3′-UTR含有IFN-γ序列中普遍存在的mRNA不稳定基序(ATTTA)3个, 而在AjIFN-γrelcDNA的3′-UTR中没有发现该基序。AjIFN-γ的C-端具有IFN-γ特征基序“T-Q-R-K-A-I-R-D-L-L-T-V”和1个NLS (RRRR), 然而, AjIFN-γrel仅具有部分IFN-γ特征基序, 并且与其他鱼类的IFN-γrel相似, AjIFN-γrel的C-端也缺乏NLS以及鲤形总目鱼类IFN-γrel中保守的“CTC”基序(图 1)。
氨基酸序列同源性分析显示, AjIFN-γ与AjIFN-γrel的一致性和相似性分别为38.88%和50.66%。PSIPRED程序预测结果显示, AjIFN-γ的成熟肽二级结构由6个α-螺旋组成, 而AjIFN-γrel仅有4个α-螺旋, 其位置与AjIFN-γ前4个螺旋分别对应(图 1)。进一步使用Phyre2程序构建了AjIFN-γ和AjIFN-γrel成熟肽的三级结构模型。结果显示, AjIFN-γ由6个长短不一的α-螺旋反向平行构成(图 2A), 而AjIFN-γrel则仅由4个α-螺旋反向平行构成(图 2B)。
2.2 脊椎动物IFN-γ氨基酸序列多重比对
选取人类、小鼠、鸡的IFN-γ, 以及硬骨鱼类IFN-γ和IFN-γrel的氨基酸全长序列进行多重比对。结果显示, 虽然脊椎动物IFN-γ氨基酸序列一致性较低,但是, 都具有特征基序“[I/V]-Q-X-[K/Q]-A-X2-E-[L/F]-X2-[I/V]”和由4个连续精氨酸或赖氨酸组成的NLS; 鱼类IFN-γrel均缺乏该类NLS, 此外, 鲤形总目鱼类IFN-γrel氨基酸序列的近1/2处有一个“CTC”基序, 而AjIFN-γrel和绿河鲀IFN-γrel则无(图 1)。N-糖基化位点预测结果显示, 爪蟾和大西洋鳕均缺少N-糖基化位点, 而褐牙鲆有3个, 其他IFN-γ和IFN-γrel均具有1—2个, 其中AjIFN-γ和AjIFN-γrel均具有2个糖基化位点(图 1、表 2)。
2.3 AjIFN-γ同源性分析
为了研究日本鳗鲡II型IFN与其他脊椎动物II型IFN的同源性, 使用ClustalW软件和SIAS程序(http://imed.med.ucm.es/Tools/sias.html)分析其氨基酸成熟肽序列的一致性和相似性。AjIFN-γ与其他脊椎动物IFN-γ的一致性均相对稍高于AjIFN-γrel: AjIFN-γ与高等脊椎动物IFN-γ的一致性为14.3%—20%, 而AjIFN-γrel仅为9%—17.5%; AjIFN-γ与其他鱼类IFN-γ的一致性为20%—30.7%, AjIFN-γrel为16.8%—27%。除斑点叉尾和绿河鲀外, AjIFN-γrel与其他鱼类IFN-γrel的相似性高于其与同种鱼类IFN-γ的相似性。鲇形目和鲑形目鱼类IFN-γ与日本鳗鲡II型IFN的同源性相对高于其他鱼类:在所比较的鱼类IFN-γ中, 与AjIFN-γ的同源性最高者为大西洋鲑, 与AjIFN-γrel同源性最高的是斑点叉尾; 与AjIFN-γ和AjIFN-γrel的同源性最低的均为大黄鱼IFN-γ (表 2)。
2.4 AjIFN-γ基因结构分析
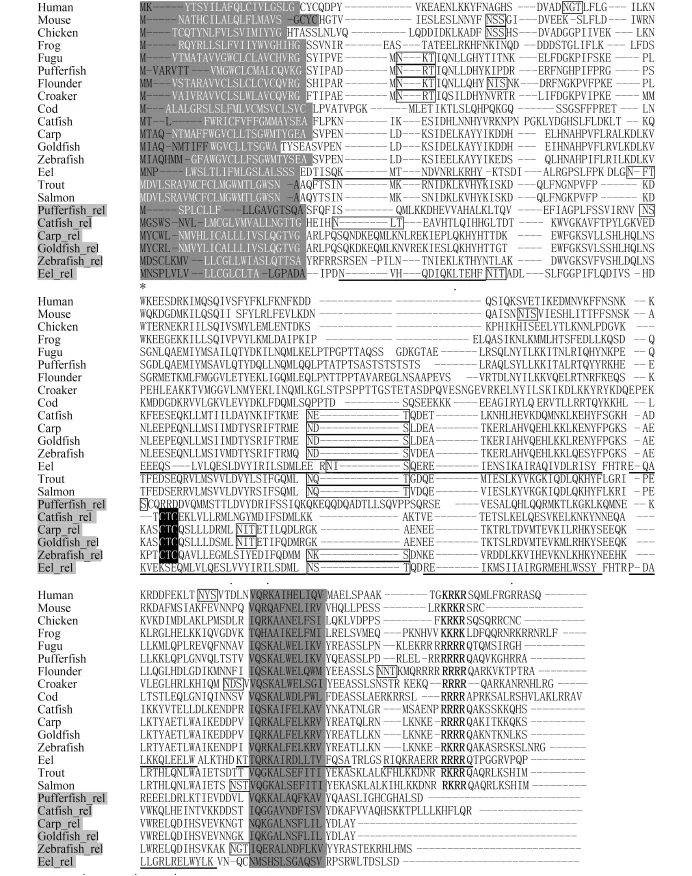
图 1 氨基酸序列多重比对Fig. 1 Multiple alignment of deduced amino acid sequence of IFN-γs and IFN-γrels“–”表示空缺; “*”表示一致, “.”或“:”表示相似; 粗体为NLS; 灰底黑字为IFN-γ特征基序, 灰色名称为IFN-γrel; 下划线为预测的α-螺旋;灰底白字为信号肽序列; 方框内为预测的N-糖基化位点; 黑色背景为鲤科鱼类中保守的“CTC”基序”Dashes (–) indicate gaps in the alignment, asterisks (*) indicate identity and dots (or :) indicate similarity. Nuclear localization signals are in bold and the conserved IFN-γ family signatures are depicted in dark grey. Sequences underlined represent the predicted α-helices. The predicted signal peptides are in italic. The predicted N-glycosylation sites are showed in boxes. The conserved “CTC” motif is shaded in black
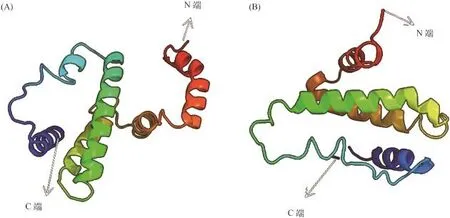
图 2 AjIFN-γ(A)和AjIFN-γrel(B)成熟肽的三级结构Fig. 2 3D structures for AjIFN-γ (A) and AjIFN-γrel (B) mature peptides
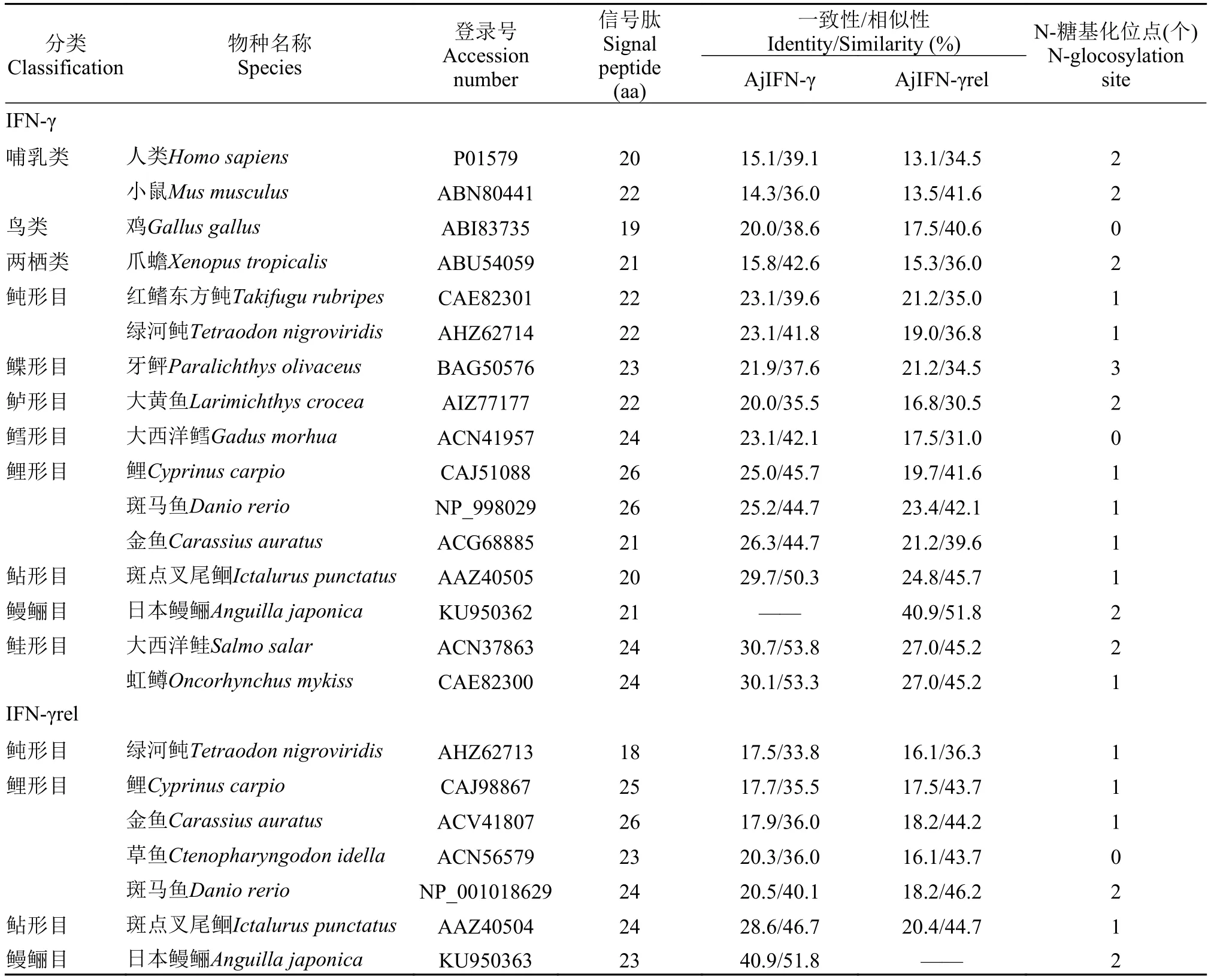
表 2 日本鳗鲡Ⅱ型IFN与其他脊椎动物Ⅱ型IFN的同源性Tab. 2 Protein homology between type Ⅱ IFN from Japanese eel and other vetebrates
使用Spidey程序(http://www.ncbi.nlm.nih.gov/ spidey/)比对AjIFN-γ和AjIFN-γrel的cDNA序列和基因组序列, 获得其基因结构; 同时, 从Ensembl数据库中提取人类、小鼠、鸡、爪蟾、斑马鱼以及青鳉的IFN-γ以及斑马鱼和草鱼IFN-γrel的基因数据;使用GeneMaper 2.5 绘制基因结构图。结果显示,虽然不同物种间IFN-γ的总核苷酸数量差异较大(817—5601 bp), 但是其基因结构相似, 都由4个外显子和3个内含子组成, 所有内含子均为0相位, 4个外显子的相对大小比较保守, 最小的均为外显子2,最大的均为外显子3 (图 3A), 表明IFN-γ的基因结构在进化过程中较为保守。
斑马鱼和草鱼的IFN-γrel与脊椎动物IFN-γ的基因结构相似, 也是由4个外显子和3个内含子组成,最小和最大的外显子也分别是外显子2和外显子3,但是, 其外显子1都大于IFN-γ的外显子1, 而外显子4均小于IFN-γ外显子4。值得注意的是, 与斑马鱼和草鱼IFN-γrel不同, AjIFN-γrel的基因结构仅由3个外显子和2个内含子组成, 且外显子3的大小约为其他物种的IFN-γ或IFN-γrel的外显子3和外显子4的大小之和(图 3B)。
2.5 AjIFN-γ基因簇基因共线性分析
低等脊椎动物与哺乳动物的染色体存在共线性, 许多细胞因子基因在不同动物染色体上的位置相对保守[5,25,26]。同时, 由于鱼类细胞因子与哺乳动物的同源基因间同源性低, 因此, 染色体共线性分析已成为寻找鱼类细胞因子基因的常用方法之一。利用该方法, 已在鱼类中发现了许多细胞因子基因, 包括IL-10、IL-22、IFN-γ、IL-2和IL-21等[5,6,27—29]。本研究将克隆获得的AjIFN-γ和AjIFN-γrel基因cDNA序列在日本鳗鲡基因组数据(Gen-Bank登录号: AVPY00000000.1)中进行比对, 发现AjIFN-γ位于日本鳗鲡基因组数据库Scoffold_1498,运用FGENESH网站(http://www.softberry.com/berry. phtml)对Scoffold_1498进行基因预测, 发现6个基因, 其中前5个分别为MDM1、IL-22、IL-26、IFN-γ和IFN-γrel, 第6个为假定基因。我们选择了人类、小鼠、爪蟾、斑马鱼和绿河鲀与日本鳗鲡的IFN-γ基因簇进行共线性分析, 结果显示, 与人类、非洲爪蟾和斑马鱼一致, AjIFN-γ上游基因主要包括MDM1、IL-22、IL-26, 但是在非洲爪蟾IL-22和MDM1之间插入了一个MDM4基因, 而在绿河鲀的IL-22与MDM1基因之间则插入了一个RAP1B基因,该基因在人类和小鼠中均位于MDM1基因的上游。小鼠IFN-γ上游有两个IL-22基因(IL-22a和IL-22b),但是其IL-26基因为假基因, 在其序列中间插入了一段由长散布核元件(Long Interspersed Nuclear Elements, LINE)和长末端重复序列(Long Terminal Re-peat, LTR)组成的(LTR)-LINE-LTR结构[30]。与其他脊椎动物相比, 鱼类IFN-γ的上游额外增加一个IFN-γrel基因, 因此, 认为该基因应为鱼类所特有。除了小鼠IL-22b和非洲爪蟾MDM1以外, IFN-γ基因簇上各基因的转录方向均相同(图 4)。

图 3 IFN-γ (A)和IFN-γrel (B)基因结构比较分析Fig. 3 Comparative analysis of IFN-γ (A) and IFN-γrel (B) genomic structures线条表示内含子, 斜线框表示外显子, 数字表示内含子或外显子的碱基数; 基因结构数据来源于Ensembl基因组数据库Exons are showed as slash boxes and introns are presented as lines. Numbers above boxes and below lines indicate the size of exons and introns, respectively. The data are derived from Ensembl database
2.6 AjIFN-γ在鱼体中的组织分布
采用实时荧光定量方法检测AjIFN-γ在日本鳗鲡皮肤、鳃、肠、胃、肝脏、脾脏、心脏、鳔、头肾、体肾、性腺和血液共12个组织/器官中的表达量。结果表明, AjIFN-γ在健康日本鳗鲡的所有检测组织中均有不同程度的低水平转录表达, 表达量约为β-actin的1‰, 其中, AjIFN-γ肝脏中的转录表达量最高, 其次是皮肤和头肾, 在胃、性腺和鳔中的转录表达量最低(图 5)。
2.7 Poly I:C刺激能诱导AjIFN-γ基因的转录表达
为了研究Poly I:C刺激对重要免疫器官中AjIFN-γ转录表达的诱导作用, 本研究对健康日本鳗鲡腹腔注射Poly I:C, 注射后8h、16h、24h以及72h, 采用实时荧光定量的方法分别检测AjIFN-γ在鳃、脾脏、头肾和体肾中的表达量, 对照组注射等体积PBS。结果显示, Poly I:C能够显著上调AjIFN-γ基因的转录表达水平(P<0.05)。除脾脏以外, 其他3个组织中AjIFN-γ基因的转录表达量均在Poly I:C刺激后8h极显著上调(P<0.01)。在鳃和体肾中, 表达量在16h达到最高, 而后下降, 在24h仍显著高于对照组, 72h恢复到与对照组相似水平。在脾脏中, AjIFN-γ基因的转录表达量在刺激后各个时间点均无显著变化(P>0.05)(图 6)。
2.8 迟缓爱德华氏菌感染能诱导AjIFN-γ基因的转录表达
迟缓爱德华氏菌是日本鳗鲡常见的致病菌之一, 本研究检测了健康日本鳗鲡腹腔注射迟缓爱德华氏菌后8h、16h、24h和72h, AjIFN-γ在鳃、脾脏、头肾和体肾中的转录表达量, 对照组注射等体积PBS。结果显示, 迟缓爱德华氏菌感染可显著诱导AjIFN-γ在鳃、头肾、体肾和脾脏中的表达(P<0.05)。在鳃、头肾和体肾中, 迟缓爱德华氏菌感染8h后, AjIFN-γ的表达量先略为下调, 而后在16h显著上调(P<0.05), 在24h达到最高水平; 在体肾中, 刺激后72h, AjIFN-γ的表达量仍极显著高于对照组(P<0.001), 而在鳃和头肾中则与对照组无差异; 在脾脏中, AjIFN-γ的表达量在8h、16h和24h均显著下调(P<0.05), 但是呈现逐步上升的趋势, 在72h表达量达到最高, 且极显著高于对照组(P<0.001)(图 7)。
2.9 AjIFN-γ基因启动子区转录因子结合位点和删节突变体活性分析
扩增日本鳗鲡AjIFN-γ基因5′-侧翼区长为1536 bp的序列, 预测其转录起始位点(TSS)位于起始密码子上游155 bp处, TATA-box位于TSS上游24 bp处; AjIFN-γ基因5′-侧翼区的转录因子结合位点(TFBS)包括GATA-1 (GATA结合因子-1)、SRF(血清应答因子)、AP-1(激活蛋白-1)、IRF-1 (干扰素调节因子-1)、C/EBPβ (CCAAT增强子结合蛋白β)、NF-κB (核因子-κB)等(图 8A)。
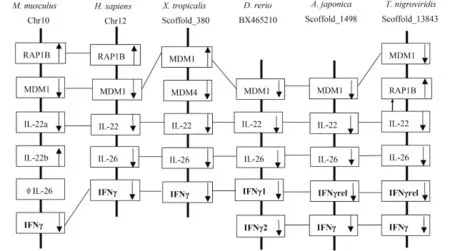
图 4 AjIFN-γ基因座的基因共线性比较分析Fig. 4 Gene synteny analysis of the AjIFN-γ gene locus with that from human, mouse, zebrafish and Xenopus箭头指示基因的转录方向, 方框内为基因名称。“ψ”表示假基因Arrows indicate gene transcription orientation. ψ represents pseudo gene
为研究AjIFN-γ启动子活性的调控序列, 以pGL3-basic载体, 构建了6个5′-端删节片段的突变体质粒(P1—P6), 并分别转染AJSB细胞, 测定各删节突变体的相对荧光素酶活性。以pGL3-basic为对照组。结果显示, 6个AjIFN-γ启动子删节突变体的相对活性都显著高于pGL3-basic。P6(–240/+ 136)活性约为pGL3-basic的40倍; P3(–1062/+136)活性最高, 为pGL3-basic的157倍, 并且显著高于P2(–1252/+136)和P4(–814/+136), 而P1、P2和P4之间相对活性无显著差异, 但其活性显著高于pGL3-basic, 为后者的48—68倍(图 8B)。
3 讨论
哺乳动物、鸟类和两栖类的Ⅱ型IFN成员均只有1个, 而鱼类的Ⅱ型IFN包括IFN-γ和IFN-γrel。本研究克隆获得了日本鳗鲡IFN-γ基因(AjIFN-γ)的cDNA序列, 并与本实验室已经克隆的日本鳗鲡IFN-γrel的cDNA序列进行了比较分析, 同时, 对AjIFN-γ和AjIFN-γrel进行了基因结构和共线性分析。此外, 研究了AjIFN-γ在健康日本鳗鲡中的组织分布及Poly I:C诱导和迟缓爱德华氏菌感染诱导其转录表达的效应, 初步揭示其在抗菌和抗病毒方面的作用。此外, 我们还克隆了AjIFN-γ的5′调控区序列,预测其中的TFBS, 并研究了其启动子活性。
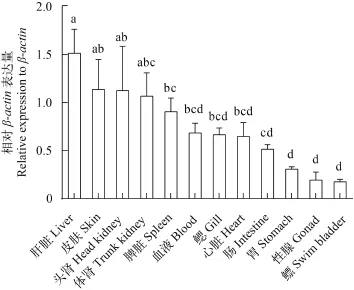
图 5 健康日本鳗鲡不同组织/器官中AjIFN-γ相对β-actin的表达量Fig. 5 Relative expressions of AjIFN-γ in various tissues of healthy Japanese eel relative to β-actin数值通过1000×(AjIFN-γ/β-actin)公式计算; 纵轴表示平均值±SEM, N=6; 相同字母表示没有显著性差异, P>0.05The value is present by 1000×(AjIFN-γ/β-actin). Vertical bars represent mean±SEM, N=6. Same letter above the bars means no significant difference. P>0.05
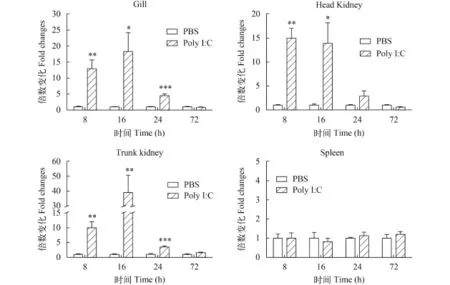
图 6 腹腔注射Poly I:C后AjIFN-γ相对表达量Fig. 6 The relative expression of AjIFN-γ in various tissues after intraperitoneal injection with Poly I:CPoly I:C刺激后8h、16h、24h、72h时, 采用real-time PCR分析不同组织中AjIFN-γ的转录本数量; 以每个组织中β-actin作为内参比; 纵轴表示平均值± SEM, N=6 (鳃16h组, N=5); *P<0.05, **P<0.01, ***P<0.001, t-test; 下同Real-time PCR was used to analyze the amount of AjIFN-γ transcript in different tissues at 8h, 16h, 24h, 72h after being stimulated by Poly I:C in vivo. Copies of β-actin in each tissue was used as an internal control for normalized different samples. Vertical bars represented the mean±SEM, N=6 (for gill at 16h, N=5) *P<0.05, **P<0.01 and ***P<0.001, t-test; the same applies below

图 7 腹腔注射E. tarda后AjIFN-γ的相对表达量Fig. 7 The relative expression of AjIFN-γ after intraperitoneal injection with E. tardaE. tarda 刺激后8h、16h、24h、72h时, 采用real-time PCR分析不同组织中AjIFN-γ的转录本数量Real-time PCR was used to analyze the amount of AjIFN-γ transcript in different tissues at 8h, 16h, 24h and 72h after being challenged by E. tardain in vivo
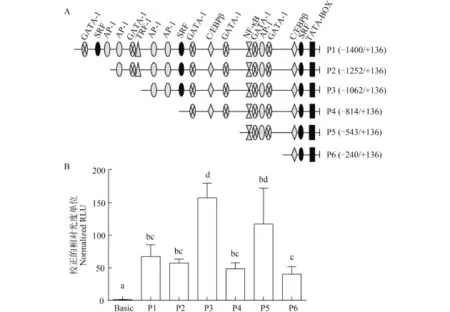
图 8 AjIFN-γ启动子删节突变体的相对活性Fig. 8 Relative luciferase activities of promoter 5′-deletion mutants of AjIFN-γA. 用于构建AjIFN-γ启动子5′-端删节突变体的插入序列及其预测的转录因子结合位点示意图; B. AjIFN-γ启动子5′-端删节突变体在AJSB细胞中的相对活性; 启动子活性以经海肾荧光素酶活性校正的相对光度单位(RLU)表示A. Schematic diagram of the promoter deletion mutants in different length and the TFBSs on them; B. Relative luciferase activities of promoter 5′-deletion mutants of AjIFN-γ in AJSB cells
3.1 AjIFN-γ具有鱼类Ⅱ型IFN的典型特征
虽然AjIFN-γ与其他物种IFN-γ的一致性较低,但是, 具有IFN-γ的典型特征, 包括特征基序“[I/V]-Q-X-[K/Q]-A-X2-E-[L/F]-X2-[I/V]”、C端保守的NLS (“RRRR”)以及6个α-螺旋构成的高级结构。迄今, 仅在鱼类中发现了第二个Ⅱ型IFN, 即IFN-γrel,其最初被命名为IFN-γ1[6], 随后, 发现其与鱼类IFN-γ同源性不高, 且与高等脊椎动物IFN-γ的同源性低, 更名为IFN-γrel[31]。与其他鱼类IFN-γrel相似,日本鳗鲡IFN-γrel也缺少鱼类IFN-γ的C端高度保守的由连续4个赖氨酸或精氨酸组成的NLS (为区分,以下记为NLS1)。尽管有研究认为虹鳟IFN-γ缺失NLS1, 其生物活性受影响[13], 然而, Grayfer等[21]研究发现, 缺乏NLS1的重组金鱼IFN-γrel, 仍然具有诱导单核细胞反应性氧中间物(ROI)的产生、激活巨噬细胞的吞噬作用、通过诱导iNOS基因的表达增加NO的产生、诱导前炎症细胞因子和趋化因子的表达以及STAT1的磷酸化作用等生物活性。最近, Shibasaki等[12]在鲫和金鱼中分别克隆到2个IFN-γrel, 部分鱼类IFN-γrel的C端具有一个富含碱性氨基酸的NLS(记为NLS2, 在金鱼和鲫中均为“KHHHR”), NLS2也能够发挥核定位作用。此外,他们根据IFN-γrel中是否含有NLS2分为IFN-γrel1,主要包括鲤科鱼类; 缺乏NLS2的IFN-γrel为IFN-γrel2。本实验室克隆到的AjIFN-γrel与Shibasaki等[12]所描述的IFN-γrel2相似, 也缺少NLS2。此外, AjIFN-γrel和绿河鲀IFN-γrel均缺乏鲤形总目鱼类中高度保守的“CTC”基序, 推测该基序只存在于鲤形总目鱼类中。
3.2 AjIFN-γ主要参与日本鳗鲡的抗病毒免疫过程
早期采用半定量PCR方法, 在健康斑马鱼和虹鳟的多个组织中均未发现IFN-γ的转录表达[6,13],而斑点叉尾IFN-γ仅在头肾和脾脏中检测到一定的表达[7]。而采用灵敏度更高的荧光定量PCR方法后, 发现IFN-γ在健康鱼体的不同组织中均有表达, 尤其是鳃和脾脏。此外, 金鱼和南亚野鲮(Labeo rohita) IFN-γ在脾脏中的表达量均高于其他组织[9,32], 鲤IFN-γ在肠和鳃中的表达量最高[8], 大西洋鳕IFN-γ在鳃和脾脏中表达量最高[14]。本研究中AjIFN-γ在所检测的12个组织中均低水平表达, 其表达模式与大黄鱼IFN-γ相似, 主要在肝脏以及主要免疫组织(皮肤和头肾)中表达, 暗示其可能参与机体的免疫过程。
与哺乳动物相似, 鱼类IFN-γ参与机体的抗病毒反应。经Poly I:C诱导后, 虹鳟(头肾白细胞、肾和脾脏)、斑马鱼(鳃和肠)、大西洋鳕(头肾)、金鱼(肾白细胞)IFN-γ的转录表达量均上调[6,9,13,14]。在鲤科疱疹病毒三型感染的每一个阶段, 鲤IFN-γ和IFN-γrel的转录表达量均显著上调[16]。Stolte等[8]发现鲤IFN-γrel主要由IgM+细胞产生, 表明其主要调节体液免疫[8,33]。在本研究中, Poly I:C刺激后, AjIFN-γ在鳃、头肾和体肾中的表达量显著上调, 尤其是体肾中, 上调约39倍, 而头肾和体肾是硬骨鱼类抗体产生的重要器官[34], 鱼类鳃中也含有抗体分泌细胞[35,36]以及T淋巴细胞[37], 并且能高水平表达T淋巴细胞相关的基因[38,39], 推测AjIFN-γ也可能参与日本鳗鲡抗病毒的体液免疫和(或) T淋巴细胞介导的细胞免疫反应。
爱德华氏菌病是严重危害日本鳗鲡养殖的主要疾病之一, 流行范围广, 给日本、中国台湾和大陆的养鳗业造成重大经济损失[40]。有研究发现, 褐牙鲆(Paralichthys olivaceus)重组IFN-γ可激活宿主的抗迟缓爱德华氏菌感染的免疫反应[41]。在本研究中, AjIFN-γ在鳃、头肾和体肾中的转录表达量均在迟缓爱德华氏菌感染16h开始显著上调, 24h表达水平达到最高, 而脾脏中在72h也显著上调。该结果表明, AjIFN-γ可参与日本鳗鲡抵抗迟缓爱德华氏菌感染的免疫反应。
3.3 AjIFN-γ启动子活性分析
哺乳动物IFN-γ的转录表达受一系列转录因子的调控, 尤其是NFAT、AP-1和NF-κB家族的转录因子[42—48]。AjIFN-γ的启动子区存在AP-1、NF-κB、GATA-1、SRF、IRF-1、C/EBPβ等TFBS。对删节突变体的启动子活性的分析结果显示, P6(–240/+ 136)的相对活性显著高于pGL3-basic, 表明上游–240/+136区域中含有AjIFN-γ启动子的关键转录调控元件, 可起始AjIFN-γ的转录, 该区域内含有我们所预测的TSS和TATA-box, 说明该TSS可能是AjIFN-γ转录起始的位置。此外, 研究结果还显示, P3(–1062/+ 136)相对活性最高, 且显著高于P2(–1252/+136)和P4(–814/+136), 说明在–1062/–814区域中可能存在提高AjIFN-γ转录活性的TFBS, 而在–1252/–1062区域可能含有抑制AjIFN-γ转录活性的TFBS。进一步分析发现–1062/–814区域中含有2个AP-1和1个SRF,–1252/–1062区域中包括AP-1、GATA-1和IRF-1,然而, 究竟是哪个TFBS正向调控或负向调控AjIFN-γ的转录表达, 有待进一步研究。值得一提的是, Lai等[49]研究发现, 在草鱼肾脏细胞中过表达IRF-1可以提高Ⅰ型IFN的启动子活性, 而有关IRF-1对Ⅱ型IFN启动子活性的调控作用, 未见相关报道。
综上所述, 本研究克隆获得了日本鳗鲡IFN-γ基因, 命名为AjIFN-γ; 研究了AjIFN-γ在日本鳗鲡中的表达情况, 发现其在肝脏、皮肤和脾脏中的转录表达水平较高; 研究其对不同免疫刺激的响应, Poly I:C刺激和迟缓爱德华氏菌感染都能诱导AjIFN-γ的转录表达; 克隆并分析AjIFN-γ的上游调控区序列, 通过构建不同长度启动子区删节突变体并分析其启动子活性, 发现其启动子区–1062/–814区域可能存在AjIFN-γ的转录正调控元件。后续我们将通过体外重组表达获得AjIFN-γ的重组蛋白, 对其免疫功能进行深入研究, 并继续细化分析AjIFN-γ的启动子区–1062/–814区域, 进一步研究其主要转录调控机制。
[1]Schroder K, Hertzog P J, Ravasi T, et al. Interferongamma: an overview of signals, mechanisms and functions [J]. Journal of Leukocyte Biology, 2004, 75(2): 163—189
[2]Frucht D M, Fukao T, Bogdan C, et al. IFN-gamma production by antigen-presenting cells: mechanisms emerge [J]. Trends in Immunology, 2001, 22(10): 556—560
[3]Shtrichman R, Samuel C E. The role of gamma interferon in antimicrobial immunity [J]. Current Opinion in Microbiology, 2001, 4(3): 251—259
[4]Arts J A, Tijhaar E J, Chadzinska M, et al. Functional analysis of carp interferon-gamma: evolutionary conservation of classical phagocyte activation [J]. Fish & Shellfish Immunology, 2010, 29(5): 793—802
[5]Zou J, Yoshiura Y, Dijkstra J M, et al. Identification of an interferon gamma homologue in Fugu, Takifugu rubripes [J]. Fish & Shellfish Immunology, 2004, 17(4): 403—409
[6]Igawa D, Sakai M, Savan R. An unexpected discovery of two interferon gamma-like genes along with interleukin (IL)-22 and -26 from teleost: IL-22 and -26 genes have been described for the first time outside mammals [J]. Molecular Immunology, 2006, 43(7): 999—1009
[7]Milev-Milovanovic I, Long S, Wilson M, et al. Identification and expression analysis of interferon gamma genes in channel catfish [J]. Immunogenetics, 2006, 58(1): 70—80
[8]Stolte E H, Savelkoul H F J, Wiegertjes G, et al. Differential expression of two interferon-gamma genes in common carp (Cyprinus carpio L.) [J]. Developmental & Comparative Immunology, 2008, 32(12): 1467—1481
[9]Grayfer L, Belosevic M. Molecular characterization, expression and functional analysis of goldfish (Carassius aurutus L.) interferon gamma [J]. Developmental & Comparative Immunology, 2009, 33(2): 235—246
[10]Chen W Q, Xu Q Q, Chang M X, et al. Molecular characterization and expression analysis of the IFN-gamma related gene (IFN-gamma rel) in grass carp Ctenopharyngodon idella [J]. Veterinary Immunology and Immunopathology, 2010, 134(3—4): 199—207
[11]Yang K, Zhang S, Chen D, et al. IFN-gamma-activated lymphocytes boost nitric oxide production in grass carp monocytes/macrophages [J]. Fish & Shellfish Immunology, 2013, 35(5): 1635—1641
[12]Shibasaki Y, Yabu T, Araki K, et al. Peculiar monomeric interferon gammas, IFNgammarel 1 and IFNgammarel 2, in ginbuna crucian carp [J]. The FEBS Journal, 2014, 281(4): 1046—1056
[13]Zou J, Carrington A, Collet B, et al. Identification and bioactivities of IFN-γ in rainbow trout Oncorhynchus mykiss: the first Th1-type cytokine characterized functionally in fish [J]. The Journal of Immunology, 2005, 175(4): 2484—2494
[14]Furnes C, Seppola M, Robertsen B. Molecular characterisation and expression analysis of interferon gamma in Atlantic cod (Gadus morhua) [J]. Fish & Shellfish Immunology, 2009, 26(2): 285—292
[15]Chen R N, Su Y Q, Wang J, et al. Molecular characterization and expression analysis of interferon-gamma in the large yellow croaker Larimichthys crocea [J]. Fish & Shellfish Immunology, 2015, 46(2): 596—602
[16]Sunarto A, Mccoll K A. Expression of immune-related genes of common carp during cyprinid herpesvirus 3 infection [J]. Diseases of Aquatic Organisms, 2015, 113(2): 127—135
[17]Robertsen B, Zou J, Secombes C, et al. Molecular and expression analysis of an interferon-gamma-inducible guanylate-binding protein from rainbow trout (Oncorhynchus mykiss) [J]. Developmental & Comparative Immunology, 2006, 30(11): 1023—1033
[18]Sun B, Skjaeveland I, Svingerud T, et al. Antiviral activity of salmonid gamma interferon against infectious pancreatic necrosis virus and salmonid alphavirus and its dependency on type I interferon [J]. Journal of Virology, 2011, 85(17): 9188—9198
[19]Peng W, Lu D Q, Li G F, et al. Two distinct interferongamma genes in Tetraodon nigroviridis: Functional analysis during Vibrio parahaemolyticus infection [J]. Molecular Immunology, 2016, 70: 34—46
[20]Robertsen B, Bergan V T, Larsen R, et al. Atlantic salmon interferon genes: cloning, sequence analysis, expression, and biological activity [J]. Journal of Interferon & Cytokine Research, 2003, 23(10): 601—612
[21]Grayfer L, Garcia E G, Belosevic M. Comparison of macrophage antimicrobial responses induced by type II interferons of the goldfish (Carassius auratus L. ) [J]. Journalof Biological Chemistry, 2010, 285(31): 23537—23547
[22]Guo S L. The research situation of eel bacterial disease [J]. Scientific Fish Aquaculture, 2011, 263(7): 48—49 [郭松林. 鳗鲡细菌性疾病的研究概况. 科学养鱼, 2011, 263(7): 48—49]
[23]Duan M Z, Huang B, Liang Y, et al. Molecular cloning and expression analysis of a liver expressed antimicrobial peptide-2 in Japanese eel, Anguilla japonica [J]. Acta Hydrobiologica Sinica, 2016, 30(2): 252—260 [段明珠, 黄贝, 梁英, 等. 日本鳗鲡肝脏表达抗菌肽2基因的克隆与表达. 水生生物学报, 2016, 30(2): 252—260]
[24]Wang T, Gao Q, Nie P, et al. Identification of suppressor of cytokine signalling (SOCS) 6, 7, 9 and CISH in rainbow trout Oncorhynchus mykiss and analysis of their expression in relation to other known trout SOCS [J]. Fish & Shellfish Immunology, 2010, 29(4): 656—667
[25]Qi Z T, Nie P. Comparative study and expression analysis of the interferon gamma gene locus cytokines in Xenopus tropicalis [J]. Immunogenetics, 2008, 60(11): 699—710
[26]Shi J T, Li Z, Gui J F, et al. The cloning and expression analysis of zebrafish fem-1c, a member of fem-1 family [J]. Acta Hydrobiologica Sinica, 2015, 39(3): 459—467 [侍建涛, 李志, 桂建芳, 等. 斑马鱼fem-1c cDNA克隆与表达分析. 水生生物学报, 2015, 39(3): 459—467]
[27]Zou J, Clark M S, Secombes C J. Characterisation, expression and promoter analysis of an interleukin 10 homologue in the puffer fish, Fugu rubripes [J]. Immunogenetics, 2003, 55(5): 325—335
[28]Yoshiura Y, Kiryu I, Fujiwara A, et al. Identification and characterization of Fugu orthologues of mammalian interleukin-12 subunits [J]. Immunogenetics, 2003, 55(5): 296—306
[29]Bird S, Zou J, Kono T, et al. Characterisation and expression analysis of interleukin 2(IL-2) and IL-21 homologues in the Japanese pufferfish, Fugu rubripes, following their discovery by synteny [J]. Immunogenetics, 2004, 56(12): 909—923
[30]Schoenborn J R, Wilson C B. Regulation of interferon-γ during innate and adaptive immune responses [J]. Advances in Immunology, 2007, 96: 41—101
[31]Zou J, Secombes C J. Teleost fish interferons and their role in immunity [J]. Developmental & Comparative Immunology, 2011, 35(12): 1376—1387
[32]Parhi J, Sahoo L, Choudhury J, et al. Molecular characterization and expression analysis of interferon γ (IFN-γ) gene in Labeo rohita (Ham. ) [J]. Aquaculture Reports, 2015, 2: 97—105
[33]Zou J, Secombes C J. Teleost fish interferons and their role in immunity [J]. Developmental & Comparative Immunology, 2011, 35(12): 1376—1387
[34]Zhang Y A, Sun B J, Nie P. Immune tissues and cells of fish: a review [J]. Acta Hydrobiologica Sinica, 2000, 24(6): 648—654 [张永安, 孙宝剑, 聂品. 鱼类免疫组织和细胞的研究概况. 水生生物学报, 2000, 24(6): 648—654]
[35]Davidson G A, Lin SH, Secombes C J, et al. Detection of specific and ‘constitutive’ antibody secreting cells in the gills, head kidney and peripheral blood leucocytes of dab (Limanda limanda) [J]. Veterinary Immunology and Immunopathology, 1997, 58(3—4): 363—374
[36]Salinas I, Zhang YA, Sunyer J O. Mucosal immunoglobulins and B cells of teleost fish [J]. Developmental & Comparative Immunology, 2011, 35(12): 1346—1365
[37]Scapigliati G, Romano N, Abelli L. Monoclonal antibodies in fish immunology: identification, ontogeny and activity of T- and B-lymphocytes [J]. Aquaculture, 1999, 172(1—2): 3—28
[38]Ortiz N N, Gerdol M, Stocchi V, et al. T cell transcripts and T cell activities in the gills of the teleost fish sea bass (Dicentrarchus labrax) [J]. Developmental & Comparative Immunology, 2014, 47(2): 309—318
[39]Boschi I, Randelli E, Buonocore F, et al. Transcription of T cell-related genes in teleost fish, and the European sea bass (Dicentrarchus labrax) as a model [J]. Fish & Shellfish Immunology, 2011, 31(5): 655—662
[40]Chen C Z. Edwardsiella and edwardsiellasis of fish [J]. Journal of Hebei Normal University of Science & Technology, 2004, 18(3): 70—76 [陈翠珍. 爱德华氏菌及鱼类爱德华氏菌病. 河北科技师范学院学报, 2004, 18(3): 70—76]
[41]Jung C Y, Hikima J, Ohtani M, et al. Recombinant interferon-gamma activates immune responses against Edwardsiella tarda infection in the olive flounder, Paralichthys olivaceus [J]. Fish & Shellfish Immunology, 2012, 33(2): 197—203
[42]Penix L A, Weaver W M, Pang Y, et al. Two essential regulatory elements in the human interferon gamma promoter confer activation specific expression in T cells [J]. The Journal of Experimental Medicine, 1993, 178(5): 1483—1496
[43]Penix L A, Sweetser M T, Weaver W M, et al. The proximal regulatory element of the interferon-γ promoter mediates selective expression in T cells [J]. Journal of Biological Chemistry, 1996, 271(50): 31964—31972
[44]Aune T M, Penix L A, Rincon M R, et al. Differential transcription directed by discrete gamma interferon promoter elements in naive and memory (effector) [J]. Molecular and Cellular Biology, 1997, 17(1): 199—208
[45]Sweetser M T, Hoey T, Sun A L, et al. The roles of nuclear factor of activated T cells and ying-yang 1 in activation-induced expression of the interferon-γ promoter in T cells [J]. The Journal of Biological Chemistry, 1998, 273(52): 34775—34783
[46]Kiani A, García-Cózar F J, Habermann I, et al. Regulation of interferon-γ gene expression by nuclear factor ofactivated T cells [J]. Blood, 2001, 98(5): 1480—1488
[47]Tato C M, Villarino A, Caamaño J H, et al. Inhibition of NF-κB activity in T and NK cells results in defective effector cell expansion and production of IFN-γ required for resistance to Toxoplasma gondii [J]. The Journal of Immunology, 2003, 170(6): 3139—3146
[48]Young H A, Bream J H. IFN-γ recent advances in understanding regulation of expression, biological functions, and clinical applications [J]. Microbial Cell Factories, 2007, 316: 97—117
[49]Lai Q, Lin G, Ma M, et al. IRF-1 acts as a positive regulator in the transcription of grass carp (Ctenopharyngodon idella) IFN gene [J]. Fish & Shellfish Immunology, 2013, 34(6): 1432—1438
CHARACTERIZATION, EXPRESSION PATTERN AND PROMOTER ACTIVITY ANALYSIS OF INTERFERON-GAMMA IN JAPANESE EEL, ANGUILLA JAPONICA
PENG Xi-Xia1, HUANG Bei1, DUAN Li-Peng1, LI Chun-Yan1, LIANG Ying1, NIE Pin1,2and HUANG Wen-Shu1,3,4
(1. College of Fisheries, Jimei University, Xiamen 361021, China; 2. State Key Laboratory of Freshwater Ecology and Biotechnology, Institute of Hydrobiology, Chinese Academy of Sciences, Wuhan 430072, China; 3. Engineering Research Center of the Modern Technology for EelIndustry, Ministry of Education PRC, Xiamen 361021, China; 4. Fujian Collaborative Innovation Center for Development and Utilization of Marine Biological Resources, Xiamen 361005, China)
IFN-γ is a cytokine that is critical for innate and adaptive immunity against viral, some bacterial and protozoal infections. In this study, an interferon-γ gene, named AjIFN-γ, was cloned and characterized from Japanese eel, Anguilla japonica. The AjIFN-γ shared some common features with its vertebrate homologues including a 4-exon/3-intron gene structure, a typical IFN-γ characteristic motif and a predicted nuclear localization site in the predicted protein. AjIFN-γ mRNA could be detected in all the tested tissues from healthy Japanese eel with the highest in liver, followed by skin and head kidney by real-time RT-PCR. A significantly increased expression of AjIFN-γ could be found in gill, head kidney, trunk kidney and (or) spleen post intraperitoneal injection with Poly I:C or Edwardsiella tarda, which indicated a role in defense of Japanese eel against both viruses and bacteria. Furthermore, luciferase reporter assay demonstrated that the sequence from –240 bp to +136 bp in the 5′ flanking region of AjIFN-γ gene was essential for initiating the transcription of AjIFN-γ, and the sequence form –1062 bp to –814 bp may contain some positive transcriptional regulatory elements while the sequence from –1252 bp to –1062 bp may contain negative transcriptional regulatory elements. This study provided the basis for further investigation of the expanding functions of IFN-γ molecules in immunity and other physiological processes in teleost and other animals.
IFN-γ; Anguilla japonica; Transcriptional expression; Promoter
Q344+.1
A
1000-3207(2017)03-0589-14
10.7541/2017.76
2016-05-18;
2016-08-21
国家自然科学基金(31402329、31174238和U1205123); 福建省自然科学基金(2014J05042、2012J06008和JK2014026); 鳗鲡工程中心科研基金资助 [Supported by the National Natural Science Foundation of China (31402329, 31174238 and U1205123); the Natural Science Foundation of Fujian Province (2014J05042, 2012J06008 and JK2014026); a Fund from Engineering Research Center of the Modern Technology for Eel Industry]
彭喜霞(1990—), 女, 江西吉安人; 硕士; 主要研究方向为鱼类免疫学。E-mail: pengxixia1990@163.com
聂品, E-mail: pinnie@ihb.ac.cn; 黄文树, E-mail: wshuang@jmu.edu.cn

