CRISPR/ Cas9基因编辑技术在病原微生物中的应用
安鼎杰,康元环,陈 龙,张海月,张冬星,田佳鑫,贾俊鹏,孙武文,单晓枫,钱爱东
CRISPR/ Cas9基因编辑技术在病原微生物中的应用
安鼎杰,康元环,陈 龙,张海月,张冬星,田佳鑫,贾俊鹏,孙武文,单晓枫,钱爱东
规律成簇间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats, CRISPR)广泛存在于古细菌及细菌中是细菌在长期进化过程中形成的一种获得性免疫系统。近年来以该系统为基础,经过人工改造形成的一种新型基因编辑技术—CRISPR/Cas9在基因工程领域的应用越发广泛;该技术与前两代编辑技术相比,具有结构简单、成本低廉、实用价值较高等优点。自2012年在基因研究领域成功应用后,已成为当前关注度较高的基因编辑工具;该技术在多种真核生物的基因修饰中已得到成功应用,但在病原微生物上却报道较少。本文将从CRISPR/Cas9系统的结构、作用机制及其在病原微生物基因功能研究中的应用等几个方面进行综述为其深入研究奠定基础。
基因编辑;CRISPR/Cas9;病原生物;脱靶效应
基因编辑技术是指对特定的DNA片段编辑,如:敲除、插入、干扰以及沉默等。最初的基因编辑技术是利用注射或电转的方法将外源DNA导入细胞中,通过发生同源重组将导入的DNA片段插入到细胞基因组中[1];但是较低的插入效率使得该项技术在应用上受到了一定程度的限制。随着锌指核酸酶(Zinc finger nuclease,ZFN)和类转录激活因子效应物核酸酶(Transcription activator-like effector nuclease,TALEN)技术的出现,不仅实现了对基因的精准编辑而且在效率上有显著的提高;然而这两项技术从一开始出现就被少数几家公司垄断,在后续的实验过程中这两项技术存在的弊端也逐渐显露,例如:系统构建复杂、准备周期长、成本较高、脱靶造成的基因突变不可控等;目前急需一种更为简单,高效且能满足复杂实验要求的新方法。
CRISR/Cas9基因编辑技术是继ZFN和TALEN技术之后出现的第3代基因编辑技术,该技术是古细菌和细菌中存在的II型CRISPR/Cas获得性免疫系统经人工改造而来[2]。CRISPR/Cas9系统由可以整合外源DNA片段的规律成簇间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPR)和发挥剪切作用的Cas9核酸内切酶构成;在一段特殊的RNA序列引导下对目的片段进行切割。由于该项技术较前两代基因编辑技术相比具有结构简单、易于操作、特异性较强等优点,目前已经成功在人类细胞、小鼠、斑马鱼、食蟹猴、果蝇、以及拟南芥等动植物上应用,对其相应基因进行修饰加工,从而得到理想的相关生物学模型;这也意味着该技术在真核生物相关的研究领域内发展日趋成熟;近年报道表明在原核生物基因功能研究方面也已经开始使用该技术。本文将主要从CRISPR/Cas9系统结构、作用机制和在病原生物基因功能研究中的应用进展等几个方面进行综述为其深入研究奠定基础。
1 CRISPR/Cas系统的介绍
1.1 CRISPR/Cas系统发现历程 CRISPR系统是细菌长期进化过程中抵御噬菌体入侵和外源质粒转移而形成的一种获得性免疫系统,该系统的发现历程最早可追溯到1987年,日本科学家Y.Ishino[3]等在K12大肠杆菌中首次发现CRISPR序列,但无法确定这段序列的功能,因此并未引起科研人员的足够重视;1995年西班牙科学家在对地中海极嗜盐菌(Haloferaxediaterranei)和沃尔卡尼极嗜盐菌(Haleferaxvolcanii)研究中再次发现类似结构[4]。随着科学技术的进步和对该序列研究程度的加深,2002年Jansed[5]等正式将发现的这种串联重复序列命名为CRISPR(Clustered regularly interspaced short palindromic repeats)序列,自此之后CRISPR系统逐渐进入人们的视野,但对其具体功能仍尚不明确;直到2005年3个研究小组发现CRISPR序列中的间隔序列与入侵细菌的噬菌体和质粒有较高的同源性,据此推测该序列可能参与到抵御噬菌体入侵和外源质粒转移的过程中[6]。2012年该技术开始应用于基因工程领域的研究中[7],并逐步在原核和真核生物基因编辑中获得成功,特别是最近几年,该技术发展更为迅猛并在动物建模,新药开发及疾病治疗等不同领域中逐步得到应用,并展现出良好的应用前景。
1.2 CRISPR/Cas系统的结构组成及类型 根据已收录的CRISPR序列信息,通过统计分析发现大约40%的细菌和90%的古细菌中均存在CRISPR系统,且至少存在一个CRISPR位点,但有些细菌则含有十几个或上百个[8]; CRISPR位点通常位于细菌的染色体上,有些则存在于细菌胞内的质粒上。CRISPR序列的主体是由发挥不同作用的Cas蛋白基因序列,前导序列以及CRISPR基因座构成。其中功能不同的Cas蛋白基因序列在适应性免疫的不同阶段转录、翻译不同的蛋白执行各自特有的功能;而位于CRISPR位点上游的前导序列末端存在启动子,可在适应性免疫的第二阶段发挥着至关重要的作用;CRISPR基因座由启动子区域、多段同向的重复序列和间隔序列构成的R-S结构组成;其中重复序列并非严格意义上的保守序列,甚至在同一个细菌内不同的CRISPR基因座中也存在差异,但这段序列的5′端和3′端尤为保守,分别为GTTT/g和GAAAC。重复序列还包含回文结构,转录出来的RNA能形成稳定且保守的二级结构,可能在与Cas蛋白结合形成相关核糖蛋白复合物的过程中发挥作用[9-12]。间隔序列不仅在数量上存在较大的差异,而且由于每次入侵细菌的噬菌体和质粒不同,被整合到R-S结构中的序列信息也各不相同,因此不存在相同或较为相近的间隔序列。
最初的研究结果显示只有附近存在Cas基因的CRISPR基因座才有活性,但后续的研究证明该观点并不十分准确,某些附近不存在Cas基因的CRISPR基因座也可被转录并与其他位点表达的Cas蛋白相互作用从而行使其自身功能[12-13];由此可见Cas蛋白是CRISPR/Cas系统功能的执行者,并在实现这一功能的过程中发挥着重要的作用。由于Cas蛋白多种多样,目前对其分类尚不统一,但其中一种分类方法依照Cas基因序列的保守程度不同,将其分为核心Cas基因、亚特异型Cas基因及重复序列功能未知蛋白(Repeat associated nysterious proteins,RAMP)[14];而Makarova[15]等参照保守性Cas蛋白间的进化关系和Cas基因操纵子的组成方式等,将CRISPR/Cas系统分为2个独立的子系统,第1个系统由核心Cas1蛋白和Cas2蛋白将外源基因整合到CRISPR序列中;第2个子系统负责crRNA的加工、外源基因的识别及降解。根据这种分类法将CRISPR/Cas系统分为3种不同类型的系统:Type I、Type II 和、Type III。
这3种类型的系统中存在一些共有蛋白基因,同时也含有各自特有的Cas蛋白基因,其功能的不同主要体现在对于crRNA的加工、外源基因的识别以及降解这一过程中。
I型CRISPR/Cas系统在细菌和古细菌中均有分布,是3类系统中亚型最多的系统,其编码的Cas蛋白种类繁多,其中由多个Cas蛋白形成的CRISPR相关病毒防御复合物Cascade(CRISPR associated complex for antiviral defense)不仅参与crRNA的加工,而且还可与之形成能指导Cas3蛋白降解外源基因的核糖核蛋白复合体[16]。目前该系统在大肠杆菌(Escherichiacoli)和绿脓假单胞菌(Pseudomonasaeruginosa)中研究较多[16-20],其特有蛋白为Cas3蛋白。
II型CRISPR/Cas系统仅存在于细菌内,为三类系统中结构最为简单的系统,特有蛋白为Cas9蛋白,这种蛋白是一种大分子多功能蛋白,该蛋白不仅可降解外源基因序列,而且还参与了crRNA的成熟过程[21]。根据系统所含有的操纵子类型不同,将II型系统分为两个亚型,一种是含有Csn2基因的TypeII-A亚型,另一种是含有Cas4基因的TypeII-B亚型。在Cas9蛋白降解外源基因序列过程中,不仅仅需要crRNA的参与,后续的研究表明除了crRNA之外,反式激活crRNA(transactivating crRNA,tracrRNA)及RNaseIII均参与了该过程协助Cas9蛋白降解外源基因[22]。
III型CRISPR/Cas系统大多存在于古细菌中,仅有少量的存在于细菌中。特有蛋白分别是Cas10和Cas6蛋白[23],但功能尚不十分明确,推测这两类蛋白均可能参与crRNA的加工,而Cas10蛋白可对靶序列进行降解。除了特有蛋白不同之外,该系统与前两类系统最大的区别在于对靶序列的识别机制,其通过crRNA5′末端与靶序列错配来区分外源基因[24]。目前该型系统存在两个亚型:TypeIII-A和TypeIII-B型,二者干扰的靶标并不相同,分别为mRNA和DNA。
1.3 CRISPR/Cas系统的作用机制 在漫长的进化过程中细菌为了抵御病毒入侵和外源性质粒的转移,已进化出多种防御机制,以CRISPR/Cas为核心的适应性免疫系统就是其中之一。其作用机制可分为三个阶段:第一阶段是新间区的获取,第二阶段是CRISPR基因座的表达,第三阶段是对入侵核酸的干扰。图1为:CRISPR/Cas系统抵御噬菌体入侵的机制示意图,具体机制如下。
1.3.1 第一阶段:新间区的获取 当外源性基因进入带有CRISPR系统的细菌时,该系统首先对外源基因进行扫描识别后,而后通过具有DNA内切酶活性Cas1蛋白将外源基因切割成17~48 bp大小的目的片段,剪切后的片段被具有核糖核酸内切酶活性Cas2蛋白识别并插入到CRISPR/Cas基因座中的前导序列与第一个重复序列之间,伴随着新间隔序列插入的同时,重复序列也进行复制,进而形成了新的R-S结构,将外源基因的序列信息存储在CRISPR系统中,为适应性免疫奠定了结构基础。与这段间隔序列具有较高同源性的噬菌体或质粒序列被称为原间隔(protospacer);而在原间隔5′或3′端有一段长度大约为2~5 bp左右的保守序列叫做原间隔相关基序(Protospacer associated motif,PAM)[25],该段序列在对外源基因序列剪切的过程中起到相应靶点的作用,不同的菌属间或同属不同菌株间PAM序列也不近相同,正因为PAM序列的存在,才避免了CRISPR系统对自身序列的切割,但只有I和II型系统通过PAM序列识别外源基因。
1.3.2 第二阶段:CRISPR基因座表达 正常情况下CRISPR基因座表达水平很低,当那些与间隔序列具有同源性的噬菌体或质粒再次入侵细菌时,CRISPR基因座表达量迅速上调[15],转录起始于前导序列末端的启动子,生成CRISPR前体RNA(CRISPR precursor RNA,pre-crRNA),但是其还不具备与Cas蛋白结合的能力,随后该RNA被进一步加工成含有一个间隔序列和部分重复序列的小段成熟crRNA,并与特异的Cas蛋白结合形成相关核糖核蛋白复合物,在干扰阶段发挥作用。
1.3.3 第三阶段:对入侵核酸的干扰 干扰是CRISPR/Cas系统抵御外源基因入侵中关键性的步骤,crRNA与外源DNA通过碱基配对结合后,核糖核蛋白复合物通过识别PAM序列来定位切割的目的片段,在核酸酶活性的作用下对外源基因进行降解;早期的研究认为crRNA与原间隔序列匹配程度决定了能否对外源基因成功进行干扰,但后期的研究表明这仅仅是干扰过程中的先决条件[26-27],PAM序列的存在才是能否成功实施干扰的关键,倘若不存在PAM序列或其发生突变,即使crRNA与原间隔序列完美匹配,CRISPR/Cas系统也不能够进行正常的切割活动[21,28-29]。
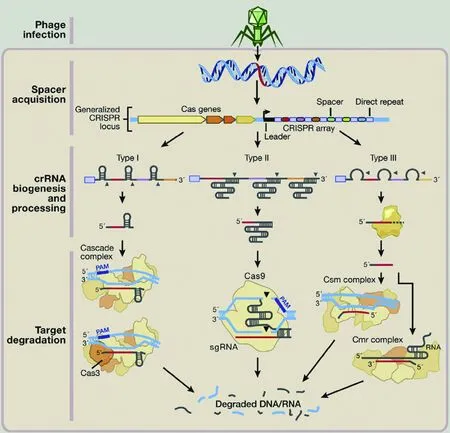
图1 CRISPR/Cas抵御噬菌体侵染的过程[30]Fig.1 Resistance to phage infection by CRISPR/Cas
2 CRISPR/Cas9在病原微生物中的应用
目前CRISPR/Cas9技术已成功应用于人类细胞、小鼠、斑马鱼、食蟹猴、非洲爪蟾、果蝇、烟草、水稻及拟南芥等真核生物的基因编辑中,并取得一系列重要的科研成果且部分成功已被应用到实际生产中。近年来,该技术也逐步被用于对病原微生物的研究中,利用CRISPR/Cas9系统可对病原微生物的基因进行敲除,并通过相关实验推测基因功能,而且该系统特异性较高,在效率上已明显优于传统方法。除了在敲除方面的应用外,该系统经过改造还可以对基因进行插入,沉默和定向调控,虽然目前该系统在病原微生物研究中的应用相对较少,但也已取得了一定的研究成果。
2.1 CRISPR/Cas9系统在寄生虫上的应用 在对寄生虫基因功能的研究中,明确相应基因的具体功能至关重要,CRISPR/Cas9技术除了较高的编辑效率之外,可对所要研究的基因进行精准的敲除,从而实现对具体功能的研究。Shen等[31]最先利用CRISPR/Cas9敲除了弓形虫的rop18基因并获得缺失型GT-1I型虫株,结果表明与RHI型虫株相比GT-1I型虫株毒力有所降低,并且验证了该方法在敲除效率上明显高于同源重组法。Zheng等[32]利用CRISPR/Cas9敲除亮氨酸基肽酶基因后的缺失型虫株转染非洲绿猴肾细胞,揭示了亮氨酸基肽酶在弓形虫入侵和附着中的作用,并证明了亮氨酸基肽酶对虫体生长有一定影响。
袁晶[33]利用了CRISPR/Cas9对疟原虫Pyser1,Py03652和Pyhsp70基因分别进行敲除,插入和等位基因的置换,其效率分别达到了100%、20%和45%。
Noelia Lander[34]利用该技术对克氏锥虫虫体表面糖蛋白GP72基因和鞭毛棒状蛋白PFR1和PFR2基因进行了研究,揭示了这3个基因在鞭毛吸附细胞和虫体运动中所发挥的作用,并发现对克氏锥虫内源性基因进行编辑不产生明显毒性作用。
2.2 CRISPR/Cas9系统在细菌上的应用 由于细菌之间差异较大,相较于其他领域的研究,CRISPR/Cas9技术在细菌上的应用才刚刚起步,但已被应用于对肺炎链球菌和大肠杆菌的基因编辑中,并取得了较高的编辑效率,同时其在食品生产中也展现了巨大的应用潜力。Jiang等[35]对肺炎链球菌和大肠杆菌中多个基因进行了编辑,突变效率较高在肺炎链球菌中达到100%,在大肠杆菌中达到65%。Michall E.Pyne等[36]将CRISPR/Cas9系统和Red重组系统融合后对大肠杆菌中的单链寡聚核苷酸基因进行重组,同时对染色体上多基因进行置换。研究结果再次验证了CRISPR/Cas9技术在基因编辑上的高效性,同时为其功能的拓展提供了新的方向。此外,在食品发酵生产的过程中,该技术可被用来提高发酵菌株抵抗噬菌体侵染的能力[37]。鉴于该技术所具有的优势,本实验室拟采用CRIPR/Cas9技术对维氏气单胞菌(Averomonasveronii)相关基因进行研究。
2.3 CRISPR/Cas9系统在病毒上的应用 在艾滋病的治疗过程中除了抑制病毒基因组的表达外,怎样能够让免疫系统识别并清除处于转录抑制状态的整合病毒基因组成为治疗的难点所在,CRISPR/Cas9技术的出现为艾滋病的治疗带来一丝曙光。长末端重复序列(Long terminal repeats,LTR)参与HIV病毒RNA的复制和翻译,Hirotaka Ebina等[38]针对该序列设计相应的sgRNA,与hCas9共转染整合有HIV-1的Jurkat细胞,经过3轮转染,流式分析GFP阳性细胞比例由 97.8%下降到35.5%,成功限制了原病毒的激活和表达。Hu等[39]构建了靶向HIV-1 LTR U3 启动子的gRNA-Cas9 质粒,并转染整合有单轮次感染的HIV基因的小胶质细胞和T4细胞,也可阻断病毒基因组的复制和表达,同时发现Cas9/gRNA在宿主细胞内不产生细胞毒性也不造成脱靶效应。Zhu等[40]设计10个针对HIV-1病毒的基因的CRISPR/Cas9靶点,并对已感染病毒的Jarkat细胞进行基因插入,其中第2个外显子位点的突变可使HIV-1的基因表达量和病毒产物较插入前减小20倍。 Prajit Limsirichai[41]利用基于乙酰转移酶的CRISPR系统介导HIV-1的整合基因表达上调,结果显示该系统可高效诱导病毒表达,与抗逆转录病毒疗法结合可以抑制HIV-1感染。
在乙肝治疗方面仅仅抑制病毒复制和表达是不够的,在抑制其复制和表达的同时运用相应的技术清除侵入的病毒基因组才是有望治愈乙肝的有效途径。Lin等[42]将HBV体外表达质粒和靶向 HBV 基因组的 gRNA-Cas9 质粒共转染 Huh-7 细胞,检测发现 HBV 核心蛋白和表面蛋白的表达量明显降低,明显抑制了病毒基因的表达;将 HBV表达质粒和靶向 HBV 基因组的 gRNA-Cas9 质粒共注射到 HBV 肝炎耐受小鼠的尾静脉,检测发现小鼠血清表面抗原水平较对照组明显降低,进一步说明CRISPR/Cas9技术具有应用于临床治疗的潜质。Christoph Seegar等[43]转染含有NTPC受体的Hep G2 细胞,测试了不同的gRNA,证明了Cas9蛋白可对cDNA造成突变或敲除相应片段,同时干扰素不会对CRISPR/Cas9的抗病毒活性产生明显影响。Wang等[44]针对HBV A-D的4个基因型设计了15种gRNA分别靶向HBV中不同的调控区域,其中11套系统为双股RNA。结果显示所用gRNAs均可在相应的调控区内抑制病毒基因组的表达,双股RNA系统通过剪切2个RNAs之间的片段来破坏病毒的表达模板,在效率上明显优于单股RNA;而gRNA-5和gRNA-12组合使用不仅高效抑制了HBsAg和HBeAg的表达,同时也能够清除HepAD38细胞中的cccDNA存储库;此项研究为乙肝的治疗方面提供了相应的理论基础。
此外,CRISPR/Cas9技术还被用于构建相关病毒的弱毒株,Tiffant A.Russell[45]利用CRISPR/Cas9系统对单纯疱疹毒株的UL26和UL27基因之间进行基因插入,结果显示插入相应的目的片段可有效减弱病毒的毒力,构建的弱毒株可作为工程毒株用于后续研究。Xu等[46]基于CRISPR/Cas9技术建立了一种高效构建基因缺失伪狂犬病毒株的方法,并对伪狂犬病毒(Pseudorabies Virus,PRV)EP0、UL50基因进行单缺失的同时又对EP0/US3基因进行双缺失,从而揭示急性早幼粒细胞白血病蛋白(Promyelocytic leukemia,PML)抵抗PRV的作用机制,构建的基因缺失型PRV为后期的疫苗研发奠定了基础。
3 CRISPR/Cas9的优缺点
与前两代技术相比CRISPR/Cas9打靶位点分布广泛,不仅可对单个基因进行修饰,而且通过设计不同引物还可一次性对多个基因进行修饰,进而减少了后期筛选的工作量,在提升效率的同时又缩短了实验周期;该技术的系统结构简单,易于构建;经合理的人工改造后,添加相应的功能组件可对特定的基因表达进行调控[47-48]。而与其他技术融合使用还可执行不同的功能,极大地拓展了该技术的使用范围,为其他领域的研究提供一条全新的途径。
当然,CRISPR/Cas9技术也存在一定的不足:在基因编辑过程中造成的脱靶效应会扰乱基因组中其他基因的稳定性,其次由脱靶效应引起的非正常突变不可控,会带来意想不到的结果。除脱靶效应外,Cas9蛋白作为一种菌体蛋白,应用于真核生物基因编辑中是否会产生相应的细胞毒性或者带有一定的免疫原性还尚无定论,有待进一步的研究取证。
4 展 望
随着科学技术的发展,人们对于基因的了解程度逐渐加深,但受到科学技术的制约,对大部分基因的功能还没有做到真正意义上的了解;CRISPR/Cas9技术因其具有的独特魅力,自出现开始便在基因编辑领域受到广泛的关注,该系统已被广泛应用于对基因功能的研究中并取得了丰硕的科研成果。伴随着该系统使用范围的拓展,其在遗传育种,药物开发,临床医疗上的潜力被逐步发掘;虽然CRISPR/Cas9系统还存在一些不足之处,相信通过不断的改进和完善,该系统能在不同层次的研究上得到更广阔的发展和应用。
[1] Capecchi MR. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century[J]. Nat Rev Genetics, 2005, 6(6): 507-512.
[2] Liu ZG. Research progress on CRISPR/Cas9 mediated genome editing[J]. Acta Vet Et Zootech Sci, 2014, 45(10): 1567-1583.
刘志国. CRISPR/Cas9系统介导基因组编辑的研究进展[J].畜牧兽医学报, 2014, 45(10): 1567-1583.
[3] Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion inEscherichiacoli, and identification of the gene product[J]. J Bacteriol, 1987, 169(12): 5429-5433.
[4] Mojica FJM, Ferrer C, Juez G, et al. Long stretches of short tandem repeats are present in the largest replicons of the ArchaeaHaloferaxmediterraneiandHaloferaxvolcaniiand could be involved in replicon partitioning[J]. Mol Microbiol, 1995, 17(1): 85-93.
[5] Jansen R, Embden J, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565-1575.
[6] Bolotin A, Quinquis B, Sorokin A, et al. Clustered regularly interspaced short palindrome repeats (CRISPRs) have spacers of extrachromosomal origin[J]. Microbiology, 2005, 151(8): 2551-2561.
[7] Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity[J]. Science, 2012, 337(6096): 816-821.
[8] Rousseau C, Gonnet M, Le Romancer M, et al. CRISPI: a CRISPR interactive database[J]. Bioinformatics, 2009, 25(24): 3317-3318.
[9] Jansen R, Embden J, Gaastra W, et al. Identification of genes that are associated with DNA repeats in prokaryotes[J]. Mol Microbiol, 2002, 43(6): 1565-1575.
[10] Deveau H, Garneau JE, Moineau S. CRISPR/Cas system and its role in phage-bacteria interactions[J]. Ann Rev Microbiol, 2010, 64: 475-493.
[11] Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J]. Genome Biol, 2007, 8(4): 1.
[12] Godde JS, Bickerton A. The repetitive DNA elements called CRISPRs and their associated genes: evidence of horizontal transfer among prokaryotes[J]. J Mol Evolut, 2006, 62(6): 718-729.
[13] Horvath P, Coúté-Monvoisin AC, Romero DA, et al. Comparative analysis of CRISPR loci in lactic acid bacteria genomes[J]. Intl J Food Microbiol, 2009, 131(1): 62-70.
[14] Haft DH, Selengut J, Mongodin EF, et al. A guild of 45 CRISPR-associated (Cas) protein families and multiple CRISPR/Cas subtypes exist in prokaryotic genomes[J]. PLoS Comput Biol, 2005, 1(6): e60.
[15] Makarova KS, Haft DH, Barrangou R, et al. Evolution and classification of the CRISPR-Cas systems[J]. Nat Rev Microbiol, 2011, 9(6): 467-477.
[16] Wiedenheft B, van Duijn E, Bultema JB, et al. RNA-guided complex from a bacterial immune system enhances target recognition through seed sequence interactions[J]. Proc Nat Acad Sci, 2011, 108(25): 10092-10097.
[17] Wiedenheft B, Zhou K, Jinek M, et al. Structural basis for DNase activity of a conserved protein implicated in CRISPR-mediated genome defense[J]. Structure, 2009, 17(6): 904-912.
[18] Haurwitz RE, Jinek M, Wiedenheft B, et al. Sequence-and structure-specific RNA processing by a CRISPR endonuclease[J]. Science, 2010, 329(5997): 1355-1358.
[19] Jore MM, Lundgren M, van Duijn E, et al. Structural basis for CRISPR RNA-guided DNA recognition by Cascade[J]. Nat Structural Mol Biol, 2011, 18(5): 529-536.
[20] Brouns SJJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science, 2008, 321(5891): 960-964.
[21] Garneau JE, Dupuis ME, Villion M, et al. The CRISPR/Cas bacterial immune system cleaves bacteriophage and plasmid DNA[J]. Nature, 2010, 468(7320): 67-71.
[22] Deltcheva E, Chylinski K, Sharma CM, et al. CRISPR RNA maturation by trans-encoded small RNA and host factor RNase III[J]. Nature, 2011, 471(7340): 602-607.
[23] Anantharaman V, Iyer LM, Aravind L. Presence of a classical RRM-fold palm domain in Thg1-type 3′-5′nucleic acid polymerases and the origin of the GGDEF and CRISPR polymerase domains[J]. Biol Direct, 2010, 5(1): 1.
[24] Mojica FJM, García-Martínez J, Soria E. Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements[J]. J Mol Evolut, 2005, 60(2): 174-182.
[25] Mojica FJM, Diez-Villasenor C, Garcia-Martinez J, et al. Short motif sequences determine the targets of the prokaryotic CRISPR defence system[J]. Microbiology, 2009, 155(3): 733-740.
[26] Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709-1712.
[27] Deveau H, Barrangou R, Garneau JE, et al. Phage response to CRISPR-encoded resistance inStreptococcusthermophilus[J]. J Bacteriol, 2008, 190(4): 1390-1400.
[28] Terns MP, Terns RM. CRISPR-based adaptive immune systems[J]. Curr Opinion Microbiol, 2011, 14(3): 321-327.
[29] Sapranauskas R, Gasiunas G, Fremaux C, et al. TheStreptococcusthermophilusCRISPR/Cas system provides immunity inEscherichiacoli[J]. Nucl Acids Res, 2011: gkr606.
[30] Hsu PD, Lander ES, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering[J]. Cell, 2014, 157(6): 1262-1278.
[31] Shen B, Brown KM, Lee TD, et al. Efficient gene disruption in diverse strains ofToxoplasmagondiiusing CRISPR/CAS9[J]. MBio, 2014, 5(3): e01114-14.
[32] Zheng J, Jia H, Zheng Y. Knockout of leucine aminopeptidase inToxoplasmagondiiusing CRISPR/Cas9[J]. Intl J Parasitol, 2015, 45(2): 141-148.
[33] Zhang C, Xiao B, Jiang Y, et al. Efficient editing of malaria parasite genome using the CRISPR/Cas9 system[J]. MBio, 2014, 5(4): e01414-14.
[34] Lander N, Li ZH, Niyogi S, et al. CRISPR/Cas9-induced disruption of paraflagellar rod protein 1 and 2 genes inTrypanosomacruzireveals their role in flagellar attachment [J]. MBio, 2015, 6(4): e01012-15.
[35] Jiang W, Bikard D, Cox D, et al. RNA-guided editing of bacterial genomes using CRISPR-Cas systems[J]. Nat Biotechnol, 2013, 31(3): 233-239.
[36] Pyne ME, Moo-Young M, Chung DA, et al. Coupling the CRISPR/Cas9 system with lambda red recombineering enables simplified chromosomal gene replacement inEscherichiacoli[J]. Appl Environmental Microbiol, 2015, 81(15): 5103-5114.
[37] Barrangou R, Horvath P. CRISPR: new horizons in phage resistance and strain identification[J]. Ann Rev Food Sci Technol, 2012, 3: 143-162.
[38] Ebina H, Misawa N, Kanemura Y, et al. Harnessing the CRISPR/Cas9 system to disrupt latent HIV-1 provirus[J]. Scientif Reports, 2013, 3.
[39] Hu W, Kaminski R, Yang F, et al. RNA-directed gene editing specifically eradicates latent and prevents new HIV-1 infection[J]. Proc Natl Acad Sci, 2014, 111(31): 11461-11466.
[40] Zhu W, Lei R, Le Duff Y, et al. The CRISPR/Cas9 system inactivates latent HIV-1 proviral DNA[J]. Retrovirology, 2015, 12(1): 1.
[41] Limsirichai P, Gaj T, Schaffer DV. CRISPR-mediated activation of latent HIV-1 expression[J]. Mol Ther, 2015.
[42] Lin SR, Yang H C, Kuo YT, et al. The CRISPR/Cas9 system facilitates clearance of the intrahepatic HBV templates in vivo[J]. Mol Ther Nucl Acids, 2014, 3(8): e186.
[43] Seeger C, Sohn JA. Targeting hepatitis B virus with CRISPR/Cas9[J]. Mol Ther Nucl Acids, 2014, 3(12): e216.
[44] Wang J, Xu ZW, Liu S, et al. Dual gRNAs guided CRISPR/Cas9 system inhibits hepatitis B virus replication[J]. World J Gastroenterol, 2015, 21(32): 9554.
[45] Russell TA, Stefanovic T, Tscharke DC. Engineering herpes simplex viruses by infection-transfection methods including recombination site targeting by CRISPR/Cas9 nucleases[J]. J Virologic Methods, 2015, 213: 18-25.
[46] Xu A, Qin C, Lang Y, et al. A simple and rapid approach to manipulate pseudorabies virus genome by CRISPR/Cas9 system[J]. Biotechnol Lett, 2015, 37(6): 1265-1272.
[47] Miller JC, Tan S, Qiao G, et al. A TALE nuclease architecture for efficient genome editing[J]. Nat Biotechnol, 2011, 29(2): 143-148.
[48] Zhang F, Cong L, Lodato S, et al. Efficient construction of sequence-specific TAL effectors for modulating mammalian transcription[J]. Nat Biotechnol, 2011, 29(2): 149-153.
Advance and application of CRISPR/ Cas9 mediated genome editing technique on pathogenic microorganism
AN Ding-jie, KANG Yuan-huan, CHEN Long, ZHANG Hai-yue, ZHANG Dong-xing, TIAN Jia-xin, JIA Jun-peng, SUN Wu-wen, SHAN Xiao-feng, QIAN Ai-dong
(CollegeofAnimalScienceandTechnology,JilinAgriculturalUniversity,Changchun130118,China)
Clustered regularly interspaced short palindromic repeats (CRISPR) is an acquired immune system existing in archaea and bacteria with the long-term process of evolutionary. CRISPR/Cas9 gene editing system is a new type of gene editing technology developed based on the system. CRISPR/Cas9 is a more efficient method for gene targeting than the previous methods. It has been successfully applied for gene-modified of eukaryotes since 2012, but the reports about pathogenic microorgaisms are rarely. Here, the research progress in the structure, mechanism of CRISPR/Cas9 system and its applications on pathogenic microorgaisms is reviewed.
geneome editing; CRISPR/Cas9; pathogenic microorgaisms; off-target effect Supported by the National Natural Science Foundation of China(No.31201927) and the Key Scientific and Technological Project of Jilin Provincial Science & Technology Department (No. 20150204065NY).
s: Shan Xiao-feng, Email: sxf1997@163.com
10.3969/j.issn.1002-2694.2017.03.017
国家自然科学基金项目(No.31201927)和吉林省重点科技攻关项目(No.20150204065NY)联合资助
单晓枫,Email: sxf1997@163.com
吉林农业大学动物科学技术学院,长春 130118
R372
A
1002-2694(2017)03-0280-07
2016-09-26 编辑:梁小洁
——一道江苏高考题的奥秘解读和拓展

