产黄曲霉毒素真菌多重PCR检测体系的建立
李 慧 蔡 军 胡梦龙 傅 洋 欧静堃
(中粮营养健康研究院;营养健康与食品安全北京市重点实验室,北京 102209)
产黄曲霉毒素真菌多重PCR检测体系的建立
李 慧 蔡 军 胡梦龙 傅 洋 欧静堃
(中粮营养健康研究院;营养健康与食品安全北京市重点实验室,北京 102209)
依据标准SN/T 2582—2010《产黄曲霉毒素真菌PCR检测方法》推荐的产黄曲霉毒素调节基因aflR、柄曲霉素转甲氧基酶基因omt-1、杂色曲霉素A脱氢酶基因ver-1及真菌共有的5.8S rDNA的ITS序列分别设计4对特异性引物,对4对引物进行多重PCR反应体系构建与优化,建立快速检测产黄曲霉毒素真菌的多重PCR方法,并应用10株试验菌株对所建立的检测方法进行验证。结果表明,建立的多重PCR方法具有灵敏度高、特异性强、方便快捷的优点,为产黄曲霉毒素真菌快速检测试剂盒的研发及应用提供了重要技术保障。
多重PCR 产黄曲霉毒素真菌 快速检测 特异性 灵敏度
目前全球每年约有25%的农产品遭受真菌毒素污染,造成高达数百亿美元的经济损失[1]。对农业、畜牧业危害较严重的真菌毒素主要有黄曲霉毒素(AFT)、赭曲霉毒素A(OTA)、玉米赤霉烯酮(ZEN)、脱氧雪腐镰刀菌烯醇(DON)、伏马毒素B(FB)和T-2毒素等[2-4]。其中,黄曲霉毒素对环境污染最严重、对人畜危害最大,能够广泛污染花生、玉米、大豆、小麦、坚果等农作物及其制品[5-6]。黄曲霉毒素来源于曲霉属、青霉属、毛霉属、镰孢霉属和根霉属等真菌[7],曲霉属的黄曲霉(A.flavus)和寄生曲霉(A.parasiticus)是2种主要的黄曲霉毒素产生菌[8-9]。因此,能否及时有效地预防与控制黄曲霉毒素危害,首要条件就是能够快速准确地检测判断粮食是否被黄曲霉(A.flavus)、寄生曲霉(A.parasiticus)等产黄曲霉毒素真菌污染[10-11]。
过去对产黄曲霉毒素真菌的检测通常采用形态学方法、免疫学方法等传统检测方法,主要基于真菌形态、生理特征、抗原特异性等特征进行分类鉴别,通常需要经过纯化培养、形态观察、生理生化鉴定等的过程,存在检测周期长、操作程序繁琐、专业知识要求高、鉴别水平不够准确等缺点,难以满足企业和政府监管部门对大批量农作物及其制品进行产黄曲霉毒素真菌快速、准确检测的需求[12]。随着遗传学、分子生物学等学科的发展,分子生物学鉴定方法,尤其是PCR技术在产黄曲霉毒素真菌的检测与鉴别方面得到了深入的研究与发展。如孙长坡等[13]应用PCR-RFLP技术成功鉴别了不同黄曲霉毒素产毒菌株,Mahmoud[14]应用实时PCR(RT-PCR)技术成功检测了储藏花生中污染的黄曲霉(A.flavus)菌株及其产黄曲霉毒素基因的表达水平,Ahmad等[8]应用PCR-RFLP技术成功检测并区分了黄曲霉毒素污染花生中的黄曲霉(A.flavus)、寄生曲霉(A.parasiticus)菌株,Midorikawa等[15]建立了曲霉属特异的PCR检测方法。此外,基于普通PCR技术发展起来的能够在同一反应体系中同时检测多个目标DNA的多重PCR技术,由于具备操作简便、成本低廉和快速高效等优点,也被广泛用于有害微生物检测[16-17]。然而,除秦文彦等[18]应用多重PCR技术对可污染食品及饲料的产黄曲霉毒素真菌进行检测外,国内关于应用多重PCR技术进行产黄曲霉毒素真菌快速检测的研究鲜见报道。
本研究针对标准SN/T 2582—2010《产黄曲霉毒素真菌PCR检测方法》[19]推荐的黄曲霉毒素生化合成途径中关键调控基因aflR、omt-1和ver-1及真菌共有的5.8S rDNA的ITS序列,通过生物信息学手段设计多重PCR检测引物,同时优化多重PCR反应体系及扩增程序,建立了更为灵敏特异、快捷高效的产黄曲霉毒素真菌的多重PCR检测体系,并成功应用于人工污染样品的检测。本研究为产黄曲霉毒素真菌的多重PCR检测奠定了良好研究基础,为其他类型的有害微生物的多重PCR检测提供了一定的技术借鉴,为农作物及其制品的科学储藏和质量安全控制提供了有力的检测技术手段。
1 材料与方法
1.1 材料和仪器
1.1.1 供试菌株
试验菌株部分购自中国普通微生物菌种保藏管理中心CGMCC菌种库,部分为本实验室分离鉴定后保藏。菌株目录如表1。

表1 供试菌株
1.1.2 试剂和仪器
马铃薯-葡萄糖琼脂培养基(PDA):北京陆桥技术有限责任公司;萨氏培养液(SDB):蛋白胨1.0%,葡萄糖4.0%及酵母粉1.0%,pH 6.0±0.2,蒸馏水配制,121℃、20 min灭菌备用;YPD固体培养基:酵母粉1.0%,蛋白胨2.0%,葡萄糖2.0%,蒸馏水配制,121℃、20 min灭菌备用。
dNTPs、Taq酶及DNA Marker等:Takara公司;真菌基因组DNA提取试剂盒:北京索莱宝科技有限公司。
MJ-150-Ⅱ霉菌培养箱、HZQ-X300C恒温振荡器及DK-8D三孔电热恒温水槽:上海一恒科学仪器有限公司;Veriti96孔快速热循环仪:美国ABI公司;PowerPac通用电泳仪及GelDoc XR System凝胶成像系统:美国Bio-rad公司;NanoDrop 2000超微量分光光度计:美国ThermoFisher公司。
1.2 方法
1.2.1 菌株培养与基因组DNA提取
各菌株接种于马铃薯-葡萄糖琼脂培养基(PDA)平板上,28℃活化培养3~5 d(酵母菌使用YPD琼脂培养基);挑取少量相应菌株菌丝体分别接种于萨氏培养液(SDB),28℃、150 r/min培养2~3 d(酵母菌使用YPD培养基);离心收集菌体,采用真菌基因组DNA提取试剂盒对所收集的菌体进行基因组DNA提取。
1.2.2 目标基因检测引物的设计
依据标准推荐的产黄曲霉毒素调节基因aflR[19]、柄曲霉素转甲氧基酶基因omt-1、杂色曲霉素A脱氢酶基因ver-1及真菌共有的5.8S rDNA的ITS序列,采用Primer 5.0软件设计相应特异性多重PCR引物,引物由英潍捷基(上海)贸易有限公司合成,引物序列如表2。

表2 产黄曲霉毒素真菌多重PCR检测所用目的基因及其引物序列
1.2.3 单重PCR检测特异性及检测灵敏度测定
以表1所列菌株基因组DNA为模板,分别PCR扩增ITS、aflR、ver-1和omt-1目的基因片段,测定各基因PCR扩增特异性,同时采用标准推荐方法进行对比验证[21]。经优化的反应体系为:5.0 μL 10×PCR buffer,1.0 μL 10 mmol/L dNTP,各引物0.4 μmol/L,Taq酶0.3 μL(5 U/μL),2 μL DNA模板(10 ng/μL),加ddH2O至50 μL。经优化的扩增程序为:94℃预变性2 min;94℃变性20 s,55℃退火30 s,72℃延伸40 s,35个循环;72℃延伸7 min。经凝胶电泳观察分析相应PCR扩增产物后,分别进行纯化测序,并与GenBank中相应序列进行比对。
以黄曲霉菌株CGMCC 3.4410基因组DNA为模板,按终浓度10 ng/μL~1 pg/μL进行10倍梯度稀释,采用上述经优化的反应体系及扩增程序分别进行PCR检测,经凝胶电泳观察分析相应单重PCR扩增的检测灵敏度。
2 结果与分析
2.1 单重PCR检测特异性分析
应用所设计的4对引物,分别对表1中11株菌株的ITS、aflR、ver-1和omt-1目的基因进行单重PCR特异性分析。采用本研究提供的4对引物进行单重PCR检测结果(图1b)与采用标准[21]推荐方法检测结果(图1a)相一致,5、6、8、10号菌株基因组DNA中均能扩增出ITS、aflR、ver-1和omt-1目的基因片段,表明这4株菌株可能都具有产黄曲霉毒素能力,同理论结果一致。此外,2号样品采用本研究提供方法扩增产物中未出现明显非特异性扩增。表明本研究所提供的单重PCR检测方法具有较高的检测特异性,能够满足产黄曲霉毒素真菌特异性检测的要求。
1.2.4 多重PCR检测特异性与检测灵敏度测定
在不改变扩增程序的前提下,对多重PCR反应体系主要因素进行优化,确定多重PCR反应体系:5.0 μL 10×PCR buffer,1.0 μL 10 mmol/L dNTP,各引物0.2μmol/L,Taq酶0.3μL(5 U/μL),2 μL DNA模板(10 ng/μL),加ddH2O至50 μL。经凝胶电泳观察分析相应PCR扩增产物后,分别回收纯化相应扩增DNA片段进行测序比对验证。
以黄曲霉菌株CGMCC 3.4410基因组DNA为模板,按终浓度10 ng/μL~1 pg/μL进行10倍梯度稀释,采用上述经优化的多重PCR反应体系及扩增程序进行多重PCR检测,经凝胶电泳观察分析相应多重PCR扩增的特异性及灵敏度。同时采用基于标准[19]构建的多重PCR检测方法进行对比验证。
1.2.5 人工污染样品的多重PCR检测
取表1中1~10号菌株新鲜SDB培养物各1 mL,分别充分混入10~15 g经121℃、20 min灭菌处理的玉米和花生中,制备成20份人工污染样品,于28℃、相对饱和湿度60%的条件下培养10 d。分别加入20 mL TE缓冲液,涡旋振荡120~180 s,分离上清。离心收集菌体(和孢子),加1 mL TE缓冲液漂洗后离心,重复2~3次,收集菌体,采用真菌基因组DNA提取试剂盒进行基因组DNA提取。调整基因组DNA浓度至10~50 ng/μL,按1.2.4所建立的多重PCR反应体系及扩增程序进行多重PCR检测,凝胶电泳观察分析多重PCR体系扩增的特异性及稳定性。
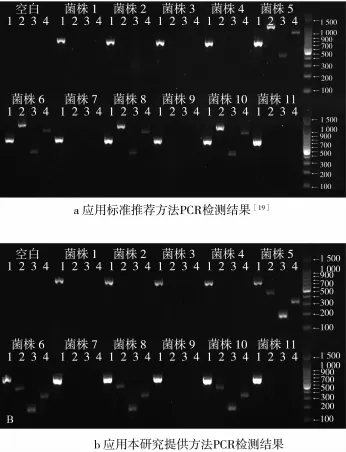
图1 11株菌株ITS、aflR、ver-1和omt-1目的基因单重PCR检测
2.2 单重PCR检测灵敏度分析

图2 以ITS、aflR、ver-1和omt-1基因为目的基因的单重PCR检测灵敏度检测
以黄曲霉菌株CGMCC 3.4410基因组DNA为模板,分别对各对引物的PCR检测灵敏度进行了分析。图2显示,应用标准推荐方法[19],加入基因组DNA浓度最低至100 pg/μL时,能看到清晰的ITS、ver-1和omt-1基因扩增条带,加入基因组DNA浓度最低至1 ng/μL时,能看到清晰的aflR基因扩增条带;应用本研究提供的单重PCR方法,加入基因组DNA浓度最低至10 pg/μL时,能看到清晰的ver-1和omt-1基因扩增条带,加入基因组DNA浓度最低至100 pg/μL时,能看到清晰的ITS和aflR基因扩增条带。表明本研究提供的单重PCR检测方法对aflR、ver-1和omt-1基因的检测灵敏度均优于标准推荐方法的检测灵敏度[19]。
2.3 多重PCR检测特异性和检测灵敏度分析
本研究在不改变扩增程序的前提下,对反应体系主要因素进行优化,确定了多重PCR反应体系。应用优化的多重PCR检测体系及扩增程序,以黄曲霉菌株CGMCC 3.4410基因组DNA为模板,对多重PCR检测特异性及灵敏度进行了分析。图3显示,应用基于标准[19]构建的多重PCR检测方法,加入基因组DNA浓度最低至1 ng/μL时,能看到清晰的4条目的扩增条带;应用本研究提供的多重PCR检测方法,加入基因组DNA浓度最低至100 pg/μL时,能看到清晰的4条目的扩增条带,并且未出现非特异性扩增。表明本研究所提供的多重PCR检测方法在检测特异性和灵敏度方面均优于基于标准[19]构建的多重PCR检测方法,能够满足产黄曲霉毒素真菌多重PCR检测的要求。

图3 ITS、aflR、ver-1和omt-1目的基因的多重PCR检测特异性和检测灵敏度检测
2.4 人工污染样品的多重PCR检测分析
应用本研究提供的多重PCR检测方法对20份人工污染样品检测结果显示,由4株产黄曲霉毒素真菌分别污染的4份花生样品及4份玉米样品的检测结果中4条目的扩增条带均清晰可见,被判定为样品被可能具有产黄曲霉毒素能力菌株污染;而其他12份样品的检测结果中4条目的扩增条带均未同时出现,被判定为样品未被产黄曲霉毒素菌株污染。本研究建立的多重PCR方法符合产黄曲霉毒素真菌快速、高通量检测的要求。
3 结论
以SN/T 2582—2010推荐的黄曲霉毒素生化合成途径中关键调控基因aflR、omt-1和ver-1及真菌共有的5.8S rDNA的ITS序列为目标检测基因,通过生物信息学手段设计多重PCR检测引物,优化反应体系及扩增程序,建立了产黄曲霉毒素真菌多重PCR快速检测方法。该方法对aflR、ver-1和omt-1基因具有较高的检测灵敏度,能够很好地满足实际样品的检测需求。此外,该方法具有操作简便、成本低廉和快速高效等优点,更能符合当前高通量、快速检测的需求。
[1]杜英秋,张瑞英,杨焕春,等.国内外真菌毒素检测标准现状及分析[J].粮食与油脂,2014(7):60-66 Du Y Q,Zhang R Y,Yang H C,et al.Current status and analysis of domestic and international mycotoxin determination standard[J].Cereals and Oils,2014(7):60-66
[2]Marin S,Ramos A J,Cano-Sancho G,et al.Mycotoxins:occurrence,toxicology,and exposure assessment[J].Food and Chemical Toxicology,2013,60(10):218-237
[3]Rocha M E B D,Freire F D C O,Maia F E F,et al.Mycotoxins and their effects on human and animal health[J]. Food Control,2014,36:159-165
[4]秦健,朱珠,于海华,等.粮油饲料原料中外源性真菌毒素的危害与防控[J].粮食与食品工业,2015,22(1):61-64 Qin J,Zhu Z,Yu H H,et al.The harm and prevention&control of exogenous mycotoxins in raw material of grain and oil feedstuff[J].Cereal and Food Industry,2015,22(1):61-64
[5]Razzaghi-Abyaneh M,Chang P,Shams-Ghahfarokhi M,et al.Global health issues of aflatoxins in food and agriculture:challenges and opportunities[J].Frontiers in Microbiology,2014,5(8):420
[6]Hassan Yazdanpanah D,Eslamizad S.Aflatoxins[J].Toxinology,2014,42(4):1-16
[7]Abrar M,Anjum F M,Butt M S,et al.Aflatoxins:biosynthesis,occurrence,toxicity,and remedies[J].Critical Reviews in Food Science&Nutrition,2013,53(8):862-874
[8]Ahmad M M,Ahmad M,Ali A,et al.Detection of Aspergillus flavus and Aspergillus parasiticus from aflatoxin-contaminated peanuts and their differentiation using PCR-RFLP[J].Animals of Microbiology,2014,64(4):1597-1605
[9]Gourama H,Bullerman L B,Gourama H.Aspergillus flavus and Aspergillus parasiticus:aflatoxigenic fungi of concern in foods and feeds:a review[J].Journal of Food Protection,1995,58(12):1395-1404
[10]杨生瑞,屈凌波,孙长坡,等.黄曲霉菌株的分离、鉴定及产毒能力分析[J].中国粮油学报,2012,27(6):110-114 Yang S R,Qu L B,Sun CP,et al.Isolation and identification of aspergillus olavus strains and analysis of toxin-producing ability[J].Journal of the Chinese Cereals and Oils Association,2012,27(6):110-114
[11]Ahmad M M,Alam P,Z.Abdin M,et al.Panfungal PCR method for detection of aflatoxigenic molds[J].Fungal Biology,2013:495-499
[12]Sudini H,Srilakshmi P,Vijay K K K,et al.Detection of aflatoxigenic Aspergillus strains by cultural and molecular methods:A critical review[J].African Journal of Microbiology Research,2015,9(8):484-491
[13]孙长坡,常晓娇,伍松陵,等.利用PCR-RFLP方法鉴别黄曲霉毒素产毒菌株[J].中国农业科学,2014,47(18):3675-3683 Sun C P,Chang X J,Wu S L,et al.Identification of aflatoxin producing strains by using PCR-RFLP method[J]. Scientia Agricultura Sinica,2014,47(18):3675-3683
[14]Mahmoud,M A.Detection of Aspergillus flavus in stored peanuts using real-time PCR and the expression of aflatoxin genes in toxigenic and atoxigenic A.flavus isolates[J]. Foodborne Pathogens and Disease,2015,12(4):289-296
[15]Midorikawa G E,Sousa M D L M D,Silva O F,et al.Characterization of Aspergillus species on Brazil nut from the Brazilian Amazonian region and development of a PCR assay for identification at the genus level[J].Bmc Microbiology, 2014,14:138
[16]蔡军,李慧,欧静堃,等.3种食源性致病菌多重PCR检测体系的建立[J].食品科技,2015,40(3):324-329 Cai J,Li H,Ou J K,et al.Establishment of multiplex PCR for the detection of three foodbornebacterial pathogens[J]. Food Science and Technology,2015,40(3):324-329
[17]Yan W.Multiplex PCR primer design for simultaneous detection of multiple pathogens[J].Methods in Molecular Biology,2015,1275:91-101
[18]秦文彦,程洁,应盛华,等.可污染食品及饲料的产黄曲霉毒素真菌的多重PCR检测[J].菌物学报,2007,26(3):448-454 Qin WY,Cheng J,Ying S H,et al.Multiplex PCR detection of aflatoxigenic fungi able to contaminate food and feed[J].Microsystem,2007,26(3):448-454
[19]SN/T 2582—2010产黄曲霉毒素真菌PCR检测方法[S]SN/T 2582—2010 Detection of aflatoxigenic strains of Aspergillus by PCR[S]
[20]GB/T 18979—2003食品中黄曲霉毒素的测定免疫亲和层析净化高效液相色谱法和荧光光度法[S]GB/T 18979—2003 Determination aflatoxins content in foodcleanup by immunoaffinity chromatographyand determination by high-performance liquid chromatography and fluorometer[S]
[21]Kumeda Y,Asao T.Single-strand conformation polymorphism analysis of PCR-amplified ribosomal DNA internal transcribed spacers to differentiate species of Aspergillus section Flavi[J].Applied and Environmental Microbiology,1996,62(8):2947-2952.
Establishment of Multiplex PCR for the Detection of Aflatoxigenic Fungi
Li Hui Cai Jun Hu Menglong Fu Yang Ou Jingkun
(Nutrition&Health Research Institute,COFCO Corporation;Beijing Key Laboratory of Nutrition,Health&Food Safety,Beijing 102209)
To establish a multiplex PCR method for rapid and high sensitivity detection of aflatoxigenic fungi,4 pairs of specific primers were designed according to the specific genes recommended by the standard of SN/T 2582—2010 Detection of aflatoxigenic strains of Aspergillus by PCR,including aflatoxin biosynthesis regulatory gene aflR,versicolorin A dehydrogenase gene ver-1,sterigmatocystin o-methyltransferase gene omt-1,and the internal transcribed spacer(ITS)of fungal 5.8S rDNA,a multiplex PCR reaction system for aflatoxigenic fungi was established and optimized,and then verified by detection of 10 testing stains.The results showed that this multiplex PCR method had many advantages,such as high sensitivity,strong specificity,convenient and efficient to operation.In conclusion,this method provided a key technical support for developing and applying of rapid detection kit of aflatoxigenic fungi.
multiplex PCR,aflatoxigenic fungi,rapid detection,specificity,sensitivity
Q93-31
A
1003-0174(2017)01-0125-05
国家科技支撑计划(2012BAK08B04-01)
2015-06-05
李慧,女,1971年出生,博士,生物技术
蔡军,男,1985年出生,博士,微生物学

