线粒体DNA D-环区单核苷酸多态性与非霍奇金淋巴瘤患者预后的关系研究
赵桂敏,刘丽宏,高玉环,吴晓琳,高 哲,刁兰萍
·论著·
线粒体DNA D-环区单核苷酸多态性与非霍奇金淋巴瘤患者预后的关系研究
赵桂敏,刘丽宏,高玉环,吴晓琳,高 哲,刁兰萍*
背景 线粒体DNA(mtDNA)D-环区单核苷酸多态性(SNP)的积累可能与患癌风险和疾病预后有关。目的 探讨mtDNA D-环区SNP与非霍奇金淋巴瘤(NHL)患者预后的关系。方法 选取2000—2007年河北医科大学第四医院血液科接受治疗的NHL患者190例(NHL组),其中弥漫大B细胞淋巴瘤(DLBCL)108例(DLBCL亚组),T细胞淋巴瘤(TCL)82例(TCL亚组);同时收集体检健康者159例为对照组。记录NHL组患者性别、年龄、Ann Arbor分期、病理类型(B细胞型、T细胞型)、有无B症状、乳酸脱氢酶(LDH)水平、国际预后指数(IPI),检测NHL组与对照组mtDNA D-环区SNP。NHL患者预后采用多因素Cox比例风险回归模型进行分析。结果 不同Ann Arbor分期、病理类型、有无B症状、不同LDH、IPI NHL患者5年生存率比较,差异均有统计学意义(P<0.05)。对照组与DLBCL亚组患者73、200、315位点等位基因频率比较,差异均有统计学意义(P<0.05)。对照组与TCL亚组患者73、16362、249、315位点等位基因频率比较,差异均有统计学意义(P<0.05)。不同146、199、309、315、16304位点等位基因频率NHL患者5年生存率比较,差异均有统计学意义(P<0.05)。多因素Cox比例风险回归模型结果显示,年龄〔HR=2.104,95%CI(1.095,4.403)〕、IPI〔HR=1.827,95%CI(1.098,3.034)〕、16304位点〔HR=0.523,95%CI(0.276,0.889)〕与NHL患者预后有回归关系(P<0.05)。结论 mtDNA D-环区SNP 16304位点与NHL患者预后相关,可通过检测mtDNA D-环区SNP 16304位点预测NHL患者预后。
淋巴瘤,非霍奇金;DNA,线粒体;多态性,单核苷酸;预后
非霍奇金淋巴瘤(NHL)是第5位最常见的肿瘤[1],2003—2007年我国NHL占所有肿瘤相关死亡的2.15%[2]。目前认为,免疫相关因素、家族血液肿瘤组织学、体质指数与患癌风险有关[3-4]。氧化应激通路基因多态性也被确定与NHL的风险有关[5-6]。罹患肿瘤的风险与氧化应激增加,从而导致DNA损伤有关[7]。虽然NHL可以治愈,但仍有部分患者常规化疗后未能达到完全缓解[8]。目前常用预后指数如国际预后指数(IPI)、滤泡IPI(FIPI)等不能准确预测预后[1]。许多肿瘤标志物已确定为NHL的预测因子,但很少应用在临床常规检查[9]。
人类线粒体基因组是一个16 kb闭环双分子,包含37个基因,其中包括两个核糖体RNA和一套完整的22个转移RNA[10]。线粒体DNA(mtDNA)与核DNA相比更容易受到损伤和突变,归因于其高浓度的活性氧(ROS)、缺乏保护组蛋白和mtDNA修复能力有限[11]。体细胞mtDNA突变与各种退行性疾病和肿瘤有关[12],在许多肿瘤中,体细胞mtDNA突变和多态性位于一个所谓mtDNA D-环区非编码区[13]。这个部位重要的作用是调控mtDNA的复制和表达,因为其包含复制的前导链起源和转录的主要启动子[14]。肿瘤患者D-环区序列发生变化已被广泛研究[15],但只有少数单核苷酸多态性(SNP)被选定可预测肿瘤的风险,而且其预测价值仍不明确[16]。D-环区包含1个1 122 bp长度的核苷酸(核苷酸16024~16569 和1~576)。本研究检测NHL患者mtDNA D-环区SNP,并评估其对NHL预后的预测价值。
1 资料与方法
1.1 临床资料 选取2000—2007年河北医科大学第四医院血液科接受治疗的NHL患者190例为NHL组,其中男121例,女69例;年龄32~74岁,平均年龄(57.6±9.2)岁;弥漫大B细胞淋巴瘤(DLBCL)108例(DLBCL亚组),T细胞淋巴瘤(TCL)82例(TCL亚组)。同时收集体检健康者159例为对照组,其中男86例,女73例;年龄20~73岁,平均年龄(58.7±10.0)岁。本研究获得河北医科大学第四医院人体组织研究委员会的监督和批准,受试者入组前均获得知情同意。
本研究背景:
淋巴瘤是原发于淋巴结和结外淋巴组织的血液系统恶性肿瘤。近年来,淋巴瘤的治疗模式已经从单纯的放化疗进入靶向药物治疗的时代,随着单克隆抗体药物(如针对抗CD20的利妥昔单抗)的应用,使弥漫大B细胞淋巴瘤等B细胞淋巴瘤的缓解率明显提高,但仍有部分患者缓解率低或缓解后短期复发。对于T细胞淋巴瘤,目前临床尚无特异性靶向药物应用,预后较B细胞淋巴瘤差。对于弥漫大B细胞淋巴瘤及外周T细胞淋巴瘤两种最常见类型淋巴瘤,目前采用国际预后指数(IPI)来评价疾病预后,即年龄>60岁、Ann Arbor分期Ⅲ/Ⅳ期、结外病变>1处、乳酸脱氢酶(LDH)水平升高、行为状态ECOG评分≥2分,是5个不良的IPI指标,评分越高,预后越差,但临床实践证明,IPI不能准确预测预后,尤其是“利妥昔单抗时代”。近年来,通过对线粒体DNA(mtDNA)功能的普遍认识及与肿瘤发生、发展相关研究的逐渐丰富,发现mtDNA单核苷酸多态性(SNP)与淋巴瘤预后相关。因此有关mtDNA SNP与淋巴瘤预后的研究可以从基因水平提供思路,来寻找新的可能影响预后的标志,以便更好评价预后、寻求新的治疗手段。
1.2 观察指标 记录NHL组患者性别、年龄、Ann Arbor分期、病理类型(B细胞型、T细胞型)、有无B症状(发热、盗汗、体质量减轻)、乳酸脱氢酶(LDH,速率法L→P,参考范围120~250 U/L)、IPI(0或1为低危、2为低中危、3为中高危、4或5为高危)[1]。检测NHL组与对照组mtDNA D-环区SNP。
1.3 样本采集 入院后空腹采集肘静脉血5 ml,置于试管〔乙二胺四乙酸(EDTA)抗凝〕,放置于-4 ℃冰箱保存备用。将血标本倒入离心管中,加入蒸馏水至45 ml,吹打混匀,将离心管置于离心机,2 500 r/min离心15 min(离心半径20 cm)。取出离心管,弃去上清液,加入少量0.1% Triton-X,用吸管进行吹打混匀,后加0.1% Triton-X至45 ml,再次将离心管置于离心机,2 500 r/min离心15 min(离心半径20 cm)。弃上清液,可见沉淀物,此为白细胞。
1.4 基因组DNA提取 加入3 ml DNA提取液,吹打混匀,加入10%十二烷基硫酸钠(SDS)150 ml,微振,再加入400 μl PK液,微振,水浴过夜。加入5 mol/L氯化钠溶液(NaCl)1 200 μl,2 500 r/min离心15 min(离心半径20 cm)。倒上清液入已装有8 ml的无水乙醇的玻璃瓶中,数秒后看到DNA絮状物析出。将絮状物(即DNA)析出完全,将其移至1.5 ml的经高压灭菌的EP管中(已盛有70%乙醇溶液1 ml)。12 000 r/min离心5 min(离心半径20 cm),弃乙醇溶液,倒置EP管,晾干。
1.5 聚合酶链式反应(PCR) 根据引物合成的Tm值,寻找退火温度的范围,浮动范围不超过10 ℃,设多个温度梯度,调整Taq DNA聚合酶、上下游引物和DNA模板剂量,经多次预实验,最后经凝胶电泳,选择条带最明亮,无非特异扩增条带时的体系为最佳反应条件。试剂采用PCR无色体系预混试剂盒(美国Promega公司),本实验反应体系为50 μl,组成如下:Master Mix 25 μl,Primer F 2.5 μl,Primer R 2.5 μl,Template DNA 2 μl,Water 18 μl。反应条件:95 ℃ 5 min解链→(95 ℃ 30 s,60 ℃ 30 s,72 ℃ 1 min)×40个循环→72 ℃ 5 min延伸。基因组DNA使用向导基因组DNA提取工具立即提取(Promega,Madison,WI)并存储在-20 ℃冰箱。利用上游引物5′-CCCCATGCTTACAAGCAAGT-3′(核苷酸16190~16209)和下游引物5′-GCTTTGAGGAGGTAAGCTAC-3′(核苷酸602~583)从mtDNA D-环区扩增成一个982 bp产物。PCR按照PCR混合试剂盒协议进行测序前纯化。用染料终结者循环测序反应试剂盒进行循环测序,然后把产物置于ABIPRISM基因分析仪3100上分离(Applied Biosystem)。通过反复分析两链多态性被证实。
1.6 随访 电话随访截至2012-10-01,随访时间3~60个月,主要终点事件为死亡。
1.7 统计学方法 采用SPSS 18.0统计软件进行统计学处理,不同临床病理特征患者5年生存率比较采用χ2检验;采用Kaplan-Meier法绘制生存曲线,组间比较采用Log-rank检验;采用多因素Cox比例风险回归模型进行多因素分析,以风险比(HR值)及其95%CI表示。以P<0.05为差异有统计学意义。
2 结果
2.1 不同临床病理特征NHL患者5年生存率比较 不同性别、年龄NHL患者5年生存率比较,差异均无统计学意义(P>0.05);不同Ann Arbor分期、病理类型、有无B症状、不同LDH、IPI NHL患者5年生存率比较,差异均有统计学意义(P<0.05,见表1)。
表1 不同临床病理特征NHL患者5年生存率比较(%)
Table 1 Comparison of five-year survival rates of NHL patients with different clinicopathological features
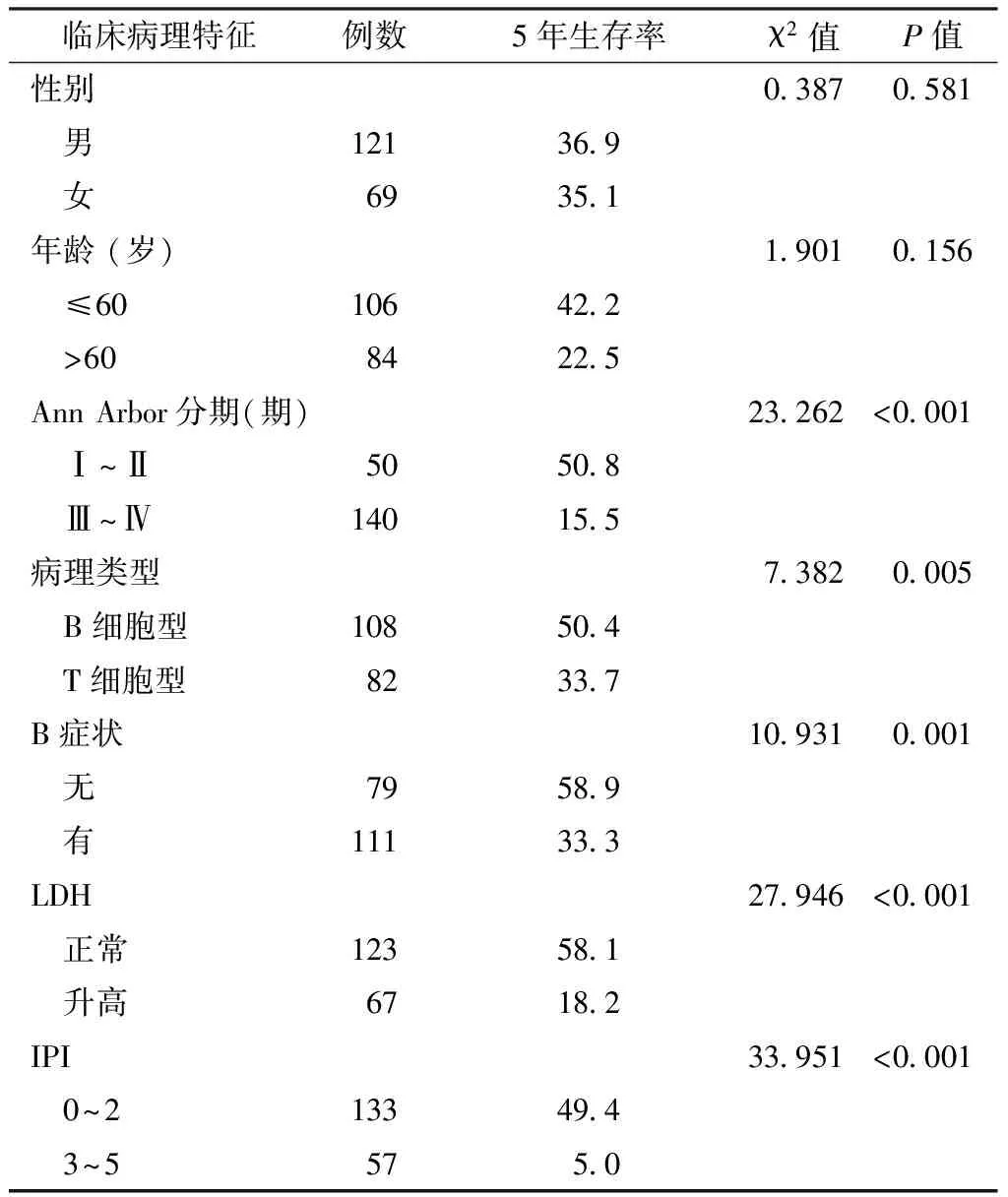
临床病理特征例数5年生存率χ2值P值性别03870581 男121369 女69351年龄(岁)19010156 ≤60106422 >6084225AnnArbor分期(期)23262<0001 Ⅰ~Ⅱ50508 Ⅲ~Ⅳ140155病理类型73820005 B细胞型108504 T细胞型82337B症状109310001 无79589 有111333LDH27946<0001 正常123581 升高67182IPI33951<0001 0~2133494 3~55750
注:LDH=乳酸脱氢酶,IPI=国际预后指数
2.2 对照组与DLBCL、TCL亚组患者SNP位点等位基因频率比较 从对照组和NHL组患者血液样本的982 bp mtDNA D-环区检测到140个位点存在SNP,其中26个位点微小等位基因频率高于5%。对照组与DLBCL亚组患者73、200、315位点等位基因频率比较,差异均有统计学意义(P<0.05,见表2)。对照组与TCL亚组患者73、16362、249、315位点等位基因频率比较,差异均有统计学意义(P<0.05,见表3)。
2.3 不同SNP位点等位基因频率NHL患者5年生存率比较 26个SNP位点在序列数据中均检测到同质的单峰值,预后与加入参考资源库AC_000021 26个SNP之间的关系被检测。依据每个SNP位点的等位基因将NHL患者分为两组,采用Kaplan-Meier法绘制生存曲线。不同146、199、309、315、16304位点等位基因频率NHL患者5年生存率比较,差异均有统计学意义(P<0.05,见图1、表4)。
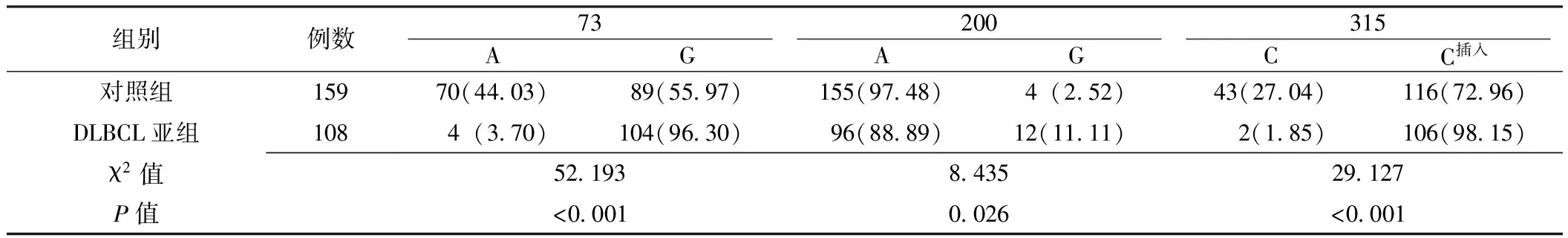
表2 对照组与DLBCL亚组患者SNP位点等位基因频率比较〔n(%)〕
注:DLBCL=弥漫大B细胞淋巴瘤
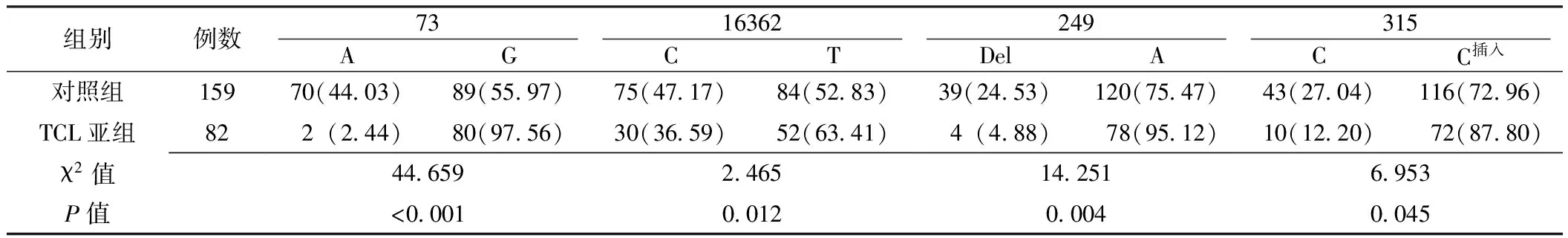
表3 对照组与TCL亚组患者SNP位点等位基因频率比较〔n(%)〕
注:TCL= T细胞淋巴瘤
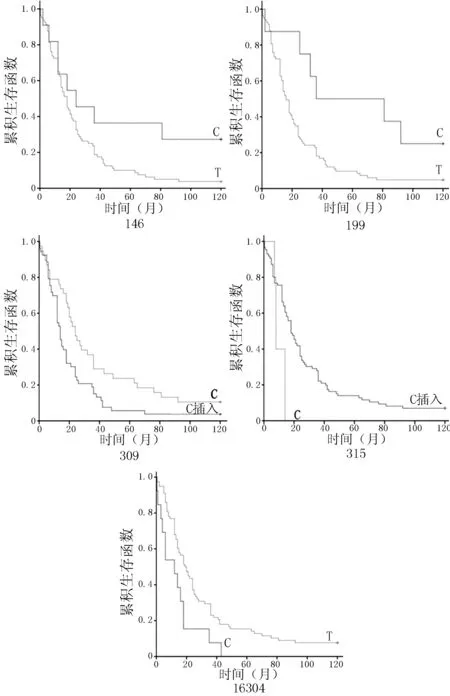
图1 不同SNP位点NHL患者生存曲线
Table 4 Comparison of five-year survival rates of NHL patients with different SNP sites allele frequencies
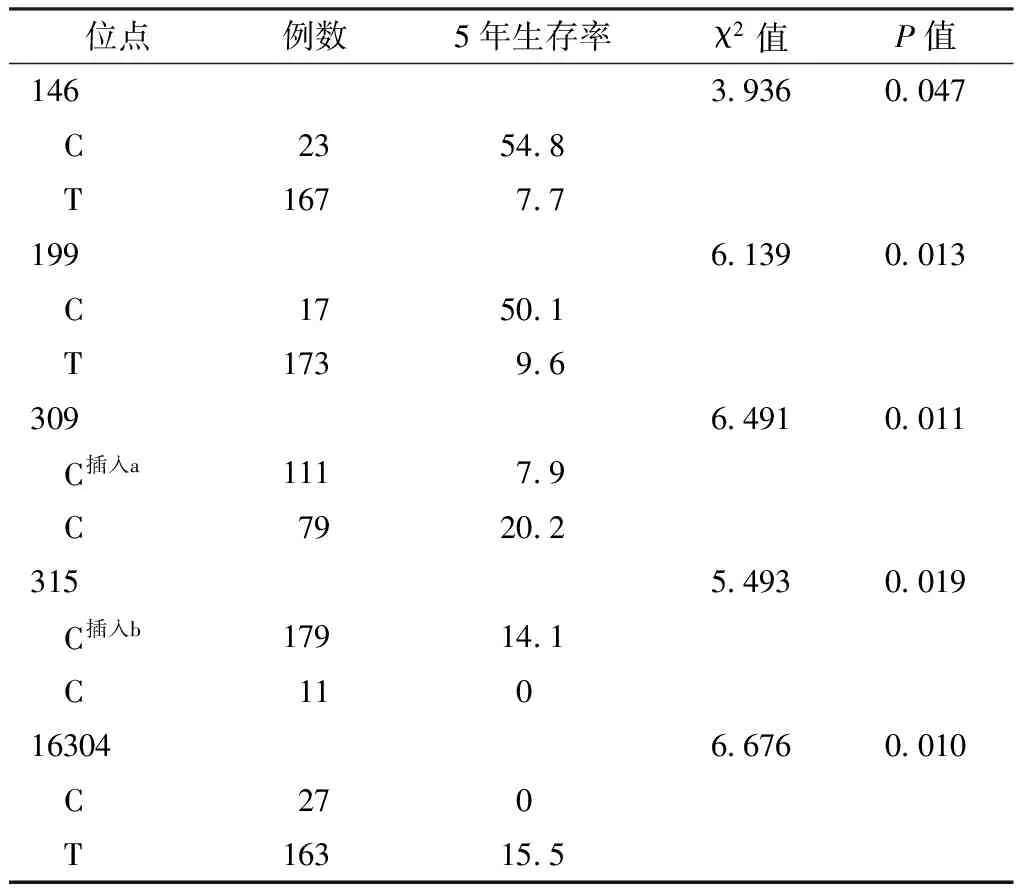
位点例数5年生存率χ2值P值14639360047 C23548 T1677719961390013 C17501 T1739630964910011 C插入a11179 C7920231554930019 C插入b179141 C1101630466760010 C270 T163155
注:a包括1 C、2 C、3 C插入,b包括 1 C、2 C插入
2.4 多因素Cox比例风险回归分析 以NHL患者预后(赋值:生存=0,死亡=1)为因变量,以性别(男=0,女=1)、年龄(≤60岁=0,>60岁=1)、Ann Arbor分期(Ⅰ~Ⅱ期=0,Ⅲ~Ⅳ期=1)、病理类型(B细胞型=0,T细胞型=1)、B症状(无=0,有=1)、LDH(正常=0,升高=1)、IPI(0~2=0,3~5=1)、146位点(C=0,T=1)、199位点(C=0,T=1)、309位点(C插入=0,C=1)、315位点(C插入=0,C=1)、16304位点(C=0,T=1)为自变量,代入多因素Cox比例风险回归模型,结果显示,年龄、IPI、16304位点与NHL患者预后有回归关系(P<0.05,见表5)。
表5 NHL患者预后影响因素的多因素Cox比例风险回归分析
Table 5 Multivariate Cox proportional hazards regression analysis of prognostic factors in NHL patients
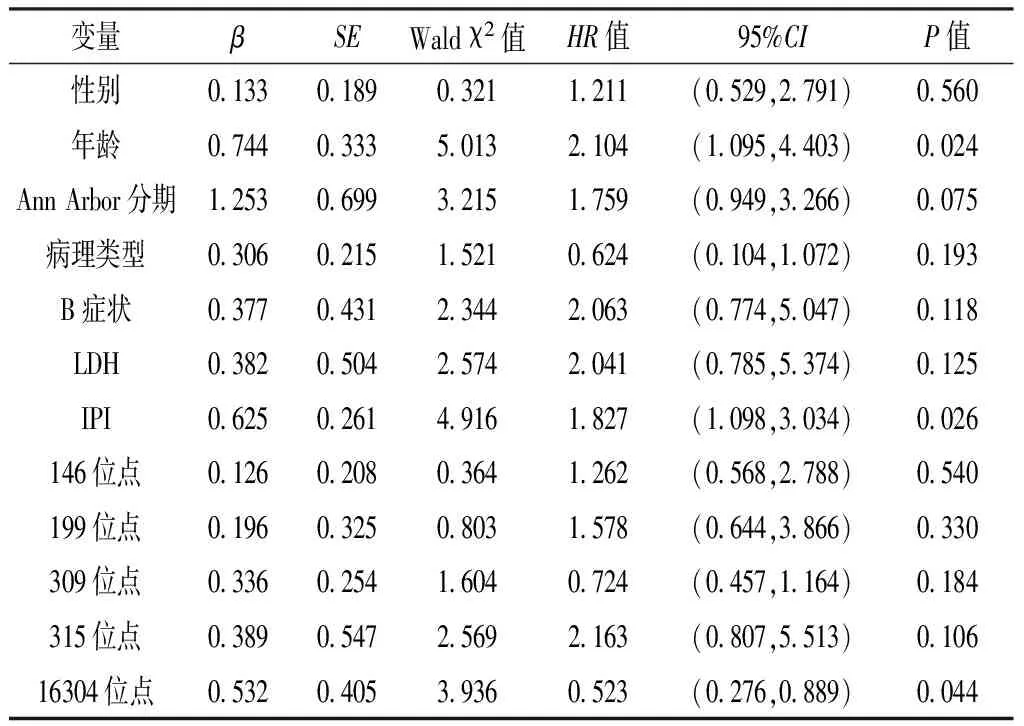
变量βSEWaldχ2值HR值95%CIP值性别0133018903211211(0529,2791)0560年龄0744033350132104(1095,4403)0024AnnArbor分期1253069932151759(0949,3266)0075病理类型0306021515210624(0104,1072)0193B症状0377043123442063(0774,5047)0118LDH0382050425742041(0785,5374)0125IPI0625026149161827(1098,3034)0026146位点0126020803641262(0568,2788)0540199位点0196032508031578(0644,3866)0330309位点0336025416040724(0457,1164)0184315位点0389054725692163(0807,5513)010616304位点0532040539360523(0276,0889)0044
3 讨论
本研究发现不同性别、年龄NHL患者5年生存率无明显差异,提示性别、年龄与NHL预后无关。但不同Ann Arbor分期、病理类型、有无B症状、不同LDH、IPI NHL患者5年生存率差别较大,提示分期晚、T细胞型、有B症状、LDH水平升高、高IPI为NHL患者不良预后的因素。既往研究在某些肿瘤中已经确定了与肿瘤风险和预后相关的mtDNA D-环区SNP[17]。本研究通过检测mtDNA D-环区SNP,以评估其预测NHL风险的能力。对照组与NHL患者比较73 A、315 C等位基因频率增高,提示核苷酸微小等位基因73、315位点与NHL风险降低有关,这表明携带这些等位基因可抵抗患NHL风险。对照组与DLBCL亚组患者比较200 G等位基因频率明显减低,提示200位点特异性与DLBCL的风险增加有关。对照组与TCL亚组患者比较16362 C、249 Del等位基因频率增高,提示16362、249位点特异性与TCL的风险降低相关。mtDNA复制和表达受mtDNA D-环区调控,因为其是复制起始位点。D-环区转录调控区SNP如何增加肿瘤风险的真正机制仍然未知,这个区域的SNP可能影响mtDNA复制,导致电子传递链的改变,这引起高ROS释放和核基因组损伤以及肿瘤发生和发展[18]。
本研究进一步选择mtDNA D-环区SNP评估其预测NHL患者预后能力。不同146、199、309、315、16304位点等位基因频率NHL患者5年生存率有明显差异,提示mtDNA D-环区SNP影响NHL患者预后。进一步进行多因素Cox比例风险回归分析,结果显示年龄、IPI、16304位点为NHL患者的独立预后影响因素。单因素分析患者年龄与NHL预后无关,可能是因为一部分年轻患者为侵袭性更高的TCL,从而影响NHL患者生存率,多因素分析提示年龄与NHL预后有关,年龄越大预后越差。IPI作为NHL常用预后指数,目前广泛应用于临床,本研究也进一步验证了IPI与预后相关。同时发现新的预后因素16304位点,但尚需在临床实践中进一步验证。研究显示,16304位点SNP,食管鳞癌发生体细胞突变[19],16304位点SNP属于控制16192-16270-16304-150区域主题区分线粒体单倍型U5亚型[20]。不同mtDNA D-环区SNP位点似乎表现出独特的致癌作用和肿瘤预后,这种现象也在肝细胞癌和非小细胞肺癌观察到,每种肿瘤与预后和肿瘤风险相关的SNP是不同的[21]。与肿瘤风险及预后相关的SNP位点均位于高变区(HV),生殖系突变热点和肿瘤mtDNA优先发生突变[22]。核苷酸16362、16304位点属于HV-Ⅰ而其他属于HV-Ⅱ。HV的功能性意义尚不清楚,但是其对肿瘤发展起重要作用。
总之,mtDNA D-环区SNP与NHL患癌风险及预后有关,通过分析D-环区SNP可能有助于识别发展为NHL风险较高及高危预后的患者亚组,从而有助于改善这些患者的治疗决策。
作者贡献:赵桂敏进行文章的构思与设计,研究的实施与可行性分析,进行结果的分析与解释,撰写论文;吴晓琳进行数据收集、整理;高哲进行统计学处理;刘丽宏、刁兰萍负责论文的修订;高玉环负责文章的质量控制及审校;刁兰萍对文章整体负责,监督管理。
本文无利益冲突。
[1] GEYER S M,MORTON L M,HABERMANN T M,et al.Smoking,alcohol use,obesity,and overall survival from non-Hodgkin lymphoma:a population-based study[J].Cancer,2010,116(12):2993-3000.DOI:10.1002/cncr.25114.
[2] 张敏,李广灿,张玉玲.2003~2007年中国恶性淋巴瘤发病与死亡分析[J].中国肿瘤,2012,21(3):190-196. ZHANG M,LI G C,ZHANG Y L.An analysis of the incidence and mortality with malignant lymphoma in China during 2003—2007 [J].China Cancer,2012,21(3):190-196.
[3] CHATTERJEE N,HARTGE P,CERHAN J R,et al.Risk of non-Hodgkin′s lymphoma and family history of lymphatic,hematologic,and other cancers[J].Cancer Epidemiol Biomarkers Prev,2004,13(9):1415-1421.
[4] LAN Q,WANG S S,MENASHE I,et al.Genetic variation in Th1/Th2 pathway genes and risk of non-Hodgkin lymphoma:a pooled analysis of three population-based case-control studies[J].Br J Haematol,2011,153(3):341-350.DOI:10.1111/j.1365-2141.2010.08424.x.
[5] LIGHTFOOT T J,SKIBOLA C F,SMITH A G,et al.Polymorphisms in the oxidative stress genes,superoxide dismutase,glutathione peroxidase and catalase and risk of non-Hodgkin′s lymphoma[J].Haematologica,2006,91(9):1222-1227.
[6] WANG S S,DAVIS S,CERHAN J R,et al.Polymorphisms in oxidative stress genes and risk for non-Hodgkin lymphoma[J].Carcinogenesis,2006,27(9):1828-1834.DOI:10.1093/carcin/bgl013.
[7] LOFT S,SVOBODA P,KAWAI K,et al.Association between 8-oxo-7,8-dihydroguanine excretion and risk of lung cancer in a prospective study[J].Free Radic Biol Med,2012,52(1):167-172.DOI:10.1016/j.freeradbiomed.2011.10.439.
[8] KUBE D,HUA T D,VON BONIN F,et al.Effect of interleukin-10 gene polymorphisms on clinical outcome of patients with aggressive non-Hodgkin′s lymphoma:an exploratory study[J].Clin Cancer Res,2008,14(12):3777-3784.DOI:10.1158/1078-0432.CCR-07-5182.
[9] DIAO L P,MA H,WEI G C,et al.Matrix metalloproteinase-2 promoter and tissue inhibitor of metalloproteinase-2 gene polymorphisms in non-Hodgkin′s lymphoma[J].Int J Cancer,2012,131(5):1095-1103.DOI:10.1002/ijc.26483.
[10] LAWLESS M W,O′BYME K J,Gray S G.Oxidative stress induced lung cancer and COPD:opportunities for epigenetic therapy[J].J Cell Mol Med,2009,13(9A):2800-2821.DOI:10.1111/j.1582-4934.2009.00845.x.
[11] DIMAURO S,SCHON E A.Mitochondrial DNA mutations in human disease[J].Am J Med Genet,2001,106(1):18-26.
[12] WALLACE D C.Mouse models for mitochondrial disease[J].Am J Med Genet,2001,106 (1):71-93.DOI:10.1002/ajmg.1392.
[13] SANCHEZ-CESPEDES M,PARRELLA P,NOMOTO S,et al.Identification of a mononucleotide repeat as a major target for mitochondrial DNA alterations in human tumors[J].Cancer Res,2001,61(19):7015-7019.
[14] TAANMAN J W.The mitochondrial genome:structure,transcription,translation and replication[J].Biochim Biophys Acta,1999,1410(2):103-123.
[15] YONEYAMA H,HARA T,KATO Y,et al.Nucleotide sequence variation is frequently in the mitochondrial DNA displacement loop region of individual human tumor cells[J].Mol Cancer Res,2005,3(1):14-20.
[16] ZHANG R,WANG R,ZHANG F,et al.Single nucletide polymorphisms in the mitochondrial displacement loop and outcome of esophageal squamous cell carcinoma[J].J Exp Clin Cancer Res,2010,29:155.DOI:10.1186/1756-9966-29-155.
[17] ZHANG R,ZHANG F,WANG C,et al.Identification of sequence polymorphisms in the D-Loop region of mitochondrial DNA as a risk factor for hepatocellular carcinoma with distinct etiology[J].J Exp Clin Cancer Res,2010,29:130.DOI:10.1186/1756-9966-29-130.
[18] GILLE J J,JOENJE H.Cell culture models for oxidative stress:superoxida and hydrogen peroxidative versus normobaric heperoxia[J].Mutant Res,1992,275(3/4/5/6):405-414.
[19] KUMIMOTO H,YAMANE Y,NISHIMOTO Y,et al.Frequent somatic mutations of mitochondrial DNA in esophageal squamous cell carcinoma[J].Int J Cancer,2004,108(2):228-231.DOI:10.1002/ijc.11564.
[20] PALA M,ACHILLI A,OLIVIERI A,et al.Mitochondrial haplogroup U5b3:a distant echo of the epipaleolithic in Italy and the legacy of the early Sardinians[J].Am J Hum Genet,2009,84(6):814-821.DOI:10.1016/j.ajhg.2009.05.004.
[21] WANG C,ZHANG F,FAN H,et al.Sequence polymorphisms of mitochondrial D-Loop and hepatocellular carcinoma outcome[J].Biochem Biophys Res Commun,2011,406(3):493-496.DOI:10.1016/j.bbrc.2011.02.088.
[22] STONEKING M.Hypervariable sites in the mtDNA control region are mutational hotspots[J].Am J Hum Genet,2000,67(4):1029-1032.DOI:10.1086/303092.
(本文编辑:陈素芳)
Single Nucleotide Polymorphisms in the D-Loop Region of Mitochondrial DNA and Prognosis of Non-Hodgkin Lymphoma Patients
ZHAOGui-min,LIULi-hong,GAOYu-huan,WUXiao-lin,GAOZhe,DIAOLan-ping*
DepartmentofHematology,FourthHospitalofHebeiMedicalUniversity,Shijiazhang050000,China
*Correspondingauthor:DIAOLan-ping,Chiefphysician;E-mail:diaolanping@163.com
Background Accumulation of single nucleotide polymorphisms (SNP) in the displacement loop (D-Loop) of mitochondrial DNA (mtDNA) might be associated with cancer risk and disease prognosis.Objective To discuss the relationship between the prognosis of non-Hodgkin lymphoma (NHL) patients and the SNP in D-Loop of mtDNA.Methods We enrolled 190 NHL patients(NHL group) who received treatment in Department of Hematology,Fourth Hospital of Hebei Medical University from 2000 to 2007,including 108 diagnosed with diffuse large B-cell lymphoma (DLBCL),the DLBCL subgroup,and 82 with T-cell lymphoma (TCL),the TCL subgroup.At the same time 159 healthy controls undergoing physical examination in the hospital during the same period were selected as the control group.The patient′s gender,age,Ann Arbor staging,type of pathology (B-cell type,T-cell type),B symptoms,lactate dehydrogenase (LDH) levels,and value of international prognostic index (IPI) in NHL group were recorded.The SNP in D-Loop of mtDNA in NHL group and control group were detected.The prognosis of NHL patients was analyzed by using a Cox proportional hazard regression model.Results The differences in 5-year survival rate among different Ann Arbor staging,type of pathology,B symptoms,LDH levels and value of IPI in NHL patients were statistically significant(P<0.05).The differences in 73,200 and 315 sites allele frequencies between the control group and the DLBCL subgroup were statistically significant (P<0.05).The differences in 73,16362,249,315 sites alleles frequencies between the control group and the TCL subgroup were statistically significant (P<0.05).The differences in 5-year survival rate among different 146,199,309,315 and 16304 sites allele frequencies in NHL patients were statistically significant (P<0.05).Multivariate Cox proportional hazards regression analysis showed that age〔HR=2.104,95%CI(1.095,4.403)〕,value of IPI〔HR=1.827,95%CI(1.098,3.034)〕,and 16304 site〔HR=0.523,95%CI(0.276,0.889)〕 were significantly associated with the prognosis of NHL patients (P<0.05).Conclusion The 16304 SNP site in D-Loop of mtDNA is associated with the prognosis of NHL patients,which can be used to predict the prognosis of NHL patients.
Lymphoma,non-Hodgkin;DNA,mitochondrial;Polymorphism,single nucleotide;Prognosis
河北省科技支撑计划资助项目(135200)
R 733.41
A
10.3969/j.issn.1007-9572.2017.11.012
2016-08-18;
2017-01-20)
050000河北省石家庄市,河北医科大学第四医院血液科
*通信作者:刁兰萍,主任医师;E-mail:diaolanping@163.com
赵桂敏,刘丽宏,高玉环,等.线粒体DNA D-环区单核苷酸多态性与非霍奇金淋巴瘤患者预后的关系研究[J].中国全科医学,2017,20(11):1335-1340.[www.chinagp.net]
ZHAO G M,LIU L H,GAO Y H,et al.Single nucleotide polymorphisms in the D-Loop region of mitochondrial DNA and prognosis of non-Hodgkin lymphoma patients[J].Chinese General Practice,2017,20(11):1335-1340.

