两种花吊丝竹叶片蛋白提取方法的2-DE比较
沈少炎, 吴玉香, 荣俊冬, 何天友, 郑郁善1,
(1.福建农林大学园林学院;2.福建农林大学林学院;3.福建农林大学竹类研究所,福建 福州 350002)
两种花吊丝竹叶片蛋白提取方法的2-DE比较
沈少炎1,3, 吴玉香1,3, 荣俊冬2,3, 何天友1,3, 郑郁善1,2,3
(1.福建农林大学园林学院;2.福建农林大学林学院;3.福建农林大学竹类研究所,福建 福州 350002)
以2年生花吊丝竹的扦插苗为研究材料,对2种方法(TCA/酚提结合法和PEG法)提取的蛋白质的2-DE差异进行研究.结果表明:PEG法的5个组分的条带差异明显,全蛋白得到了有效分离,在F3中富集到了2条高丰度蛋白带;2种方法的2-DE图谱的均一性分析发现,PEG法中F3富集了高丰度蛋白,提高了2-DE中蛋白质的检测率和分辨率,PEG法中F1-F5共检测的蛋白总数超过了1700个,但是相邻组存在一定的重叠率,而TCA丙酮/酚提结合法只检测到约571个蛋白点,5个组与NF平均有439个蛋白点匹配,达到77.9% 以上;质谱鉴定了5种差异蛋白,生物信息学分析发现这5种蛋白是糖酵解、糖异生和ATP合成的重要辅酶.2种方法的比较发现,PEG法能够将花吊丝竹叶片全蛋白中高丰度蛋白单一富集在16%的组分中,从而显著提高了2-DE的分辨率,再通过质谱技术能够检测到更多的影响生物体生长发育的低丰度蛋白,所以PEG法是一种切实可行的且较为理想的蛋白质提取方法.
花吊丝竹; TCA/酚提结合法; PEG法; 双向电泳; 生物信息学
随着分子生物技术的迅猛发展,双向聚丙烯凝胶电泳(two-dimensional gel electrophoresis technology,2-DE)和质谱技术已经成为研究植物蛋白组学的重要技术手段[1].2-DE技术的高分辨率及高重复性对于分离和显现复杂的蛋白质混合物尤为适用[2].但是,蛋白样品制备方法的优劣严重影响了第一向等电聚焦(isoelectric focusing electrophoresis, IEF)的结果,进而影响了蛋白的质量和2-DE分析的准确性[3].植物叶片组织中包含了大量的非蛋白物质,如脂类、有机酸、多糖多酚、植物细胞壁组分及次生代谢产物,这些物质的存在严重影响了2-DE的效果,造成蛋白点分离不清晰,进而影响下游的生物学分析[4-6].尽管如液相层析法等蛋白分离生物新技术有了长足的发展,但是利用传统方法提取和分离蛋白并结合质谱技术依然是蛋白组学研究的主要手段.
酚提法或TCA/丙酮法已成功应用,但是在植物叶片蛋白组分析中有一些缺陷.虽然TCA/丙酮法已成功应用于玉米(Zeamays)叶片[7]以及木本植物组织[8],但是对于一些其他的植物组织(如成熟果实、橄榄叶等)无法制备出良好的蛋白质样品,且蛋白质无法完全裂解是该方法的固有弊端[9].酚提法也成功应用于植物组织全蛋白的提取实验[10],尤其是植物果实组织中蛋白质的提取[11],但是该方法提取的全蛋白数量级较小,且像植物纤维、根系等组织也无法较好地去除干扰的混合物[12].TCA/酚提结合法是上述两种方法的结合,是一种较新的蛋白质提取方法,能够有效地提取植物全蛋白[13],该方法是在提取成年橄榄叶中蛋白质的研究中发展的,该方法通过SDS提取液和TCA丙酮结合的酚提法,去除了橄榄叶中大量的多酚化合物,许多在TCA/丙酮法中无法溶解的蛋白质,在经过SDS溶液提取后可以得到较好的解决.此外,通过SDS提出的蛋白质还可以用饱和酚进一步纯化[9].TCA丙酮/酚提结合法也成功地在植物蛋白提取试验中得到验证,研究者已经通过该方法提取出了竹类叶片、橄榄老叶以及红树(Rhizophoraapiculata)叶片的蛋白质[13].然而,上述提及的三种蛋白提取方法都无法检测到植物组织中的低丰度蛋白,致使蛋白组分析的分辨率较低,主要原因是高丰度蛋白的遮挡效应(如1,5-二磷酸核酮糖羧化酶/加氧酶Rubisco)[14]以及IPG胶条的低上样量[15].TCA/丙酮法提取的蛋白溶解率低,无法完全排除核酸的干扰,造成了2-DE的低分辨率[16];Sarma et al[17]利用改良酚提法提取了不同大豆组织中的全蛋白,蛋白溶解率较高,但是干扰物质较多,呈现的2-DE图谱条纹背景较严重,此外,大豆叶片中高丰度的Rubisco遮挡了较多的低丰度蛋白.所以在含有大量Rubisco及其他干扰物的植物体中,采用TCA/酚提结合法也无法获得较多低丰度蛋白及高分辨率的2-DE图谱[1].
然而,聚乙二醇(polyethylene glycol, PEG)分级法利用不同PEG浓度富集原理,对分子质量不同的蛋白质进行分级制备[18].在拟南芥(Arabidopsisthaliana)中,采用16% PEG富集高丰度蛋白Rubisco[15],之后研究人员又利用多个浓度的PEG富集拟南芥蛋白组中的Rubisco[19].此外,多种草本植物都采用15% PEG富集高丰度的蛋白[1,20,21].通过富集高丰度的蛋白,可以针对性地研究低丰度蛋白在生物生长发育过程中的调节功能,是当今蛋白组学的研究趋势[22].
花吊丝竹(Dendrocalamusminorvar.amoenus)为竹亚科(Bambusoideae)牡竹属(Dendrocalamus)植物,为吊丝竹(Dendrocalamusminor)变种,杆较原种矮小,节间浅黄并有若干条深绿色纵条纹,竹竿颜色奇异、条纹美丽,是园林观赏的重要竹种之一[23].但是花吊丝竹在成笋期容易受到病虫侵害,影响花吊丝竹的使用价值.而植物体对于病虫害的防御受到蛋白质相互作用的影响[24].采用TCA丙酮/酚提结合法[9]以及PEG分级法[15]提取花吊丝竹叶片全蛋白进行2-DE比较,发现2种方法的图谱分辨率差异显著,PEG法通过富集高丰度蛋白使得低丰度蛋白得以显现,表现出更好的分辨率和分离效果.对2种提取方法2-DE中表现出的显著差异蛋白点进行MS质谱分析,并对这些蛋白进行生物信息学分析.通过对两种方法不同方面差异的比较,探讨竹类植物适宜的蛋白质提取方法体系,为竹类植物蛋白组学研究中的样品制备提供一定的试验基础,同时PEG法能够鉴定更多的低丰度蛋白,为其他植物蛋白质的提取以及功能性蛋白的研究提供依据.
1 材料与方法
1.1 材料
花吊丝竹叶片取自福建农林大学GAP中心.选取2年生长势相同的植株3株,每株剪取若干侧枝顶部叶片,贮存于液氮罐中,并存储于-80 ℃冰箱中.试验中样品的制备和2-DE均重复3次,以保证试验的重复性和精确性.
1.2 蛋白样品制备
1.2.1 TCA/酚提结合法 采用Wang et al[13]改进的TCA丙酮/酚提结合的蛋白提取方案,将离心的转速和时间调整为15 000g,5 min.
1.2.2 PEG分级法 参照Xi et al[15]方法并略有修改,将全蛋白分为5个组分.将全部5个组分加入5倍体积预冷的10% TCA丙酮溶液(质量分数10% TCA和体积分数0.07% β-巯基乙醇),-20 ℃静置过夜.
1.3 双向电泳(2-DE)
1.3.1 蛋白的溶解及定量 将2种方法提取的蛋白加入适量样品裂解溶液(7 mol·L-1尿素,2 mol·L-1硫脲,4% CHAPS,40 mmol·L-1MDTT),完全溶解后采用Bradford法[25]对蛋白溶液进行标准定量.
1.3.2 蛋白样品的SDS-PAGE电泳 垂直电泳采用Mini-PROTEAN®(BIO-RAD, USA),用来分析2种提取方法中花吊丝竹叶片蛋白质分子质量条带的区别.实际电泳操作参照BIO-RAD公司Mini-PROTEAN操作指导手册.电泳结束后立即用考马斯亮蓝(coomassie brilliant blue, CBB)染色[26].凝胶谱图用UMAX Power Look 2100XL扫描,PDQUEST 8.0.1(BIO-RAD, USA)分析.
1.3.3 2-DE 选用pH 5~8 IPG胶条(BIO-RAD, USA)进行样品的水化,采用被动水化方式,上样量及上样体积分别为30 μg和150 μL.第一向等电聚焦(IEF)采用PROTEAN®i12TM(BIO-RAD, USA),IEF参数设为:250 V(30 min)线性,500 V(30 min)快速,4 000 V(3 h)快速,4 000 V(20 000 Vh),500 V保持.IEF结束后立即进行2次平衡,随后进行SDS-PAGE电泳,聚丙烯酰胺凝胶的浓度设为12%.电泳后采用银染法染色,但不添加戊二醛,以免造成后续质谱的不兼容[27].所有水化、IEF、SDS-PAGE的试验操作参照BIO-RAD的2-DE双向电泳操作指导手册.凝胶的蛋白点通过UMAX Power Look 2100XL扫描获得,并用PDQUEST 8.0.1分析.
1.4 胶内酶解和质谱鉴定
银染后的凝胶图谱经过软件分析后,能够匹配到2种方法表现出的差异蛋白点.选取显著变化的若干点(包括低丰度蛋白点和差异点)进行MALDI-TOF-MS质谱分析[28],步骤为:蛋白点的挖取、质谱样品制备、肽段酶解、质谱仪检测、数据库比对[29,30].主要的分析参数设置:数据库选用NCBI和Swissprot;分类系统选用NCBI_Poaceae和NCBI_Viridiplantae;搜索类型设为Combined(MS+MS/MS);采用Trypsin进行酶解;固定修饰Carbamidomethyl(C);可变修饰Oxidation(M);蛋白质分子质量无限制:Unrestricted;肽段量的误差范围设为±0.4 u;Mass value Monoisotopic.最后得到鉴定的蛋白质结果.质谱后得到原始文件利用MASCOT 2.2(Matrixscience)程序进行肽段图谱(PMF)的数据库检索;为确保鉴定结果更为可信,每个质谱数据的最小离子分值可信度应不低于95.
1.5 生物信息学分析
通过MALDI-TOF-MS质谱鉴定的蛋白可进行生物信息学分析,通过PANTHER、PID、KEGG及GENE ONTOLOGY等分析工具进一步分析差异蛋白的生物功能,为更深层次的花吊丝竹蛋白组研究提供依据.
2 结果与分析
2.1 2种提取方法蛋白条带的SDS-PAGE分析
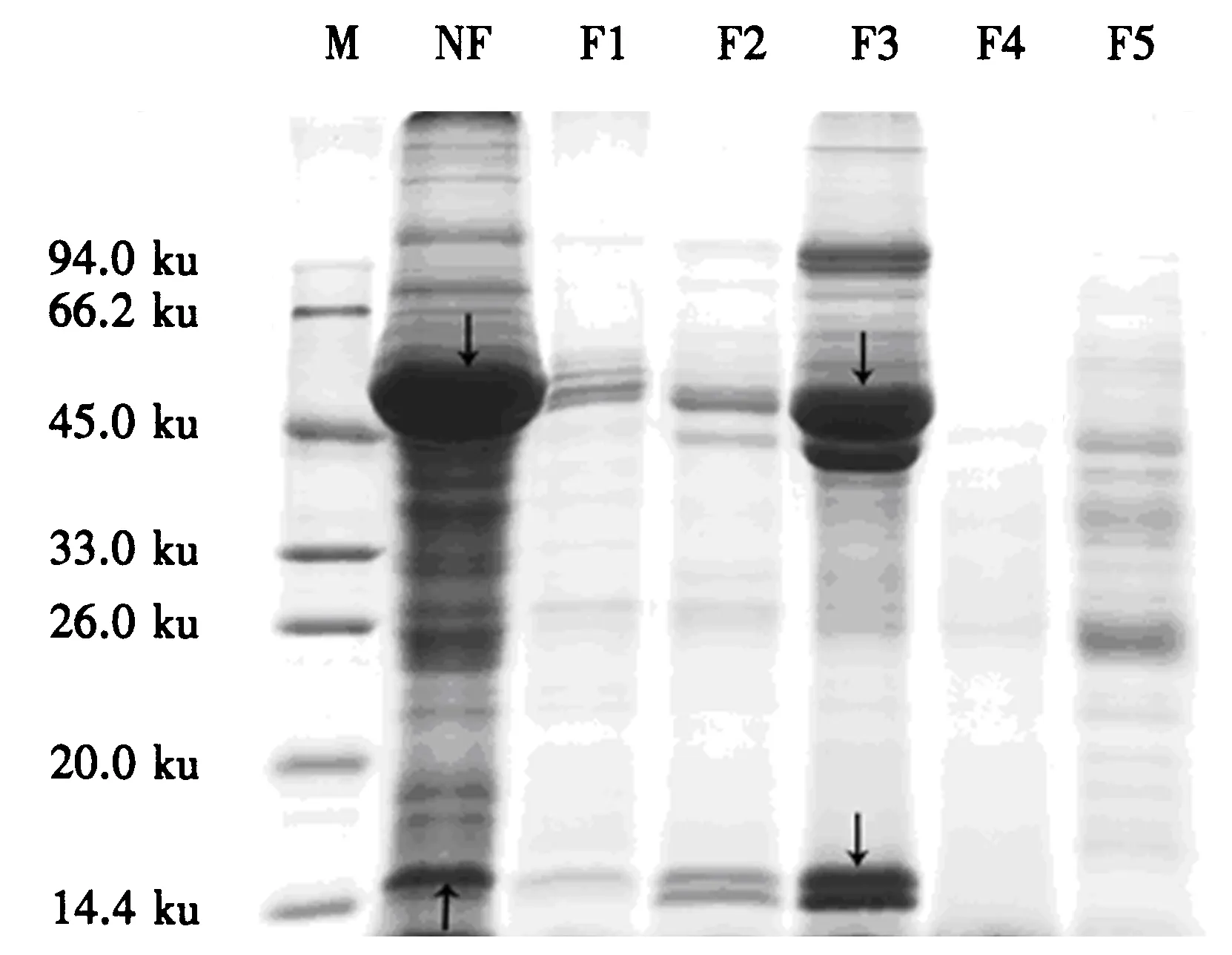
M:Marker protein.图1 TCA丙酮/酚提法(NF)和PEG法(F1~F5)的SDS-PAGE分析Fig.1 SDS-PAGE analysis of proteins from TCA/phenol (NF) and PEG fractionation (F1~F5)
对2种花吊丝竹叶片蛋白提取方法进行比较,每组试验重复3次.即将TCA丙酮/酚提结合法的全蛋白NF和PEG法的5个组分(F1~F5) 进行SDS-PAEG分析(图1).结果表明:PEG法提出的5个组分(F1~F5)的条带存在明显差异,表明了花吊丝竹叶片全蛋白通过PEG法得到了有效分离,16% PEG浓度的F3富集了叶片中大多数的高丰度蛋白.NF中有2条颜色较深的高丰度蛋白带(15 ku上箭头和60 ku下箭头),而F3中有2条深色的高丰度蛋白条带,条带颜色较深,在15 ku上箭头和60 ku下箭头附近各有1条深色的蛋白条带,而F4及F5中蛋白的分布较均匀,相同区域的蛋白条带较浅或没有,尤其是F4没有明显的深色条带.表明了2种高丰度蛋白被富集在F3中,而F2、F4中显现了较多的低丰度蛋白,对后续2-DE的效果影响较大.
2.2 2种提取方法2-DE的均一性分析
将TCA丙酮/酚提结合法的全蛋白NF和PEG法的F1~F5进行2-DE比较(图2). 由图2可以看到,在TCA丙酮/酚提结合法提取的NF中有2部分的高丰度蛋白点(圆圈范围内),染色较深,覆盖范围大,低丰度蛋白极易被遮盖,尤以大圆圈内的高丰度点的遮盖最为明显.而PEG法提取的蛋白质2-DE图谱(图2-F3、F4、F5)中,NF中的高丰度蛋白由于被富集分离,显现在F3组分中.因为高丰度蛋白富集在F3组中以及IPG胶条有限的上样量,导致F1、F2组分图像不清晰,都未显示出高丰度蛋白的富集和低丰度蛋白.F4及F5这2个组分高丰度蛋白含量明显降低,其中,F4中高丰度蛋白基本没有在凝胶中显现.2种提取方法对比分析表明,在相同水化上样量的条件下,F4和F5 组分中的低丰度蛋白含量明显提高,加大了对低丰度蛋白的检测.
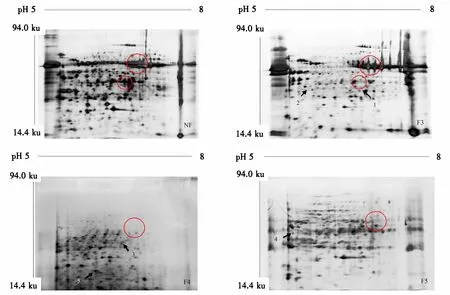
图2 TCA丙酮/酚提法(NF)和PEG法(F3、F4、F5)的2-DE分析Fig.2 2-DE analysis of whole protein (NF) and PEG fraction of F3, F4, F5
通过3组重复试验的对比,PEG法中5个组的2-DE凝胶图谱中蛋白点有部分重叠,利用PDQUEST 8.0.1软件对5个组进行相邻组的重叠匹配分析,计算出蛋白点的重叠率,结果表明,F1/F2、F2/F3的重叠率相对较高(表1).
通过PDQUEST对5个组分和NF进行进一步的均一性匹配(表2).PEG法之后能够检测到的蛋白点显著增多,较多的低丰度蛋白显现在凝胶上.虽然5个组分的相邻组存在一定的重叠蛋白点,但是F1~F5可检测的蛋白总数超过1 700个,而TCA丙酮/酚提结合法(NF)检测到的蛋白点只有约571个,各组分中的蛋白点与NF平均有439个点匹配,重叠率达到77.9% 以上,表明PEG法之后既保证了相对完整的花吊丝竹叶片全蛋白,与此同时又显著提高了2-DE的分辨率.表明通过16% PEG能够单一富集花吊丝竹叶片中的高丰度蛋白,同时PEG 法可以作为提取、检测花吊丝竹叶片全蛋白的改良方案.
2.3 差异蛋白点的MS鉴定
在PDQUEST8.0.1中选择Analyze下的Analysis Set Manager分析组管理器,新建1个Qualitative定性分析类型.以NF为Master胶,F1~F5为比较分析胶,选择Method: On in F1~F5(只在F1~F5中有表达).完成后点击Save运行计算,显示只在F1~F5中表达的点.选取5个胶上清晰的差异点1、2、3、4、5进行质谱鉴定(表3),以期发现这些差异的蛋白对植物生长发育的作用.在得到原始峰图后进行比对.
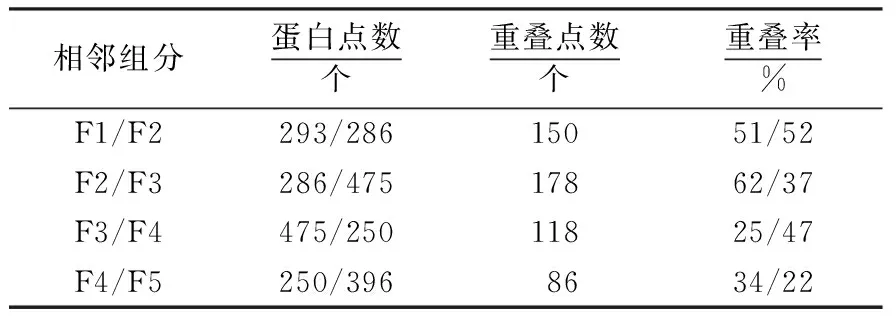
表1 5个组分间的蛋白点重叠率Table 1 Overlapping rate of 5 adjacent component proteins
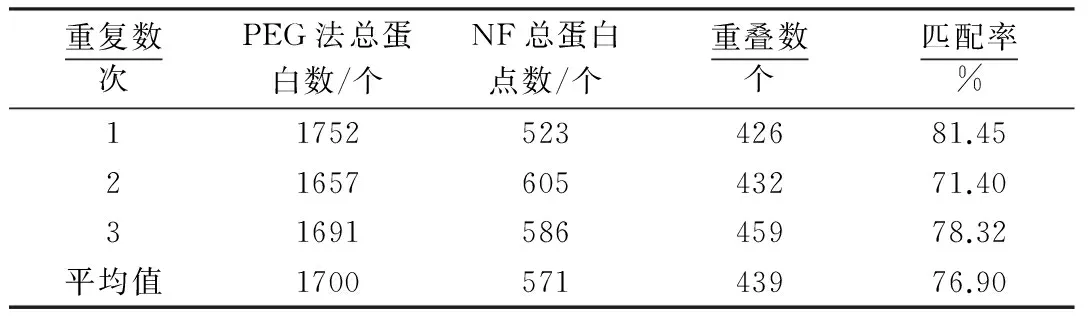
表2 5个组分的总蛋白点(F1-F5)与全蛋白点(NF)数量分析Table 2 The number of protein points of 5 components between F1-F5 and NF

表3 MS质谱鉴定结果Table 3 Differential protein spots identified using MS
2.4 生物信息学分析
在车间的一角,有个硕大的池子,里面装满红褐色的液体,闻那气味估计是一种化工原料,木材加工厂老板收购来的一部分质地坚硬的木材,都被加工成木方,浸泡在里面。刚开始贾鹏飞不懂这是做什么的,后来听工友介绍说浸泡的木材,经过高温蒸干定型,再拿去打造高档的红木家具,由于材质细密,内外木质泛红,与真的红木就没有什么差别了。想想商场几万元一张的餐桌,这“红木家具”真是暴利。
通过验证低丰度蛋白对于植物个体生长发育的作用,对质谱鉴定成功的5种蛋白质进行生物信息学分析,进一步探讨PEG法对于研究植物生长发育中起重要作用的低丰度蛋白的重要性,表明PEG法对于蛋白组研究的重要性.
2.4.1 差异蛋白的GO功能分类 甘油醛-3-磷酸脱氢酶通过GENE ONTOLOGY数据库的比对分析,得到的GO功能分类包括:分子功能中有甘油醛-3-磷酸脱氢酶(NAD+)(磷酸化)活性(GO0004365)催化D-甘油醛-3-磷酸形成3-磷酸-D-甘油酰基磷酸和NADH,NAD结合(GO0051287)与烟酰胺腺嘌呤二核苷酸形成非共价结合,可以是氧化形式(NAD+)也可以是还原形式(NADH+),NADP结合(GO0050661)与NAD结合类似可以是氧化形式(NADP+)也可以是还原形式(NADPH);生物过程中有葡糖糖代谢过程(GO0006006)和糖酵解过程(GO0006096);细胞组成中有细胞质(GO0005737).
ATP合成酶CF1β亚基通过GENE ONTOLOGY数据库的比对分析,得到的GO功能分类包括:分子功能中有ATP结合(GO0005524)与ATP及腺苷-5-磷酸形成非共价键是重要的辅酶和酶活调节剂,质子转运ATP合成酶活性及旋转机制(GO00046933)通过旋转机制跨膜转运质子;生物过程有水解耦合质子转运(GO0015991)通过ATP的水解逆电化学梯度转运质子,合成耦合质子转运(GO0015986)跨膜转运质子形成电化学梯度形成ATP;细胞组成中有叶绿体类囊体膜(GO0009535)及质子转运ATP合成酶复合物催化中心F1(GO0045261).
磷酸丙糖异构酶通过GENE ONTOLOGY数据库的比对分析,得到的GO功能分类包括:分子功能中有磷酸丙糖异构酶活性(GO0004807)催化甘油-3-磷酸脱氢酶形成磷酸甘油酮;生物过程中有糖异生(GO0006094)由丙酮酸、氨基酸、甘油等非糖类前体形成葡萄糖,糖酵解(GO0006096)将葡萄糖分解为丙酮酸,且将NAD还原成NADH生成少量的ATP,磷酸戊糖支路(GO0006098)将葡萄糖-6-磷酸分解生成NADPH及核糖-5-磷酸并释放CO2;细胞组成中有细胞质(GO0005737).
假定蛋白OsI_20531和假定蛋白TRIUR3_08890在KEGG数据库及GO功能分类中未找到同源分类.
2.4.2 差异蛋白的信号通路分析 磷酸丙糖异构酶通过KEGG数据库比对后,得到参与的生物通路包括:糖异生和糖酵解途径(map00010).
甘油醛-3-磷酸脱氢酶通过KEGG数据库的比对分析,得到参与的生物通路有:糖酵解途径(map00010)中的第一步亚模块(M00001)将D-甘油醛3-磷酸合成甘油醛,是碳水化合物降解过程中的关键作用酶.
3 讨论与结论
3.1 讨论
优良的蛋白制备是蛋白质组学中2-DE的关键,由于个体、器官、组织的化学成分组成不同造成了植物蛋白的提取方法也是多种多样,研究者通常需要根据研究材料和设备的不同做出针对性的蛋白提取方案[10,31,32].同时,植物体内蛋白的丰度变化较大,达到106,由于现阶段技术设备的局限,2-DE可检测的丰度在104以下[22,33].本研究通过对花吊丝竹叶片全蛋白2种提取方法的2-DE差异比较,更深层次地研究竹类蛋白组学.从2-DE结果(图2)中可以看出,TCA丙酮/酚提结合法得到的蛋白明显少于PEG法,尤其是F4中低丰度的蛋白显现较多,但是TCA丙酮/酚提结合法有较高的净化能力,极大地降低了蛋白间的互作[34],表现的图谱背景干扰较低.同时,PEG法中的5个组(F1-F5)中的总蛋白数超过了1 700,是TCA丙酮/酚提结合法(NF)的3倍,重复3次的试验结果基本类似.图2 F3中利用16%的PEG成功地富集了单一的高丰度组分,本试验得到的结果与在拟南芥及其他植物的蛋白PEG试验类似[35],表明利用PEG提取植物不同丰度蛋白是切实可行的.TCA丙酮/酚提结合法改善了传统TCA方法耗时及低pH制备的弊端,同时用甲醇和80%丙酮清除残余的TCA和污染物,减少制备时间明显提高了蛋白质的提取质量,而且采用甲醇清洗沉淀能够清除植物组织中的多酚物质.酚/SDS混合溶液能够提取干扰物质较多的植物样本[5],可以将酚类及其他干扰物质与提取的蛋白共沉淀至离心管中.而在PEG法中,通过去除总蛋白中的高丰度蛋白(如Rubisco),各组分的蛋白质点数增加.5个组分的蛋白显著提高了IPG胶条的样品吸收量,从而显著提高2-DE的分辨率.高丰度蛋白(如Rubisco)是高拷贝基因产物,广泛存在于植物中,尤其是光合组织中,所以PEG法可以延伸至与分子生物学相关联的应用研究中.但是,PEG法提取的蛋白无法与NF完全匹配,首先可能是因为2种提取方法的操作步骤过于繁琐,而且提取的原理并不相同,造成不同的蛋白损失;其次可能是因为不同的蛋白丰度造成IEF时间和效果的不同;最后因为5个组分的同时聚焦,采用相同的参数可能会对各个组分造成一定影响.
甘油醛-3-磷酸脱氢酶是高等植物糖异生及糖酵解途径中重要的酶,同时由于亚细胞定位的不同,甘油醛-3-磷酸脱氢酶的类型也不同,在拟南芥、烟草(Nicotianatabacum)中均发现同源性较高的甘油醛-3-磷酸脱氢酶类型[36,37,38].拟南芥的研究表明,在甘油醛-3-磷酸脱氢酶缺失的突变体中,糖酵解途径和三羧酸循环发生了改变,影响了细胞内能量的变化,进而影响了植物的呼吸作用及个体的正常发育.此外,在植物响应各种非生物胁迫时会产生氧化物质,而甘油醛-3-磷酸脱氢酶是植物抗氧化胁迫及响应氧化还原信号重要的转导因子[37,39,40].ATP合成酶CF1β亚基是ATP合成的重要酶,参与细胞内光合磷酸化过程,研究表明,在应对镉胁迫时,紫苏(Perillafrutescens)[41]、秋茄(Kandeliacandel)[42]、水稻(Oryzasativa)[43]中CF1β亚基的含量显著上升,ATP的合成能力同时增强,加强了植物体对镉的抗性,同样也是植物体应对非生物胁迫及生长中的重要蛋白质.磷酸丙糖异构酶是植物体糖酵解过程中关键的酶,在多种植物组织中均有发现,能够催化D-甘油醛-3-磷酸与二羟丙酮磷酸之间发生可逆转化[44].在不同蛋白质提取方法中,TCA丙酮/酚提结合法不能较好地鉴定出3种蛋白质,而在PEG中显现较为明显,说明利用PEG法可以鉴定出对植物体生长作用极其重要的蛋白质,对花吊丝竹深层次的蛋白组学研究提供依据.
3.2 结论
通过TCA丙酮/酚提结合法和PEG法的2-DE比较分析表明,传统的TCA丙酮/酚提结合法无法检测出花吊丝竹叶片中的低丰度蛋白质,而PEG法通过不同浓度的PEG将花吊丝竹叶片全蛋白分为5个组,其中16%的PEG(F3)能够单一地富集较多的高丰度蛋白,提高了其他组的蛋白检测率(F4、F5),从而显著提高了2-DE的分辨率.试验从F3、F4及F5中鉴定出5个差异蛋白:1号(甘油醛-3-磷酸脱氢酶)、2号(ATP合成酶CF1β亚基)、3号(假定蛋白OsI_20531)、4号(假定蛋白TRIUR3_08890)、5号(磷酸丙糖异构酶).通过生物信息学分析发现,其中1、2、5号蛋白质对植物体的能量代谢、生物通路及生长发育有重要作用.综上,利用PEG法能够有效地提高花吊丝竹叶片蛋白的提取率及2-DE的分辨率,能够检测到更多的影响生物体生长发育的低丰度蛋白,是一种可以拓展到其他植物体蛋白质组研究中的试验方法.
[1] ALAM I, SHARMIN S A, KIM K H, et al. An improved plant leaf protein extraction method for high resolution two-dimensional polyacrylamide gel electrophoresis and comparative proteomics[J]. Biotechnic & Histochemistry, 2013,88(2):61-75.
[2] ANGELIKA G, CHRISTIAN O, GUNTHER B, et al. The current state of two-dimensional electrophoresis with immobilized pH gradients[J]. Electrophoresis, 2000,21(6):1 037-1 053.
[3] SHAW M M, RIEDERER B M. Sample preparation for two-dimensional gel electrophoresis[J]. Proteomics, 2003,3(8):1 408-1 417.
[4] RABILLOUD T, LELONG C. Two-dimensional gel electrophoresis in proteomics: a tutorial[J]. Proteomics, 2011,74(10):1 829-1 841.
[5] WANG W, SCALI M, VIGNANI R, et al. Protein extraction for two dimensional electrophoresis from olive leaf, a plant tissue containing high levels of interfering compounds[J]. Electrophoresis, 2003,24(14):2 369-2 375.
[6] ISAACSON T, DAMASCENO C M B, SARAVANAN R S, et al. Sample extraction techniques for enhanced proteomic analysis of plant tissues[J]. Nature Protocol, 2006,1(2):769-774.
[7] MONIKA B, DANA H, LUKAF, et al. The physiology and proteomics of drought tolerance in maize: early stomatal closure as a cause of lower tolerance to short-term dehydration?[J]. Plos One, 2012,7(6):e38017. doi:10.1371/journal.pone.0038017.
[8] PLOMION C, LALANNE C. Protein extraction from woody plants[J]. Methods in Molecular Biology, 2007,355:37-41.
[9] WU X L, XIONG E H, WANG W, et al. Universal sample preparation method integrating trichloroacetic acid/acetone precipitation with phenol extraction for crop proteomic analysis[J]. Nature Protocols, 2014,9(2):362-374.
[10] CARPENTIER S C, WITTERS E, LAUKENS K, et al. Preparation of protein extracts from recalcitrant plant tissues: an evaluation of different methods for two-dimensional gel electrophoresis analysis[J]. Proteomics, 2005,5(10):2 497-2 507.
[11] DELPHINE V, WHEATLEY M D, CRAMER G R. Optimization of protein extraction and solubilization for mature grape berry clusters[J]. Lectrophoresis, 2006,27(9):1 853-1 865.
[12] YAO Y, YANG Y W, LIU J Y. An efficient protein preparation for proteomic analysis of developing cotton fibers by 2-DE[J]. Electrophoresis, 2006,27(22):4 559-4 569.
[13] WANG W, VIGNANI R, SCALI M, et al. A universal and rapid protocol for protein extraction from recalcitrant plant tissues for proteomic analysis[J]. Electrophoresis, 2006,27(13):2 782-2 786.
[14] CORTHALS G L, WASINGER V C, HOCHSTRASSER D F, et al. The dynamic range of protein expression: a challenge for proteomic research[J]. Electrophoresis, 2000,21(6):1 104-1 115.
[15] XI J H, WANG X, LI S Y, et al. Polyethylene glycol fractionation improved detection of low-abundant proteins by two-dimensional electrophoresis analysis of plant proteome[J]. Phytochemistry, 2006,67(21):2 341-2 348.
[16] KOMATSU S, AHSAN N. Soybean proteomics and its application to functional analysis[J]. Journal of Proteomics, 2009,72(3):325-336.
[17] SARMA A D, OEHRLE N W, EMERICH D W. Plant protein isolation and stabilization for enhanced resolution of two-dimensional polyacrylamide gel electrophoresis[J]. Analytical Biochemistry, 2008,379(2):192-195.
[18] ATHA D H, INGHAM K C. Mechanism of precipitation of proteins by polyethylene glycols. analysis in terms of excluded volume[J]. Journal of Biological Chemistry, 1981,256(23):12 108-12 117.
[19] ARYAL U K, KROCHKO J E, ROSS A R. Identification of phosphoproteins inArabidopsisthalianaleaves using polyethylene glycol fractionation,immobilized metal-ion affinity chromatography, two-dimensional gel electrophoresis and mass spectrometry[J]. Journal of Proteome Research, 2011,11(1):425-437.
[20] AHSAN N, LEE D G, LEE S H, et al. A comparative proteomic analysis of tomato leaves in response to waterlogging stress[J]. Physiologia Plantarum, 2007,131(4):555-570.
[21] WALLIWALAGEDARA C, KEULEN H V, CUTRIGHT T, et al. Comparison of sample preparation methods for the resolution of metal-regulated proteins in helianthus annuus by 2-dimensional gel electrophoresis[J]. Open Proteomics Journal, 2010(3):20-25.
[22] ANGELIKA G, WALTER W, Michael J D. Current two-dimensional electrophoresis technology for proteomics[J]. Proteomics, 2004,4(12):3 665-3 685.
[23] 耿伯介.中国植物志第9卷第1分册[M].北京:科学出版社,1996.
[24] KIM S T, YU S, KIM S G, et al. Proteome analysis of rice blast fungus (Magnaporthegrisea) proteome during appressorium formation[J]. Proteomics, 2004,4(11):3 579-3 587.
[25] BRADFORD M M. A rapid and sensitive for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding[J]. Analytical Biochemistry, 1976,25(1):248-256.
[26] NEUHOFF V, AROLD N, TAUBE D, et al. Improved staining of proteins in polyacrylamide gels including isoelectric focusing gels with clear background at nanogram sensitivity using Coomassie Brilliant Blue G-250 and R-250[J]. Electrophoresis, 1988,9(6):255-262.
[27] Yan J X, WAIT R, BERKELMAN T, et al. A modified silver staining protocol for visualization of proteins compatible with matrix-assisted laser desorption/ionization and electrospray ionization-mass spectrometry[J]. Electrophoresis, 2000,21(17):3 666-3 672.
[28] 王旭.PEG分级沉淀方法及其在拟南芥低温胁迫蛋白组学研究中的应用[D].吉林:吉林大学,2007.
[29] ALAM I, LEE D G, KIM K H, et al. Proteome analysis of soybean roots under waterlogging stress at an early vegetative stage[J]. Journal of Biosciences, 2010,35(1):49-62.
[30] HUGHES S M, RAWSON P M, JOLLY R D, et al. Submitochondrial distribution and delayed proteolysis of subunit c of the H+-transporting ATP-synthase in ovine ceroid-lipofuscinosis[J]. Electrophoresis, 2001,22(9):1 785-1 794.
[31] ALAM M, GHOSH W. Optimization of a phenol extraction-based protein preparation method amenable to downstream 2-DE and MALDI-MS based analysis of bacterial proteomes[J]. Proteomics, 2014,14(2-3):216-221.
[32] WANG W, TAI F J, CHEN S N. Optimizing protein extraction from plant tissues for enhanced proteomics analysis[J]. Journal of Separation Science, 2008,31(11):2 032-2 039.
[33] RABILLOUD T. Two-dimensional gel electrophoresis in proteomics: old, old fashioned, but it still climbs up the mountains[J]. Proteomics, 2002,2(1):3-10.
[34] SARAVANAN R S, ROSE J K C. A critical evaluation of sample extraction techniques for enhanced proteomic analysis of recalcitrant plant tissues[J]. Proteomics, 2004,4(9):2 522-2 532.
[35] KIM S T, CHO K S, JANG Y S, et al. Two-dimensional electrophoretic analysis of rice proteins by polyethylene glycol fractionation for protein arrays[J]. Electrophoresis, 2001,22(10):2 103-2 109.
[36] BEDHOMME M, ADAMO M, MARCHAND H C, et al. Glutathionylation of cytosolic glyceraldehyde-3-phosphate dehydrogenase from the model plantArabidopsisthalianais reversed by both glutaredoxins and thioredoxinsinvitro[J]. Biochemi J, 2012,445(3):337-347.
[37] RIUS P S, CASATI P, IGLESIAS A A, et al. Characterization ofArabidopsislines deficient in GAPC-1, a cytosolic NAD-Dependent glyceraldehyde-3-phosphate dehydrogenase[J]. Plant Physiology, 2008,148(3):1 655-1 667.
[38] WAWER I, BUCHOLC M, ASTIER J, et al. Regulation ofNicotianatabacumosmotic stress-activated protein kinase and its cellular partner GAPDH by nitric oxide in response to salinity[J]. Biochemical Journal, 2010,429(1):73-83.
[39] 卢倩,弭晓菊,崔继哲.植物甘油醛-3-磷酸脱氢酶作用机制的研究进展[J].生物技术通报,2013,8:1-6.
[40] ZHANG X H, RAO X L, SHI H T, et al. Overexpression of a cytosolic glyceraldehyde-3-phosphate dehydrogenase gene OsGAPC3 confers salt tolerance in rice[J]. Plant Cell Tissue Organ Culture, 2011,107(1):1-11.
[41] 谢慧玲,刘杰,陈珊,等.紫苏叶片响应镉胁迫的蛋白质差异表达分析[J].中国生态农业学报,2014,22(10):1 207-1 213.
[42] 翁兆霞.镉胁迫下秋茄(Kandeliacandel)根系蛋白质组变化及差异表达蛋白功能研究[D].福州:福建农林大学,2011.
[43] 张国君.水稻钙调蛋白对镉胁迫的分子响应[D].福州:福建农林大学,2013.
[44] 刘江红,石毅,周筠梅.磷酸丙糖异构酶的折叠及稳定性研究[J].生物化学与生物物理进展,2006,33(5):465-472.
(责任编辑:吴显达)
Comparison of two protein extraction protocol ofDendrocalamusminorvar.amoenusleaves with two-dimensional electrophoresis
SHEN Shaoyan1,3, WU Yuxiang1,3, RONG Jundong2,3, HE Tianyou1,3, ZHENG Yushan1,2,3
(1.College of Landscape, Fujian Agriculture and Forestry University; 2.College of Forestry, Fujian Agriculture and Forestry University; 3.Institute of Bamboo, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China)
Two years old cutting seedlings ofDendrocalamusminorvar.amoenuswere used to analyze protein spots and matching rates. The proteins extracted by TCA/phenol method and PEG methods were separated using 2-DE technology. The result showed that the differences of protein bands between the 5 fractions generated by the PEG method were significant in the SDS-PAGE analysis and 2 highly abundant proteins bands enriched in F3, indicating an effectively separated total proteins. The homogeneity analysis of the 2-DE images by the 2 methods revealed that a highly abundant protein extracted by PEG method was enriched in fraction F3, which significantly increased the detection rate and resolution of 2-DE protein spots. In PEG method, the number of protein detected in F1-F5 was more than 1 700, but a certain degree of overlapping occurred between adjacent fractions. However, TCA/phenol method could only detect about 571 protein spots, and on average 439 protein spots were matched between the 5 fractions and NF, resulting in a matching rate of more than 77.9%. Five different protein spots between PEG method and TCA acetone/phenol were identified by MALDI-TOF MS. Referring to database matching and bioinformatic analysis, these 5 proteins turned out to be essential coenzyme for plant involved in glycolysis, gluconeogenesis and ATP synthesis. To summarize, traditional TCA acetone/phenol method was unable to detect low-abundant proteins inDendrocalamusminorvar.amoenusleaves. However, the total proteins could be separated into 5 fractions by PEG method and 16% of proteins (F3) could be enriched in a single fraction of high-abundant proteins, which resulted in higher detection rate and significantly improved resolution of 2-DE. More low-abundant proteins that affect growth and development of organism can be detected by mass spectrometry. The PEG method is a practical and ideal proteomics research method for bamboos or other plants.
Dendrocalamusminorvar.amoenus; TCA/phenol method; PEG methods; 2-DE; bioinformations
2016-06-21
2016-07-31
福建省科技重大专项资助(2013NZ0001).
沈少炎(1992-),男,硕士研究生.研究方向:竹类蛋白组研究与园林植物.Email:1075455791@qq.com.通讯作者郑郁善(1960-),男,教授,博士生导师.研究方向:森林培育与园林植物.Email:zys1960@163.com.
S718.43
A
1671-5470(2017)01-0050-08
10.13323/j.cnki.j.fafu(nat.sci.).2017.01.009

