长链非编码RNA 在肌肉生成与脂肪发育过程中的研究进展
张思欢,朱熙春,许 晗,党瑞华,黄永震,雷初朝,陈 宏,蓝贤勇
(西北农林科技大学动物科技学院,陕西 杨凌 712100)
基因组DNA经过转录可以产生两大类RNA,一类是可以被翻译成蛋白质的编码RNA,另一类是不具有或具有较低编码能力的非编码RNA(noncoding RNA,ncRNA)。2012年发布的ENCODE研究数据显示,75 %的人类基因组序列能够转录形成RNA, 其中约26 %的转录产物为蛋白编码RNA,其余大部分均为非编码RNA[1]。ncRNA包括普遍存在的转运RNA(tRNA)、核糖体RNA(rRNA),以及具有组织表达特异性和时间表达特异性的siRNA(small interfering RNA)、miRNAs(microRNA)、circRNA(circle RNA)、lncRNA(long nocoding RNA)等[2]。近年来,长链非编码RNA(lncRNA)受到了越来越多的关注,其多样的功能机制正逐渐被人们所揭示。
肌肉和脂肪是组成机体的重要组织,二者均可由间充质干细胞分化而来,其分化与增值水平与生物体的生长发育密切相关[3,4]。肌肉和脂肪的发育会影响家畜的生长速度,产肉量以及肉质等,同时在人上还会导致肥胖等疾病。研究肌肉和脂肪的发育机制对于以上问题的深入理解具有重要意义。因此,本文将对动物lncRNA的基本特征及功能机制进行介绍,综述lncRNA对肌肉和脂肪组织发育的调控机制,并对lncRNA未来的研究进行展望,为研究者在lncRNA方面的研究提供有力的参考。
1 长链非编码RNA的基本特征及功能机制
lncRNA是指长度大于200个核苷酸,具有调控功能的非编码RNA。但最新研究发现,一些被认为不具有蛋白编码能力的RNA也可以被翻译成小肽,进而发挥其调控功能[5,6,7]。20世纪末,第一个调控性的lncRNA-Xist被发现,从此揭开了lncRNA的面纱[8]。大多数的lncRNA都具有明显的时空表达特异性[9,10],并且在不同的生物过程中,还会形成不同的转录本,从而动态的调控生物学过程;相对于蛋白编码基因在物种间保守的特征,lncRNAs在物种间的序列保守性较低[11,12,13,14]。lncRNA可以直接与编码蛋白的基因或其它种类的ncRNA(例如miRNA等)结合,直接或间接调控功能靶基因的表达[15,16]。同时,一些lncRNA可以形成复杂的二级结构,为许多蛋白分子提供结合位点,通过与转录因子、染色质重构复合物等蛋白结合,调控相关基因的表达[17,18]。根据lncRNA在不同层面对相关基因的调控作用,本文将lncRNA的作用机制大致分为转录水平调控、转录后水平调控及表观遗传学水平调控三类进行介绍。
1.1 转录水平的调控作用
LncRNA可以通过多种方式从转录水平调控邻近基因及非邻近基因的表达水平[19]。(1) 调控邻近功能基因的转录。当lncRNA位于蛋白编码基因的上游临近区域时,lncRNA的转录可能影响转录因子与下游蛋白编码基因启动子区的结合,例如酵母中lncRNA SRG1的转录延长,占据了下游SER3启动子区,阻碍了转录因子与SER3启动子的结合,导致SER3不能正常转录[20,21];当lncRNA位于蛋白编码基因启动子区时,在其转录延长的过程中,可以诱导组蛋白的修饰作用,进而阻止下游蛋白编码基因的转录,例如酵母的GAL1-10基因簇[22]。(2) 调控非邻近基因的转录水平。有一些lncRNA可以直接与功能基因启动子区序列、Pol II或者转录因子结合,阻止Pol II或其它转录因子与功能基因启动子区结合[23]。例如由CDKN1A 启动子区转录而来的lncRNA PANDA 可以和转录因子NF-YA互作,限制促凋亡基因的表达[24]。
1.2 转录后水平的调控作用
转录后水平上的调控主要分为调控mRNA前体的选择性剪接、控制mRNA输出细胞核以及一些通过miRNA的调控作用等[25,26]。(1) lncRNA调控选择性剪接。Serine/arginine(SR)丝氨酸-精氨酸剪接因子是真核生物中组织细胞内特异的剪接因子,其发挥功能依赖于自身浓度和磷酸化水平。LncRNA MALAT1可以调控SR在细胞核中的分布以及SR的磷酸化水平,进而调控细胞内的选择性剪接[27]。(2)lncRNA调控蛋白的亚细胞定位。LncRNA NRON可以调节核质交换,从而抑制NFAT转录因子在细胞核中的积累,进而调控NFAT所调控的基因的转录水平[28]。(3)LncRNA与miRNA的调控关系。LncRNA可以作为竞争性内源RNA(ceRNA),通过与靶基因竞争性的结合miRNA,降低miRNA对靶基因的抑制作用。例如lncRNA H19[29,30];同时,H19的第一外显子可以转录形成两个保守的miRNAs:miR-675-3p以及miR-675-5p,它们可以直接靶向Smad1, Smad5和DNA复制起始因子Cdc6,从而发挥调控作用[31,32];除此之外,lncRNA还可以直接与靶基因结合,从而阻止miRNA对靶基因的抑制作用。例如从lncRNA Sirt1 AS,它可以通过与Sirt1 mRNA结合,从而解除miRNA对Sirt1 mRNA的抑制作用,抑制肌细胞的分化(图1a)[33]。
1.3 表观遗传学水平的调控作用
从表观遗传学调控层面上,lncRNA参与印记基因沉默,X染色体失活等重要的生物学过程。第一个被发现的lncRNA Xist在X染色体失活的过程中发挥了重要的作用。由X染色体基因转录产生的Xist覆盖在X染色体上,使染色体大量组蛋白被甲基化,从而导致X染色体失活[34,35]。另外,lncRNA还可以通过招募表观激活因子,调控基因的表达。例如lncRNA Mira在基因组上的位置位于Hoxa6和Hoxa7中间,lncRNA Mira可以招募染色体修饰的表观激活因子MLL1到Mira基因的位置,进而促进Hoxa6和Hoxa7基因的表达[36]。LncRNA作为具有调控功能的非编码RNA,除了具有上述的作用机制外,还有很多其它的作用方式有待我们进一步的挖掘。
2 lncRNA调控肌肉与脂肪的发育
lncRNA可以通过多种作用机制在生物体内发挥了各种各样的生物学功能,例如影响癌细胞的发生、干细胞分化多潜能性的维持以及个体肌肉脂肪的发育等[33,37,38,39]。肌肉和脂肪的发育水平是影响家畜的生长速度、产肉量、肉质等重要经济性状的重要因素,同时,还与肌肉萎缩,过度肥胖等疾病密切相关。因此,阐明lncRNA调控肌肉和脂肪发育的机制对于畜牧业的发展以及肌肉脂肪发育相关疾病的治疗具有重要的意义。
2.1 lncRNA调控肌肉发育
目前已经发现一些lncRNA对于肌肉的发育具有重要作用,例如linc-MD1,lnc-mg,Linc-RAM等。linc-MD1是被发现的第一个具有肌肉组织特异表达的lncRNA,它含有1个miR-133和2个miRNA-135的结合位点,这两个miRNAs的靶基因分别是MAML-1和MEF2C,它们是激活肌细胞分化晚期相关基因表达的重要转录因子[40]。在肌细胞分化时期,linc-MD1 可以竞争性的结合这两个miRNA,解除他们对靶基因的抑制作用,促进肌细胞的分化(图1b)[41]。lnc-mg是一个在骨骼肌中特异性高表达的lncRNA,在肌细胞分化和骨骼肌发育过程中具有重要的调控作用。lnc-mg 可以作为ceRNA与IGF2竞争性的结合miR-125b,从而影响IGF2的表达丰度。在小鼠中,在骨骼肌中特异性的敲除lnc-mg,将导致肌肉萎缩,而过表达lnc-mg将导致肌肉肥大。体外试验分析发现,在原代骨骼肌分化的过程中,lnc-mg的表达水平逐渐上升,过表达lnc-mg将促进肌细胞的分化[42]。Linc-RAM在肌肉生成的过程中也发挥重要作用。体内研究发现,Linc-RAM敲除小鼠由于肌卫星细胞分化缺陷导致肌肉再生受损。同时Linc-RAM可以直接靶向于MyoD,促进MyoD-Baf60c-Brg1复合物在调控因子上的组装,进而调控肌肉发育相关靶基因的表达(图1c)[43]。
除此之外,转录组测序技术逐渐被应用于肌肉发育过程中重要的lncRNAs的挖掘和深入研究。2016年Sun等利用Ribo-Zero RNA-Seq的方法对秦川牛胎牛、犊牛、成年牛的骨骼肌进行测序,发现了一批在不同时期牛肌肉组织中差异表达的lncRNAs。其中,lncRNA lncMD在肌细胞分化的过程中表达水平逐渐上升,过表达lncMD促进肌细胞的分化,抑制 lncMD的表达导致肌细胞分化相关标志基因MHC和MyoG的表达量下降。进一步分析发现,lncMD可以通过吸附miR-125b来调控IGF2基因的表达,进而影响肌肉的发育(图1d)[44]。近期,有研究对牛背最长肌、肩胛肌、臀肌以及肋间肌中的lncRNA进行测序分析,共鉴定得到7188个lncRNAs。分析结果显示,牛骨骼肌中的lncRNAs的特征在很多方面与已知的其它哺乳动物中的lncRNAs类似,例如表达水平比mRNA表达水平低等。同时,作者对部分的lncRNAs进行了lncRNAs-miRNAs-mRNAs的互作分析,为研究骨骼肌的发育提供了重要的研究方向[45]。
除以上的例子外,还有其它的lncRNAs被发现具有调控肌肉发育的作用。例如Yam-1等[32,46,47]。尽管目前已经发现一些lncRNAs与肌肉的发育密切相关,但该方面的研究仍然较少,因此更多调控肌肉发育的lncRNAs急需被挖掘。
2.2 lncRNA调控脂肪发育
脂肪生成是一个受多种因子共同调控的复杂而精细的过程,近年来,随着ncRNA研究的快速发展,调控脂肪生成的lncRNAs逐渐被揭示出来[48]。SRA(steroid receptor RNA activator)是被发现最早的对脂肪代谢具有重要调控功能的lncRNA。脂肪中大量的SRA能与PPARγ结合并增强PPARγ活性,在ST2间质前体细胞中过表达SRA促进细胞向脂肪细胞分化,敲低3T3-L1前体脂肪细胞中的内源性SRA将抑制细胞的分化[49]。同时,高脂日粮诱导的肥胖小鼠白色脂肪组织中SRA的表达升高,而SRA基因敲除小鼠可以抵制高脂饮食引起的肥胖[50]。PU.1的反义转录本PU.1 AS lncRNA(antisense lncRNA),转录自功能基因PU.1的反义链,它能够直接结合到PU.1的mRNA上,形成mRNA/lncRNA复合体,对PU.1的翻译起到阻遏作用,调控脂联素等脂肪因子的释放,进而调控脂肪细胞分化和脂肪沉积过程[51,52]。沉默3T3-L1前体脂肪细胞中PU.1 AS lncRNA的表达时,细胞中PU.1蛋白表达增强,抑制分化标志性基因PPARγ和CEBPα的表达,从而抑制生脂过程[51]。
2015年Xiao等利用RNA芯片筛选在人骨髓间充质干细胞(hMSCs)向脂肪细胞诱导分化过程中差异表达的lncRNAs时,发现一个对脂肪生成具有调控作用的lncRNA-Adipogenic differentiation induced noncoding RNA(ADINR)[53]。ADINR是从C/EBPα基因上游450 bp处转录产生的lncRNA,在脂肪细胞分化的过程中,ADINR能与PA1特异结合,募集MLL3/4组蛋白甲基化转移酶复合体,从而提高C/EBPα位点的H3K4me3甲基化水平,促进C/EBPα表达,增强脂肪生成。沉默ADINR后,脂肪细胞分化相关标记基因表达下降,脂滴沉积减少,成脂分化受到抑制[53]。2016年Li等对秦川牛分化前后的脂肪细胞进行测序,筛选出16个在分化前后差异表达的lncRNAs,其中lncRNR ADNCR在分化前表达量显著高于分化后。试验验证发现,ADNCR对脂肪的分化具有抑制作用,它可以与SIRT1竞争性的结合miR-204,从而调控脂肪细胞的分化[54]。
以上对肌肉和脂肪发育过程中具有调控功能的关键lncRNA分别进行了综述。然而,肌肉和脂肪细胞均来源于间充质干细胞,并且在生物体发育过程中二者又密切相关。目前已经发现一些功能基因以及lncRNA同时对肌肉和脂肪的生成具有调控作用,例如Sirt1和Klf5基因,以及lncRNA HOTAIR[33,55,56,57]。HOTAIR可以通过影响组蛋白修饰复合物的位置调控组蛋白的甲基化状态,从而调控肌肉发育过程[58]。同时,HOTAIR参与人皮下脂肪组织前体脂肪细胞的分化过程,在腹部前体脂肪细胞中表达HOTAIR,能促进脂肪细胞的分化[59]。
综上所述,lncRNA可以通过多种机制调控生物学过程,但目前关于lncRNA调控肌肉和脂肪细胞增殖、分化的报道特别少仍然较少。因此,需要有更多的研究关注lncRNA调控肌肉与脂肪发育的机制,为lncRNA在畜牧业和疾病治疗中的应用奠定基础。
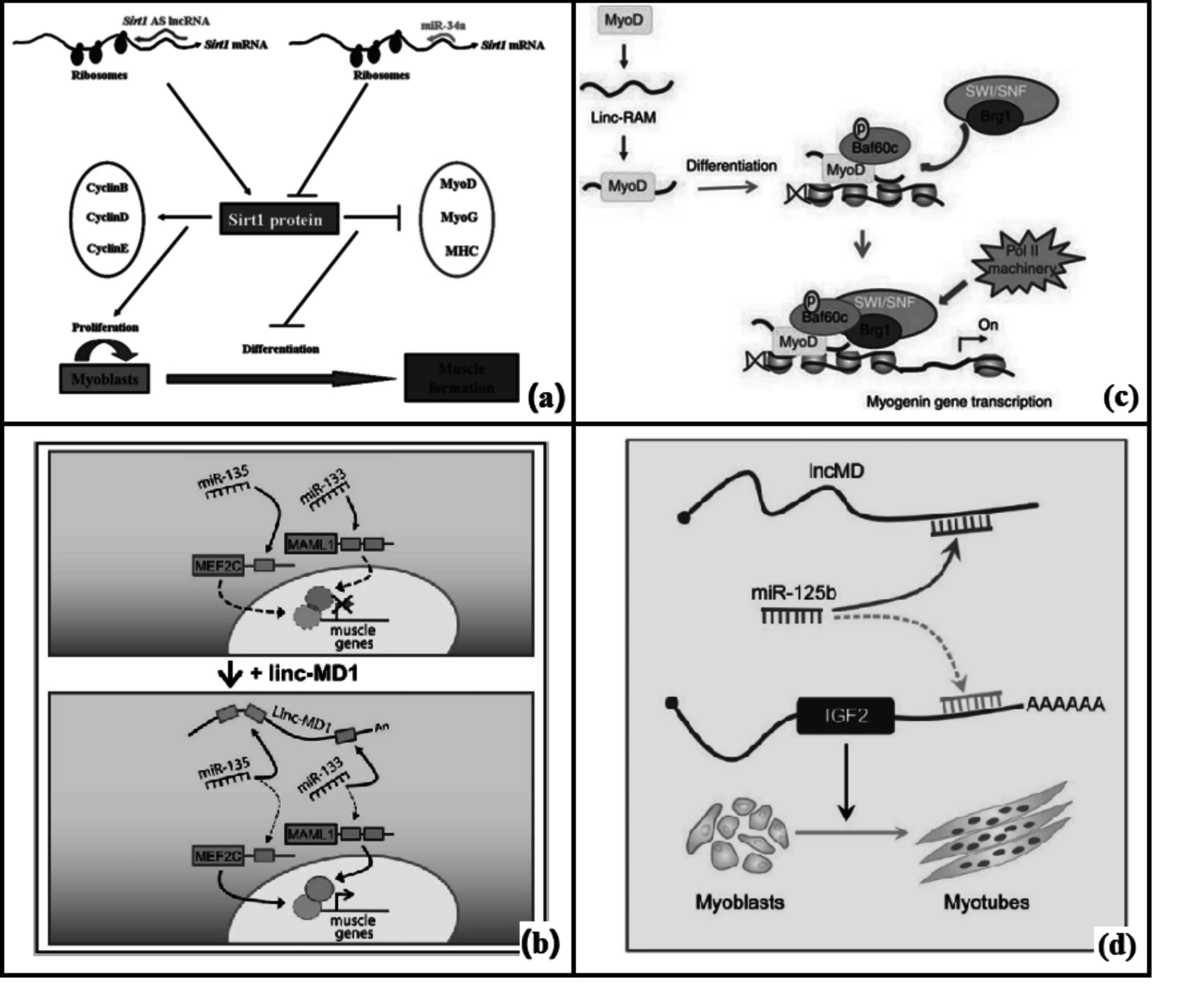
图1 lncRNA调控基因表达的分子机制注:(a) Sirt1 AS lncRNA结合与 miR34a竞争结合Sirt1 mRNA调控肌肉形成 (Wang et al.2016);(b) linc-MD1调控肌细胞分化 (Shen et al.2006);(c) Linc-RAM发挥功能的可能的模型 (Yu et al.2017); (d) lncMD通过调控miR-125b控制Igf2表达水平的分子机制 (Sun et al.2016)。
4 展望
关于lncRNA的研究是自2012年以来生命科学的前沿之前沿,进展迅速,但是目前人们对lncRNA的认识仍然只是冰山一角。随着新一代高通量测序技术应用的普及,生物信息学分析的发展,lncRNA数据库的完善,大量lncRNAs在各类生物体中不断被发现,极大的吸引着人们对其生物学功能及其调控机理的探寻。2013年,Weikard等利用RNA-Seq技术,发现牛皮中存在4848个潜在的lncRNA[14]。2013年,Sun等鉴定了脂肪生成过程中全基因组范围内的lncRNAs的表达特征和功能,通过比较棕色、白色前体脂肪细胞与成熟脂肪细胞的转录组表达,发现了175个特异性调节的lncRNAs[60]。2015年,Alvarez-Dominguez等利用RNA-seq技术,鉴定出127个棕色脂肪组织相关的lncRNAs[61]。所有这些研究,将为相关疾病的治疗,遗传资源的开发,畜牧业生产效益的提高奠定基础。但相关研究才刚起步,更多在肌肉和脂肪发育过程中的关键lncRNA急需被挖掘与研究,对于lncRNA功能的研究还有很长的路要走。
[1] 杨峰, 易凡, 曹慧青, 等.长链非编码RNA研究进展[J].遗传, 2014, 2014(5): 456-468.
[2] 周凤燕, 杨青, 朱熙春, 等.环状RNA的分子特征、作用机制及生物学功能[J].农业生物技术学报, 2017, 25(3): 485-501.
[3] 方洪松, 周建林, 彭昊, 等.不同来源间充质干细胞生物学特性差异[J].中国组织工程研究, 2015, 19(32):5243-5248.doi:10.3969/j.issn.2095-4344.2015.32.029.
[4] Jeong BC, Kang IH, Hwang YC,etal.MicroRNA-194 reciprocally stimulates osteogenesis and inhibits adipogenesis via regulating COUP-TFII expression[J].Cell Death Dis, 2014, 20;5:e1532.doi: 10.1038/cddis.2014.485.
[5] Anderson DM, Anderson KM, Chang CL,etal.A micropeptide encoded by a putative long noncoding RNA regulates muscle performance[J].Cell, 2015, 160(4):595-606.
[6] Cai B, Li Z, Ma M,etal.LncRNA-Six1 Encodes a Micropeptide to Activate Six1 in Cis and Is Involved in Cell Proliferation and Muscle Growth[J].Front Physiol, 2017, 8:230.
[7] Nelson BR, Makarewich CA, Anderson DM,etal.A peptide encoded by a transcript annotated as long noncoding RNA enhances SERCA activity in muscle[J].Science, 2016, 351(6270):271-275.
[8] Penny GD, Kay GF, Sheardown SA,etal.Requirement for Xist in X chromosome inactivation[J].Nature, 1996, 379(6561): 131-137.
[9] Cabili MN, Trapnell C, Goff L,etal.Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses[J].Genes Dev, 2011, 25, 1915-1927.
[10] Zhu J, Fu H, Wu Y,etal.Function of lncRNAs and approaches to lncRNA-protein interactions[J].Science China Life Sciences, 2013, 56, 876-885.
[11] 于红.表观遗传学:生物细胞非编码RNA调控的研究进展[J].遗传, 2009, 31(11): 1077-1086.
[12] Huang W, Long N, Khatib H.Genome-wide identification and initial characterization of bovine long non-coding RNAs from EST data[J].Anim Genet, 2012, 43(6): 674-682.
[13] Koufariotis LT, Chen YP, Chamberlain A,etal.A catalogue of novel bovine long noncoding RNA across 18 tissues[J].PLoS One, 2015, 10(10): e0141225.
[14] Weikard R, Hadlich F, Kuehn C.Identification of novel transcripts and noncoding RNAs in bovine skin by deep next generation sequencing[J].BMC Genomics, 2013, 14: 789.
[15] Ben AB, Wirth S, Merchan F,etal.Novel long non-protein coding RNAs involved in Arabidopsis differentiation and stress responses[J].Genome Research, 2009, 19: 57-69.
[16] Ma X, Shao C, Jin Y,etal.Long non-coding RNAs: a novel endogenous source for the generation of dicer-like 1-dependent small RNAs in Arabidopsis thaliana[J].RNA Biology, 2014, 11: 373-390.
[17] Bai Y, Dai X, Harrison AP,etal.RNA regulatory networks in animals and plants: a long noncoding RNA perspective[J].Briefingsin Functional Genomics, 2015, 14: 91-101.
[18] Wierzbicki AT.The role of long non-coding RNA in transcriptional gene silencing[J].Current Opinion in Plant Biology, 2012, 15: 517-522.
[19] Sheik MJ, Gaughwin PM, Lim B,etal.Conserved long noncoding RNAs transcriptionally regulated by Oct4 and Nanog modulate pluripotency in mouse embryonic stem cells[J].RNA, 2010, 16: 324-337.
[20] Petruk S, Sedkov Y, Riley KM,etal.Transcription of bxd noncoding RNAs promoted by trithorax represses Ubx in cis by transcriptional interference[J].Cell, 2006, 127: 1209-1221.
[21] Pruneski JA, Hainer SJ, Petrov KO,etal.The Paf1 complex represses SER3 transcription in Saccharomyces cerevisiae by facilitating intergenic transcription-dependent nucleosome occupancy of the SER3 promoter[J].Eukaryot Cell, 2011, 10(10): 1283-1294.
[22] Houseley J, Rubbi L, Grunstein M,etal.A ncRNA modulates histone modification and mRNA induction in the yeast GAL gene cluster[J].Mol Cell, 2008, 32(5): 685-695.
[23] Martianov I, Ramadass A, Serra Barros A,etal.Repression of the human dihydrofolate reductase gene by a non-coding interfering transcript[J].Nature, 2007, 445(7128): 666-670.
[24] Hung T, Wang Y, Lin MF,etal.Extensive and coordinated transcription of noncoding RNAs within cell-cycle promoters[J].Nat Genet, 2011, 43(7): 621-629.
[25] Beltran M, Puig I, Pena C,etal.A natural antisense transcript regulates Zeb2/Sip1 gene expression during Snail1induced epithelial-mesenchymal transition[J].Genes & Dev, 2008, 22: 756-769.
[26] Wilusz JE, Sunwoo H, Spector DL.Long noncoding RNAs: functional surprises from the RNA world[J].Genes Dev, 2009, 23(13): 1494-1504.
[27] Tripathi V, Ellis JD, Shen Z,etal.The nuclear-retained noncoding RNA MALAT1 regulates alternative splicing by modulating SR splicing factor phosphorylation[J].Mol Cell, 2010, 39(6): 925-238.
[28] Willingham AT, Orth AP, Batalov S,etal.A strategy for probing the function of noncoding RNAs finds a repressor of NFAT[J].Science, 2005, 309: 1570-1573.
[29] Peng F, Li TT, Wang KL,etal.H19/let-7/LIN28 reciprocal negative regulatory circuit promotes breast cancer stem cell maintenance[J].Cell Death Dis, 2017, 8(1): e2569.
[30] Yang Q, Wang X, Tang C,etal.H19 promotes the migration and invasion of colon cancer by sponging miR-138 to upregulate the expression of HMGA1[J].Int J Oncol, 2017, 50(5): 1801-1809.
[31] Cai X, Cullen BR.The imprinted H19 noncoding RNA is a primary microRNA precursor[J].RNA, 2007, 13(3): 313-316.
[32] Dey BK, Pfeifer K, Dutta A.The H19 long noncoding RNA gives rise to microRNAs miR-675-3p and miR-675-5p to promote skeletal muscle differentiation and regeneration[J].Genes Dev, 2014, 28(5): 491-501.
[33] Wang GQ, Wang Y, Xiong Y,etal.Sirt1 AS lncRNA interacts with its mRNA to inhibit muscle formation by attenuating function of miR-34a[J].Sci Rep, 2016, 6: 21865.
[34] Stavropoulos N, Lu N, Lee JT.A functional role for Tsix transcription in blocking Xist RNA accumulation but not in X-chromosome choice[J].Proc Natl Acad Sci U S A, 2001, 98(18): 10232-10237.
[35] Zhao J, Sun BK, Erwin JA,etal.Polycomb proteins targeted by a short repeat RNA to the mouse X chromosome[J].Science, 2008, 322(5902): 750-756.
[36] Bertani S, Sauer S, Bolotin E,etal.The noncoding RNA Mistral activates Hoxa6 and Hoxa7 expression and stem cell differentiation by recruiting MLL1 to chromatin[J].Mol Cell, 2011, 43(6): 1040-1046.
[37] 夏天, 肖丙秀, 郭俊明.长链非编码RNA的作用机制及其研究方法[J].遗传, 2013, 35(3): 269-280.
[38] Zhang C, Peng G.Non-coding RNAs: an emerging player in DNA damage response[J].Mutat Res Rev Mutat Res, 2015, 763: 202-211.
[39] Zhou J, Zhang S, Wang H,etal.LncFunNet: an integrated computational framework for identification of functional long noncoding RNAs in mouse skeletal muscle cells[J].Nucleic Acids Res, 2017, doi: 10.1093/nar/gkx232.
[40] Shen H, McElhinny AS, Cao Y,etal.The Notch coactivator, MAML1, functions as a novel coactivator for MEF2C-mediated transcription and is required for normal myogenesis[J].Genes Dev, 2006, 20(6): 675-688.
[41] Cesana M, Cacchiarelli D, Legnini I,etal.A long noncoding RNA controls muscle differentiation by functioning as a competing endogenous RNA[J].Cell, 2011, 147(2): 358-369.
[42] Zhu M, Liu J, Xiao J,etal.Lnc-mg is a long non-coding RNA that promotes myogenesis[J].Nat Commun, 2017, 8:14718.
[43] Yu X, Zhang Y, Li T,etal.Long non-coding RNA Linc-RAM enhances myogenic differentiation by interacting with MyoD[J].Nat Commun, 2017, 8: 14016.
[44] Sun X, Li M, Sun Y,etal.The developmental transcriptome sequencing of bovine skeletal muscle reveals a long noncoding RNA, lncMD, promotes muscle differentiation by sponging miR-125b[J].Biochimica Et Biophysica Acta, 2016, 1863(11): 2835-2845.
[45] Liu XF, Ding XB, Li X,etal.An atlas and analysis of bovine skeletal muscle long noncoding RNAs[J].Anim Genet, 2017, 48(3): 278-286.
[46] Lu L, Sun K, Chen X,etal.Genome-wide survey by ChIP-seq reveals YY1 regulation of lincRNAs in skeletal myogenesis[J].EMBO J, 2013, 32(19): 2575-2588.
[47] Kallen AN, Zhou XB, Xu J,etal.The imprinted H19 lncRNA antagonizes let-7 microRNAs[J].Mol Cell, 2013, 52(1): 101-112.
[48] Li Z, Jin C, Chen S,etal.Long non-coding RNA MEG3 inhibits adipogenesis and promotes osteogenesis of human adipose-derived mesenchymal stem cells via miR-140-5p[J].Mol Cell Biochem, 2017, doi: 10.1007/s11010-017-3015-z.
[49] Xu B, Gerin I, Miao H,etal.Multiple roles for the non-coding RNA SRA in regulation of adipogenesis and insulin sensitivity[J].PLoS One, 2010, 5, e14199.
[50] Liu S, Sheng L, Miao H,etal.SRA gene knockout protects against diet-induced obesity and improves glucose tolerance[J].J Biol Chem, 2014, 289, 13000-13009.
[51] Pang WJ, Lin LG, Xiong Y,etal.Knockdown of PU.1 AS lncRNA inhibits adipogenesis through enhancing PU.1 mRNA translation[J].J Cell Biochem, 2013, 114, 2500-2512.
[52] Wei N, Wang Y, Xu RX,etal.PU.1 antisense lncRNA against its mRNA translation promotes adipogenesis in porcine preadipocytes[J].Anim Genet, 2015, 46, 133-140.
[53] Xiao T, Liu L, Li H,etal.Long noncoding RNA ADINR regulates adipogenesis by transcriptionally activating C/EBPalpha[J].Stem Cell Rep, 2015, 5, 856-865.
[54] 李明勋.长链非编码RNA ADNCR通过竞争性结合miR-204抑制牛脂肪细胞分化.[D].陕西杨凌: 西北农林科技大学,2016.
[55] Li M, Sun X, Cai H,etal.Long non-coding RNA ADNCR suppresses adipogenic differentiation by targeting miR-204[J].Biochim Biophys Acta, 2016, 1859(7): 871-882.
[56] Hayashi S, Manabe I, Suzuki Y,etal.Klf5 regulates muscle differentiation by directly targeting muscle-specific genes in cooperation with MyoD in mice[J].Elife, 2016, 5, pii: e17462.
[57] Oishi Y, Manabe I, Tobe K,etal.Krüppel-like transcription factor KLF5 is a key regulator of adipocyte differentiation[J].Cell Metabolism, 2005, 1: 27-39.
[58] Tsai MC, Manor O, Wan Y,etal.Long noncoding RNA as modular scaffold of histone modification complexes[J].Science, 2010, 329(5992): 689-693.
[59] Divoux A, Karastergiou K, Xie H,etal.Identification of a Novel lncRNA in Gluteal Adipose Tissue and Evidence for Its Positive Effect on Preadipocyte Differentiation[J].Obesity, 2014, 22(8): 1781-1785.
[60] Sun L, Goff LA, Trapnell C,etal.Long noncoding RNAs regulate adipogenesis[J].P Natl Acad Sci USA , 2013, 110(9), 3387-3392.
[61] Alvarez-Dominguez JR, Bai Z, Xu D,etal.De Novo Reconstruction of Adipose Tissue Transcriptomes Reveals Long Non-coding RNA Regulators of Brown Adipocyte Development[J]. Cell Metab, 2015, 21(5), 764-776.

中国牛业科学2017,43(5):44-46;52ChinaCattleScience专 论

