盐胁迫下甘蓝型油菜发芽期下胚轴和根长的全基因组关联分析
张蕊,邓文亚,杨柳,王亚萍,肖芳枝,禾健,卢坤
盐胁迫下甘蓝型油菜发芽期下胚轴和根长的全基因组关联分析
张蕊1,邓文亚1,杨柳1,王亚萍1,肖芳枝2,禾健2,卢坤1
(1西南大学农学与生物科技学院,重庆 400715;2西南大学神农班,重庆 400715)
【目的】解析甘蓝型油菜发芽期根和下胚轴发育及耐盐性的调控位点,筛选油菜耐盐性相关的候选基因,可为油菜耐盐性改良提供依据。【方法】以317份具有代表性的甘蓝型油菜自交系为材料,在正常生长和盐胁迫条件下进行沙培鉴定,利用芸薹属60K SNP芯片和全基因组关联分析鉴定正常生长与盐胁迫下甘蓝型油菜发芽期根和下胚轴长度显著关联的SNP,并确定其连锁不平衡区间。通过区间内基因的功能注释及盐胁迫下油菜幼苗根和叶片转录组差异表达基因筛选连锁不平衡区间内的重要候选基因,并以实时荧光定量PCR分析候选基因的组织特异性和盐胁迫诱导表达模式,提高候选基因筛选的准确性。【结果】正常生长和盐胁迫下甘蓝型油菜发芽期下胚轴和根长在不同材料间变异较大,频次分布表明目标性状均为数量性状,受多基因调控。全基因组关联分析模型比较表明,MLM+P+K模型为最优模型。以此模型对目标性状进行全基因组关联分析,检测到45个显著关联SNP,其中40个与下胚轴长度显著关联,5个与根长显著关联,单个SNP解释的表型变异分别为9.12%—14.46%和7.67%—8.93%。重复检测的显著相关SNP中,值得注意的是C04染色体的rs8970,同时与4个性状显著关联,表型贡献率为7.67%—12.35%,是唯一在下胚轴长和根长间重复检测到的显著关联SNP。11个重要关联SNP中有6个位于10—442 kb的连锁不平衡区块中。转录组分析表明,11个连锁不平衡区间共包含447个基因,其中15个受盐胁迫诱导表达。转录组和基因功能注释综合分析表明,、、、、、、、、、和很可能为各自区间的候选基因。实时荧光定量PCR结果证实除外,其他基因均在根或下胚轴中受盐胁迫诱导上调表达。组织特异性分析还发现、和主要在萌发的根和下胚轴中特异表达,、和虽然呈组成型表达,但在萌发阶段的下胚轴中表达量最高,证实这些基因很可能参与油菜发芽期根和下胚轴生长发育及耐盐性的调节。【结论】全基因组关联分析共鉴定出45个控制油菜发芽期根和下胚轴发育及耐盐性的显著关联SNP。连锁不平衡、转录组和基因功能注释综合分析初步鉴定出11个重要候选基因。
甘蓝型油菜;萌发;耐盐性;全基因组关联分析;转录组
0 引言
【研究意义】盐害是危害农业生产的主要因素之一。作为典型的非生物胁迫,盐害会破坏作物的离子平衡,引起离子毒性、高渗和氧化胁迫等,从而影响种子萌发和幼苗生长,导致产量和品质下降[1]。诸多研究表明,甘蓝型油菜耐盐能力较强,因此,挖掘油菜耐盐调控位点和基因,培育耐盐油菜品种,以开发利用盐碱地,对缓解中国植物油供应压力、维护国家食物安全具有重要意义[2]。【前人研究进展】目前,油菜耐盐性的遗传和分子机制报道较少[3-4]。龙卫华等[5]对芥菜型油菜()、白菜型油菜()和甘蓝型油菜()共203份种质资源进行盐胁迫比较研究,筛选出1份白菜型油菜和2份甘蓝型油菜材料用于培育耐盐油菜品种。刘国红等[2]发现盐胁迫对油菜光合作用的抑制主要来自气孔因素,但在长期高盐条件下,非气孔因素对光合抑制的贡献也很明显。在遗传定位方面,荐红举等[6]利用高世代重组自交系群体和SNP遗传图谱定位到11个盐胁迫下油菜种子发芽率相关的QTL。Yong等[7]对85个油菜自交系开展了基于多样性芯片技术(diversity arrays technology,DArT)和DArTseq基因型分析的研究,利用GWAS检测到62个耐盐性、地上部生物量及离子平衡相关的QTL,并鉴定出可能的候选基因。【本研究切入点】尽管甘蓝型油菜耐盐相关QTL有少量报道,但盐胁迫下油菜发芽期根长和下胚轴长度的GWAS研究尚未见报道。【拟解决的关键问题】为解析控制油菜发芽期根和下胚轴发育及耐盐性的关键位点和候选基因,本研究采用GWAS鉴定目标性状显著关联SNP,结合转录组分析,筛选连锁不平衡(linkage disequilibrium,LD)区间内的盐胁迫诱导差异表达基因,初步确定控制性状变异的候选基因。以实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)分析候选基因的组织特异性和盐胁迫诱导表达模式,提高候选基因筛选的准确性。本研究将为分子标记辅助选择和基因工程改良等方式培育耐盐油菜新品种提供理论依据和新的基因资源。
1 材料与方法
1.1 试验材料
供试材料共317份(电子附表1),均为国内外广泛收集的代表性油菜资源,其中,国内材料283份,主要来自重庆、湖北和湖南等地,国外材料34份,主要来自德国和加拿大。所有材料均由重庆市油菜工程技术研究中心保存。2015年收获的当年自交种用于本研究。
1.2 胁迫处理和性状调查
试验于2015年在西南大学油菜培养间进行,整个试验重复2次,生长条件为昼夜温度25℃,光照/黑暗时间16 h/8 h,光照强度5 000 lx。从供试材料中挑选大小均匀、健康饱满的种子各120粒,将种子浸泡24 h后分为2份,分别播种在100 mmol·L-1NaCl和对照蒸馏水充分浸润的蛭石上,覆盖保鲜膜。播种5 d后,选取长势一致的10株幼苗测定根长(root length,RL)和下胚轴长(hypocotyl length,HL),正常萌发条件下的RL和HL以RLW和HLW表示,盐胁迫下萌发的RL和HL以RLN和HLN表示。两性状差值(RLD和HLD)为对照与盐胁迫环境测定值之差。以SPSS 20.0(美国IBM公司)软件对表型数据进行描述性统计分析和Kolmogorov-Smirnov正态分布检验。利用R脚本(www.eXtension.org/pages/61006)对2次重复试验的性状值进行基于线性模型的最佳线性无偏预测。
1.3 基因型分析及SNP标记定位
参考Qu等[8]的方法,利用芸薹属60K SNP芯片进行SNP检测,去除最小基因型频率(minor allele frequency,MAF)低于0.05和SNP得率(call frequency)小于80%的标记。为确定SNP在染色体上的位置,利用探针序列与基因组进行BLASTN比对[9-10],E-value设为1e-5。去除Bit-Score相同的SNP,最终获得31 468个在基因组上具有唯一位置的高质量SNP用于后续分析。
1.4 群体结构分析与全基因组关联分析
在TASSEL 5[11]中进行主成分分析(PCA矩阵),计算亲缘关系矩阵(K)。群体结构Q值矩阵采用Qu等[8]的结果。以K、Q和PCA矩阵为协变量,采用基于一般线性模型(general linear model,GLM)的GLM+P和GLM+Q模型及混合线性模型(mixed linear model,MLM)的MLM+K和MLM+P+K和MLM+Q+K共5种模型进行GWAS。显著关联SNP宽松阈值设为1/31468=3.18e-5,严谨阈值设为0.05/31468=1.59e-6。根据所有SNP的–log10()观察值和期望值,以ggplot2[12]绘制Quantile-Quantile散点图,确定GWAS最佳模型。基于最优模型的关联分析结果,利用qqman[13]绘制曼哈顿图。
1.5 LD区间确定及候选基因分析
以Haploview 4.2[14]计算显著关联SNP所在染色体的LD,HW阈值(Hardy Weinberg-value cutoff)设定为0.001;非缺失标记的比例为75%,MAF设定为0.05;单倍型块(haplotype block)分析采用Four Gamete Rule算法,第4配子频率(4thgamete frequency)>0.1。将显著关联SNP所在单倍型块作为候选基因所在LD区间。若显著关联SNP不属于任何单倍型块,则以SNP标记上下游各100 kb侧翼序列区间作为LD区间。候选基因筛选利用Yong等[15]的转录组数据,将LD区间内的盐胁迫诱导表达基因作为候选基因。主要分析流程为利用STAR 2.4.2a将reads比对至油菜参考基因组[16],采用Cufflinks中的cuffdiff筛选差异表达基因(differentially expressed genes,DEGs)[17],筛选标准为差异表达倍数大于2,错误发现率(false discovery rate,FDR)-value小于0.05。
1.6 候选基因组织特异及诱导表达模式
为分析候选基因组织特异性及盐胁迫诱导表达模式,于2015年盛花期取常规品种中双11的根、茎、成熟叶片和蕾,灌浆期取花后7、14、21、30和40 d种子和角果皮。按照1.2的方法进行砂培,并分别取正常和盐胁迫处理72 h的根和下胚轴。总RNA提取参照卢坤等[18]的方法。cDNA模板采用PrimeScript RT reagent Kit(宝生生物工程有限公司,大连)合成。利用SYBR(宝生生物工程有限公司,大连)在CFX96定量PCR系统(美国Bio-Rad公司)上按照MIQE国际化标准进行qRT-PCR[19],内参为和[20]。引物采用Geneious Pro 8.1.5设计(表1)。为保证扩增片段特异性,所有引物与同源基因至少相差2—3个碱基。
2 结果
2.1 油菜萌发相关性状表型数据
正常和盐胁迫条件下,幼苗平均根长分别为4.92和3.94 cm,变异系数为16.86%和42.48%(表2),胁迫处理前后根长差值平均为1.01 cm,变异系数为199.57%。正常萌发条件下,下胚轴平均长4.48 cm,变异系数为25.75%;盐胁迫处理后降至3.03 cm,变异系数为31.41%;胁迫处理前后下胚轴长差值平均为1.49 cm,变异系数为67.07%。结果表明,不同材料间的萌发性状差异较大,为GWAS分析奠定基础。
正态分布检验表明(表2),RLW、HLW、HLN和HLD的-value大于0.05,符合正态分布。RLN和RLD的-value为0,但其频次分布呈单峰偏态或双峰分布,表现为主基因+多基因的数量遗传特征(图1)。上述结果表明,本研究目标性状均为数量性状,受多基因调控。
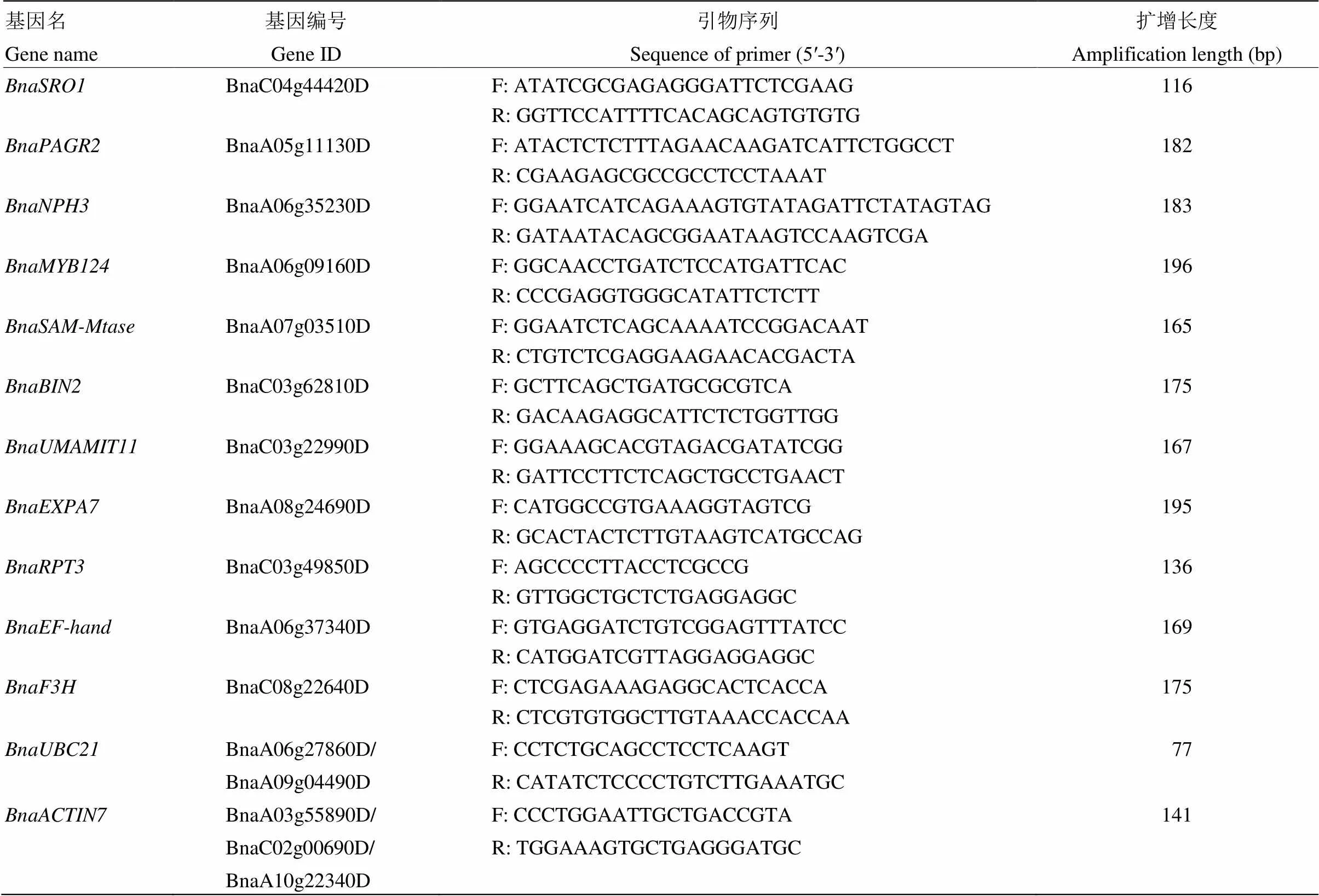
表1 本研究采用的qRT-PCR鉴定引物

表2 盐胁迫下甘蓝型油菜发芽期幼苗根长和下胚轴长的统计分析
RLW和HLW:正常萌发的根长和下胚轴长;RLN和HLW:NaCl处理条件的根长和下胚轴长;RLD和HLD:处理前后根长和下胚轴长的差值;“1”和“2”表示同一性状的第1和第2次试验;**表示在0.01水平差异显著
RLW and HLW: RL and HL under normal germination conditions; RLN and HLW: RL and HL under salt stress germination conditions; RLD and HLD: difference between trait value under normal condition and salt stress condition; “1” and “2” represent experiment replicate 1 and replicate 2 of the same trait; ** indicates significance level at 0.01

图1 盐胁迫下甘蓝型油菜发芽期幼苗根长和下胚轴长的频次分布
2.2 关联分析模型选择
GWAS最优模型分析表明,GLM模型控制假阳性的能力较差,与MLM模型相比,GLM+P和GLM+Q模型均严重偏离期望值。3种MLM模型对根长的解析能力差别不大,但在下胚轴长的GWAS分析中,MLM+P+K模型检测到的-value较其他模型更接近期望值,能更好地控制假阳性(图2),因此,采用MLM+P+K模型进行GWAS分析。
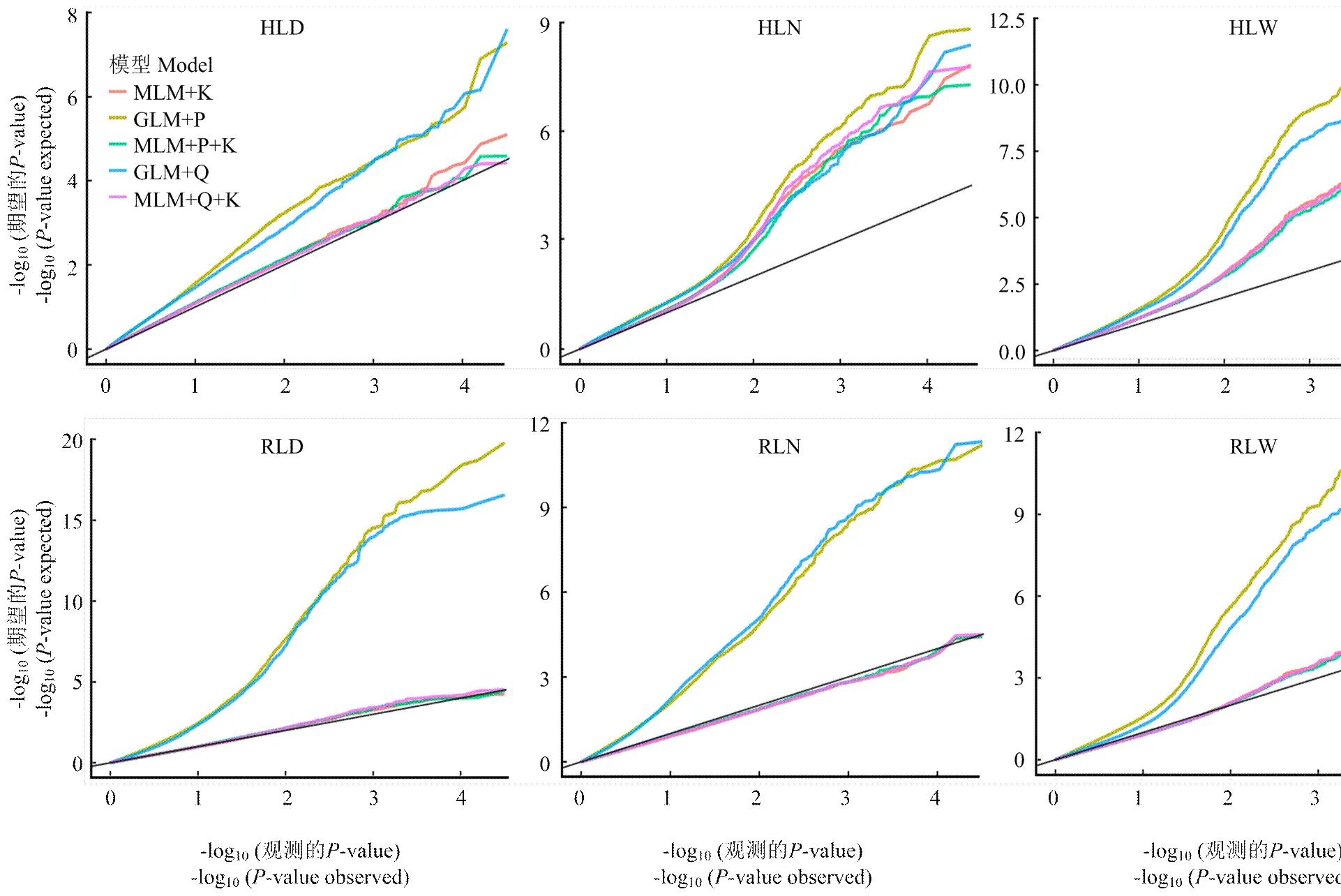
图2 不同性状5种模型比较的QQ图
2.3 萌发性状的全基因组关联分析
利用MLM+P+K模型对目标性状进行GWAS分析。由于HLW和HLN检出位点较多,筛选显著关联SNP时采用严谨阈值,其他4个性状采用宽松阈值(表3和图3)。结果表明,共检测到45个显著关联SNP,其中独立SNP 36个,重复检测SNP 7个。与HL显著关联SNP共40个,其中HLW和HLN存在17和21个显著相关SNP,C03染色体紧密相邻的2个SNP与HLD显著关联。与RLW、RLN和RLD显著关联的SNP各有3个、1个和1个。HL和RL的单一SNP表型贡献率分别为9.12%—14.46%和7.67%—8.93%。
重复检测的显著相关SNP中,6个为HLW和HLN间的重复,位于A05、A06、A07和C03染色体,其很可能与控制下胚轴发育的基因紧密连锁。A08染色体上分别与HLN和HLW显著关联的rs18442和rs18448为相邻SNP,其可能同时与HLN和HLW紧密连锁。值得注意的是C04染色体的rs8970,其同时与HLW、HLN、RLW和RLD 4个性状显著关联,表型贡献率为7.67%—12.35%,是唯一在HL和RL间重复检测到的显著关联SNP。
2.4 盐胁迫下油菜幼苗叶片和根部差异表达基因分析
转录组分析表明,盐胁迫处理12 h后幼叶和根部有大量基因诱导表达,其中上调表达基因分别为1 304和1 334个,下调表达基因为220和336个。在叶片和根部同时上调和下调表达的基因分别为202和12个,而在叶片和根部特异上调表达的基因分别为1 125和1 097个。
2.5 LD区间内候选基因的筛选
利用Haploview 4.2[14]分析各性状贡献率最高或性状间重复检测到的显著关联SNP所在LD区块。结果表明,11个重要的显著关联SNP中有6个位于LD区块中,长度分别为10—442 kb(表4)。GWAS与差异表达基因比较分析发现,在11个LD区间的447个基因中有15个受盐胁迫诱导表达,但无显著关联SNP位于这些基因内。此外,分析与HLW和HLN相关的6个LD区段时,重点关注苗期发育调控基因。剩余区段重点选择与非生物胁迫相关基因。最终初步筛选的候选基因中与盐胁迫和发育调控相关,、、、、、和与发芽阶段的发育过程有关,、和可能与油菜发芽期的盐胁迫适应性相关。
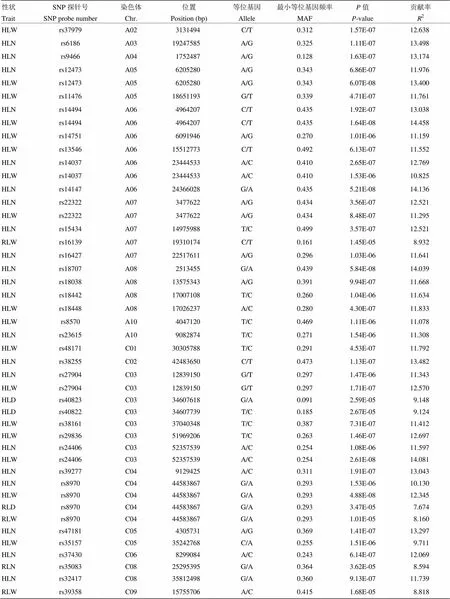
表3 盐胁迫下甘蓝型油菜发芽期幼苗下胚轴长和根长的的显著关联SNPs
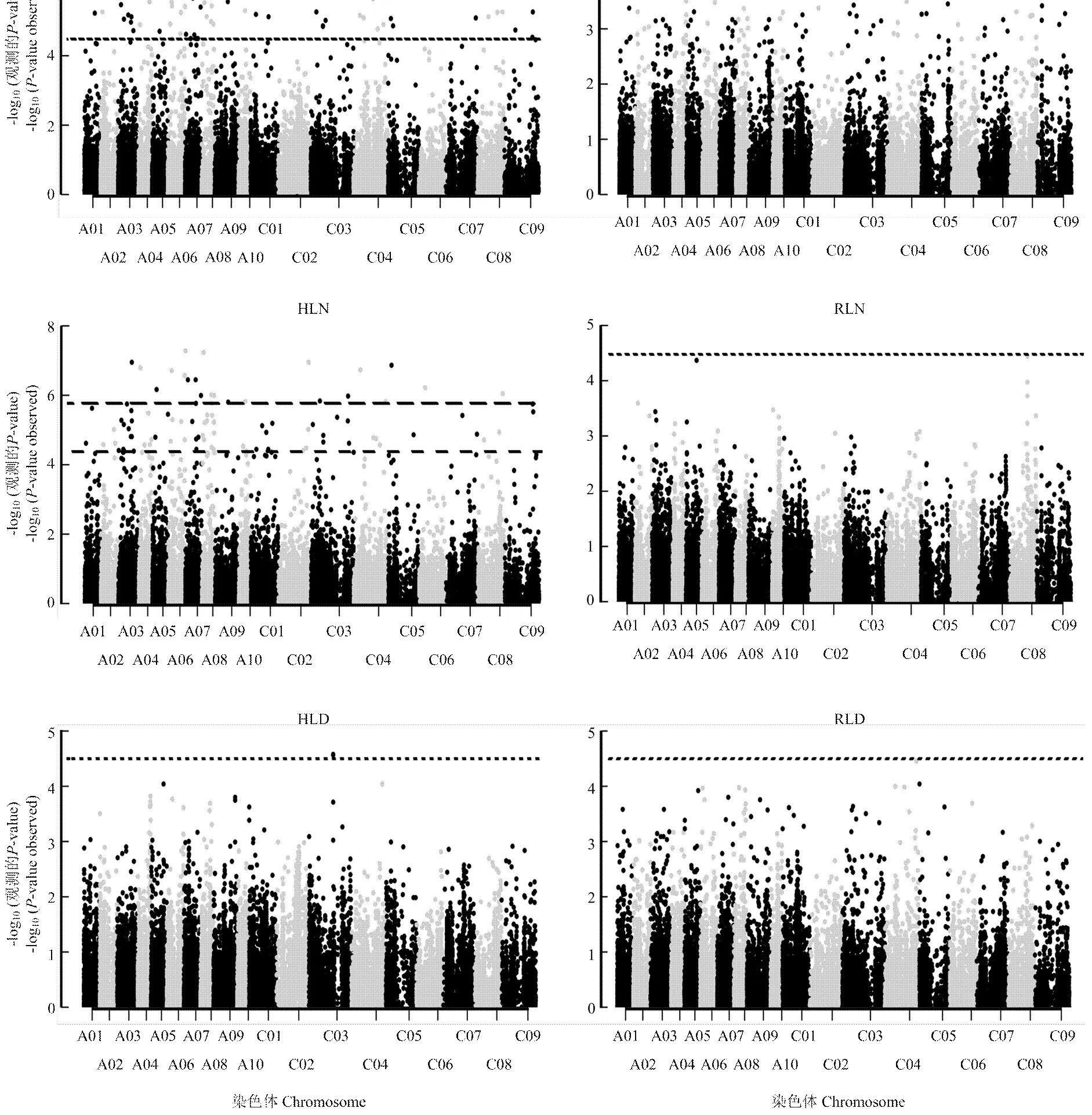
图3 盐胁迫下甘蓝型油菜发芽期下胚轴长和根长全基因组关联分析的曼哈顿图
2.6 候选基因的组织特异和诱导表达模式
qRT-PCR检测结果表明(图4),11个基因均在萌发后72 h的根和下胚轴中有不同程度表达,、、和在萌发后72 h根的表达量高于下胚轴,而、、和则刚好相反。盐胁迫处理后,、、、、和在根部显著上调表达,、、、和在下胚轴中显著上调表达。此外,、和在萌发的根和下胚轴中特异表达,表明其可能与油菜萌发阶段根和下胚轴的发育调控有关。、和虽然呈组成型表达,但在下胚轴中表达量最高。在萌发阶段的根和花后40 d种子中表达量较高,说明该基因同时参与种子成熟及萌发。、和在除萌发阶段的根和下胚轴外的其他组织器官中也有较高的转录水平,表明其除了在萌发阶段发挥功能以外,还参与油菜叶片、蕾、种子和角果皮的发育调控。
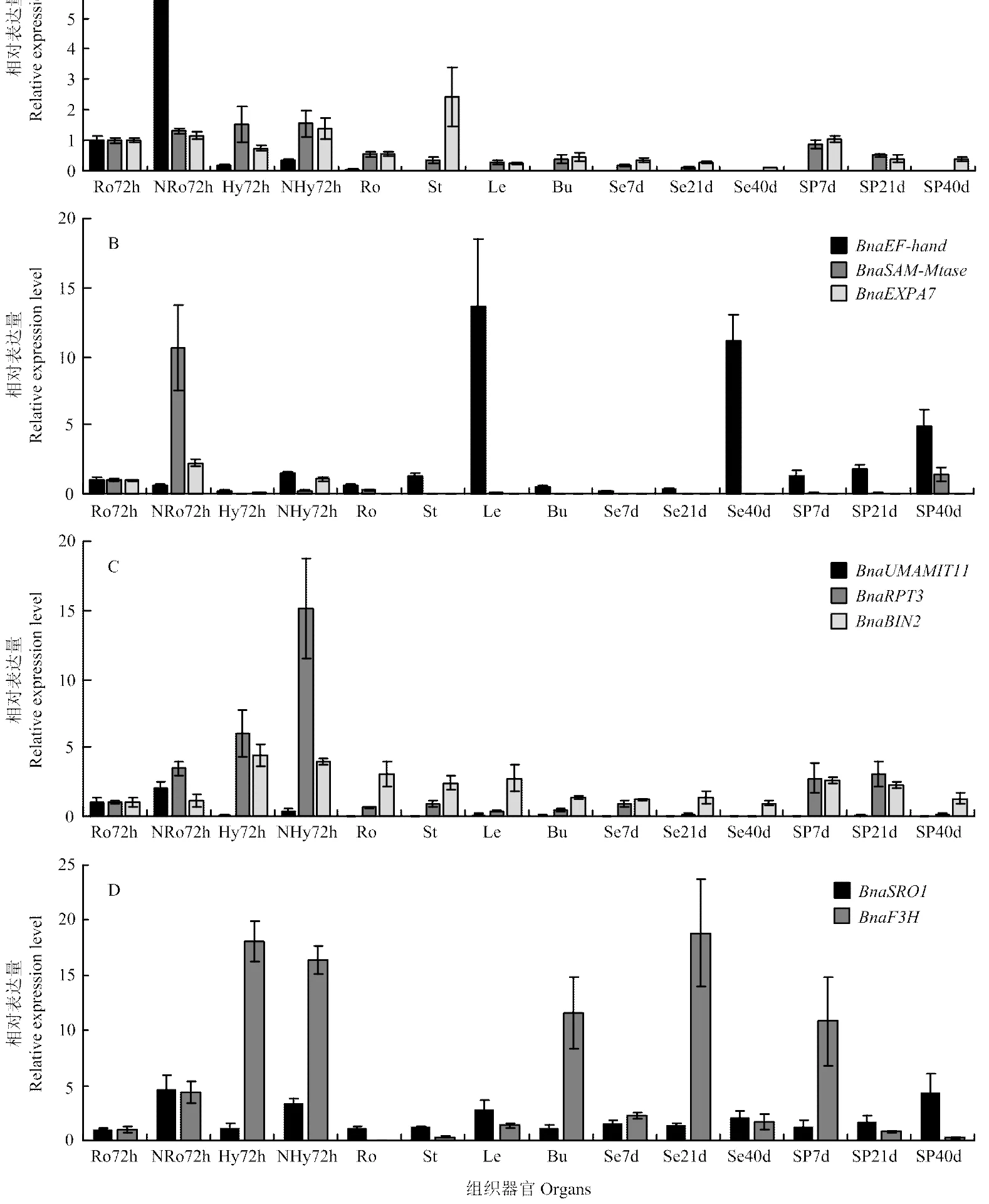
A:BnaPAGR2、BnaMYB124和BnaNPH3的表达模式;B:BnaEF-hand、BnaSAM-Mtase和BnaEXPA7的表达模式;C:BnaUMAMIT11、BnaRPT3和BnaBIN2的表达模式;D:BnaSRO1和BnaF3H的表达模式;Ro72h:发芽72 h的根;NRo72h:盐胁迫下发芽72 h的根;Hy72h:发芽72 h的下胚轴;NHy72h:盐胁迫下发芽72 h的下胚轴;Ro:花期的根;St:花期的茎;Le:花期成熟叶片;Bu:蕾;Se7d:花后7 d种子;Se21d:花后21 d种子;Se40d:花后40 d种子;SP7d:花后7 d荚果皮;SP21d:花后21 d荚果皮;SP40d:花后40 d荚果皮。误差线表示平均值的标准差(n=3)

表4 显著关联标记所在LD区间及候选基因
3 讨论
甘蓝型油菜基因组序列的公布及SNP芯片的开发促进了油菜重要数量性状的定位研究[9]。目前,有学者已开展了油菜种子重量和品质[21]、干旱胁迫抗性[22]、种子萌发和活力[23]、收获指数[24]、株高、一次分枝数[25]、单株产量、生物产量和开花时间[26]等性状的GWAS分析,鉴定了大量控制油菜重要性状的位点。本研究利用砂培试验,对正常和盐胁迫萌发条件下幼苗下胚轴长和根长性状进行GWAS分析,鉴定出45个显著关联的SNP,分布在油菜基因组的16条染色体,其中A03染色体上的rs6816与荐红举等[6]利用重组自交系群体检测到的控制盐胁迫下油菜种子发芽率的QTL位点非常接近,仅相距348 kb,很可能为同一QTL区间。ZHANG等[22]对模拟干旱胁迫条件下油菜萌发的根长、茎长、根茎长度比和鲜重进行了GWAS分析,鉴定出的显著关联标记Bn-A10-p3063220、Bn-scaff_16394_2- p1222322、Bn-scaff_17623_1-p657238和Bn-scaff_ 16445_1-p1834116与笔者在相同染色体发现的显著关联SNP均在1 Mb范围内。由于ZHANG等[22]的GWAS群体仅包含66个自交系,表型数据较少,关联分析准确性受影响,因此,其鉴定的位点与本研究结果可能一致,但还需要进一步比较研究。HATZIG等[23]利用GWAS对油菜种子萌发和种子活力进行了解析,其中A05和C08染色体的显著关联SNP与本研究定位结果一致。虽然,本研究检测到的位点与前人定位QTL部分重叠,但仍有多个显著关联标记无法与其他研究对应,可能与鉴定方法及调查性状不同有关,这些位点可作为新的候选基因区域作进一步研究。
不同油菜品种对盐胁迫的抗(耐)性存在显著差异[2],这是解析油菜耐盐性关键遗传因子和耐盐基因的基础。本研究利用GWAS和转录组综合分析方法,最终确定了11个重要LD区间的候选基因。其中,显著关联SNP rs22322所在LD区间的候选基因()位于A07染色体,编码S-腺苷基甲硫氨酸依赖的甲基转移酶(S- adenosyl-L-methionine-dependent methyltransferase),其被证实对维持细胞功能,提高植株抗氧化胁迫能力具有重要作用[27]。本研究中在根和下胚轴均受盐胁迫显著上调表达,提示其可能也以类似的方式提高油菜发芽期下胚轴和根部的耐盐性。而rs18442和rs18448标记LD区间的候选基因编码α-扩张蛋白7(BnaEXPA7),在根和下胚轴中特异表达,并在下胚轴中受盐胁迫诱导上调表达,由于α-扩张蛋白家族主要具有松弛细胞壁,决定器官大小和形态建成的作用,因此其对油菜下胚轴伸长和盐胁迫适应性可能也起到了重要作用[28]。()是最值得关注的候选基因,其位于显著关联SNP rs8970的LD区段内,与HLN、HLW、RLD和RLW 4个性状均紧密连锁,编码具有多聚(ADP-核糖)聚合酶催化结构域和WWE蛋白互作结构域的RCD1类蛋白(SIMILAR TO RCD ONE1,SRO1)。有研究表明,RCD1能与AP2/ERF、NAC和bHLH(basic helix-loop-helix)转录因子互作,调节植物的发育 和胁迫响应[29]。此外,RCD1还能质膜Na+/H+逆向转运蛋白SOS1互作,提高拟南芥氧化胁迫适应能力[30]。本研究发现,在根和下胚轴中受盐胁迫诱导表达,其转录水平在叶和花后40 d角果皮中也具有较高的转录水平,提示其不仅对油菜发芽期下胚轴的发育及盐胁迫适应性具有重要作用,可能也参与叶片和角果皮的发育调控。类黄酮合成途径的关键蛋白黄烷酮3-羟化酶(flavanone 3- hydroxylase gene,F3H)具有提高植物抵抗干旱、盐胁迫和紫外辐射的能力[31]。位于显著关联标记rs35083 LD区间内的()在油菜发芽期根部受盐胁迫诱导上调表达,提示其可能对油菜发芽期根部的盐胁迫适应也具有重要作用。除上述4个基因外,基因表达模式和基因功能注释分析显示另外7个候选基因很可能也参与了油菜发芽阶段根和下胚轴的发育调节或与苗期盐胁迫适应相关。因此,深入开展上述候选基因的基因功能研究将有助于准确揭示油菜发芽期根和下胚轴的发育及盐胁迫适应机制,为提高油菜耐盐性和选育耐盐品种奠定基础。
4 结论
GWAS分析检测到45个与油菜发芽期根和下胚轴发育及盐胁迫适应性显著关联的SNP,其中40个和5个分别与下胚轴长和根长显著关联。重要显著关联SNP的11个LD区间共含447个基因,转录组分析和基因功能注释综合分析进一步筛选出11个重要候选基因。除,其余10个候选基因均在根或下胚轴中受盐胁迫诱导上调表达,部分基因具有根或下胚轴特异表达特性,说明这些基因很可能参与油菜发芽期根和下胚轴生长发育及耐盐性的调节。
References
[1] Yamaguchi T, Blumwald E. Developing salt-tolerant crop plants: Challenges and opportunities., 2005, 10: 615-620.
[2] 刘国红, 姜超强, 刘兆普, 梁明祥, 殷祥贞, 郑青松. 盐胁迫对油菜幼苗生长和光合特征的影响. 生态与农村环境学报, 2012, 28(2): 157-164.
Liu G H, Jiang C Q, Liu Z P, Liang M X, Yin X Z, Zheng Q S. Effects of salt stress on growth and photosynthetic traits of canola seedlings., 2012, 28(2): 157-164. (in Chinese)
[3] Ashraf M, McNeilly T. Salinity tolerance inoilseeds., 2004, 23(2): 157-174.
[4] Zhang X, Lu G, Long W, Zou X, Li F, Nishio T. Recent progress in drought and salt tolerance studies incrops., 2014, 64(1): 60-73.
[5] 龙卫华, 浦惠明, 陈松, 张洁夫, 戚存扣, 张学昆. 油菜3个栽培种发芽期耐盐性评价. 植物遗传资源学报, 2014, 15(1): 32-37.
LONG W H, Pu H M, Chen S, Zhang J F, Qi C K, Zhang X K. Evaluation for salt tolerance of three cultivated species of rapeseeds at germination stage., 2014, 15(1): 32-37. (in Chinese)
[6] 荐红举, 肖阳, 李加纳, 马珍珍, 魏丽娟, 刘列钊. 利用SNP遗传图谱定位盐、旱胁迫下甘蓝型油菜种子发芽率的QTL. 作物学报, 2014, 40(4): 629-635.
Jian H J, Xiao Y, Li J N, Ma Z Z, Wei L J, Liu L Z. QTL mapping for germination percentage under salinity and drought stresses inL. using a SNP genetic map., 2014, 40(4): 629-635. (in Chinese)
[7] Yong H Y, Wang C, Bancroft I, Li F, Wu X, Kitashiba H, Nishio T. Identification of a gene controlling variation in the salt tolerance of rapeseed (L.)., 2015, 242: 313-326.
[8] Qu C M, Li S M, Duan X J, Fan J H, Jia L D, Zhao H Y, Lu K, Li J N, Xu X F, Wang R. Identification of candidate genes for seed glucosinolate content using association mapping inL.., 2015, 6: 1215-1229.
[9] Chalhoub B, Denoeud F, Liu S, Parkin I. a. P, Tang H, Wang X, Chiquet J, Belcram H, Tong C, Samans B, Correa M, Da Silva C, Just J, Falentin C, Koh C S, Le Clainche I, Bernard M, Bento P, Noel B, Labadie K, Alberti A, Charles M, Arnaud D, Guo H, Daviaud C, Alamery S, Jabbari K, Zhao M, Edger P P, Chelaifa H, Tack D, Lassalle G, Mestiri I, Schnel N, Le Paslier M C, Fan G, Renault V, Bayer P E, Golicz a a, Manoli S, Lee T H, Thi V H D, Chalabi S, Hu Q, Fan C, Tollenaere R, Lu Y, Battail C, Shen J, Sidebottom C H D, Canaguier A, Chauveau A, Berard A, Deniot G, Guan M, Liu Z, Sun F, Lim Y P, Lyons E, Town C D, Bancroft I, Meng J, Ma J, Pires J C, King G J, Brunel D, Delourme R, Renard M, Aury J M, Adams K L, Batley J, Snowdon R J, Tost J, Edwards D, Zhou Y, Hua W, Sharpe A G, Paterson A H, Guan C Wincker P. Early allopolyploid evolution in the post-Neolithicoilseed genome., 345(6199): 950-953.
[10] Altschul S F, Madden T L, Schäffer A A, Zhang J, Zhang Z, Miller W, Lipman D J. Gapped BLAST and PSI-BLAST: A new generation of protein database search programs., 1997, 25: 3389-3402.
[11] Bradbury P J, Zhang Z, Kroon D E, Casstevens T M, Ramdoss Y, Buckler E S. TASSEL: Software for association mapping of complex traits in diverse samples., 2007, 23(19): 2633-2635.
[12] Ginestet C. ggplot2: elegant graphics for data analysis.:, 2001, 174: 245-246.
[13] Turner S D. qqman: an R package for visualizing GWAS results using Q-. Q and manhattan plots., 2014. http://dx.doi.org/ 10.1101/005165
[14] Barrett J C, Fry B, Maller J, Daly M J. Haploview: Analysis and visualization of LD and haplotype maps., 2005, 21(2): 263-265.
[15] Yong H Y, Zou Z, Kok E P, Kwan B H, Chow K, Nasu S, Nanzyo M, Kitashiba H, Nishio T. Comparative transcriptome analysis of leaves and roots in response to sudden increase in salinity inby RNA-seq., 2014, 2014: 467395.
[16] Dobin A, Davis C A, Schlesinger F, Drenkow J, Zaleski C, Jha S, Batut P, Chaisson M, Gingeras T R. STAR: Ultrafast universal RNA-seq aligner., 2013, 29(1): 15-21.
[17] Trapnell C, Roberts A, Goff L, Pertea G, Kim D, Kelley D R, Pimentel H, Salzberg S L, Rinn J L, Pachter L. Differential gene and transcript expression analysis of RNA-seq experiments with TopHat and Cufflinks., 2012, 7(3): 7: 562-578.
[18] 卢坤, 张琳, 曲存民, 梁颖, 唐章林, 李加纳. 利用RNA-Seq鉴定甘蓝型油菜叶片干旱胁迫应答基因. 中国农业科学, 2015, 48(4): 630-645.
Lu K, Zhang L, Qu C M, Liang Y, Tang Z L, LI J N. Identification of drought stress-responsive genes in leaves ofusing RNA sequencing technology., 2015, 48(4): 630-645. (in Chinese)
[19] Bustin S A, Benes V, Garson J A, Hellemans J, Huggett J, Kubista M, Mueller R, Nolan T, Pfaffl M W, Shipley G L. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments., 2009, 55(4): 611-622.
[20] Qu C, Fu F, Liu M, Zhao H, Liu C, Li J, Lu K. Comparative transcriptome analysis of recessive male sterility (RGMS) in sterile and fertilelines., 2015, 10(12): e0144118.
[21] Li F, Chen B, Xu K, Wu J, Song W, Bancroft I, Harper A L, Trick M, Liu S, Gao G, Wang N, Yan G, Qiao J, Li J, Li H, Xiao X, Zhang T, Wu X. Genome-wide association study dissects the genetic architecture of seed weight and seed quality in rapeseed (L.)., 2014, 21: 355-367.
[22] Zhang J, Mason A S, Wu J, Liu S, Liu S, Zhang X, Luo T, Redden R, Batley J, Hu L, Yan G. Identification of putative candidate genes for water stress tolerance in canola ()., 2015, 6: 1058.
[23] Hatzig S V, Frisch M, Breuer F, Nesi N, Ducournau S, Wagner M H, Leckband G, Abbadi A, Snowdon R J. Genome-wide association mapping unravels the genetic control of seed germination and vigor in., 2015, 6: 221.
[24] Luo X, Ma C, Yue Y, Hu K, Li Y, Duan Z, Wu M, Tu J, Shen J, Yi B, Fu T. Unravelling the complex trait of harvest index in rapeseed (L.) with association mapping., 2015, 16: 379.
[25] Li F, Chen B, Xu K, Gao G, Yan G, Qiao J, Li J, Li H, Li L, Xiao X, Zhang T, Nishio T, Wu X. A genome-wide association study of plant height and primary branch number in rapeseed ()., 2016, 242: 169-177.
[26] Li L, Long Y, Zhang L, Dalton-Morgan J, Batley J, Yu L, Meng J, Li M. Genome wide analysis of flowering time trait in multiple environments via high-throughput genotyping technique inL.., 2015, 10(3): 1-18.
[27] Kunstmann B, Osiewacz H D. Over‐expression of an S‐adenosylmethionine‐dependent methyltransferase leads to an extended lifespan of Podospora anserina without impairments in vital functions., 2008, 7: 651-662.
[28] Sampedro J, Cosgrove D J. The expansin superfamily., 2005, 6: 1.
[29] Jaspers P, Blomster T, Brosche M, Salojärvi J, Ahlfors R, Vainonen J P, Reddy R A, Immink R, Angenent G, Turck F, Overmyer K, Kangasjärvi J. Unequally redundant RCD1 and SRO1 mediate stress and developmental responses and interact with transcription factors., 2009, 60: 268-279.
[30] Katiyar-Agarwal S, Zhu J, Kim K, Agarwal M, Fu X, Huang A, Zhu J K. The plasma membrane Na+/H+antiporter SOS1 interacts with RCD1 and functions in oxidative stress tolerance in., 2006, 103: 18816-18821.
[31] Walia H, Wilson C, Condamine P, Liu X, Ismail A M, Zeng L, Wanamaker S I, Mandal J, Xu J, Cui X, Close T J. Comparative transcriptional profiling of two contrasting rice genotypes under salinity stress during the vegetative growth stage., 2005, 139: 822-835.
(责任编辑 李莉)
附表1 本研究所用的植物材料
Supplementary Table 1 Plant materials used in this study

材料编号Accession number材料名Accession name国家,地区Country, region亚群Sub-population B1Ganyou5中国重庆 Chongqing, ChinaP1 B2Nonglin42中国重庆 Chongqing, ChinaP1 B4Huaiyou6中国重庆 Chongqing, ChinaP1 B5Yan81-2中国重庆 Chongqing, ChinaP1 B628887中国重庆 Chongqing, ChinaP1 B7Qianyou4中国重庆 Chongqing, ChinaP1 B8Qianyou331中国重庆 Chongqing, ChinaP1 B10Niuerduo中国重庆 Chongqing, ChinaP1 B14SWU43中国重庆 Chongqing, ChinaP1 B16SWU45中国重庆 Chongqing, ChinaP1 B17SWU46中国重庆 Chongqing, ChinaP1 B18SWU47中国重庆 Chongqing, ChinaP1 B19SWU48中国重庆 Chongqing, ChinaP1 B20SWU52中国重庆 Chongqing, ChinaP1 B21SWU53中国重庆 Chongqing, ChinaP1 B23SWU59中国重庆 Chongqing, ChinaP1 B24SWU65中国重庆 Chongqing, ChinaP1 B25SWU82中国重庆 Chongqing, ChinaP1 B26SWU83中国重庆 Chongqing, ChinaP1 B27SWU92中国重庆 Chongqing, ChinaP1 B28SWU101中国重庆 Chongqing, ChinaP1 B29SWU106中国重庆 Chongqing, ChinaP1 B30SWU108中国重庆 Chongqing, ChinaP1 B31Chuanyou20中国四川 Sichuan, ChinaP1 B33CY12NY-7中国四川 Sichuan, ChinaP1 B35CY12Q8-7中国四川 Sichuan, ChinaP1 B36CY12QSZ06中国四川 Sichuan, ChinaP1 B38CY12Q95108中国四川 Sichuan, ChinaP1 B39CY12Q21535-N3中国四川 Sichuan, ChinaP1 B40CY12PXW-4中国四川 Sichuan, ChinaP1 B41CY12PXW-6中国四川 Sichuan, ChinaP1 B43CY13PXW-17中国四川 Sichuan, ChinaP1 B44CY14PXW-18中国四川 Sichuan, ChinaP1 B45CY15PXW-31中国四川 Sichuan, ChinaP1 B46CY16PXW-35中国四川 Sichuan, ChinaP1 B47CY17PXW-58中国四川 Sichuan, ChinaP1 B49CY19PXW-65中国四川 Sichuan, ChinaP1 B52CY12GJ-1中国四川 Sichuan, ChinaP1 B53wx1025中国湖南 Hunan, ChinaP1 B54wx10213中国湖南 Hunan, ChinaP1 B55wx10296中国湖南 Hunan, ChinaP1 B56wx10315中国湖南 Hunan, ChinaP1 B5710-1043中国湖南 Hunan, ChinaP1 B6010-1070中国湖南 Hunan, ChinaP1 B6110-804中国湖南 Hunan, ChinaP1 B6210-1358中国湖南 Hunan, ChinaP1 B64Xiangyou13中国湖南 Hunan, ChinaP1 B65Xiangyou15中国湖南 Hunan, ChinaP1 B66Xiangyou11中国湖南 Hunan, ChinaP1 B67740中国湖南 Hunan, ChinaP1 B69613中国湖南 Hunan, ChinaP1 B70783中国湖南 Hunan, ChinaP1 B71782中国湖南 Hunan, ChinaP1 B72YB3中国湖南 Hunan, ChinaP1 B731360中国湖南 Hunan, ChinaP1 B75WX10329中国湖南 Hunan, ChinaP1 B76santana中国湖南 Hunan, ChinaP1 B771281中国湖南 Hunan, ChinaP1 B791368中国湖南 Hunan, ChinaP1 B801322中国湖南 Hunan, ChinaP1 B811252中国湖南 Hunan, ChinaP1 B821321中国湖南 Hunan, ChinaP1 B8407094中国湖北 Hubei, ChinaP1 B8507016中国湖北 Hubei, ChinaP1 B869F087中国湖北 Hubei, ChinaP1 B8797096中国湖北 Hubei, ChinaP1 B8897097中国湖北 Hubei, ChinaP1 B9007191中国湖北 Hubei, ChinaP1 B9107037中国湖北 Hubei, ChinaP1 B92RQ011中国湖北 Hubei, ChinaP1 B9796063中国湖北 Hubei, ChinaP1 B9801111中国湖北 Hubei, ChinaP1 B9901570中国湖北 Hubei, ChinaP1 B10202354中国湖北 Hubei, ChinaP1 B10593205中国湖北 Hubei, ChinaP1 B10693210中国湖北 Hubei, ChinaP1 B107Nca中国湖北 Hubei, ChinaP1 B108Zhongshuang4中国湖北 Hubei, ChinaP1 B109Zhongshuang9中国湖北 Hubei, ChinaP1 B110Zhongshuang11中国湖北 Hubei, ChinaP1 B1112011-6200中国湖北 Hubei, ChinaP1 B1152012-3448中国湖北 Hubei, ChinaP2 B1162012-3546中国湖北 Hubei, ChinaP1 B1182012-5086中国湖北 Hubei, ChinaP1 B1192012-5113中国湖北 Hubei, ChinaP1 B1202012-8327中国湖北 Hubei, ChinaP1 B1212012-8355中国湖北 Hubei, ChinaP1 B1222012-8380中国湖北 Hubei, ChinaP1 B1242012-9323中国湖北 Hubei, ChinaP1 B1272012-9478中国湖北 Hubei, ChinaP1 B1282012-9542中国湖北 Hubei, ChinaP1 B1292012-K8053中国湖北 Hubei, ChinaP1 B130R2中国湖北 Hubei, ChinaP1 B131Xiwang106中国湖北 Hubei, ChinaP1 B132Yangguang198中国湖北 Hubei, ChinaP1 B133Yangguang2009中国湖北 Hubei, ChinaP1 B134Zhongshuang10中国湖北 Hubei, ChinaP1 B135Zhongshuang12中国湖北 Hubei, ChinaP1 B136Zhongshuang4中国湖北 Hubei, ChinaP1 B137Zhongshuang6中国湖北Hubei, ChinaP1 B138Zhongshuang7中国湖北 Hubei, ChinaP1 B139Zhongyou589中国湖北 Hubei, ChinaP1 B140Zhongyou821中国湖北 Hubei, ChinaP1 B141Huayou2中国湖北 Hubei, ChinaP1 B142Major中国湖北 Hubei, ChinaP1 B143Huashuang2中国湖北 Hubei, ChinaP1 B144Aurora中国湖北 Hubei, ChinaP1 B145Huayou13中国湖北 Hubei, ChinaP1 B146Rucabo中国湖北 Hubei, ChinaP2 B147Huayou3中国湖北 Hubei, ChinaP1 B149Ningyou1中国湖北 Hubei, ChinaP1 B15111-9-700中国湖北 Hubei, ChinaP1 B15311-9-702中国湖北 Hubei, ChinaP1 B15411-9-703中国湖北 Hubei, ChinaP1 B15511-9-704中国湖北 Hubei, ChinaP1 B15611-9-705中国湖北 Hubei, ChinaP1 B15711-9-706中国湖北 Hubei, ChinaP1 B15811-9-707中国湖北 Hubei, ChinaP1 B15911-P63-5中国湖北 Hubei, ChinaP1 B16011-P63-8中国湖北 Hubei, ChinaP1 B16309-P64-1中国湖北 Hubei, ChinaP1 B16410-Cong23中国湖北 Hubei, ChinaP1 B16510-Cong24中国湖北 Hubei, ChinaP1 B16610-Cong25中国湖北 Hubei, ChinaP1 B16710-Cong29中国湖北 Hubei, ChinaP1 B16810-Cong32中国湖北 Hubei, ChinaP1 B16910-Cong33中国湖北 Hubei, ChinaP1 B17010-Cong34中国湖北 Hubei, ChinaP1 B17110-Jiangpeng2中国湖北 Hubei, ChinaP1 B17210-Jiangpeng3中国湖北 Hubei, ChinaP1 B17311-7-103中国湖北 Hubei, ChinaP1 B17411-7-117中国湖北 Hubei, ChinaP1 B17511-7-125中国湖北 Hubei, ChinaP1 B1787-7766-74中国湖北 Hubei, ChinaP1 B18064Peng-10中国湖北 Hubei, ChinaP1 B181Shengguang77中国湖北 Hubei, ChinaP1 B182Jiayu17中国湖北 Hubei, ChinaP1 B183Jiayu25中国湖北 Hubei, ChinaP1 B184Jiayu16中国湖北 Hubei, ChinaP1 B185Jiayu31中国湖北 Hubei, ChinaP1 B186Huashuang5中国湖北 Hubei, ChinaP1 B187Huashuang4中国湖北 Hubei, ChinaP1 B188Jia972中国湖北 Hubei, ChinaP1 B189Huashuang128中国湖北 Hubei, ChinaP1 B190Jia904中国湖北 Hubei, ChinaP1 B192JiaPF190中国湖北 Hubei, ChinaP1 B195Jia951中国湖北 Hubei, ChinaP1 B196Jia917中国湖北 Hubei, ChinaP1 B197Jia923中国湖北 Hubei, ChinaP1 B198Jia931中国湖北 Hubei, ChinaP1 B200Jia963中国湖北 Hubei, ChinaP2 B201Huyou17中国上海 Shanghai, ChinaP1 B202Huyou15中国上海 Shanghai, ChinaP1 B204Ningyou18中国江苏 Jiangsu, ChinaP1 B206Ningyou14中国江苏 Jiangsu, ChinaP1 B209Shilifeng中国江苏 Jiangsu, ChinaP1 B211Yangyou5中国江苏 Jiangsu, ChinaP1 B212Zhenyou3中国浙江 Zhejiang, ChinaP1 B213Hongyou3中国江苏 Jiangsu, ChinaP1 B215Zheyou18中国浙江 Zhejiang, ChinaP1 B216Zheshuang72中国浙江 Zhejiang, ChinaP1 B217Zheshuang8中国浙江 Zhejiang, ChinaP1 B218Zheyou758中国浙江 Zhejiang, ChinaP1 B219Huyou14中国上海 Shanghai, ChinaP1 B220Huyou18中国上海 Shanghai, ChinaP1 B221Huyou19中国上海 Shanghai, ChinaP1 B222Zheyou19中国浙江 Zhejiang, ChinaP1 B223Zheyou21中国浙江 Zhejiang, ChinaP1 B224Zheshuang6中国浙江 Zhejiang, ChinaP1 B226Wanyou16中国安徽 Anhui, ChinaP1 B227Wanyou20中国安徽 Anhui, ChinaP1 B228Wanyou29中国安徽 Anhui, ChinaP1 B241Weijie加拿大 CanadaP2 B243Zhizun加拿大 CanadaP1 B244Haisheng加拿大 CanadaP2 B245D2丹麦 DenmarkP2 B262P310中国陕西 Shanxi, ChinaP1 B263P312中国陕西 Shanxi, ChinaP2 B264P668中国陕西 Shanxi, ChinaP1 B265P685中国陕西 Shanxi, ChinaP1 B266A117中国陕西 Shanxi, ChinaP1 B267A172中国陕西 Shanxi, ChinaP1 B268B250中国陕西 Shanxi, ChinaP1 B269B265中国陕西 Shanxi, ChinaP1 B271B285中国陕西 Shanxi, ChinaP2 B273GY270中国陕西 Shanxi, ChinaP1 B274GY282中国陕西 Shanxi, ChinaP1 B275GY284中国陕西 Shanxi, ChinaP1 B279B414中国新疆 Xinjiang, ChinaP1 B29111-P30中国湖北 Hubei, ChinaP1 B30303LF1中国甘肃 Gansu, ChinaP2 B313SWU01中国湖北 Hubei, ChinaP1 B326Nonglin43中国重庆 Chongqing, ChinaP1 B327SWU25中国西藏 Tibet, ChinaP1 B328Youyan2中国重庆 Chongqing, ChinaP1 B330SWU41中国重庆 Chongqing, ChinaP1 B333SWU57中国重庆 Chongqing, ChinaP1 B334SWU60中国重庆 Chongqing, ChinaP1 B337SWU63中国重庆 Chongqing, ChinaP1 B339SWU66中国重庆 Chongqing, ChinaP1 B340SWU67中国重庆 Chongqing, ChinaP1 B341SWU68中国重庆 Chongqing, ChinaP1 B342SWU69中国重庆 Chongqing, ChinaP1 B343SWU70中国重庆 Chongqing, ChinaP1 B345SWU74中国重庆 Chongqing, ChinaP1 B346SWU75中国重庆 Chongqing, ChinaP1 B349SWU80中国重庆 Chongqing, ChinaP1 B350SWU81中国重庆 Chongqing, ChinaP1 B351SWU84中国重庆 Chongqing, ChinaP1 B354SWU88中国重庆 Chongqing, ChinaP1 B357SWU93中国重庆 Chongqing, ChinaP1 B358SWU94中国重庆 Chongqing, ChinaP1 B360SWU96中国重庆 Chongqing, ChinaP1 B361SWU99中国重庆 Chongqing, ChinaP1 B362SWU100中国重庆 Chongqing, ChinaP1 B363SWU102中国重庆 Chongqing, ChinaP1 B365SWU104中国重庆 Chongqing, ChinaP1 B366SWU105中国重庆 Chongqing, ChinaP1 B367SWU107中国重庆 Chongqing, ChinaP1 B368SWU110中国重庆 Chongqing, ChinaP1 B369SWU111中国重庆 Chongqing, ChinaP1 B370SWU112中国重庆 Chongqing, ChinaP1 B371SWU113中国重庆 Chongqing, ChinaP1 B373Zhongshuang10中国湖北 Hubei, ChinaP1 B375Zhongshuang7中国湖北 Hubei, ChinaP1 B376Zhongshuang12中国湖北 Hubei, ChinaP1 B377Zhongyou589中国湖北 Hubei, ChinaP1 B378Fuyou4中国湖北 Hubei, ChinaP1 B380Zhen2609中国江苏 Jiangsu, ChinaP1 B381HX0352中国江苏 Jiangsu, ChinaP1 B382Yangguang2009中国湖北 Hubei, ChinaP1 B383Huyou21中国上海 Shanghai, ChinaP1 B384Zheshuang3中国浙江 Zhejiang, ChinaP1 B385Zheyou21中国浙江 Zhejiang, ChinaP1 B386Wanyou20中国安徽 Anhui, ChinaP1 B387Wanyou12中国安徽 Anhui, ChinaP1 B389Hongyou3中国江苏 Jiangsu, ChinaP1 B390Zhenyou5中国湖北 Hubei, ChinaP1 B391Yangyou4中国湖北 Hubei, ChinaP1 B392Huyou15中国上海 Shanghai, ChinaP1 B393Huyou16中国上海 Shanghai, ChinaP1 B395Huyou18中国浙江 Zhejiang, ChinaP1 B396Zheshuang72中国浙江 Zhejiang, ChinaP1 B397Zheshuang8中国浙江 Zhejiang, ChinaP1 B398Zheyou50中国浙江 Zhejiang, ChinaP1 B399Suyou1中国江苏 Jiangsu, ChinaP1 B400Zhongshuang4中国湖北 Hubei, ChinaP1 B401Zhongshuang11中国湖北 Hubei, ChinaP1 B402Yangguang198中国湖北 Hubei, ChinaP1 B403Huahang901中国湖北 Hubei, ChinaP1 B404YangJ6711中国江苏 Jiangsu, ChinaP1 B406Yangjian8中国江苏 Jiangsu, ChinaP1 B407Xiwang106中国湖北 Hubei, ChinaP1 B408Zheyou17中国浙江 Zhejiang, ChinaP1 B409Zhongshuang5中国湖北 Hubei, ChinaP1 B410Zhongyou821中国湖北 Hubei, ChinaP1 B411Qinyou1中国湖北 Hubei, ChinaP1 B412Weilong88中国湖北 Hubei, ChinaP1 B414Qinyou5中国湖北 Hubei, ChinaP1 B415Suyou4中国江苏 Jiangsu, ChinaP1 B416Fengyou9中国湖北 Hubei, ChinaP1 B424Ningyou10中国湖北 Hubei, ChinaP1 B425Helios中国湖北 Hubei, ChinaP1 B427Mian96-203中国青海 Qinghai, ChinaP1 B428Qing662A中国湖北 Hubei, ChinaP1 B429699中国湖北 Hubei, ChinaP1 B430Canada2中国湖北 Hubei, ChinaP1 B432Zhongshuang9中国湖北 Hubei, ChinaP1 B433WH-12中国湖北 Hubei, ChinaP1 B435WH-17中国湖北 Hubei, ChinaP1 B436WH-19中国湖北 Hubei, ChinaP1 B437WH-20中国湖北 Hubei, ChinaP1 B438WH-23中国湖北 Hubei, ChinaP1 B439WH-24中国湖北 Hubei, ChinaP1 B440WH-25中国湖北 Hubei, ChinaP1 B442WH-27中国湖北 Hubei, ChinaP1 B443WH-28中国湖北 Hubei, ChinaP1 B445WH-30中国湖北 Hubei, ChinaP1 B446WH-31中国湖北 Hubei, ChinaP1 B447WH-33中国湖北 Hubei, ChinaP1 B451WH-42中国湖北 Hubei, ChinaP2 B452WH-43中国湖北 Hubei, ChinaP2 B453WH-45中国湖北 Hubei, ChinaP2 B454WH-49中国湖北 Hubei, ChinaP1 B455WH-50中国湖北 Hubei, ChinaP2 B458WH-57中国湖北 Hubei, ChinaP1 B459WH-58中国湖北 Hubei, ChinaP1 B460WH-59中国湖北 Hubei, ChinaP1 B466WH-83中国湖北 Hubei, ChinaP1 B475NY7中国湖北 Hubei, ChinaP1 B479Cubs root中国湖北 Hubei, ChinaP1 B482Shengli中国湖北 Hubei, ChinaP1 B486Daichousen中国湖北 Hubei, ChinaP1 B487comet中国湖北 Hubei, ChinaP2 B488Niklas中国湖北 Hubei, ChinaP2 B491WESBROOK中国湖北 Hubei, ChinaP2 B492Suigenshu中国湖北 Hubei, ChinaP1 B493Huayou4中国湖北 Hubei, ChinaP1 B494Y1中国上海 Shanghai, ChinaP1 B499Shaoyeqing中国上海 Shanghai, ChinaP1 B501Hujizao中国上海 Shanghai, ChinaP1 B504Fengding 240中国江苏 Jiangsu, ChinaP1 B506Dahuaqiu中国江苏 Jiangsu, ChinaP1 B508Ningyou 10中国江苏 Jiangsu, ChinaP1 B509Ningyou 8中国江苏 Jiangsu, ChinaP1 B510Ningyou 6中国江苏 Jiangsu, ChinaP1 B511Duoyou 1中国江苏 Jiangsu, ChinaP1 B513Huaiyou 12中国江苏 Jiangsu, ChinaP1 B514Peixuan 170中国江苏 Jiangsu, ChinaP1 B516Guangde 8104中国安徽 Anhui, ChinaP1 B517Dangyouzao 1中国安徽 Anhui, ChinaP1 B518Guangde 761中国安徽 Anhui, ChinaP1 B521Chu 107中国安徽 Anhui, ChinaP1 B526Jie 65-1中国浙江 Zhejiang, ChinaP1 B527Shenhuang1中国上海 Shanghai, ChinaP1 B528Zheyou 601中国浙江 Zhejiang, ChinaP1 B529Sangao rapeseed中国浙江 Zhejiang, ChinaP1
Genome-wide association study of root length and hypocotyl length at germination stageunder saline conditions in
ZHANG Rui1, DENG WenYa1, YANG Liu1, WANG YaPing1, XIAO FangZhi2, HE Jian2, LU Kun1
(1College of Agronomy and Biotechnology, Southwest University, Chongqing 400715;2Shennong Class, Southwest University, Chongqing 400715)
【Objective】 Identification of the regulatory loci and candidate genes governing salt stress tolerance inat germination stage could lay the foundation for improvement ofsalt resistance. 【Method】 In this study, 317 representativeinbred lines were genotyped under normal and salt-stressed conditions in a sand culture system. Significant SNPs associated with root length and hypocotyl length inunder normal and saline stress conditions and their linkage disequilibrium (LD) were determined by genome-wide association studies (GWAS), based on the60 K SNP array. Candidate genes were selected based on the combination analyses results of functional annotation of genes within the LD blocks and transcriptome analyses of seedling roots and leaves inunder saline stress treatments. Accuracy of candidate gene selection was improved by real-time quantitative reverse transcriptase PCR (qRT-PCR). 【Result】Hypocotyl length and root length ofshowed large variation among accessions at germination stage under normal and salt-stressed conditions, and frequency distribution revealed that all the target traits were quantitative traits and controlled by polygenic genes. Comparison of different models showed that MLM+P+K model was the optimal model. Based on this model, GWAS identified 45 loci significantly associated with target traits, including 40 and 5 SNPs associated with hypocotyl length and root length, and each of SNP explained 9.12%-14.46% and 7.67%-8.93% of phenotypic variation, respectively. Among the significantly associated SNPs, rs8970 on chromosome C04 was the most notable, since it was the only SNP, which could be repeatedly detected between root length and hypocotyl length, and associated with four traits simultaneously, explaining 7.67%-12.35% of observed phenotypic variation. Of the 11 important significantly associated SNPs, 6 SNPs were distributed in 10 to 442 kb of linkage disequilibrium (LD) blocks. By combining differentially expressed genes detected by transcriptome analysis with LD block identification, 447 genes were identified within the 11 important LD intervals, of which 15 were activated by salt stress.,,,,,,,,,-andwere most likely the candidate genes within their LD blocks. Results of qRT-PCR detection showed that 10 candidate genes were induced by salt stress treatment in root or hypocotyl at germination stage, except for. In addition, tissue-specificity detection of candidate genes also showed that,andmainly expressed in the root and hypocotyl at germination stage, and,andpossessed the highest expression in hypocotyl, confirmed that these genes might be involved in development of root and hypocotyl and salt resistance ofat germination stage. 【Conclusion】 A total of 45 significantly associated SNPs controlling development and salt resistance in root and hypocotyl ofat germination stage were identified by GWAS. By combined LD block identification, transcriptome analyses and functional annotation, 11 important candidate genes were screened within different LD blocks.
; germination; salt tolerance; genome-wide association study; transcriptome
2016-07-01;接受日期:2016-09-13
国家自然科学基金(31571701)、高等学校学科创新引智计划(B12006)、中央高校基本科研业务费专项资金(XDJK2012A009和XDJK2014D011)、国家级大学生创新创业训练计划(201410635040)、西南大学本科生科技创新基金(20153701002)
张蕊,E-mail:2593709868@qq.com。邓文亚,E-mail:1534159333@qq.com。张蕊和邓文亚为同等贡献作者。通信作者卢坤,Tel:023-68251264;E-mail:drlukun@swu.edu.cn

