中国玉米审定品种标准SSR指纹库的构建
王凤格,杨扬,易红梅,赵久然,任洁,王璐,葛建镕,江彬,张宪晨,田红丽,侯振华
中国玉米审定品种标准SSR指纹库的构建
王凤格1,杨扬1,易红梅1,赵久然1,任洁1,王璐1,葛建镕1,江彬2,张宪晨2,田红丽1,侯振华1
(1北京市农林科学院玉米研究中心/玉米DNA指纹及分子育种北京市重点实验室,北京100097;2北京华生恒业科技有限公司,北京100083)
【目的】对数量庞大的已知玉米品种构建可共享的作物品种标准DNA指纹库。【方法】基于荧光毛细管电泳检测平台和植物品种DNA指纹库管理系统,利用筛选的40对SSR核心引物对3 998份中国玉米审定品种标准样品进行建库,通过多实验室、多检测平台进行建库数据的质量评估。【结果】绘制了40个玉米建库引物的等位基因频率分布图作为每个引物的特征图谱,在建库试验中发挥了相当于参照样品的作用。形成了一套十重荧光毛细管电泳组合,并在SSR指纹分析器中建立了一套系统默认PANEL作为不同实验室建库时的标准PANEL。统计玉米审定品种指纹库构建的试验情况,每份样品具有2—5套原始试验数据及对应的指纹图谱,其中61%的样品做了2组独立试验,33%的样品做了3组独立试验,最终建成的标准指纹库累计缺失和差异位点仅占0.2%,数据完整性达到99.8%。在不同实验室、不同电泳检测平台的评估结果表明,同一荧光电泳检测平台上获得SSR指纹数据一致性高,而不同的电泳检测平台获得的数据存在一定偏差,为实现不同实验室的SSR指纹数据共享,需要统一荧光引物、分析软件及电泳检测平台。对所有审定品种指纹数据进行整体两两比较,表明中国玉米审定品种之间差异比较大,品种间差异位点百分比集中在80%—95%(占78.28%),品种间差异位点百分比在50%以上的已达到99.21%,而低于20%的只有0.09%;对玉米审定品种杂合率水平进行分析,平均品种杂合率达到64%,主要集中在50%—80%(占89%)。通过玉米品种标准指纹比对服务平台(网址:http://www.maizedna.org/)实现了指纹库的共享。【结论】形成了构建作物品种SSR指纹库的标准化程序,构建了3 998份玉米审定品种的SSR指纹库,通过多实验室联合比较试验,保证建库数据的准确性和数据库的可共享性;建立了玉米品种标准指纹比对服务平台网站,实现玉米审定品种指纹库在全国种子检验系统的共享。
玉米;品种鉴定;DNA指纹库;SSR
0 引言
【研究意义】主要农作物品种审定登记制度实施以来,审定品种的数量呈逐年增多趋势,截至2013年底,玉米审定品种数量就达到了6 291个[1],对品种高效管理带来了挑战;随着玉米品种资源遗传基础日趋狭窄及少数骨干亲本被集中应用,出现了一批在原品种基础上仅做细微改良的派生品种,对品种鉴定技术提出了更高要求;品种审定后进入生产推广环节,出现标签名称与实际包装的种子不符等品种真实性问题,增加了种子市场监管的难度。构建高质量的玉米审定品种标准DNA指纹库对解决上述问题和需求具有重要意义,将为政府的品种管理、企业的品种维权、农民的利益维护提供强有力的技术支撑[2]。【前人研究进展】许多作物都开展了品种DNA指纹库构建,其中开展较早或规模较大的主要有玉米[3-4]、水稻[5]、小麦[6]、油菜[7]、马铃薯[8]、番茄[9]、月季[10]、葡萄[11]等,所用建库技术主要为SSR、SNP、INDEL、AFLP、ISSR等标记也有少量使用。早期由于SSR引物开发难度大,引物筛选评价不够充分,试验程序标准化程度较低,且大部分是基于PAGE凝胶电泳检测平台建立起来的,不同实验室数据可比性较差;近年来随着引物开发数量的增多,对引物筛选和评估的强度提高,建库程序的逐步标准化,以及荧光毛细管电泳检测平台的广泛使用,所建指纹库的质量得到一定提高。【本研究切入点】总结以往作物品种指纹库构建的工作,在以下几个方面尚待进一步完善:一是未能彻底解决不同实验室数据整合和共享的问题,不同实验室数据采集不一致情况未能提出根本的解决方案[12-13];二是所建指纹库多数为选取几十到几百个代表性品种的规模较小的研究型数据库,建库样品为自行收集的材料或种质资源[14-15],并非来自官方登记备案的标准样品;三是所建数据库停留在研究阶段,未经过大量检验实践的充分验证。【拟解决的关键问题】为从根本上解决玉米品种SSR指纹数据整合共享的问题,本研究通过对试验程序、引物特征参数的设置、引物分组的设置、检测平台、分析软件、数据库管理系统进一步规范,确立大规模构建高质量可共享指纹库的切实可行方案;构建规模达到近4 000份的玉米审定品种标准样品SSR指纹库,通过多实验室联合比较试验,保证建库数据的准确性和数据库的可共享性,并通过建立指纹比对平台网站,实现玉米审定品种指纹库在全国种子检验系统的应用。
1 材料与方法
1.1 试验材料
建库样品共3 998份,由国家种质库提供,系国家和省级农作物品种审定委员会向品种选育单位收集或在区域试验种子中留取的玉米审定品种标准样品,涉及3 806份玉米审定品种,包括3 361个普通玉米品种(3 540份样品)和445个甜糯玉米品种(460份样品),其中168个品种有2—4个不同来源的同名样品,这些品种基本涵盖了中国曾经推广或正在推广使用的玉米杂交种。
1.2 基因组DNA的提取
采取改良的CTAB法提取DNA:每份供试样品均随机抽取50粒种子形成混合样品,充分磨碎后移入2.0 mL离心管;加入700 µL CTAB提取液,65℃水浴60 min;加入等体积的三氯甲烷/异戊醇(24﹕1)并充分混合,静置10 min后12 000 r/min离心15 min;上清液加入等体积的预冷异丙醇沉淀DNA,12 000 r/min离心10 min,弃上清液;加入70%乙醇清洗2次,晾干后加入100 µL超纯水,充分溶解后备用。除了供试样品外,另外选用一份玉米DH系作为参照样品进行大量DNA提取。同一批次提取的DNA样品,随机抽取约5%进行DNA质量和浓度测定,根据测量值估算稀释成工作液的倍数后对该批DNA统一进行稀释。采用NanoDrop 2000 (Thermo Scientific)紫外分光光度计进行DNA质量和浓度测定。
1.3 SSR标记分析
40对建库引物序列信息、采用的PCR反应体系和反应程序以及荧光毛细管电泳检测程序见行业标准《玉米品种鉴定技术规程 SSR标记法》(NY/T 1432-2014)和已发表的论文[16-17]。每对引物其中一条的5′端用一种荧光染料进行标记,共选用了PET、NED、VIC、FAM 4种荧光染料(Applied Biosystems,USA公司合成)。PCR反应在Veriti 384 well Thermal cycler(Applied Biosystem)上进行,荧光毛细管电泳在ABI 3730XL DNA analyzer(Applied Biosystem)上进行,分子量内标采用LIZ 500。
采用仪器自带的Date Collection Ver.1.0软件收集原始数据并形成FSA文件。采用北京市农林科学院玉米研究中心与北京华生恒业公司合作开发的SSR指纹分析器(软件登记号:2015SR161217)对FSA文件进行分析,分析时设置的引物特征参数见表1。
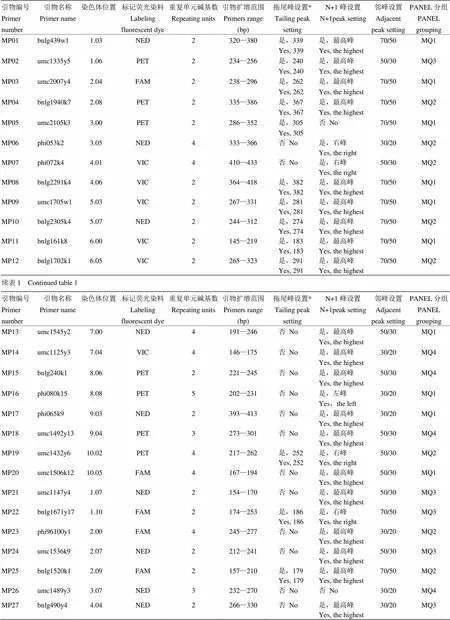
表1 建库引物指纹分析特征参数设置表
*拖尾峰设置时先设置是否存在拖尾峰,如果是,进一步确定拖尾峰的起点,用等位基因值表示
* first set the existence of tailing peak. if it is, further determine the starting point of tailing peak with the allele value
1.4 建库及数据统计分析
建库过程中采用了植物品种DNA指纹库管理系统(以下简称指纹库管理系统,登记号:2015SR085905),安排至少两组独立平行试验,选取60%以上组数的试验数据一致且试验质量较高的数据和指纹图谱进入标准指纹库,对多组试验中数据不一致而指纹图谱一致的位点,通过人工选择一组试验质量较高的数据和指纹进入标准指纹库,对数据缺失严重的样品,启动新一组独立试验,直到形成位点缺失率控制在5%以下的标准指纹库。
采用Power-Marker ver.3.25进行SSR引物位点的等位基因信息统计,采用Microsoft Excel 2010进行引物等位基因频率分布图及差异位点分布图的绘制。
1.5 建库数据的质量评估
为评估所构建SSR指纹库的质量,检验指纹库共享的可能性,开展如下几项评估试验:(1)从建成的标准指纹库中随机抽取200份样品的DNA(约占总数5%,名单略),匿名编号后提供给2个实验室进行检测并与标准指纹库的数据进行对比,采用与构建标准指纹库时相同的建库程序和电泳检测平台,只是仪器型号略有不同,评估不同实验室在同一荧光电泳检测平台上数据采集的一致性和利用指纹库进行成对比较的结果一致性。(2)从标准指纹库中挑选8份代表性样品的DNA(名单略),由北京玉米种子检测中心实验室统一进行PCR扩增,扩增产物提供给具有贝克曼GenomeLabGeXP遗传分析仪的三家实验室进行检测并与标准指纹库的指纹进行对比,评估在不同荧光电泳检测平台上数据采集的一致性和利用指纹库进行成对比较的结果一致性。
1.6 指纹比对平台网站的建立
为了实现指纹库在全国种子检验系统中的共享使用,建立玉米品种标准指纹比对服务平台,平台上可以查看玉米审定品种标准指纹库,并提供了全库比较、疑似比较、同名比较、范围内互比等便捷的比对功能。
2 结果
2.1 引物等位基因频率分布及多态性评估
对建库引物的总体多态性情况进行了全面的评估,从等位基因个数、基因型个数、PIC值、个体识别率、遗传多样性等多个参数分析,40个建库引物总体多态性较高(表2),这套核心引物是通过代表性玉米自交系和杂交种品种筛选评估后入选的高多态性的引物,表明玉米审定品种大规模建库对引物的评估结果与以往的筛选评估结果基本一致。
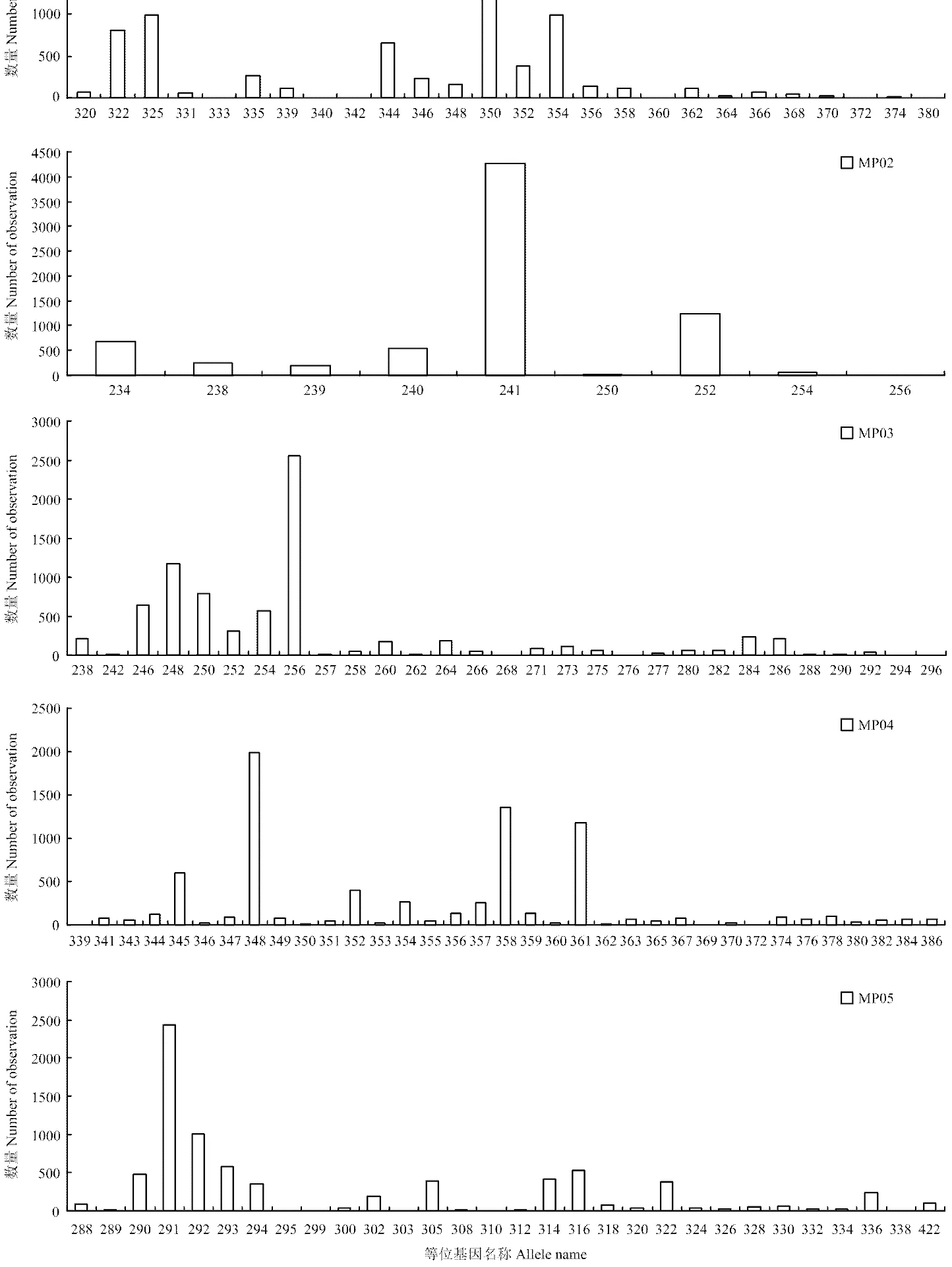
通过统计审定品种在40个引物位点上的等位基因频率分布情况,绘制了40个玉米建库引物的等位基因频率分布图(图1),由于不同引物在等位基因数、等位基因频率上具有不同的分布特征,引物的等位基因频率分布图可以作为每个引物的特征图谱。
一般情况下,荧光毛细管电泳叠加峰图与相应引物的等位基因频率分布图的图型特征应基本相似,因此,在具体建库试验中,引物等位基因频率分布图可以发挥与参照样品相似的作用。以引物MP35为例(图2),该引物的等位基因频率分布图(A)的基本特征可以概括为175、183频率高,175、180、188、189、193频率中等,178、181、186、192频率低,除此之外的其他等位基因应没有或极其稀有;未校正前的原始荧光毛细管电泳叠加峰图(B1)则178、186频率高,而175、183频率中或低,并出现了2个新等位基因。这显然与引物MP35的特征不符;对峰图进行整体向右移动3 bp的校正后的荧光毛细管电泳叠加峰图(B2)则完全符合引物MP35的等位基因频率分布图特征,表明即使没有参照样品,通过对照引物的等位基因频率分布图也可以实现数据的准确读取。
2.2 十重荧光毛细管电泳组合及PANEL的建立
基于40个玉米建库引物的引物扩增范围、等位基因分布等特征,形成了一套十重荧光毛细管电泳组合,并进一步在SSR analyzer中设置了相应的系统默认PANEL(表3),该套PANEL分4组,每组中均包括10个引物,相同颜色的荧光组合了2—3个引物。这套PANEL相当于不同实验室建库时的标准语言,有助于不同实验室建库数据的共享,并大大提高了建库的效率。
2.3 指纹库构建情况分析
统计审定品种指纹库构建的试验情况,61%的样品做了2组独立试验,33%的样品做了3组独立试验,6%的样品做了4或5组独立试验,每份样品一般具有2—5套原始试验数据及对应的指纹图谱。样品指纹负责人对多组试验数据进行审核后,缺失的位点占0.15%;不同组试验存在数据差异的位点占6.08%。分析数据差异的原因,表明大部分是由于样品在个别位点上一致性较差形成高低峰或多峰,介于阈值附近的峰在不同组试验中峰高存在细微差异,造成不同组自动采集的数据不同,对这些差异位点进一步人工比较指纹图谱,6.03%的位点指纹图谱一致并经人工审核后入标准指纹库。最终建成的审定品种标准指纹库累计缺失和差异位点仅占0.2%,数据完整性达到99.8%,标准指纹库的数据和图谱均来自至少2组指纹图谱一致的独立试验数据,数据准确性高。

表2 40个建库SSR引物多态性评估表
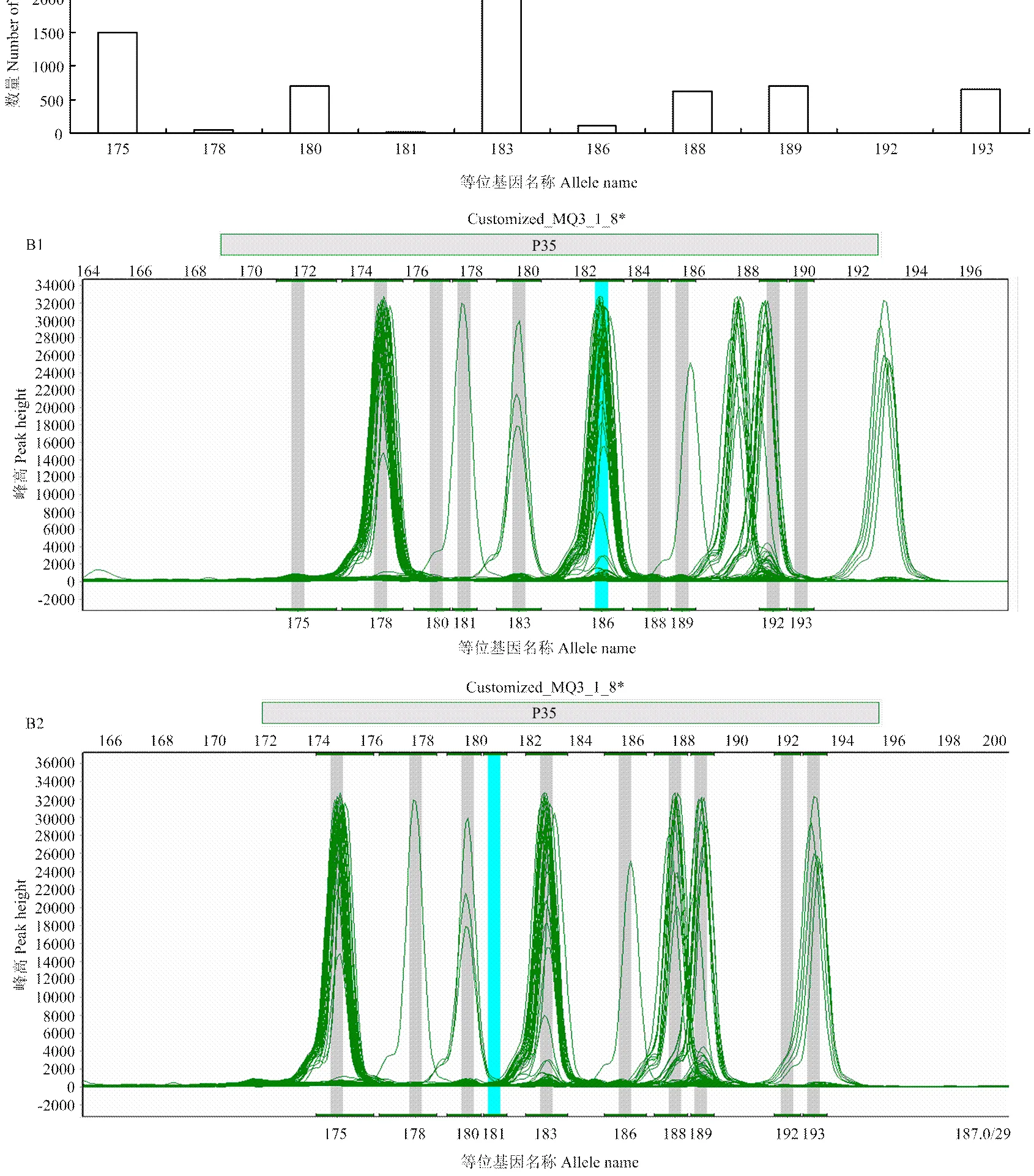
A:引物等位基因频率分布图;B1:未校正位置的荧光毛细管电泳叠加峰图;B2:校正位置后的荧光毛细管电泳叠加峰图(整体向右移动3 bp)

表3 建库SSR引物Panel组合信息表
不同实验室在相同电泳检测平台上的试验结果显示,37个建库引物表现出高度的试验稳定性和重复性,不同实验室采集数据的差异低于5%,采集的指纹图谱一致,指定样品的成对比较结果也是一致的;3个建库引物(M02、MP18、MP38)在不同实验室采集数据的差异略高于5%,今后可以考虑筛选出更好的建库引物进行替换。不同实验室在不同电泳平台上试验的结果显示:同一样品DNA在3台GenomeLabGeXP遗传分析仪上获得的原始数据的差异在0.5 bp以内;而同一样品DNA在GenomeLabGeXP遗传分析仪与ABI 3730XL遗传分析仪上获得的原始数据存在1—5 bp的差异。评估结果表明,同一荧光电泳检测平台上获得SSR指纹数据一致性高,而不同的电泳检测平台获得的数据存在一定偏差,因此,要想实现不同实验室的数据共享,获得高度一致的SSR指纹图谱和高度一致的样品成对比较结果,需要统一荧光引物、统一分析软件及统一电泳检测平台。
2.4 审定品种指纹库整体情况
对审定品种指纹数据进行两两比较,共涉及7 332 535对比较,结果表明,中国玉米审定品种之间差异比较大,品种间差异位点百分比集中在80%—95%(占78.28%),品种间差异位点百分比在50%以上的已达到99.21%,而低于20%的只有0.09%(表4)。由于这些审定品种代表了中国生产上推广的几乎全部品种,这一信息对今后的实际品种鉴定具有一定参考价值:由于中国玉米审定品种之间差异较大,因此,实际品种鉴定中当2个样品之间差异位点百分比低于50%时,就需要考察2个样品是否存在亲缘关系或具有共同亲本。
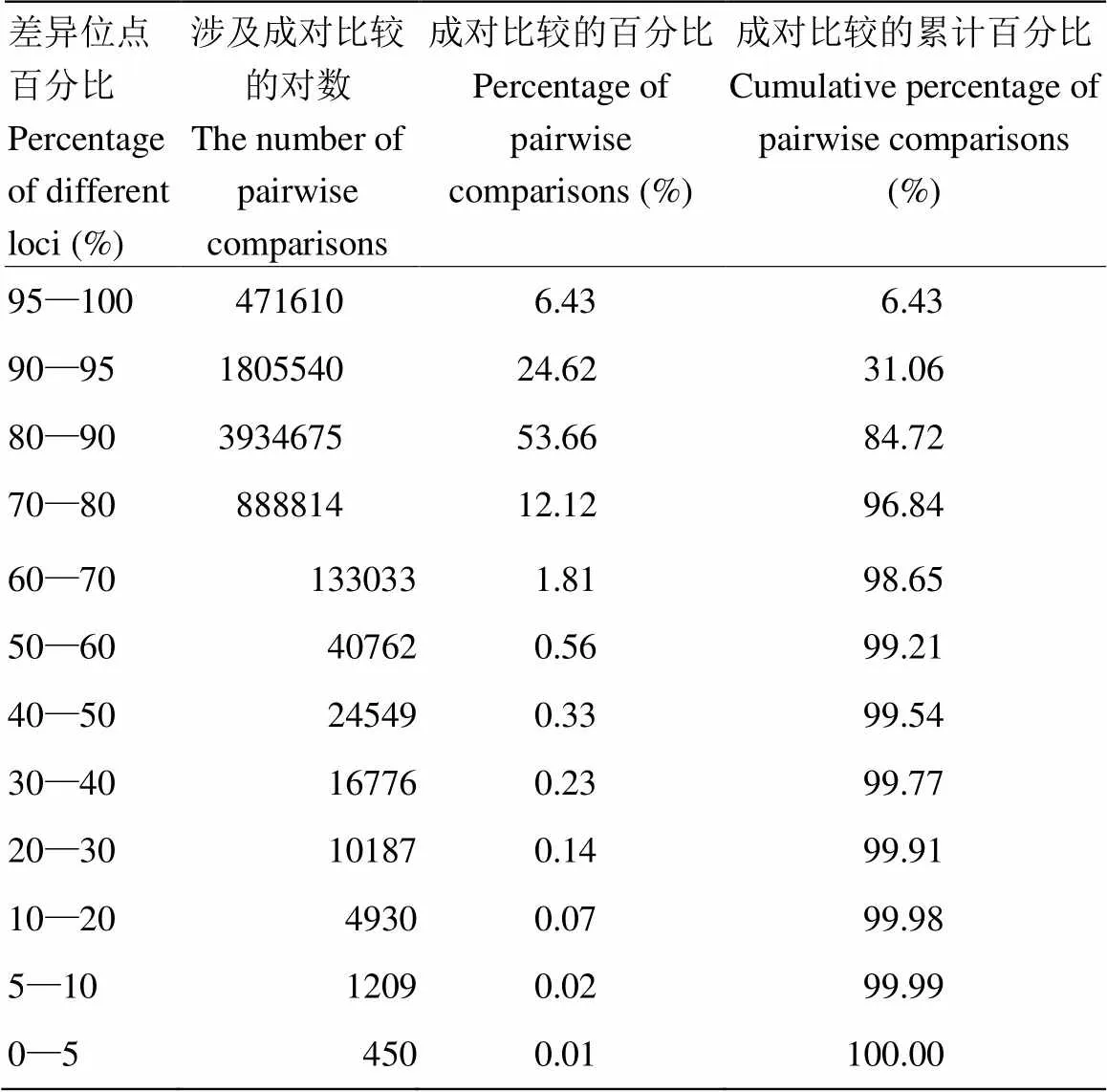
表4 玉米审定品种指纹数据两两比较情况
对玉米审定品种杂合率水平进行分析(表略),平均品种杂合率达到64%,主要集中在50%—80%(占89%),表明对大部分玉米审定品种而言,40核心引物中均能筛选到20—30个双亲互补型引物,这对利用指纹库筛选玉米审定品种的纯度鉴定引物具有重要价值。
322份样品(约占8%)存在2—3个样品指纹相同的情况,对这322份样品已经提交审定标准样品管理机构,正在进一步核实是否来自于同一品种,如果来自同一品种,应在标准样品库中将指纹相同的样品保留一份,其他进行合并或清理,如果来自不同品种,应进一步提供其他差异。标准样品库的清理工作完成后,就能够保证一个品种名称只有一个唯一的指纹图谱且相同的指纹图谱只对应一个品种名称,从而大大提高审定品种标准指纹库在真实性检测实践中的应用价值。
2.5 指纹库的应用效果
玉米品种标准指纹比对服务平台(网址:http://www.maizedna.org/)自2015年正式上线(网站界面见图4),平台上提供了已建成的全部玉米审定品种SSR指纹。该平台已向农业部授权的省级种子检测机构开放,甘肃、北京、河南等多个省级种子检测机构已经开始使用,在市场监督抽查、区试、品种权执法、企业维权等真实性检测方面已发挥了重要作用:在没有指纹比对平台之前,开展玉米真实性检测时,需要将待测样品和审定品种的标准样品一起进行成对比较试验,这意味着检测机构每次都要向种质库申请相应的标准样品进行试验,不仅会造成标准样品频繁出库,而且使室内检验的工作量翻倍;有了指纹比对平台后,则只需对待测样品进行试验,然后将待测样品指纹与平台的标准指纹进行比对就可以直接出具检测结果。

图4 指纹比对服务平台网站界面
3 讨论
3.1 指纹库数据的采集
指纹库构建时采用什么样的数据记录方式不仅影响数据库构建的难易程度,还影响数据库使用的方便程度。汇总以往指纹库构建时采用的数据记录方式,主要分为以下2种:
第一种是0/1编号,即按照电泳谱带的有无记录,有带记录为1,无带记录为0,这种方式在早期构建的指纹库中被使用过[12],现在的建库中已经较少使用。
第二种是等位基因代码,根据等位基因代码的编号方式进一步分为3类:第1类是按等位基因扩增产物大小的顺序用阿拉伯数字依次编号[18],部分研究者进一步按基因型进行了编码[19],这在小规模建库时具有简单直观的优点[20],但当建库规模扩大时面临新增等位基因无法编号的问题以及多实验室数据不易整合的问题;第2类是主要依据重复序列的重复次数对等位基因命名,这种方式具有命名严谨规范的优点,当采用多种检测平台建库时数据兼容性更强,在人类指纹库构建中采用了这种命名方式[21],选择的建库引物主要是4—5碱基重复类型的,个别作物如葡萄等通过重新开发和筛选合适的引物也开始使用这种方式[11],但这种命名方式对标记的选择比较严苛,2碱基重复类型的引物由于拖尾峰等峰型的干扰和扩增区间出现插入缺失的干扰不易准确确定碱基重复次数,不适合采用这种命名方式。由于大部分作物在筛选建库引物时主要考虑了引物多态性和试验重复稳定性,而2碱基重复类型的SSR引物的开发数量和多态性普遍高于3碱基以上的引物,造成建库引物中2碱基重复类型的SSR引物比例较高,因此,大部分作物目前不适合采用重复次数的命名方式;第3类是依据等位基因扩增产物大小对等位基因命名,这种方式不仅有利于不同实验室数据的整合,而且对标记的要求有所降低,在大规模建库中对扩增区间出现的插入缺失具有较高的容忍度,是适合目前大多数作物品种SSR指纹库构建的方式,本研究对玉米审定品种建库即采用了这种数据采集方式。
3.2 指纹库的级别
根据指纹库的实际使用价值不同,可将指纹库分为3个级别:初级指纹库仅起辅助筛查的作用;中级指纹库能够利用指纹库直接出具部分检测结果,仍有部分样品需进一步增加试验才能出具结果;高级指纹库能够利用指纹库直接出具几乎全部检测结果。
除了人类和少数动植物物种外,目前基于SSR标记建立的大部分作物指纹库仍只能起到辅助筛查的作用,而能否调取原始指纹图谱,是影响作物SSR指纹库利用价值的关键因素:与人类以个体为基础的指纹库不同,作物品种指纹库构建时存在品种内部不同个体的一致性问题,如果只采集数据,则只能反映该品种的主要等位基因信息,如果同时采集指纹图谱则不仅能反映所有等位基因及其大致比例,也能反映试验质量和数据的可靠程度,因此只有整合了数据和指纹图谱的作物品种SSR指纹库才有可能发挥直接出具检测结果的价值。本研究构建的玉米审定品种SSR指纹库通过同时采集数据和指纹图谱入库,实现了能够直接出具检测结果的目标,对其他作物品种SSR指纹库构建重要借鉴价值。
以往的研究者还在水稻[22]、高粱[15]、油菜[14]、百合[19]、大豆[23]等许多作物上开展了分子身份证的研制,通过将指纹数据转换为条形码或二维码的形式制作分子身份证,部分分子身份证还加入了商品信息等其它辅助信息[22],但尚未见综合利用分子数据和指纹图谱信息的报道。下一代的分子身份证可采取与指纹库网站建立链接的方式,建立每个品种的在网站上的二维码,通过扫描二维码,即可调取该品种的分子数据信息及指纹图谱信息。
3.3 可共享SSR指纹库的构建
SSR标记具有多等位基因的特点,这一特点对指纹库构建带来正反两方面的影响。从正面影响看,一是标记多态性高,区分品种能力强,一个SSR标记大约相当于10个SNP标记的区分能力[24],用较少标记就能对大量品种进行有效区分;二是基因型类型较多,可以采用混合样品建库,当进行样品成对比较时,只要在比较的位点上不存在共同等位基因,即使样品一致性差,也可以明确判定为差异位点,而SNP标记基因型最多只有3个,在样品一致性差的情况下无法采用混合样品建库及成对比较;三是杂交种的杂合率比较高,可以很容易筛选到双亲互补型的引物用于杂交种纯度鉴定。从负面影响看,一是SSR标记等位基因数一般在2个以上,不属于二态的标记类型,基因分型主要采用基于长度多态的电泳检测平台,检测通量一般在10个位点以内,很难实现位点高通量的检测;二是等位基因分布比较密集带来不同实验室、不同检测平台数据整合的难度。对于SSR检测通量低问题,今后通过扩增产物测序的方式有可能实现一次检测几千个SSR位点。实现几百到几千个位点的复合检测[25]。对于指纹库数据整合的问题,目前采取在建库方案、取样方案、检测平台、指纹分析工具和引物设置等方面进行统一规范的方式实现SSR数据整合[26],今后可通过开发重复次数严格按照重复单元的倍数增减的3—5碱基重复类型的玉米SSR引物,并在指纹数据采集时依据重复序列的重复次数对等位基因命名,从而大大降低共享数据库构建的难度。
在建库程序标准化的基础上,为进一步消除不同实验室或同一实验室不同时期试验的系统误差,还需要通过参照样品进行数据的校正。荧光毛细管电泳检测平台上只需提供1份参照样品就可发挥校正系统误差的作用,在玉米指纹库构建过程中,为试验设计方便,将每块电泳板的最后一个孔位作为放置参照样品扩增产物的位置,SSR指纹分析器在指纹分析过程中,依据该位置的已知标准指纹对PANEL进行左右整体位移,直到参照样品的读取指纹与其已知标准指纹一致。参照样品在作物品种共享指纹库构建中的价值已经得到了认可,然而选用什么样的参照样品,以及如何使用参照样品还有待商榷:部分研究者推荐的参照样品是从国家库中选取的一套代表建库引物的主要等位基因的标准样品[27]。从今后的检测实践看,参照样品应同时具有样品高度一致和容易大量提供的特点,由指定实验室统一繁殖并统一提供比从国家库取用更加方便,一致性高的自交系(或DH系)比杂交种更适合作参照样品。
4 结论
通过对试验程序、引物特征参数的设置、引物分组的设置、检测平台、分析软件、数据库管理系统进一步规范,形成了构建作物品种SSR指纹库的标准化程序。构建了近4 000份高质量玉米审定品种的SSR指纹库,建立了玉米品种标准指纹比对服务平台网站,实现了玉米审定品种指纹库在全国种子检验系统中的共享。
References
[1] 杨扬, 王凤格, 赵久然, 刘亚维. 中国玉米品种审定现状分析. 中国农业科学, 2014, 47(22): 4360-4370.
Yang Y, Wang F G, Zhao J R, Liu Y W. Analysis of the current situation of accredited maize varieties in China., 2014, 47(22): 4360-4370. (in Chinese)
[2] 赵久然, 王凤格, 易红梅, 田红丽, 杨扬. 我国玉米品种标准DNA指纹库构建研究及应用进展. 作物杂志, 2015, 2: 1-6.
Zhao J R, Wang F G, Yi H M, Tian H L, Yang Y. Progress of construction of chinese maize varieties standard DNA fingerprint database., 2015, 2: 1-6. (in Chinese)
[3] Van Inghelandt D V, Melchinger A E, Lebreton C, Stich B. Population structure and genetic diversity in a commercial maize breeding program assessed with SSR and SNP markers., 2010, 120(7): 1289-1299.
[4] Wang F G, Tian H L, Zhao J R, Yi H M, Wang L, Song W. Development and characterization of a core set of SSR markers for fingerprinting analysis of Chinese maize varieties., 2011, 56(1): 7-17.
[5] 庄杰云, 施勇烽, 应杰政, 鄂志国, 曾瑞珍, 陈洁, 朱智伟. 中国主栽水稻品种微卫星标记数据库的初步构建. 中国水稻科学, 2006, 20(5): 460-468.
Zhuang J Y, Shi Y F, Ying J Z, E Z G, Zeng R Z, Chen J, Zhu Z W. Construction and testing of primary microsatellite database of major rice varieties in china., 2006, 20(5): 460-468. (in Chinese)
[6] Röder M, Wendehake K, Korzun V, Bredemeijer G, Laborie D, Bertrand L, Isaac P, Rendell S, Jackson J, Cooke R, Vosman B. Construction and analysis of a microsatellite-based database of European wheat varieties., 2002, 106(1): 67-73.
[7] 赖运平, 王丽容, 何巧林, 张浙峰, 张新明, 堵苑苑, 余毅. 甘蓝型油菜核心SSR引物筛选及指纹图谱构建. 西南农业学报, 2014, 27(6): 2290-2298.
Lai Y P, Wang L R, He Q L, Zhang Z F, Zhang X M, Du Y Y, Yu Y. Screening of SSR core primers and fingerprinting construction ofL.., 2014, 27(6): 2290-2298. (in Chinese)
[8] Reid A, Hof L, Felix G, Rücker B, Tams S, Milczynska E, Esselink D, Uenk G, Vosman B, Weitz A. Construction of an integrated microsatellite and key morphological characteristic database of potato varieties on the EU common catalogue., 2011, 182(2): 239-249.
[9] Bredemeijer G, Cooke R, Ganal M, Peeters R, Isaac P, Noordijk Y, Rendell S, Jackson J, Röder M, Wendehake K, Dijcks M, Amelaine M, Wickaert V, Bertrand L, Vosman B. Construction and testing of a microsatellite database containing more than 500 tomato varieties., 2002, 105(6): 1019-1026.
[10] Smulders M, Esselink D, Voorrips R E, Vosmana B. Analysis of a database of DNA profiles of 734 hybrid tea rose varieties., 2009, 836: 169-174.
[11] Cipriani G, Spadotto A, Jurman I, Gaspero G D, Crespan M, Meneghetti S, Frare E, Vignani R, Cresti M. The SSR-based molecular profile of 1005 grapevine (L.) accessions uncovers new synonymy and parentages, and reveals a large admixture amongst varieties of different geographic origin., 2010, 121(8): 1569-1585.
[12] GEORGE M L, REGALADO E, LI W, CAO M, DAHLAN M, PABENDON M, WARBURTON M L, XIANCHUN X, HOISINGTON D. Molecular characterization of Asian maize inbred lines by multiple laboratories., 2004, 109(1): 80-91.
[13] Côté M, Leduc L, Reid A. Evaluation of simple sequence repeat (SSR) markers established in europe as a method for the identification of potato varieties grown in canada., 2013, 90(4): 340-350.
[14] 马琳, 刘海珍, 陆徐忠, 倪金龙, 张小娟, 杨剑波. 130份甘蓝型油菜种质分子身份证的构建. 中国油料作物学报, 2013, 35: 231-239.
Ma L, Liu H Z, Lu X Z, Ni J L, Zhang X J, Yang J B. Establishment molecular identity of 130varieties., 2013, 35: 231-239. (in Chinese)
[15] 王黎明, 焦少杰, 姜艳喜, 严洪冬, 苏德峰, 孙广全. 142 份甜高粱品种的分子身份证构建. 作物学报, 2011, 37(11): 1975-1983.
WANG L M, JIAO S J, JIANG Y X, YAN H D, SU D F, SUN G Q. Establishment of molecular identity in 142 sweet sorghum varieties., 2011, 37(11): 1975-1983. (in Chinese)
[16] Wang F G, Zhao J R, Dai J R, Yi H M, Kuang M, Sun Y M, Yu X Y, Guo J L, Wang L. Selection and development of representative simple sequence repeat primers and multiplex SSR sets for high throughput automated genotyping in maize., 2007, 52(2): 215-223.
[17] 王凤格, 田红丽, 赵久然, 王璐, 易红梅, 宋伟, 高玉倩, 杨国航. 中国328个玉米品种(组合)SSR标记遗传多样性分析. 中国农业科学, 2014, 47(5): 856-864.
Wang F G, Tian H L, Zhao J R, Wang L, Yi H M, Song W, Gao Y Q, Yang G H. Diversity analysis of 328 maize varieties (hybridized combinations) using SSR markers., 2014, 47(5): 856-864. (in Chinese)
[18] 王立新, 李云伏, 常利芳, 黄岚, 李宏博, 葛玲玲, 刘丽华, 姚骥, 赵昌平. 建立小麦品种DNA指纹的方法研究. 作物学报, 2007, 33(10): 1738-1740.
Wang L X, Li Y F, Chang L F, Huang L, Li H B, Ge L L, Liu L H, Yao J, Zhao C P. Method of ID constitution for wheat cultivars., 2007, 33(10): 1738-1740. (in Chinese)
[19] 徐雷锋, 葛亮, 袁素霞, 任君芳, 袁迎迎, 李雅男, 刘春, 明军. 利用荧光标记SSR构建百合种质资源分子身份证. 园艺学报, 2014, 41(10): 2055-2064.
XU L F, GE L, YUAN S X, REN J F, YUAN Y Y, LI Y N, LIU C, MING J. Using the luorescent labeled SSR markers to establish molecular identity of lily germplasms., 2014, 41(10): 2055-2064. (in Chinese)
[20] 胡振帮, 高运来, 齐照明, 蒋洪蔚, 刘春燕, 辛大伟, 胡国华, 潘校成, 陈庆山. 作物分子身份证构建软件ID analysis的编制. 中国农业科学, 2016,49(12): 2255-2266.
Hu Z B, Gao Y L, Qi Z M, Jiang H W, Liu C Y, Xin D W, Hu G H, Pan X C, Chen Q S. Software development of -ID analysis for crop molecular identity construction., 2016, 49(12): 2255-2266. (in Chinese)
[21] Bär W, Brinkmann B, Budowle B, Carracedo A, Gill P, Lincoln P, Mayr W, Olaisen B. DNA recommendations- Further report of the DNA Commission of the ISFH regarding the use of short tandem repeat systems., 1997, 87(3): 181-184.
[22] 陆徐忠, 倪金龙, 李莉, 汪秀峰, 马卉, 张小娟, 杨剑波. 利用SSR分子指纹和商品信息构建水稻品种身份证. 作物学报, 2014, 40(5): 823-829.
Lu X Z, Ni J L, Li L, Wang X F, Ma H, Zhang X J, Yang J B. Construction of rice variety indentity using SSR fingerprint and commodity information., 2014, 40(5): 823-829. (in Chinese)
[23] 高运来, 朱荣胜, 刘春燕, 李文福, 蒋洪蔚, 李灿东, 姚丙晨, 胡国华, 陈庆山. 黑龙江部分大豆品种分子ID的构建. 作物学报, 2009, 35(2): 211-218.
GAO Y L, ZHU R S, LIU C Y, LI W F, JIANG H W, LI C D, YAO B C, HU G H, CHEN Q S. Establishment of molecular id in soybean varieties in Heilongjiang, China., 2009, 35(2): 211-218. (in Chinese)
[24] Yang X, Xu Y, Shah T, Li H, Han Z, Li J, Yan J. Comparison of SSRs and SNPs in assessment of genetic relatedness in maize., 2011, 139: 1045-1054.
[25] Beadling C, Neff T L, Heinrich M C, Rhodes K, Thornton M, Leamon J, Andersen M, Corless C L. Combining highly multiplexed PCR with semiconductor-based sequencing for rapid cancer genotyping., 2013, 15(2): 171-176.
[26] Guichoux E, Lagache L, Wagner S, Chaumeil P, Le´ Ger P, Lepais O, Lepoittevin C, Malausa T, Revardel E, Salin F, Petit R J. Current trends in microsatellite genotyping., 2011, 11(4): 591-611.
[27] 郑永胜, 张晗, 王东建, 孙加梅, 王雪梅, 段丽丽, 李华, 王玮, 李汝玉. 基于荧光检测技术的小麦品种SSR鉴定体系的建立. 中国农业科学, 2014, 47(19): 3725-3735.
Zheng Y S, Zang H, Wang D J, Sun J M, Wang X M, Duan L L, Li H, Wang W, Li R Y. Development of a wheat variety identification system based on fluorescently labeled SSR markers., 2014, 47(19): 3725-3735. (in Chinese)
(责任编辑 李莉)
Construction of an SSR-Based Standard Fingerprint Database for Corn Variety Authorized in China
WANG Fengge1, YANG Yang1, YI Hongmei1, ZHAO Jiuran1, REN Jie1, WANG Lu1, GE Jianrong1, JIANG Bin2, ZHANG Xianchen2, TIAN Hongli1, HOU Zhenhua1
(1Maize Research Center, Beijing Academy of Agriculture & Forestry Sciences/Beijing Key Laboratory of Maize DNA Fingerprinting and Molecular Breeding, Beijing 100097;2Beijing TodaySoft Inc., Beijing 100083)
【Objective】 It is of great importance to construct a shareable high-quality crop variety standard DNA fingerprint database for effectively managing the huge number of known varieties.【Method】 Based on fluorescence capillary electrophoresis detection platforms and the plant variety DNA fingerprint database management system, a database containing 3 998 maize authorized accessions was built with 40 SSR primer pairs. Multi-laboratories and multi-detecting platforms were used to conduct the quality evaluation of the database.【Result】 Allele frequency distribution graphs of the 40 corn primers were plotted as characteristic spectrums of each primer, which played the role of the similarity of reference samples in the database construction. A decuplet fluorescent capillary electrophoresis combination was formed and a set of system default PANEL was established in the SSR analyzer. Statistics were conducted on the experimental conditions of the database construction. Of the total samples, 61% of them were subjected to two group independent trials and 33% of them were subjected to three group independent trials. Each sample had 2-5 sets of original experimental data and the corresponding fingerprint maps. The accumulated loss and variable sites of the final built standard fingerprint database accounted for only 0.2%, the data integrity reached 99.8%. The assessment results in different laboratories and different electrophoresis platforms showed that the SSR fingerprint data obtained high agreement on the same electrophoresis fluorescence detection platform, but showed a certain bias in different electrophoretic detection platforms. In order to realize the sharing of SSR fingerprint data in different laboratories, a unified fluorescent primer, analysis software and electrophoresis detection platform were needed. Overall pairwise comparisons were conducted on all the fingerprint data, the results showed that there existed a relative big overall difference among the certification varieties of corn in China. Percentage of different sites among the authorized varieties was mainly concentrated between 80% and 95% (accounted for 78.28%), the percentage of different sites with more than 50% reached 99.21%, and less than 20% of only 0.09%. The average hybrid rate of the authorized varieties reached 64% and mainly concentrated between 50% and 80% (accounted for 89%). By using the corn variety standard fingerprint matching service platform (URL: http: //www.maizedna.org/), a shared fingerprint database is realized. 【Conclusion】 A standard procedure in constructing crop variety SSR fingerprint database was formed in this study and the SSR fingerprint database was constructed with a scale of nearly 4 000 corn authorized varieties. Through joint multi-laboratory comparison tests, the accuracy of database building and the sharing property of database were ensured. A service platform website for corn variety standard fingerprint matching was established in this study, thus achieving sharing of corn authorized variety fingerprint database in national seed identification system, and providing an important reference for other crop species in building high-quality SSR fingerprint database.
maize; variety identification; DNA fingerprint database; SSR
2016-07-11;接受日期:2016-09-27
国家“十三五”科技支撑计划(2015BAD02B02)、北京市农林科学院院科技创新能力建设专项(KJCX20161501)
王凤格,E-mail:gege0106@163.com。杨扬,E-mail:caurwx@163.com。易红梅,E-mail:yihm47@sina.com。王凤格、杨扬和易红梅为同等贡献作者。通信作者赵久然,E-mail:maizezhao@126.com

