古籍纸张表面微生物群落组成的初步研究
周言君,钟 江
(1. 复旦大学 生命科学学院 微生物与微生物工程系,上海 200438;2. 复旦大学 中华古籍保护研究院,上海 200433)
古籍纸张表面微生物群落组成的初步研究
周言君1,2,钟 江1,2
(1. 复旦大学 生命科学学院 微生物与微生物工程系,上海 200438;2. 复旦大学 中华古籍保护研究院,上海 200433)
纸张表面的微生物直接或间接地影响古籍的保存和使用年限,全面认识纸表微生物的组成有助于古籍保护.本研究对手抄本《周本纪摘要》中的纸张表面菌斑处(stain组)和洁净处(clean组)共9处,以及环境空气进行了样品采集,并对样品进行了高通量测序,对测序结果进行了生物信息学分析.10个样品共得到高质量的细菌16S序列和真菌ITS序列共485989条.序列数据分析表明,纸表真菌可以划分为650个OTU,分属6个门,细菌可以划分3465个OTU,分属15个门,其中丰度最高的分别是细菌的糖多孢菌属(Saccharopolyspora)和真菌的曲霉属(Aspergillus).相比于clean组,stain组有显著多的糖多孢菌属和假诺卡氏菌属(Pseudonocardiaceae)细菌,以及显著多的牛肝菌目(Boletales)和爪甲团囊菌目(Onygenales)真菌.在聚类分析上,无论是细菌还是真菌部分,stain组和clean组的样品都能很好的分开,但在Alpha多样性指数上,组间差异趋势不显著.这些结果说明无论是否有菌斑,纸表微生物群落多样性程度是稳定的,但群落组成和结构有很大差异.
微生物群落; 高通量测序; 古籍保护
古籍是传统文化的重要载体,从纸张制作和印刷,到古籍流转、储藏、使用的全部过程,都有大量微生物的存在.它们消耗利用纸张的各种无机有机成分,生长和繁殖、协同与拮抗,直接或间接地影响着古籍外观和纸张寿命.古籍微生物种类繁多,功能不明,利弊难辨,更难于清除.微生物防治一直是古籍文物保护的难点之一.全面了解古籍纸张表面微生物种类、种群结构、代谢特征,就有可能明确对古籍有潜在威胁的微生物类群,指导采取有针对性的预防措施,长久地保存这些珍贵书籍,流传后世[1].
近几十年来国内外研究者对古籍纸张表面微生物进行了大量的研究,从不同材质、年代、保存状态、流转历史的古籍书页中检测到种类繁多的微生物.如有研究者发现土曲霉菌(Aspergillusterreus)能在纸张上生长并产生草酸,并与纸张中的钙离子反应形成草酸钙晶体,随着菌丝体的生长和草酸钙晶体的累积会最终导致纸张纤维的断裂[2],而生长在书画表面镉红颜料上的寡养单胞菌(Stenotrophomonas)会长出红色或黄色的菌落,从而使书画变色[3].但受限于技术手段,这些研究往往无法涵盖所有的类群,也无法全面比较不同类型纸张表面微生物的群落差异.
微生物宏基因组和高通量测序技术的迅猛发展,使我们能从一个样品中获得数万条基因序列,一次性获得样品中几乎全部微生物群落组成和相对丰度的信息[4-5],从而有机会从系统的角度看待古籍上的微生物,用全新的视角研究古籍保护中亟待解决的重要问题,促进古籍保护工作的发展.
本研究针对一本清末民初的手工皮纸抄本《周本纪摘要》中的内页菌斑、封底菌斑和内页洁净纸张这三类样品分别进行3个平行样品的采集,提取DNA,进行高通量测序和生物信息学分析,探究不同样品间微生物群落组成的异同,以期为不同类型古籍纸张表面微生物的全面深入研究奠定基础.
1 材料和方法
1.1 纸张样品
本研究选取清末民初(十九世纪末到二十世纪初)的皮纸手抄本《周本纪摘要》一册进行纸表微生物的采样.书册整体保存较完好,纸质洁白无明显酸化,内页和封底处有几处菌斑,书册及其纸张在采样前未作任何处理.
1.2 采样方法
样品采集于2016年4月,采样位置为内页菌斑处、封底菌斑处和内页洁净处,每组采3个样品.具体的采样方法见文献[6],其中擦拭方法根据研究对象做了改动,将润湿的聚酯纤维拭子用润湿液(0.15mol/L NaCl和体积分数为0.1%的Tween)湿润后,在目标区域中2cm×2cm的范围内单向轻柔擦拭20次,以不对纸张表面造成损伤为准.作为对照的空气沉降样品的采集方法为: 在采样开始时,将聚酯纤维拭子蘸取润湿液后,立于采样工作台面上,采样结束后用其他样品的处理方式收集此拭子.
1.3 DNA提取与PCR扩增
对文献[6]的方法进行部分改动,使用PowerSoil DNA Isolation Kit(MoBio)按照试剂盒说明书进行DNA提取.以提取得到的DNA作为模板,对细菌16S核糖体RNA基因(16S rDNA)和真菌核糖体RNA基因内转录间隔区(Internal Transcribed Spacer, ITS)进行PCR扩增,以确定得到的DNA样品有足够的量,可用于后续高通量测序.16S rDNA扩增引物为27F(5′-AGAGTTTGATCCTGGCTCAG-3′)和338R(5′-TGCTGCCTCCCGTAGGAGT-3′),ITS扩增选用引物为ITS1F(5′-CTTGGTCATTTAGAGGAA GTAA-3′)和ITS2(5′-GCTGCGTTCTTCATCGATGC-3′).扩增条件为94℃ 5min;94℃ 45s,50℃ 30s,72℃ 90s,32个循环;72℃ 10min.对于扩增产物在凝胶电泳中条带不够清晰的样品,可将扩增循环数增加到35个循环.
1.4 高通量测序与序列分析
委托上海华津生物科技有限公司采用Illumina公司Miseq平台针对16S rDNA V3-V4区和ITS1区进行测序.对测序得到的reads先根据overlap关系进行拼接,同时对序列质量进行质控和筛选后,得到优化序列.使用Mothur软件以序列相似性97%为标准划分操作分类单元(Operational Taxonomic Units,OTU).原始的OTU丰度矩阵中包含大量“稀有”OTU,它们丰度极低,且仅在少量样品中偶尔出现,这些OTU的存在会大大增加数据分析的复杂度,而通常情况下,去除这些稀有OTU对于解析整体菌群的影响微乎其微,因此我们在OTU划分的原始数据中,将丰度值低于全体样本测序总量0.001%的OTU去除[7],以降低数据分析的“背景噪音”.以去除了“稀有”OTU的丰度矩阵为基础进行Alpha多样性和Beta多样性指数分析和聚类分析.将每个OTU的代表序列与SILVA数据库中的16S rDNA和ITS模板序列进行比对,确定其分类学信息,并在各分类水平上进行群落组成的差异分析.
2 结果与分析
2.1 取样和扩增
共采集10个样品,3个内页菌斑样品命名为MA1、MA2、MA3,分别采自3个不同页面;3个内页洁净样品命名为MA4、MA5、MA6,均采自书本内页无明显菌斑或水迹的洁净处;3个封底菌斑样品命名为MA7、MA8、MA9,均采自本书封底,但采样区域不交叠;1个空气沉降样品,命名为ZAB1,来自采样工作台面上的空气自然沉降.对这十个样品进行DNA抽提和16S rDNA、ITS的PCR扩增,样品DNA浓度均较低,但PCR扩增32个循环时条带清晰,可供后续高通量测序所用.
2.2 高通量测序结果
测序结果经过剔除和筛选后,10个样品共得到16S rDNA高质量序列142270条,ITS高质量序列343719条.无论是初始的有效序列总数,还是高质量序列占有效序列的比例,ITS序列都高于16S rDNA序列.按照序列相似度97%的标准对高质量序列进行OTU的划分,并去除“稀有”OTU后,ITS序列共得到650个OTU,16S rDNA序列得到3465个OTU.
2.3 菌群多样性和聚类分析
为了较为全面地反映各样品微生物群落的特征,我们用多种指数来反映不同样品微生物群落的Alpha多样性(表1).不同的指数对于衡量群落多样性的侧重点不同,chao1指数和ACE指数侧重于体现群落的丰富度,即群落中OTU的数量[8-9];Shannon多样性指数和Simpson多样性指数则综合考虑群落的丰富度和均匀度,即群落中各OTU之间丰度差异的大小[10].从真菌部分来看,菌斑位置(MA1、MA2、MA3、MA7、MA8、MA9)的真菌丰富度(chao值和ACE值)均高于洁净页面(MA4、MA5、MA6)和环境空气(ZBC1),且空气沉降样品中的真菌丰富度最低.细菌的群落多样性并没有体现出这一点,而是内页中(MA1、MA2、MA3、MA4、MA5、MA6),无论是否有菌斑,细菌群落的丰富度普遍低于封底纸表(MA7、MA8、MA9)和空气(ZBC1).将群落内的OTU均匀度考虑在内之后(即Simpson指数和Shannon指数),10个样品间的多样性指数值则失去了明显的差异.
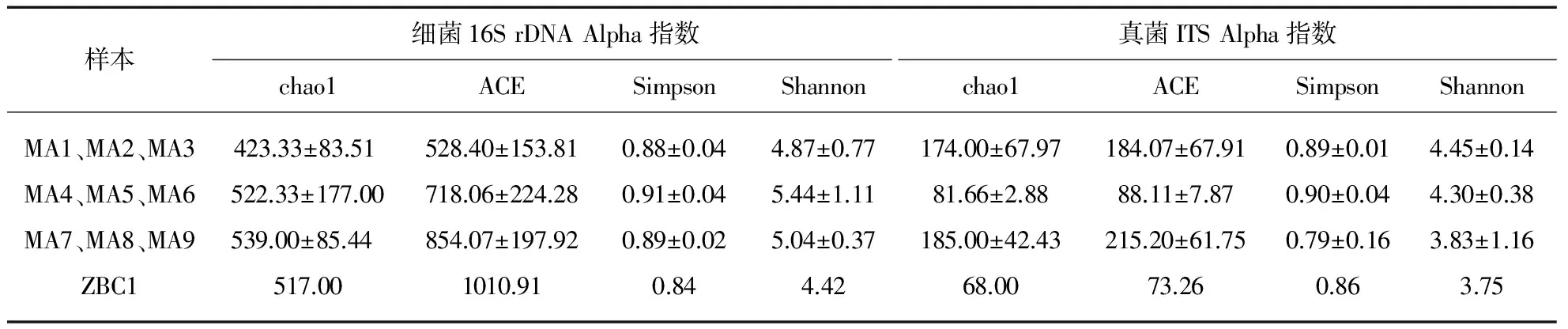
表1 不同采样位置的16S rDNA和ITS的Alpha多样性指数表
基于UniFrac距离的ITS序列UPGMA(Unweighted Pair-Group Method with Arithmetic means)样本聚类分析结果显示,洁净的纸张页面和环境空气中的真菌群落聚类十分接近,而内页菌斑处和封底菌斑处的真菌群落则并未按类型聚在一起,尤其是3个内页菌斑样品MA1、MA2和MA3的群落相似度较低(图1).这可能是由于采样时内页菌斑处的3个样品分别来自于不同页面,虽然斑点形态相似,但导致菌斑形成的菌群其实差异很大.而封底菌斑组的3个样品来源于同一纸张页面,采样范围虽未重叠,但距离较近,因此3个样品,尤其是MA7和MA9,聚类比较接近.
我们将纸张表面的9个样品按照采样类型分为两组: 菌斑组(stain组,MA1、MA2、MA3、MA7、MA8、MA9)和洁净组(clean组,MA4、MA5、MA6),以此模式进行偏最小二乘法判别分析(Partial Least Squares Discriminant Analysis, PLS-DA),结果见图2(见第710页).无论是细菌还是真菌部分,都能很好地将stain组和clean组的样品分开,且组内差异小于组间差异.其中,stain组内细菌群落能够很好地拟合,而真菌群落组内差异较大.与之相对地,clean组内细菌群落差异较大,而真菌群落能够很好地拟合.这一结果说明,在纸张表面菌斑处的真菌群落可能有很多种组成形式,而洁净纸表的真菌群落则较为相似和稳定.
2.4 微生物群落组成分析
序列分析结果表明,细菌主要来自15个门(Phylum),其中丰度最高的是变形菌门(Proteobacteria,44.4%)、放线菌门(Actinobacteria,32.3%)和厚壁菌门(Firmicutes,16.5%)(表2,见第710页).总比例较高的属为变形菌门的苍白杆菌属(Ochrobactrum)和放线菌门的糖多孢菌属(Saccharopolyspora)、红球菌属(Rhodococcus)等.真菌分别来自6个门,其中丰度(A)最高的是子囊菌门(Ascomycota),在所有样品中总比例为74.5%,其次是担子菌门(Basidiomycota),总比例为18.4%.占最主要地位的属是子囊菌门的曲霉属(Aspergillus)和拟青霉属(Simplicillium),其中曲霉属在全样品中所占的总比例为29.2%,在封底菌斑处样品MA7、MA8和MA9中的比例分别为52.0%、82.9%和53.9%(表2).主要类群与文献报道[11-14]中纸质文献表面所发现的微生物类群是基本一致的.
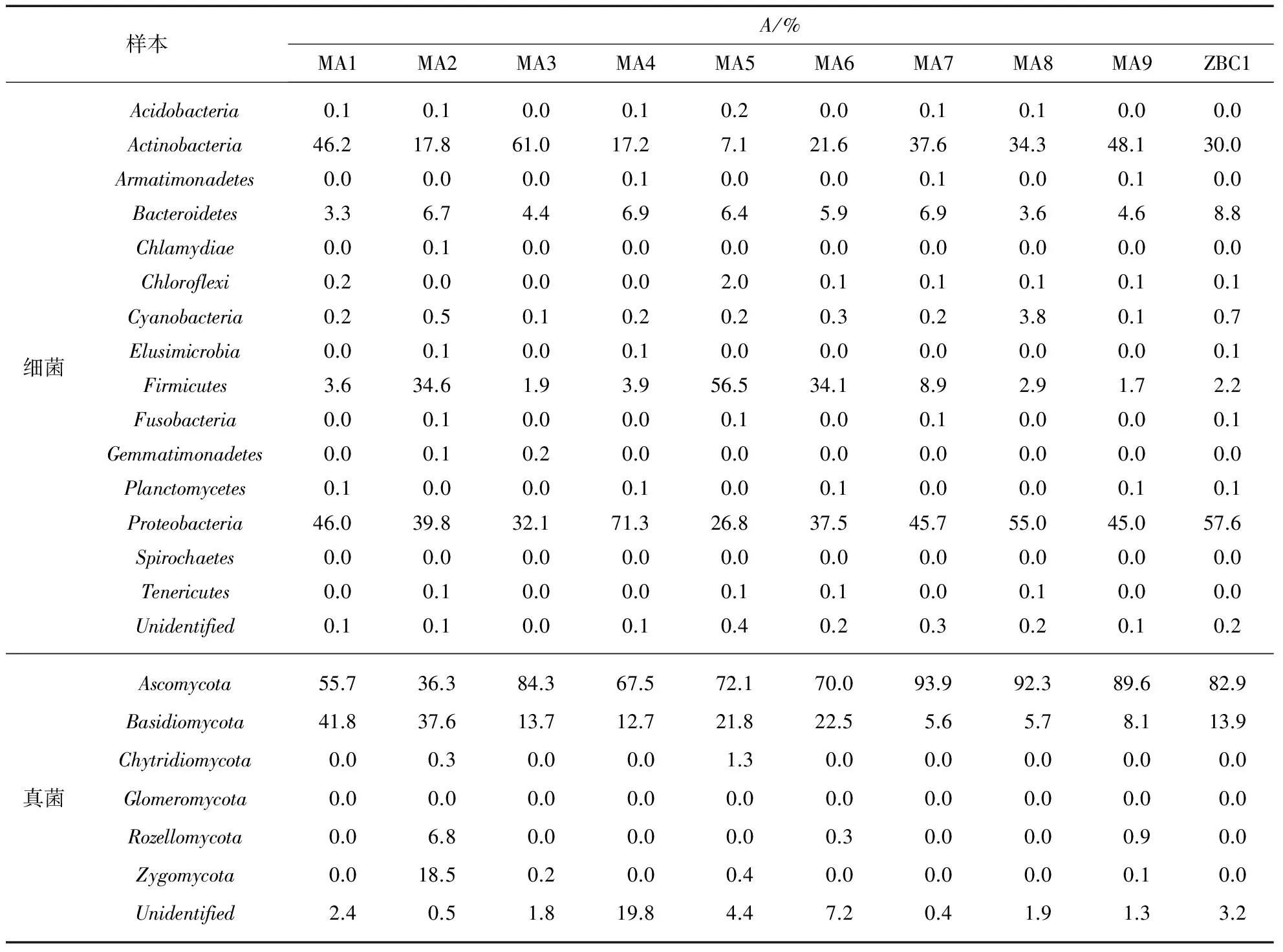
样本A/%MA1MA2MA3MA4MA5MA6MA7MA8MA9ZBC1细菌Acidobacteria0.10.10.00.10.20.00.10.10.00.0Actinobacteria46.217.861.017.27.121.637.634.348.130.0Armatimonadetes0.00.00.00.10.00.00.10.00.10.0Bacteroidetes3.36.74.46.96.45.96.93.64.68.8Chlamydiae0.00.10.00.00.00.00.00.00.00.0Chloroflexi0.20.00.00.02.00.10.10.10.10.1Cyanobacteria0.20.50.10.20.20.30.23.80.10.7Elusimicrobia0.00.10.00.10.00.00.00.00.00.1Firmicutes3.634.61.93.956.534.18.92.91.72.2Fusobacteria0.00.10.00.00.10.00.10.00.00.1Gemmatimonadetes0.00.10.20.00.00.00.00.00.00.0Planctomycetes0.10.00.00.10.00.10.00.00.10.1Proteobacteria46.039.832.171.326.837.545.755.045.057.6Spirochaetes0.00.00.00.00.00.00.00.00.00.0Tenericutes0.00.10.00.00.10.10.00.10.00.0Unidentified0.10.10.00.10.40.20.30.20.10.2真菌Ascomycota55.736.384.367.572.170.093.992.389.682.9Basidiomycota41.837.613.712.721.822.55.65.78.113.9Chytridiomycota0.00.30.00.01.30.00.00.00.00.0Glomeromycota0.00.00.00.00.00.00.00.00.00.0Rozellomycota0.06.80.00.00.00.30.00.00.90.0Zygomycota0.018.50.20.00.40.00.00.00.10.0Unidentified2.40.51.819.84.47.20.41.91.33.2
按照2.3节中提到的分组方式,运用LEfSe(Linear discriminant analysis Effect Size)方法进行组间的比较,从而找到组间在丰度上有显著性差异的分类单元,最后用线性判别分析(LDA)对数据进行降维,并评估差异显著的物种的影响力,即LDA值(取log10后),结果见图3.
其中,stain组内的细菌群落有更多的放线菌类群(尤其是糖多孢菌属)和芽孢杆菌属(Bacillus);真菌群落中有相当大比例的曲霉属和青霉属(Penicillium),但在统计学上与clean组相比有显著性差异的类群则是牛肝菌目(Boletales)、爪甲团囊菌目(Onygenales)和外担子菌纲(Exobasidiomycetes).
clean组内的细菌群落中有显著多的动胶菌属(Zoogloea)、肠杆菌属(Enterobacter)和红环菌目(Rhodocyclales);真菌群落中有显著多的链格孢属(Alternaria)和肉座菌目(Hypocreales).
3 讨 论
中华古籍的纸张成分多样,根据制作工艺和原料的不同,含有纤维素、半纤维素、木质素,同时还有少量树脂、淀粉、果胶、灰分等,这些成分都可以作为营养来源供给多种微生物的生长和繁殖.在实际的古籍善本流通和保藏过程中,微生物的影响十分显著,对其进行的各种研究一直在世界范围内进行.这些研究工作早期是应用传统的培养技术分离培养损害纸质文物的微生物,依据形态及生理生化特征对其进行鉴别分类[15-16].但由于培养条件限制、营养结构缺失、失去生态位等因素,大多数微生物在常规条件下是不可培养或难培养的,这些方法只能得到极少数菌株,且它们在古籍纸张上未必为有害菌或优势菌.随着技术手段的发展,基于PCR的分子鉴定[17-18]、DGGE[19]、基因芯片、扫描电镜(SEM)[20-21]、傅里叶变换红外光谱(FTIR)[2,22]等技术都逐渐被应用于古籍相关微生物的研究.这些方法能得到部分优势菌群,而对于整体的微生物群落,尤其是群落内丰度较低但可能对纸张的破坏起关键作用的菌群无法进行有效地分析.高通量测序技术有可能使研究者能够获得样品中几乎全部的微生物信息,为认识微生物对古籍纸张的影响提供了强大的工具.
本研究对一本手工皮纸古籍的菌斑处和洁净处分别采样,运用高通量测序技术分析其微生物群落多样性和群落结构.从9个纸张样品和1个空气沉降样品中共得到细菌16S rDNA和真菌ITS序列共485989条,稀释曲线均接近饱和,表明测序深度能够反映样品的微生物群落结构.Alpha多样性提示,在只考虑OTU数量的情况下,真菌的多样性高低与保存状况(是否有菌斑)有关,而细菌的多样性高低可能与是否暴露在空气中有关(差异不显著).但样品内OTU丰度差异较大,因此当算法考虑OTU样品均匀度时,样品间多样性指数不再有明显差异.
聚类分析发现无论是细菌还是真菌,在设定的分组下,聚类都很清晰.虽然多样性指数并不如预计——菌斑处细菌和真菌的多样性指数并不显著高于洁净纸张和空气——但两组样品的群落组成却有显著性差异,且组内聚类良好.这也说明纸张表面各种微生物群居杂生,纸表是否形成菌斑或污损,与其菌群的多样性程度并不一定相关,而更多地与菌群组成结构相关.
Zyska[11]统计了1919年至1977年发表的文献,其中从图书馆藏书上分离的真菌共84个属,234种,主要类群为曲霉属、青霉属、木霉属(Trichoderma)、链格孢属和毛壳菌属(Chaetomium)真菌,其后至今近40年的研究报道中,与纸表损伤相关的真菌类群也多为这五类[23].在本研究的所有样品中,真菌几乎都属于子囊菌门(74.5%)和担子菌门(18.4%),其中丰度最高的类群是子囊菌门的曲霉属,特别是在封底菌斑处的3个样本中,曲霉属占总真菌数的50%以上.青霉属、木霉属和毛壳菌属的比例均较低且组内差异较大,组间没有显著规律.比较特殊的是链格孢属,在总菌中比例为4.3%,在空气沉降样品中也有1.5%的丰度,但在stain组中比例均在1%以下,显著低于clean组中(图3).一个可能的原因是,链格孢属确实是纸表微生物和图书馆环境空气中的常见真菌类群,但与本研究中涉及到的菌斑形成无关.
相对于真菌而言,细菌在古籍保护领域长久以来并未受到重视.近年来,越来越多的研究者开始关注纸表细菌类群及其对纸质文物的危害.芽孢杆菌属、梭菌属(Clostridium)、假单胞菌属(Pseudomonas)和微球菌属(Micrococcus)是其中最常见的种类[12].此外放线菌属(Actinomyces)和噬纤维菌属(Cellulophaga)也常见于有关纸表细菌的报道.本研究中stain组内有显著多的芽孢杆菌和显著多的放线菌类群,如假诺卡氏菌属(Pseudonocardiaceae)、糖多孢菌属等.放线菌是公认的降解能力较强的细菌类群,能穿透不溶基质,增加它的水溶性,其中属于链霉菌属(Streptomyces)的丝状细菌在初级代谢阶段有降解木质素能力,主要是通过脱甲基、芳环断裂和侧链氧化3种途径降解木质素[24],降解率最高可达20%.另外,Pages研究发现,嗜麦芽寡养单胞菌(Stenotrophomonasmaltophilia)可以把亚硒酸盐转化成元素硒,从而使菌落颜色变为红色[25].生长在书画表面镉红颜料上的寡养单胞菌(Stenotrophomonas)会长出红色或黄色的菌落,从而使书画变色[3].许多纸质文物在久藏之后会出现各种颜色的不规则斑点,这种情况大多是由微生物导致的,多种细菌和真菌在生长过程中会产生色素,使菌落呈现肉眼可见的形状和颜色,反映在纸张上就是难以去除的色斑,根据是否可见菌丝体、是否有荧光现象等进行区分,称为“霉斑”或“狐斑”等.有研究者[23]从不同纸张的褐色斑点(狐斑)处分离到25株真菌,基本都是曲霉属和散囊菌属(Eurotium).梭菌属、假单胞菌属这两种常见于纸质文物研究的菌群在本研究所有样品中比例均在1%以下,且组间没有显著性差异,微球菌属和噬纤维菌属在本研究中没有发现,或因丰度低于0.001%被作为“稀有”OTU而剔除.
意大利的研究者在2014年的研究中发现了一套被菌斑严重侵蚀的摄影材料(正片、底片、纸质框和玻璃纸信封)中细菌和真菌在同一个生态位的共同生长,在细菌文库中变形菌门在所有的材料中都占大部分,尤其是正片中,所有细菌都是变形菌门,其他的材料中细菌的组成各有差异,其中纸质框中有58%的变形菌门,25%的放线菌门和5%的厚壁菌门细菌[13].本研究中变形菌门占44.4%,放线菌门占32.3%,厚壁菌门占16.5%,与其纸质框中的结果比较接近.不同的细菌类群对生长环境中的营养基质有其偏好,因此,这种群落结构的结果可能对以后针对不同纸张类型的菌群研究有一定的提示作用.
在纸表微生物的已有研究中,大多数研究针对的目标是真菌,尤其是丝状真菌对纸张的损害,而细菌在纸张老化中所起的作用可能被大大低估了.事实上细菌在纤维素降解、纸张酸化、色斑的产生等方面都有不可小觑的潜力[26-27].本研究中,细菌和真菌在纸表这个微环境中不仅共同存在,且都能在分组中稳定聚类,并有一些类群在组间有显著的丰度差异,这些结果说明肉眼可见的菌斑,其形成不一定是单独的真菌或细菌作用,而可能是两者的协同和共生的结果,而这些关系的发展,可能对古籍纸张的寿命起着决定性的作用.
本研究通过基本无损的采样方法和高通量测序技术初步发现古籍纸张的菌斑位置和洁净位置,细菌和真菌的群落结构有明显的差别,并对这些差别进行了生物信息学的分析.受限于样本种类和样品数量不够的问题,尚无法明确导致菌斑形成的关键微生物.希望在后续研究中,高通量测序结合生物信息学分析的方法能够在古籍相关微生物研究中得以更广泛和深入的应用,将古籍的微生物防治和保护修复工作向现代化推进.
[1] CAPPITELLI F, PASQUARIELLO G, TARSITANI G,etal. Scripta manent? Assessing microbial risk to paper heritage[J].TrendsinMicrobiology, 2010,18(12): 538-542.
[2] PINZARI F, ZOTTI M, MICO A D,etal. Biodegradation of inorganic components in paper documents: Formation of calcium oxalate crystals as a consequence ofAspergillusterreusThom growth[J].InternationalBiodeterioration&Biodegradation, 2010,64(6): 499-505.
[4] SHENDURE J, JI H. Next-generation DNA sequencing[J].NatureBiotechnology, 2008, 26(10): 1135-45.
[6] 迟 亮,谭 渊,全哲学.上海健康人群夏冬两季皮肤真菌群落组成: 基于高通量测序技术的研究[J].复旦学报(自然科学版),2014,53(6): 697-703.
[7] BOKULICH N A, SUBRAMANIAN S, FAITH J J,etal. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J].NatureMethods, 2013,10(1): 57-59.
[8] CHAO A. Non-parametric estimation of the classes in a population[J].ScandinavianJournalofStatistics, 1984,11(4): 265-270.
[9] CHAO A, YANG M C K. Stopping rules and estimation for recapture debugging with unequal failure rates[J].Biometrika, 1993,80(1): 193-201.
[10] SHANNON C E. A mathematical theory of communication[J].BellSystemTechnicalJournal, 1948,27(4): 623-656.
[11] ZYSKA B. Fungi isolated from library materials: A review of the literature[J].InternationalBiodeterioration&Biodegradation, 1997,40(1): 43-51.
[12] DUNCA S I, TANASE C, PADURARIU C,etal. Study of the contaminating microbiota of old paper supports[J].EuropeanScientificJournal, 2014: 237-251.
[15] MESQUITA N, PORTUGAL A, VIDEIRA S,etal. Fungal diversity in ancient documents: A case study on the archive of the University of Coimbra[J].InternationalBiodeterioration&Biodegradation, 2009,63(5): 626-629.
[16] GORBUSHINA A A, HEYTMAN J, DORNIEDEN T,etal. Bacterial and fungal diversity and biodeterioration problems in mural painting environments of St. Martins church (Greene-Kreiensen, Germany)[J].InternationalBiodeterioration&Biodegradation, 2004,53(1): 13-24.
[17] WANG W, MA Y, MA X,etal. Seasonal variations of airborne bacteria in the Mogao Grottoes, Dunhuang, China[J].InternationalBiodeterioration&Biodegradation, 2010,64(4): 309-315.
[18] RAKOTONIRAINY M S, HEUDE E, LAVÉDRINE B. Isolation and attempts of biomolecular characterization of fungal strains associated to foxing on a 19th century book[J].JournalofCulturalHeritage, 2007,8(2): 126-133.
[19] MICHAELSEN A, PINZARI F, BARBABIETOLA N,etal. Monitoring the effects of different conservation treatments on paper-infecting fungi[J].InternationalBiodeterioration&Biodegradation, 2013,84(100): 333-341.
[20] MANENTE S, MICHELUZ A, GANZERLA R,etal. Chemical and biological characterization of paper: A case study using a proposed methodological approach[J].InternationalBiodeterioration&Biodegradation, 2012,74(4): 99-108.
[21] JURADO V, PORCA E, PASTRANA M P,etal. Microbiological study of bulls of indulgence of the 15th-16th centuries[J].ScienceoftheTotalEnvironment, 2010,408(17): 3711-3715.
[22] ZOTTI M, FERRONI A, CALVINI P. Microfungal biodeterioration of historic paper: Preliminary FTIR and microbiological analyses[J].InternationalBiodeterioration&Biodegradation, 2008,62(2): 186-194.
[23] ARAI H. Foxing caused by fungi: Twenty-five years of study[J].InternationalBiodeterioration&Biodegradation, 2000,46(46): 181-188.
[24] CRAWFORD D L, POMETTO A L, CRAWFORD,etal. Lignin degradation byStreptomycesviridosporus: Isolation and characterization of a new polymeric lignin degradation intermediate[J].Applied&EnvironmentalMicrobiology, 1983,45(3): 898-904.
[25] PAGES D, ROSE J, CONROD S,etal. Heavy metal tolerance inStenotrophomonasmaltophilia[J].PLoSOne, 2008,3(2): e1539.
[27] EM G D P, SAADEDDIN A. The cellulolytic system ofThermobifidafusca[J].CriticalReviewsinMicrobiology, 2014,40(3): 236-247.
A Preliminary Study of the Microbial Community Composition on Old Paper Material
ZHOU Yanjun1,2, ZHONG Jiang1,2
(1.DepartmentofMicrobiologyandMicrobialEngineering,SchoolofLifeSciences,FudanUniversity,Shanghai200438,China; 2.ChineseAncientBooksPreservationandConservationInstitute,FudanUniversity,Shanghai200433,China)
The microorganism community on paper surface plays an important role in historical paper heritage in direct or indirect ways, the understanding of which may greatly enhance their conservation. In this study, we collected 9 microbial samples, including 3 from clean site and 6 from stain site, from an old manuscript namedZhoubenjizhaiyao, and one air deposition sample as blank control. These samples were subjected to high-throughput DNA sequencing and bioinformatics analysis. A total of 485989 high quality sequences of bacterial 16S rDNA or fungal ITS(internal transcribed spacer) were obtained. They could be categorized into 650 fungal operational taxonomic units (OTUs) and 3465 bacterial OTUs. Fugal OTUs were classified into 11 phyla, with the predominant genus beingAspergillus. Bacterial OTUs were classified into 15 phyla, andSaccharopolysporawas the most abundant group among all genera. Compared to clean group, the abundance ofSaccharopolysporaandPseudonocardiaceaewere significantly higher within stain group, as well asBoletalesandOnygenales. Althoght stain and clean groups were not significantly different in alpha diversity analysis, they did separate from each other quite well in clustering analysis. In conclusion, the diversity indices of microbial community on paper material was stable, with or without the spot, meanwhile the community composition and structure varies significantly.
microflora; high-throughput sequencing; old paper conservation
0427-7104(2016)06-0707-08
2016-10-15
周言君(1990—),女,博士研究生;钟 江,男,教授,通讯联系人, E-mail: jzhong@fudan.edu.cn.
Q 493
A

